基于ISSR标记的12份桃种质资源遗传多样性分析
李建辉,陈 骏,刘丽丽,郑雪良,杨海英,刘春荣
(浙江省衢州市农业科学研究院,浙江324000)
桃(Prunus persica)系蔷薇科(Rosaceae)李属(Prunus)果树,原产于中国,为我国主要水果种类之一。桃种质资源丰富,世界上约有3 000个不同品种和类型,我国大约有2 000多个品种[1]。作为浙江省的农业大市,衢州地区近年来先后从国内外引进和收集了一批优良桃品种,其中大多已成为当地主栽品种,取得了显著的经济社会效益。然而,在这些桃种质资源中,大多数品种的植株和叶片性状差异不明显,因此在外观上很难区别,从而给准确进行资源的保存及利用带来诸多不便。同时,衢州地区还有‘乌桃’和‘毛桃’等野生资源,其中‘乌桃’为高山野生水果,目前仅分布于常山县的新桥、芙蓉、金源等乡镇,但‘乌桃’和‘毛桃’与当地主栽品种的亲缘关系仍不明确,而相关研究迄今未见报道。
目前,用于遗传多样性研究的分子标记主要有限制性片段长度多态性(RFLP)、随机扩增多态DNA(RAPD)、简单重复序列区间(ISSR)以及简单序列重复(SSR)等技术[2-3]。其中ISSR作为简便、快速、易行的分子标记技术,能够比RAPD检测到更多的遗传变异[4]。近年来,ISSR标记已广泛应用于果树遗传多样性分析[5-7],并在桃种质资源研究中充分体现了该标记的重要性与高效性[1,8]。为此,本研究基于ISSR分子标记技术,对收集的12份桃种质资源进行遗传多样性分析,旨在进一步明确各材料之间的亲缘关系,为后续开展种质资源创新与利用提供参考依据。
1 材料与方法
1.1 材料
供试材料来源于衢州市农科院设立在柘川基地的桃种质资源圃,共12份(表1)。每份材料取着生有较多健康幼嫩叶片的枝条置于保鲜袋中,封口,于样品贮藏箱(由超低温冰袋保持冷藏条件)中迅速带回实验室,洗净,晾干,置于-70℃超低温冰箱中保存备用。

表1 12份桃种质资源Table 1 12 P.persica germplasm resources
1.2 方法
1.2.1 基因组DNA的提取
采用改良的CTAB法进行桃基因组DNA的提取[9]。经0.8%琼脂糖凝胶电泳分析,用Gel Doc XR凝胶成像系统(美国Bio-Rad公司)拍照定量,-20℃保存备用。
1.2.2 PCR扩增与产物鉴定
ISSR引物采用加拿大哥伦比亚大学(University of British Columbia,Set No.9,No.801—900)提供的序列(http:∕∕www.biotech.ubc.ca∕services∕naps∕primers∕Primers.pdf),由上海生工生物工程公司合成。 经优化的PCR扩增的最佳反应体系为:25 μL PCR反应体积,1×Taq酶配套缓冲液(10 mmol∕L Tris-HCl,pH 9.0,50 mmol∕L KCl,0.1%Triton X-100),0.25 μmol∕L 4 × dNTP,1.25 U Taq DNA 聚合酶,50 ng 模板DNA,0.3 μmol∕L引物,1.5 mmol∕L MgCl2。阴性对照加入除模板DNA外的以上各成分,用双蒸水代替总DNA。经优化的PCR扩增程序为:94℃预变性5 min;94℃变性45 s,55—62℃复性45 s(不同引物退火温度的详细信息见表2),72℃延伸1.5 min,共35个循环;72℃延伸10 min,产物于4℃保存。
所有反应在Mastercycler ep gradient PCR仪(德国Eppendorf公司)中进行。PCR扩增反应中所用的试剂购自上海生工生物工程公司。扩增产物在含有0.2%GoldViewTM核酸染料(北京赛百盛基因技术有限公司)的2.0%的琼脂糖凝胶中电泳,电泳缓冲液为0.5×TBE,用Gel Doc XR凝胶成像系统(美国Bio-Rad公司)拍照保存。
1.2.3 数据统计与分析
用DL2000标准分子量参照物(大连宝生物工程有限公司)做分子量标记,对照反应产物在凝胶上的相应位置,按扩增有无记录电泳带谱,有带记为“1”,无带记为“0”,得到ISSR表型数据矩阵。将矩阵输入POPGENE 32软件进行分析[10],计算多态位点百分率(P)、Shannon信息指数(I)、Nei基因多样性(h)、遗传相似度以及遗传距离。再根据遗传距离,采用算术平均数的非加权成组配对法(Unweighted pair group method arithmetic average,UPGMA)进行聚类分析。
2 结果与分析
2.1 12份桃种质资源的遗传多样性
从100个引物中筛选出在所有样品中均可扩增出清晰条带,且重复性好,同时阴性对照中无带的12个引物(表2)作为正式扩增的ISSR引物,对12份桃种质资源的DNA样品进行ISSR分析,共检测到57条清晰可辨条带,其中多态性条带22条,总多态性条带百分率P为38.60%,平均为31.87%(表3)。每条引物扩增条带数量介于3—8条,平均4.75条。引物UBC856的扩增结果如图1所示,12个样品扩增的 DNA片段集中在200—2 000 bp。12份桃种质资源总 Shannon信息指数I为0.2006,平均0.1669;总Nei基因多样性h为0.1349,平均0.1125。

图1 12份桃种质资源的ISSR扩增结果(引物UBC856)Fig.1 ISSR amplification of 12 P.persica germplasm resources(by primer UBC856)
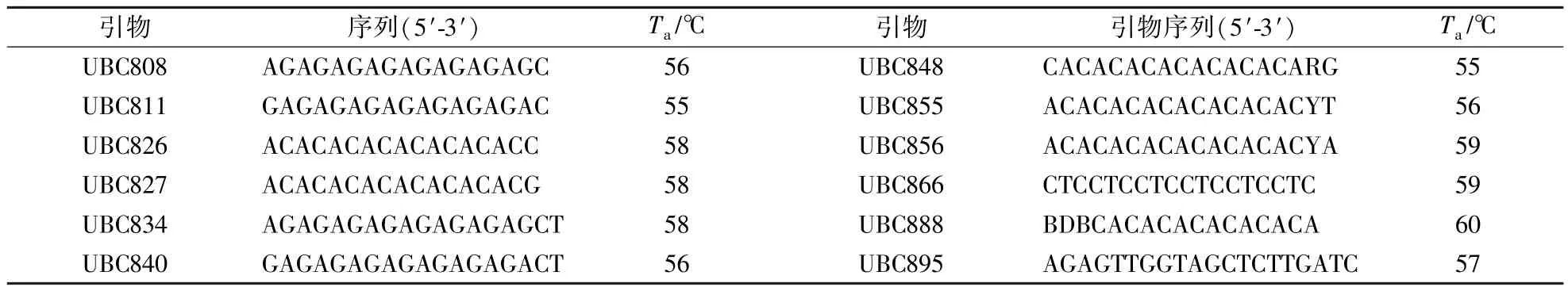
表2 用于ISSR分析的12个引物序列Table 2 Sequences of 12 primers used for ISSR analysis
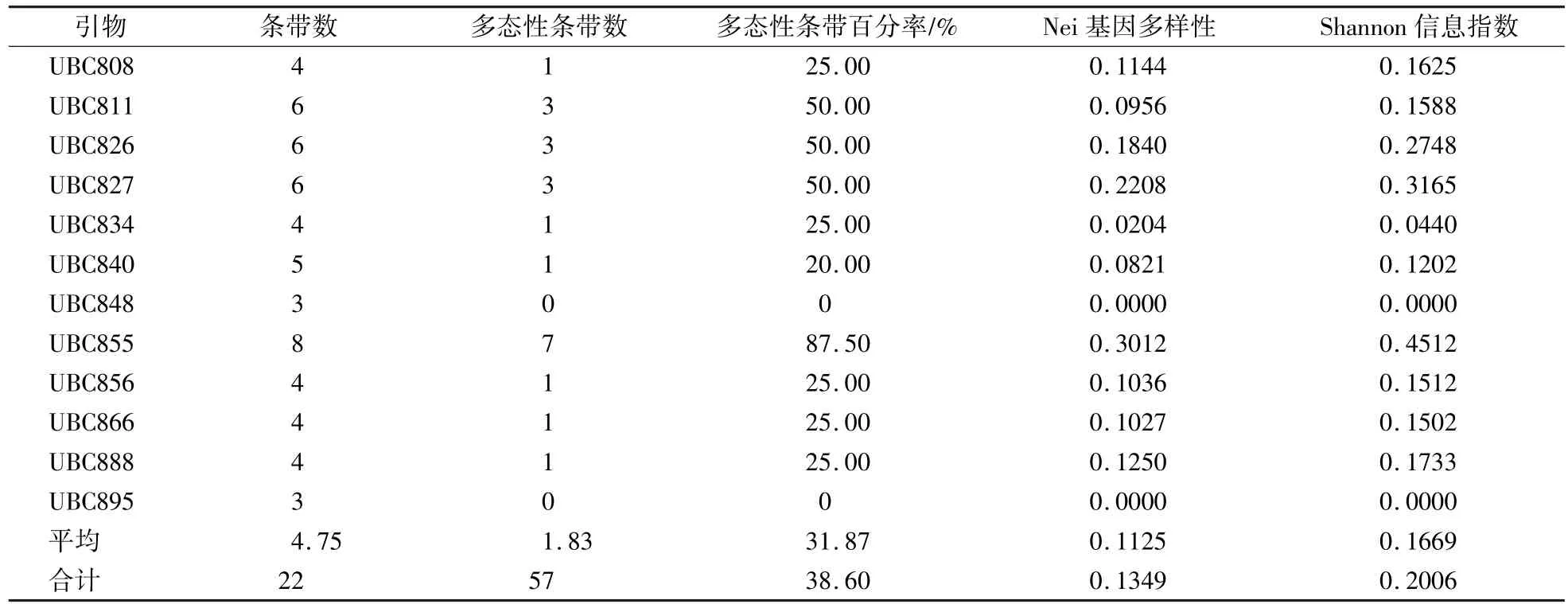
表3 12份桃种质资源的遗传多样性指数Table 3 Genetic diversity indices of 12 P.persica germplasm resources
2.2 12份桃种质资源的遗传相似度与遗传距离
采用POPGENE 32软件计算12份桃种质资源的遗传相似度与遗传距离,得到其矩阵。如表4所示,12份桃种质资源的遗传距离介于0.0357—0.2589,变幅为0.2232,平均为0.1455;遗传相似度介于0.7719—0.9649,变幅为0.1930,平均为0.8658。其中材料3和5之间的遗传距离最小,为0.0357;遗传相似度最大,为0.9649,表明材料3和5之间的亲缘关系较近,遗传差异较小。材料9和10、11以及2和10之间的遗传距离最大,为0.2589;遗传相似度最小,为0.7719,表明这些材料之间的亲缘关系较远,遗传差异较大。
2.3 12份桃种质资源的聚类分析
根据12份桃种质资源的遗传距离,采用UPGMA法进行聚类分析。根据聚类结果大致可将12份桃种质资源分为4个类群(图2):第一类群包括材料1、2、3、5、12;第二个类群包括材料4、6、7、8、11;材料 9和10各归为一类。

表4 12份桃种质资源的遗传相似度与遗传距离Table 4 Genetic similarities and genetic distances of 12 P.persica germplasm resources

图2 12份桃种质资源的聚类分析Fig.2 Cluster analysis of 12 P.persica germplasm resources by UPGMA method
3 讨论
本研究从100条ISSR引物中筛选得到12条引物对12份桃种质资源进行遗传多样性分析,从研究结果来看,这12条引物对供试样品扩增稳定,条带清晰明亮,多态性好,且几乎每条引物都能扩出一个或若干个供试材料的特殊条带,表明ISSR标记是一种经济高效的分子标记技术,可有效应用于桃种质资源遗传多样性分析,从而为进一步研究和利用这些种质资源提供了分子生物学依据。
作为农作物育种研究的重要内容,种质资源遗传多样性的高低决定了其在育种实践中的应用方向。对于果树而言,由于其育种周期较长,因而开展种质资源遗传多样性评价就显得尤为重要[11]。桃种质资源丰富,具有广泛的品种多样性。从本研究结果来看,12份桃种质资源总多态位点百分率P为38.60%,总Shannon信息指数I为0.2006,总Nei基因多样性h为0.1349,P、I、h均表明12份桃种质资源总体水平的遗传多样性较高,这与前人研究得到的结论类似[12-14],表明桃种质资源确实存在丰富的遗传多样性。
从本研究中UPGMA聚类分析结果来看,12份桃种质资源可分为4个类群,其中‘乌桃’、‘毛桃’与其他桃品种间的亲缘关系较远,单独归为一类,这可能与‘乌桃’和‘毛桃’属于较为原始的野生种质资源有关;其余10份桃品种相互之间遗传距离较小,遗传相似度较高,亲缘关系较为接近,这可能是由于桃种质资源交流频繁,同时经常在类群间进行人为杂交用于新品种或新品系的选择,从而使得育成品种的基因存在广泛的种质渗透,这与前人的研究结果基本一致[15-19]。同时,结合所用材料的生物学特征来看,早、中、晚熟桃品种的聚类结果存在相互交叉的现象,这在史红丽等[12]的研究中也有类似的报道,究其原因,一者可能是控制早、中、晚熟性状的基因只存在几个碱基的变化,而ISSR标记无法对碱基的变异进行检测;二者可能与桃遗传多样性相对复杂有关[12]。此外,白色和黄色果肉的桃品种在聚类结果中也出现了交叉现象,这可能与自然或人为因素导致的种质迁移或基因交换有关,也可能与所采用的ISSR标记产生的扩增条带不足以将它们的遗传信息加以区分有关[13]。
桃原产于中国,最远可以追溯到周朝,逐渐传播到亚洲周边地区,后传入日本。在本研究中,来自日本的‘白丽’、‘白凤’2个桃品种与我国的3个桃品种聚在一起,表明日本品种与所选国内品种亲缘关系十分接近,这与罗慧等[1]的研究结果相符。另外,从两个蟠桃品种来看,其遗传距离为0.1310,遗传相似度为0.8772,表明经过不断的栽培和变异之后,两者的亲缘关系已经逐渐变远了。葛志刚等[20]对38个蟠桃品种进行SSR标记分析,聚类结果支持蟠桃多起源假说。同时,这也可能与分子标记本身或者在自然过程中发生了外源基因片段的渗入有关[20]。
[1]罗慧,丁广文,郭启高,等.基于ISSR标记的44份桃种质资源遗传多样性分析[J].西南大学学报(自然科学版),2015,37(5):45-50.
[2]李建辉,金则新.浙江省桐庐县长叶榧居群遗传结构的ISSR分析[J].植物研究,2011,31(6):722-728.
[3]白瑞霞,彭建营.DNA分子标记在果树遗传育种研究中的应用[J].西北植物学报,2004,24(8):1547-1554.
[4]李建辉,金则新,李钧敏.濒危植物长叶榧群体不同年龄级遗传多样性的RAPD和ISSR分析[J].浙江大学学报(理学版),2010,37(1):104-111.
[5]余贤美,艾呈祥.杧果野生居群遗传多样性ISSR分析[J].果树学报,2007,24(3):329-333.
[6]潘鸿,何新华,李一伟,等.广西野生杨梅资源遗传多样性的ISSR分析[J].果树学报,2008,25(3):353-357.
[7]代红艳,郭修武,张叶,等.山楂(Crataegus pinnatifidaBge.)遗传多样性的RAPD和ISSR标记分析[J].园艺学报,2008,35(8):1117-1124.
[8]HU D,ZHANG Z,ZHANG Q,et al.Ornamental peach and its genetic relationships revealed by Inter-simple sequence repeat(ISSR)fingerprints[J].Acta Horticulturae,2006,713:113-120.
[9]ROCHE P,ALSTON F H,MALIEPAARD C,et al.RFLP and RAPD markers linked to the rosy leaf curling aphid resistance gene(Sd1)in apple[J].Theoretical&Applied Genetics,1997,94(3):528-533.
[10]YEH F C,BOYLE T J B.Population genetic analysis of codominant and dominant markers and quantitative traits[J].Belgian Journal of Botany,1997,129:157.
[11]陈巍,王力荣,朱更瑞,等.基于SSR标记和生物学性状进行桃遗传多样性的比较分析[J].植物遗传资源学报,2009,10(1):86-90.
[12]史红丽,韩明玉,赵彩平.桃遗传多样性的SRAP和SSR标记分析[J].华北农学报,2009,24(6):187-192.
[13]廖安红,杨鑫,陈红.贵州桃种质资源ISSR标记体系建立及遗传多样性分析[J].分子植物育种,2015,13(12):2773-2781.
[14]陈红,杨鑫,安华明.贵州桃种质资源遗传多样性的SCoT分析[J].西北植物学报,2014,34(8):1559-1564.
[15]程中平,黄宏文.桃不同类群的遗传多样性及其遗传结构的RAPD分析[J].武汉植物学研究,2004,22(1):27-32.
[16]程中平,陈志伟,胡春根.利用RAPD标记对桃不同类群的遗传差异比较[J].武汉大学学报(理学版),2003,49(2):266-270.
[17]王富荣,赵剑波,佟兆国,等.桃种质资源亲缘关系的研究进展[J].植物遗传资源学报,2006,7(1):118-122.
[18]方伟超,王力荣,朱更瑞.桃(Prunus persica)品种资源结果习性遗传多样性与评价指标探讨[J].果树学报,2008,25(6):801-805.
[19]陆苏瑀,俞明亮,马瑞娟,等.硬肉桃品种群SSR标记的遗传多样性分析[J].植物遗传资源学报,2010,11(3):374-379.
[20]葛志刚,俞明亮,马瑞娟,等.蟠桃种质SSR标记的遗传多样性分析[J].果树学报,2009,26(3):300-305.

