保加利亚乳杆菌D-乳酸脱氢酶同工酶基因在大肠杆菌中的表达
黄艳娜,刘振民,游春苹
(乳业生物技术国家重点实验室,上海乳业生物工程技术研究中心,光明乳业股份有限公司乳业研究院,上海 200436)
保加利亚乳杆菌D-乳酸脱氢酶同工酶基因在大肠杆菌中的表达
黄艳娜,刘振民,游春苹*
(乳业生物技术国家重点实验室,上海乳业生物工程技术研究中心,光明乳业股份有限公司乳业研究院,上海 200436)
通过对保加利亚乳杆菌ATCC11842的D-乳酸脱氢酶基因的克隆,分离获得了ldb0101、ldb0813、ldb1010、ldb1147和ldb2021五种同工酶基因,并分别构建重组质粒转化大肠杆菌JM109,实现D-LDH同工酶基因的表达。结合重组大肠杆菌的摇瓶发酵实验和D-乳酸的合成,初步确定D-乳酸脱氢酶Ldb0101和Ldb1010在保加利亚乳杆菌中对D-乳酸的合成起着重要的作用。
保加利亚乳杆菌,D-乳酸脱氢酶,D-乳酸,基因克隆表达
保加利亚乳杆菌是乳酸菌家族中的重要一员,作为发酵剂而被广泛应用于食品工业中[1-2]。众所周知,保加利亚乳杆菌以合成D-乳酸为主,基本不合成L-乳酸[3-6]。有研究表明,D-乳酸脱氢酶(D-LDH)和L-乳酸脱氢酶(L-LDH)分别来自两个完全不同的家族,因此二者的蛋白序列同源性较低,仅有27%~37%[7]。van de Guchte等报道了保加利亚乳杆菌中存在五种D-乳酸脱氢酶同工酶(Ldb0101,Ldb0813,Ldb1010,Ldb1147,Ldb2021)[8],但对其功能特性没有做详细的说明。而目前对L.bulgaricusD-DLH的报道多集中于对其蛋白结构的研究。如Holton等研究了Ldb1010酶的蛋白构象[7];Bernard等将L.bulgaricus的D-乳酸脱氢酶基因ldhA在E.coli中表达,结果表明该酶与L.caseiD-HicDH同源[9];另外,Razeto报道了L.bulgaricusD-LDH(同Ldb0101)的闭合域、底物特异性和催化活性[10]。而目前对其他D-LDH同工酶的功能与特性未见报道,且对负责L.bulgaricusD-乳酸合成的关键酶尚无明确的结论。因此,本研究的开展将有助于了解L.bulgaricus不同D-乳酸脱氢酶同工酶催化合成D-乳酸的能力,为该酶的利用或菌种的分子改造提供依据。
重组DNA技术是目前酶学研究的常用手段[11-12]。为了解保加利亚乳杆菌中不同D-LDH同工酶的功能特性,本研究以Lactobacillusdelbrueckiispp.bulgaricusATCC11842(以下简称L.bulgaricusATCC11842)为研究对象,以其基因组DNA为模板,克隆其五种D-LDH同工酶基因,以pUC18为表达载体,在大肠杆菌中过量表达,通过摇瓶发酵实验研究该酶催化合成D-乳酸的能力。
1 材料与方法
1.1 材料与仪器
大肠杆菌JM109 本实验室保存,L.bulgaricusATCC11842 中国工业微生物菌种保藏中心(CICC);表达载体pUC18 宝生物工程大连有限公司。
限制性内切酶、Taq DNA聚合酶、T4 DNA连接酶,dNTP Mixture、预染低分量蛋白Marker,DNA Marker和凝胶回收试剂盒 宝生物工程大连有限公司;细菌基因组提取试剂盒和质粒提取试剂盒 天根北京有限公司;聚丙烯酰胺凝胶电泳相关试剂、硫酸卡那霉素和异丙基-β-D-硫代半乳糖苷(IPTG) 生工生物工程上海股份有限公司;引物设计与DNA测序分析 上海生工生物工程有限公司。

表1 引物设计
MRS培养基(Oxid,Hampshire,England)用于保加利亚乳杆菌的培养;LB培养基(胰蛋白胨10 g,酵母提取物5 g,NaCl 10 g,固体培养基添加琼脂粉15 g/L)用于大肠杆菌及其重组菌的培养。
重组大肠杆菌的发酵培养基采用M9培养基。将LB活化后的重组菌按照2%的接种量接种于M9培养基中添加终浓度为100 μg/mL的氨苄青霉素,37 ℃摇床220 r/min微好氧培养20 h,测定D-乳酸和L-乳酸的合成。
Biophotometer Plus核酸蛋白测定仪 德国艾本德股份公司;5424R台式离心机 德国艾本德股份公司;Nanodrop 2000C微量分光光度计 美国赛默飞世尔科技公司;MIR-154恒温培养箱 日本三洋电器集团;MB-102恒温震荡金属浴 杭州博日科技有限公司;Proflex PCR仪 美国ABI公司;ZHWY-2102C恒温培养振荡器 上海智诚分析仪器制造有限公司。
1.2 实验方法
1.2.1 基因克隆 参照细菌基因组提取试剂盒所述抽提保加利亚乳杆菌基因组。根据Genbank中公布的L.bulgaricusATCC11842D-乳酸脱氢酶的基因序列(ldb0101,ldb0813,ldb1010,ldb1147和1db2021),设计特异性引物(表1)。PCR扩增条件为:94 ℃预变性10 min;94 ℃变性30 s,52 ℃退火30 s,72 ℃延伸l min,30个循环;72 ℃延伸10 min。琼脂糖凝胶电泳检测PCR产物。
1.2.2 重组质粒的构建 分别将PCR产物和载体pUC18按表1所示进行限制性内切酶双酶切,胶回收纯化后,采用T4 DNA连接酶16 ℃过夜连接。连接产物热激转化到E.coliJMl09感受态细胞,37 ℃复苏1 h后涂布终浓度为100 μg/mL氨苄青霉素抗性的LB平板,培养12 h后,挑单菌落至LB液体培养基中,37 ℃培养10 h,进行菌液PCR验证,并送生工生物工程上海股份有限公司测序。对重组菌进行质粒抽提,限制性内切酶双酶切验证,
1.2.3 目的基因在大肠杆菌中的表达 将过夜培养的重组菌按照1%的接种量接种至新鲜LB培养基中,培养至对数期前期,添加终浓度为0.5 mmol/L IPTG进行诱导,诱导时间为4 h,以空质粒重组菌和未诱导重组菌为对照,收集菌体进行全细胞聚丙酰胺凝胶电泳(SDS-PAGE)。
1.2.4 菌体浓度的测定 菌体浓度采用浊度法测量。将新鲜培养的菌液适当稀释后,采用Eppendorf Biophotometer Plus核酸蛋白测定仪测定细胞浓度(OD600)。
1.2.5 乳酸的测定 分别将新鲜培养的重组大肠杆菌的种子按照2%的接种量接入装有50 mL发酵培养基的100 mL蓝口瓶中,37 ℃、220 r/min培养48 h,发酵液于4 ℃,12000 r/min离心10 min,收集上清。稀释到适当浓度后,采用D-乳酸和L-乳酸检测试剂盒(Megazyme international Ireland)测定D-乳酸和L-乳酸的浓度。
1.2.6 质粒稳定性实验 根据菌体OD600将培养液样品稀释至10-6~10-7,涂在不含氨苄青霉素抗性的LB平板上,37 ℃恒温培养箱培养过夜,待长出菌落后,将单菌落分别接种于LB抗性平板上(含100 μg/mL氨苄青霉素抗性),37 ℃恒温培养过夜,统计菌落数,其中以菌落数/接种菌落数的比值为该质粒的稳定性。
1.2.7 数据统计分析 摇瓶发酵实验中每个菌株设置三次生物学重复,每个实验重复三次,采用SPSS 17.0软件进行数据的显著性分析。
2 结果与讨论
2.1D-乳酸脱氢酶的基因克隆
以保加利亚乳杆菌基因组为模板,以表1所示为引物,PCR扩增D-乳酸脱氢酶同工酶基因。琼脂糖凝胶电泳均获得特异大小的条带(图1),测序得到该片段长度为1152 bp,并与Genbank的L.bulgaricusATCC11842的ldb0101(CAI96942.1,1002 bp)、ldb0813(CAI97635.1,969 bp)、ldb1010(CAI97812.1,1002 bp)、ldb1147(CAI97949.1,957 bp)和ldb2021(CAI98759.1,1047 bp)基因序列完全一致,证明克隆的基因正确。
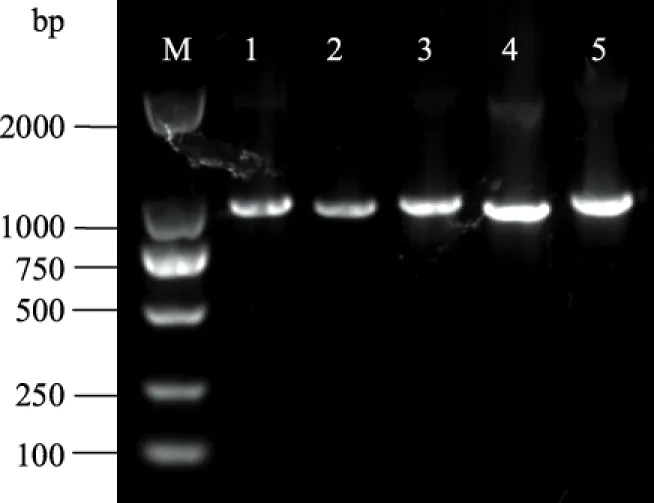
图1 D-乳酸脱氢酶基因PCR扩增电泳图Fig.1 Agarose gelelectrophoresis of D-lactate dehydrogenase genes注:M:Marker;1,ldb0101;2,ldb0813; 3,ldb1010;4,ldb1147;5,ldb2021。
2.2 表达D-乳酸脱氢酶基因的重组大肠杆菌的构建
对重组菌进行质粒抽提,并采用限制性内切酶进行双酶切验证,琼脂糖凝胶电泳结果如图2所示。从图中可看出,所抽提的重组质粒均显示了正确大小的片段,表明所挑选单克隆均为阳性。
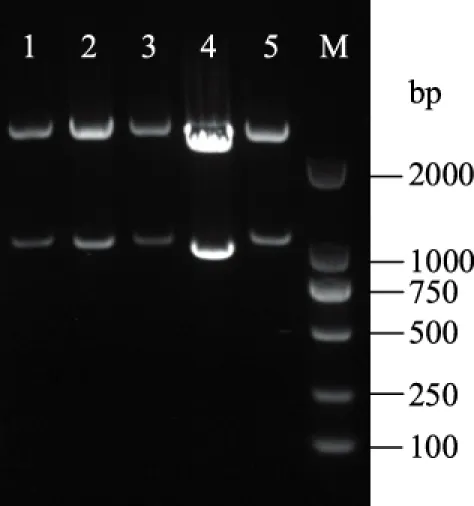
图2 重组质粒双酶切验证电泳图Fig.2 Agarose gelelectrophoresis of double restriction enzyme digestion of recombinant plasmids注:1,pUC18ldb0101;2,pUC18ldb0813;3,pUC18ldb1010; 4,pUC18ldb1147;5,pUC18ldb2021;M:Marker。
2.3 重组蛋白的聚丙烯酰氨凝胶电泳
重组菌的聚丙烯酰胺凝胶电泳结果如图3所示。与对照JM109和空质粒JM109/pUC18相比,重组菌JM109/pUC18ldb0101、JM109/pUC18ldb0813、JM109/pUC18ldb1010和JM109/pUC18ldb2021均有特异蛋白表达,且蛋白大小正确,大小为35 kDa左右。从SDS-PAGE图中可以看出,在上样量相同的情况下,五种不同的D-LDH同工酶在E.coli中的表达强度不同,以Ldb0101和Ldb1010重组蛋白的表达量最高,其次为Ldb0813和Ldb2021,而Ldb1147蛋白未见明显表达。原因尚不明确,但这也从侧面反应了各D-LDH同工酶在保加利亚乳杆菌中的表达水平。

图3 重组蛋白的聚丙烯酰胺凝胶电泳Fig.3 Extracellular protein profiles on SDS-PAGE of engineered E. coli carrying different D-LDH genes注:泳道1,JM109;泳道2,JM109/pUC18;泳道3,0 mmol/L IPTG JM109/pUC18ldb0101;泳道4,0.5 mmol/L IPTG JM109/pUC18ldb0101;泳道5,0 mmol/L IPTG JM109/pUC18ldb0813;泳道 6,0.5 mmol/L IPTG JM109/pUC18ldb0813;泳道 7,0 mmol/L IPTG JM109/pUC18ldb1147;泳道 8,0.5 mmol/L IPTG JM109/pUC18ldb1147;泳道9,0.5 mmol/L IPTG JM109/pUC18ldb2021;泳道10,0.5 mmol/L IPTG JM109/pUC18ldb1010。
2.4 重组大肠杆菌D-乳酸合成的摇瓶发酵实验
为了解不同D-乳酸脱氢酶催化丙酮酸合成D-乳酸的性能,本研究对表达D-LDH基因的重组大肠杆菌进行微好氧摇瓶发酵实验,并以表达空质粒重组菌为对照,结果如表2所示。与对照相比,表达D-LDH基因的重组菌细胞的生长均受到不同程度的抑制,其中以JM109/pUC18ldb1010细胞浓度最低,这主要是由于重组蛋白的表达所带来的代谢负荷所致[14]。而从D-乳酸的合成情况来看,重组大肠杆菌JM109/pUC18ldb0101较其他菌表现出更好的合成性能,最终D-乳酸的含量为949.6 mg/L;其次为菌株JM109/pUC18ldb1010,D-乳酸的浓度为673.9 mg/L。相比之下,其余两株重组菌D-乳酸合成能力均较低,而JM109/pUC18ldb1147和对照菌株JM109/pUC18不合成D-乳酸。从摇瓶发酵的结果发现,重组菌合成D-乳酸的能力与SDS-PAGE中重组蛋白的表达量呈正相关。因此,结合重组蛋白的表达和D-乳酸的合成,推测Ldb0101和Ldb1010在保加利亚乳杆菌合成D-乳酸的过程中起着较其他同工酶更为重要的作用。

表2 重组大肠杆菌摇瓶发酵合成乳酸的性能比较
如表2所示,各重组菌的L-乳酸的含量均较低。JM109/pUC18ldb0101和JM109/pUC18ldb1010L-乳酸的浓度较对照无显著差异,而其余三株重组菌L-乳酸含量较对照均有所下降。综合D-乳酸和L-乳酸的合成,JM109/pUC18ldb0101和JM109/pUC18ldb1010具有较其他重组菌优良的乳酸合成性能。
Hao pei等[13]对L.bulgaricus2038进行了全基因组测序和功能注释,他们认为,L-LDH在细胞中是不表达的,而D-LDH在体内表达水平较高,其中ldhD1(LBU0066)表达量最高,密码子适应指数(Condon adaption index,CAI)为0.575,而ldhD2(LBU0860)和ldhD3(LBU1637)表达水平适中,CAI分别是0.369和0.320。这也就表明了保加利亚乳杆菌中D-LDH同工酶的表达确实存在着不同程度的差异[15],这在本研究中重组D-LDH蛋白的表达中也可以得到证实。另外,从上述研究也可以发现,不同的保加利亚乳杆菌菌株含有不同的D-LDH同工酶,具有菌株差异性。
2.5 重组质粒的稳定性分析
根据本文1.2.6所述的方法,本实验测定了重组大肠杆菌的质粒稳定性(结果未显示)。结果表明,各重组菌均可以表现出较好的稳定性,好氧发酵12 h各重组菌的质粒稳定性均保持在98%以上,且差异不显著。
3 结论
为了解保加利亚乳杆菌五种D-乳酸脱氢酶同工酶基因对细胞合成D-乳酸的作用,本研究以L.bulgaricusATCC11842基因组DNA为模板,克隆得到D-LDH同工酶基因(ldb0101、ldb0813、ldb1010、ldb1147和ldb2021),构建重组质粒并转化大肠杆菌JM109,共构建五株重组菌株,实现了D-LDH同工酶基因在大肠杆菌中的表达。对重组菌进行了微好氧摇瓶发酵实验,结果显示JM109/pUC18ldb0101和JM109/pUC18ldb1010较其他重组菌合成更多的D-乳酸,其中JM109/pUC18ldb0101合成量最高,其次为JM109/pUC18ldb1010。因此,综合重组蛋白在大肠杆菌中的表达量和D-乳酸的合成两个方面,推测D-乳酸脱氢酶Ldb0101和Ldb1010对保加利亚乳杆菌D-乳酸的合成起着较为重要的作用,其他三种D-LDH同工酶的功能有待进一步研究。
[1]Salminen S,von Wright A,Ouwehand A C. Lactic acid bacteria:Microbiological and functional aspects(3rd ed.)[M]. New York:Marcel Dekker,Inc.. ISBN 0-8247-5332-1,2004.
[2]Ginovart M,Lopez D,Valls J,et al. Simulation modeling of bacterial growth in yogurt[J]. Int. J. Food Microbiol,2002,73(2-3):415-425.
[3]Manome A,Okada S,Uchimura T,et al. The ratio of L-form to D-form of lactic acid as a criteria for the identification of lactic acid bacteria[J]. J. Gen. Appl. Microbiol.,1998,44(6):371-374.
[4]Abdel-Rahman M A,Tashiro Y,Sonomoto K. Lactic acid production from lignocelluloses derived sugars using lactic acid bacteria:overview and limits[J]. J Biotechnol,2011,156(4):286-301.
[5]Abdel-Rahman M A,Tashiro Y,Sonomoto K. Recent advances in lactic acid production by microbial fermentation processes[J]. Biotechnol Adv,2013,31(6):877-902.
[6]Gasser F. Electrophoretic characterization of lactic dehydrogenases in the genus Lactobacillus[J]. J Gene Microbiol,1970,62(2):223-239.
[7]Holton S J,Anandhakrishnan M,Geerlof A,Wilmanns M:Structural characterization of a D-isomer specific 2-hydroxyacid dehydrogenase fromLactobacillusdelbrueckiissp. bulgaricus[J]. Journal of structural biology,2013,181(2):179-184.
[8]van de Guchte,M,Penaud S,Grimaldi C,et al. The complete genome sequence of Lactobacillus bulgaricus reveals extensive and ongoing reductive evolution[J]. Proc. Natl. Acad. Sci.,2006,103(24):9274-9279.
[9]Bernard,N,Johnsen K,Ferain T,et al.,NAD(+)-dependent D-2-hydroxyisocaproate dehydrogenase ofLactobacillusdelbrueckiisubsp. bulgaricus. Gene cloning and enzyme characterization[J]. Eur. J. Biochem.,1994,224(2),439-446.
[10]Razeto A,Kochhar S,Hottinger H,et al. Domain Closure,Substrate Specificity and Catalysis of D-Lactate dehydrogenase from Lactobacillus bulgaricus[J]. J. Mol. Biol.,2002,318(1):109-119.
[11]杨登峰,潘丽霞,黄日波,等. 产高纯度L-乳酸大肠杆菌基因工程菌的初步研究[J]. 现代食品科技,2010,26(2):126-128.
[12]Chang D E,Jung H C,Rhee J S,et al. Homofermentative production of D-or L-lactate in metabolically engineeredEscherichiacoliRR1[J]. Appl Environ Microb,1999,65(4):1384-1389.
[13]Hao P,Zheng H J,Yu Y,et al. Complete sequencing and pan-genomic analysis ofLactobacillusdelbrueckiisubsp. bulgaricus reveal its genetic basis for industrial yogurt production[J]. PLoSONE,2011,6(1):e15964.
[14]Wang C,Zhang J,Wu H,et al. Heterologous gshF gene expression in various vector systems inEscherichiacolifor enhanced glutathione production[J]. Journal Biotechnol 2015,214(20):63-68.
[15]Sharp P M,Li W H. The codon adaptation index-a measure of directional synonymous codon usage bias,and its potential applications[J]. Nucleic Acids Res,1987,15(3):1281-1295.
Over-expression ofD-lactate dehydrogenase isoenzyme genes fromLactobacillusdelbrueckiispp.bulgaricusinEscherichiacoli
HUANG Yan-na,LIU Zhen-min,YOU Chun-ping*
(State Key Laboratory of Dairy Biotechnology,Shanghai Engineering Research Center of Dairy Biotechnology,Dairy Research Institute,Bright Dairy & Food Co.,Ltd.,Shanghai 200436,China)
FiveD-LDH isoenzyme genes(ldb0101,ldb0813,ldb1010,ldb1147 and ldb2021)fromL.bulgaricusATCC11842 were separated and over-expressed inEscherichiacoliJM109 using pUC18 as the expression plasmid. The shake-flask fermentation of recombinantE.coliindicated that theD-LDH enzymes Ldb0101 and Ldb1010 played more important roles inD-lactic acid biosynthesis inL.bulgaricus.
Lactobacillusbulgaricus;D-lactate dehydrogenase;D-lactic acid ATCC11842;gene clone and expression
2016-09-06
黄艳娜(1980-),女,博士,工程师,研究方向:益生菌代谢工程及代谢调控,E-mail:huangyanna@brightdairy.com。
*通讯作者:游春苹(1982-),女,博士,高级工程师,研究方向:益生菌代谢工程及代谢调控,E-mail:youchunping@brightdairy.com。
国家“十二五”科技支撑计划(2013BAD18B01,2013BAD18B02);上海市闵行区领军人才项目(201443);上海科学技术委员会项目(16DZ2280600)。
TS201.3
A
1002-0306(2017)05-0159-05
10.13386/j.issn1002-0306.2017.05.021

