词袋模型在蛋白质亚细胞定位预测中的应用
赵 南, 张 梁, 薛 卫*, 王雄飞, 任守纲
(1.南京农业大学 信息科学技术学院,江苏 南京210095;2.江南大学 粮食发酵工艺与技术国家工程实验室,江苏 无锡214122)
词袋模型在蛋白质亚细胞定位预测中的应用
赵 南1, 张 梁2, 薛 卫*1, 王雄飞1, 任守纲1
(1.南京农业大学 信息科学技术学院,江苏 南京210095;2.江南大学 粮食发酵工艺与技术国家工程实验室,江苏 无锡214122)
运用词袋模型结合传统的蛋白质特征提取算法提取蛋白质序列特征,采用K-means算法构建字典,计算获得蛋白质序列的词袋特征,最终将提取的特征值送入SVM多类分类器,对数据集中蛋白质的亚细胞位置进行预测,在一定程度上提高了亚细胞定位预测的准确率。
词袋模型;K-means;支持向量机;亚细胞定位预测
人类对生命科学的研究因计算机技术的蓬勃发展发生了巨大变化,自从进入后基因组时代,人类获得了大规模的核酸和蛋白质序列数据,借助先进高效的计算机自动化数据处理技术[1]从这些海量数据中挖掘有效信息成为必然趋势。国内外学者在以往的研究中,主要采用数学方法描述提取的蛋白质序列特征信息,用高维的特征向量表示蛋白质序列,然后设计使用高效的分类器进行预测分析。
目前,用于蛋白质序列特征提取的算法主要包括:氨基酸组成(AAC)、氨基酸的物化特性、二肽及多肽组成、伪氨基酸组成(PseAAC)以及不同特征的融合等[2-6]。如Lin等[4]的蛋白质亚细胞定位预测研究采用了四肽信息;杨会芳等[5]在预测蛋白质亚细胞定位中采用了分段伪氨基酸的特征提取方法;Gao等[6]通过寻找蛋白质不同结构与物化特性的最佳组合来区分外膜蛋白。同时,在预测算法的设计方面国内外研究者开展了大量工作,统计学和机器学习方法在已有的预测算法中得到了充分应用,如陈颖丽等[7]在6类细胞凋亡蛋白的亚细胞定位研究中使用了离散增量结合支持向量机的方法;还有基于人工神经网络、马尔可夫模型和贝叶斯网络等的分类预测方法[8-9]。
总结前人研究成果不难发现,单纯采用传统的蛋白质序列特征提取算法如AAC等,进行特征提取并送入分类器进行定位预测的准确率偏低。为了改善这一问题,作者引入词袋模型 (Bag of Words Model,简称BOW模型),BOW模型源自文档处理领域,也被广泛应用于图像分类方法中。不考虑语法和词序,收集所有文档中出现过的单词,形成一本字典,然后统计获得文档中出现过的单词及其出现的频率[10],将文档表示成高维的向量。作者使用词袋模型完成序列信息的提取,实验证明结合使用BOW模型与传统序列特征提取算法AAC和PseAAC完成蛋白质序列特征的提取,并使用支持向量机分类方法进行定位预测,能有效提高识别精度。
1 材料与方法
1.1 数据集
采用两个凋亡蛋白数据集,第一个数据集由Zhou和Doctor[11]构建,该数据集包含98条凋亡蛋白质序列,分为四个亚细胞定位类别,分别是43个细胞质蛋白、30个膜蛋白、13个线粒体蛋白和12个其它类蛋白;第二个数据集是由Chen和Li[12]构建,该数据集包含317条蛋白质序列,总共有6个亚细胞定位类别,分别是112个细胞质蛋白、55个膜蛋白、34个线粒体蛋白、17个分泌蛋白、52个细胞核蛋白和47个内质网蛋白。这两个数据集的蛋白质序列均从SWISS-PROT数据库获得。
1.2 蛋白质序列的词袋特征
BOW模型描述文档的方法是用D表示一个存在的文档集合,由M个文档组成,提取M个文档中出现过的单词,假设不同的单词个数为N,由这N个单词构成字典,则每一个文档都可以被表示成一个N维的向量[13]。同理,一个蛋白数据集包含若干条蛋白质序列,连续选取每一条蛋白质序列的若干个片段,称这样的片段为序列单词,分别采用传统的序列特征提取算法AAC和PseAAC统计序列单词的氨基酸组分信息和位置信息,用向量表示,称这样的向量为序列单词特征;然后采用K-means聚类算法对所有的序列单词特征进行聚类分析,聚类分析之后所得到的所有聚类中心的集合,称为字典,字典的大小由聚类中心的个数k决定,所有的序列单词特征将映射到字典中的各个聚类中心;逐一统计每一条蛋白质序列属于各个聚类中心的序列单词个数,从而绘制出每一条蛋白质序列的序列单词直方图,计算各个聚类中心上序列单词个数占该条蛋白质序列序列单词总数的比例即可得到蛋白质序列的词袋特征,则每一条蛋白质序列都可以用一个k维向量来表示。此方法主要分为5个步骤:
1)分割数据集中所有的蛋白质序列产生若干个序列单词;
2)提取序列单词的序列单词特征;
3)对序列单词特征进行聚类分析,获得字典,字典大小为聚类中心个数k;
4)经聚类分析后序列单词特征被映射到字典中的各个聚类中心,统计每一条蛋白质序列属于各个聚类中心的序列单词个数,获得蛋白质序列的序列单词直方图;
5)对每一条蛋白质序列计算各个聚类中心上序列单词个数占该条蛋白质序列序列单词总数的比例,从而获得蛋白质序列的词袋特征,每一条蛋白质序列被表示成一个k维的向量。
词袋特征提取过程见图1。
1.2.1 序列单词特征提取 提取特征前对蛋白质序列进行分割处理,分割蛋白质序列可采用均匀分割和滑动窗口分割。均匀分割法是把每条蛋白质序列均匀分割为多个序列单词,得到的大量序列单词的集合构成构建字典的基础。滑动窗口方法则每间隔一定数量截取窗口内的蛋白质序列片段作为一个序列单词,设定不同的间隔字符个数和窗口大小可以得到不同长度的序列单词。

图1 词袋特征提取过程Fig.1 Bag of words feature extraction process
主要采用滑动窗口分割法,从序列的N端到C端每次滑动间隔固定为1,窗口大小决定序列单词的长度,选取方法如下:
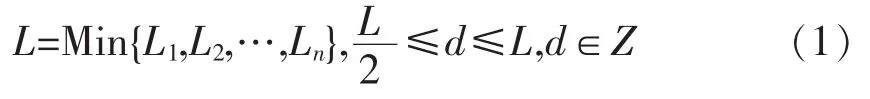
其中L1,L2,…,Ln表示数据集中所有蛋白质序列的长度,L为数据集中最短蛋白质序列的长度,d为滑动窗口大小,即序列单词长度在与L之间选取。
分割后统计序列单词的组分信息和位置信息,运用BOW模型结合已有的AAC和PseAAC算法,采用两种统计方法,分别称为BOW_AAC和BOW_PseAAC。
设序列单词P为:

其中R1R2R3R4R5表示序列单词P的第一到第五个氨基酸残基,以此类推,RL表示序列单词P的最后一个氨基酸残基。
1)BOW_AAC序列单词特征提取:P的氨基酸组分信息定义如公式(3)[2]所示:

f1f2…f20的计算用公式(4)求解:

其中,fu(u=1,2,3,…,20)表示20种氨基酸在序列单词中出现的频率,L表示一个序列单词的长度,N表示一个序列单词包含的所有氨基酸残基的总数目,A(u)表示序号u所对应的氨基酸残基。经过统计计算,所有的序列单词都可以用一个20维的向量表示,从而获得所有蛋白质序列的序列单词特征。
2)BOW_PseAAC序列单词特征提取:假设序列单词有L个氨基酸残基,表示同公式(2),任意一个氨基酸残基在同一个序列单词中与其他氨基酸残基存在不同程度的相关作用,用序列相关因子定义氨基酸残基之间的相关性[14],定义如公式(5)[15]所示:
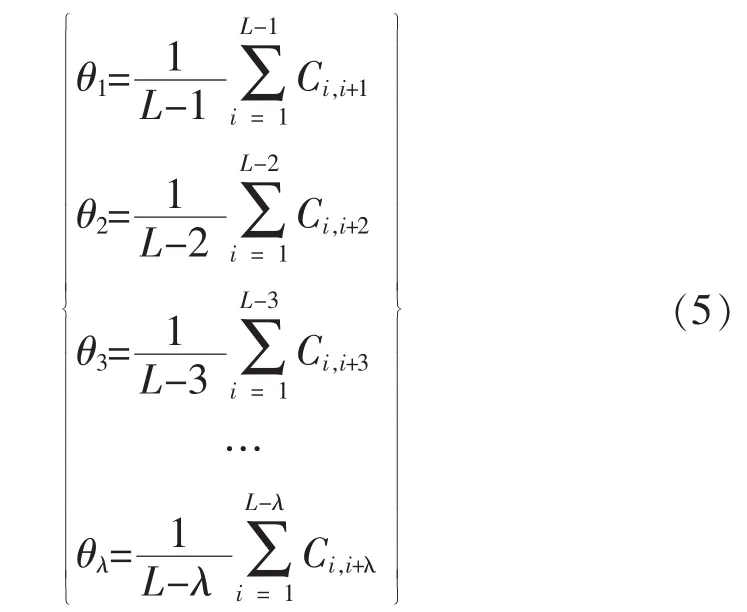
其中,θ1表示第一级相关因子,反映序列单词中相邻两个氨基酸残基之间的相关性;θ2表示第二级相关因子,反映序列单词中每间隔一个氨基酸残基的两个氨基酸残基之间的相关性;θ3表示第三级相关因子,反映序列单词中每间隔两个残基的两个氨基酸残基之间的相关性;以此类推。Ci,j是根据氨
基酸残基的疏水性、亲水性和侧链分子量构建的相关函数,定义如公式(6)[15]所示:

其中,H1(Rj)表示Rj的疏水性值,H1(Ri)表示Ri的疏水性值;H2(Rj)表示Rj的亲水性值,H2(Ri)表示Ri的亲水性值;M(Rj)表示Rj的侧链原子量,M(Ri)表示Ri的侧链原子量。然后序列单词特征可表示为:

其中
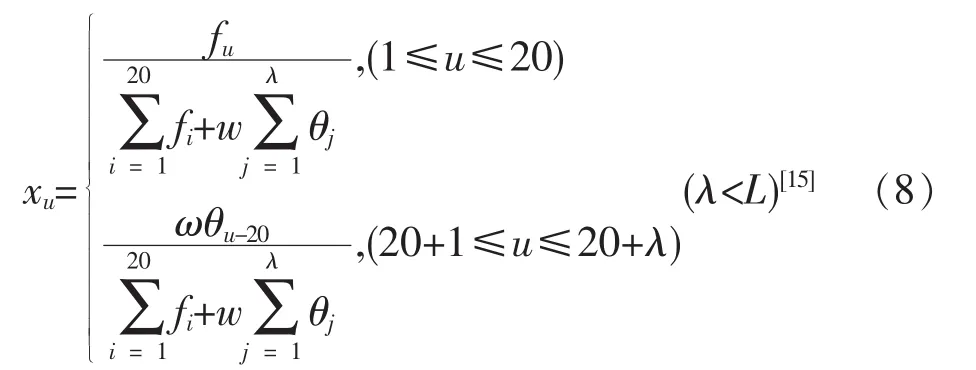
λ表示选取的相关因子类型数目,fi表示序列单词中第i种氨基酸出现的频率,w表示序列顺序效应的权重因子,θj表示序列单词中第j级序列相关因子。
1.2.2 构建字典 得到序列单词特征之后,下一步即是对这些特征值进行处理,用K-means聚类算法构建字典,聚类中心的个数即为字典的大小。核心思想是按照类内方差和最小的原则将n个序列单词特征值分为指定的k类,k的选取方法为:

即聚类中心个数从20开始逐一递增选取,结合序列单词长度d的选取,可以找到一组(d,k)使获得的词袋特征具有最高的识别精度。而类内方差和最小的定义如公式(10)[16]所示:

其中,Si(i=1,2,…,k)表示聚类中心位置是μi的第i个聚类类别,xj为属于聚类类别Si的特征值。利用K-means聚类算法构建字典的过程描述如下:
输入:DS:n个序列单词特征值组成的数据集合,k:聚类中心的个数。
输出:k个聚类中心的集合即字典。
算法:
1)从DS中任意选取k个序列单词特征值作为初始聚类中心;
2)计算每个序列单词特征值与各聚类中心的距离,按照最近距离原则将n个特征值分配到以k个初始中心为代表的聚类类别中;
3)根据步骤2得到的结果对新产生的k个类别进行中心计算,得到新的聚类中心;
4)重复步骤2~3,直至达到终止条件,如聚类中心不再变化或者已达到最大迭代次数等。
1.3 支持向量机
支持向量机(SVM)拥有坚实的理论基础,并且数学模型简单明了,在解决高维模式识别问题中具有泛化能力强、分类效率高等优点[17]。借助林智仁等开发设计的LIBSVM工具箱用一对一法构造SVM多类分类器,为任意两类样本设计一个SVM,当存在一个未知样本需要分类时,它的类别取得票最多的那个类别。基于这样的SVM分类实验,在提取出蛋白质序列的词袋特征之后,主要是选取最佳惩罚参数c和核函数参数g的问题,作者通过交叉验证选择最佳参数,调用工具箱中的SVMcgForClass函数将c和g划分网格进行搜索,最佳参数是达到最高验证分类准确率时最小参数c对应的那组c和g,如果存在多组g对应最小参数c,则最佳参数是搜索到的第一组c和g。然后将训练样本(Ci,yi)送入分类器,向量Ci表示第i组训练样本的词袋特征值,yi表示该条蛋白质序列所对应的亚细胞位置,最后送入测试样本并统计预测结果。
2 结果与讨论
为了检验方法的预测性能,采用Jackknife检验,每次仅从数据集中选取一条蛋白质序列构成测试集,训练集由剩余的蛋白质序列构成,测试次数等于数据集的大小,这种检验方法具有最小的任意性,是一种客观有效的交叉验证方法[18]。最后将本文方法BOW_AAC_SVM和BOW_PseAAC_SVM在98和317数据集上的预测结果列于表1-2。为了方便比较,将运用传统蛋白质序列特征提取算法氨基酸组成(AAC)和伪氨基酸组成(PseAAC)进行特征提取并送入SVM分类器得到的预测成功率一并列出,如表中AAC_SVM和PseAAC_SVM两行所示,同时在表 1的第一行列出了 G.P.ZHOU和K.DOCTOR[11]利用氨基酸组成提取特征值以及采用Jackknife进行检验的实验结果。
从表1可以看出,在98数据集上直接采用AAC、PseAAC特征提取算法的总体预测精度分别是80.2%和83.3%,用BOW模型结合AAC、PseAAC提取的特征值的总体识别精度达到了90.6%和91.7%,分别提高了10.4%和8.4%,对于每一个亚细胞类,也都有不同程度的提高,在传统方法预测成功率较低的Mitochondrial和Other亚细胞类上最高提升了23%~25%,尤其在最后一个亚细胞类上将AAC_CCA方法的预测成功率由 25%提高到了83.3%。通过表2的比较发现,运用BOW模型的总体预测精度也比传统方法高出6.7%和6.9%,在各个亚细胞类上也都有不同程度的提高,在Nuclear亚细胞类上分别提升了15.7%和11.8%,在Secreted上比传统方法高出23.6%。
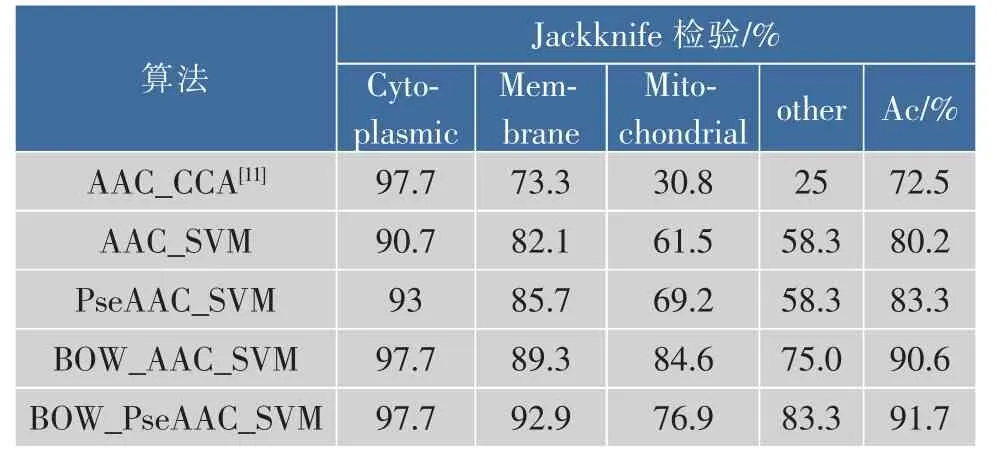
表1 98数据集结果比较Table 1 Comparison of the results of 98 data sets

表2 317数据集结果比较Table 2 Comparison of the results of 317 data sets
3 结语
作者引入词袋模型应用于蛋白质亚细胞定位预测中,主要技术包括:蛋白质序列分割——滑动窗口法,用来获得大量序列单词的集合,作为构建字典的基础;序列单词特征提取——BOW_AAC与BOW_PseAAC,运用词袋模型结合传统的蛋白质特征提取算法统计蛋白质序列的氨基酸组分信息和位置信息;构建字典——Kmeans算法,对所有的序列单词特征进行聚类分析处理,再通过统计计算获得蛋白质序列的词袋特征;亚细胞定位预测——SVM多类分类器,对数据集中蛋白的亚细胞位置进行预测。预测准确率较传统的蛋白质序列特征提取算法有所提升,最高达到了91.7%,尤其在传统方法预测准确率较低的亚细胞类上识别精度明显提高,如在98数据集other这一亚细胞分类上,预测成功率提高了25%,在317数据集Secreted这一亚细胞分类上,预测成功率也提高了20%以上,对准确预测未知蛋白质的亚细胞位置具有重要作用。此次在特征提取方面做了研究工作并取得了一些成果,接下来将在滑动窗口大小和聚类中心个数的选取方法上做一些改进,并尝试在预测算法设计方面做一些工作,重点关注集成学习以及深度学习等。
[1]QIAO Shanping,YAN Baoqiang.The research review of protein subcellular localization prediction[J].Application Research of Computers,2014,31(2):321-327.(in Chinese)
[2]CHOU Kuochen.Some remarks on protein attribute prediction and pseudo amino acid composition[J].Journal of Theoretical Biology,2011,273(1):236-247.
[3]FAN Guoliang,LI Qianzhong.Predictingprotein submitochondrialocations by combining different descriptors into the general form of Chou’s pseudo amino acid composition[J].Amino Acids,2012,43(2):545-555.
[4]LIN Hao,CHEN Wei,YUAN Lufeng,et al.Using over-represented tetrapeptides to predict protein submitochondria locations[J]. Acta Biotheoretica,2013,61(2):259-268.
[5]YANG Huifang,CHENG Yongmei,ZHANG Shaowu,et al.Based on the pseudo amino acid composition feature extractionmethod to predict protein subcellular localization[J].Acta Biophysica Sinica,2008,24(3):232-238.(in Chinese)
[6]GAO Qingbin,YE Xiaofei,JIN Zhichao,et al.Improving discrimination of outer membrane proteins by fusing different forms of pseudo amino acid composition[J].Analytical Biochemistry,2009,398(1):52-59.
[7]CHEN Yingli,LI Qianzhong,YANG Keli,et al.Based on the discrete incremental support vector machine method of apoptosis protein subcellular location prediction[J].Acta Biophysica Sinica,2007,23(3):192-198.(in Chinese)
[8]ZOU Lingyun,WANG Zhengzhi,HUANG Jiaomin.Prediction of subcellular localization of eukaryotic proteins using position-specific profiles and neural network with weighted inputs[J].Journal of Genetics and Genomics,2007,34(12):1080-1087.
[9]ZHANG Shubo,LAI Jianhuang.Machine learning-based prediction of subcellular localization for protein[J].Computer Science,2009,36(4):29-33,49.(in Chinese)
[10]ZHAO Chunhui,WANG Ying,Masahide KANEKO.An optimized method for image classification based on bag of words model [J].Journal of Electronics&Information Technology,2012,34(9):2064-2070.(in Chinese)
[11]ZHOU Guoping,DOCTOR Kutbuddin.Subcellular location prediction of apoptosis proteins[J].Proteins,2002,50(1):44-48.
[12]CHEN Yingli,LI Qianzhong.Prediction of the subcellular location of apoptosis proteins[J].Journal of Theoretical Biology,2006,245(4):775-783.
[13]YANG Quan,PENG Jinye.Chinese sign language recognition research using SIFT-BoW and depth image information[J]. Computer Science,2014,41(2):302-307.(in Chinese)
[14]MA Junwei,GAO Xinzhong,ZHANG Jie.Study on the sequence encoding method of protein subcellular location prediction[J]. Computer Science,2012,39(11A):283-287,312.(in Chinese)
[15]CHOU Kuochen.Prediction of protein cellular attributes using pseudo-amino acid composition[J].Proteins,2001,43(3):246-255.
[16]LEI Xiaofeng,XIE Kunqing,LIN Fan.An efficient clustering algorithm based on local optimality of K-Means[J].Journal of Software,2008,19(7):1683-1692.(in Chinese)
[17]GU Yaxiang,DING Shifei.Advances of support vector machines[J].Computer Science,2011,38(2):14-17.(in Chinese)
[18]WANG Wei,ZHENG Xiaoqi,DOU Yongchao,et al.Prediction of protein subcellular location using optimal cleavage site[J]. Bioinformatics,2011,9(2):171-175,180.(in Chinese)
Application of Bag of Words Model in the Prediction of Protein Subcellular Location
ZHAO Nan1, ZHANG Liang2, XUE Wei*1, WANG Xiongfei1, REN Shougang1
(1.School of Information Science and Technology,Nanjing Agricultural University,Nanjing 210095,China;2. National Engineering Laboratory for Cereal Fermention Technology,Jiangnan University,Wuxi 214122,China)
Predecessors have done a lot of work in the feature extraction of protein and subcellular localization prediction.Previous studies showed that prediction accuracy obtained by traditional feature extraction algorithm is low.In order to improve accuracy,bag of words model combined with traditional protein features extraction algorithm is used to extract feature of protein sequence in this study.Firstly,K-means algorithm is used to construct feature dictionary.Then bag of words features of protein sequences are counted by dictionary.Finally extracted feature is inputted into SVM classifier to forecast the protein subcellular location.Results showed that predictionaccuracy of subcellular localization has been improved.
bag of words model,K-means,support vector machine,subcellular localization prediction
TP 391.4
A
1673—1689(2017)03—0296—06
2015-03-10
中央高校基本科研业务费专项资金项目(KYZ201668);江苏省自然科学基金项目(BK2012363,BK2011153);江苏省博士后科研计划项目(1302038B)。
*通信作者:薛 卫(1979—),男,江苏南通人,理学博士,副教授,硕士研究生导师,主要从事生物信息、模式识别方面的研究。
E-mail:xwsky@njau.edu.cn
赵南,张梁,薛卫,等.词袋模型在蛋白质亚细胞定位预测中的应用[J].食品与生物技术学报,2017,36(03):296-301.

