利用拮抗、ITS和RAPD技术对灵芝属菌株分类的研究
[KH-*3D]王锦锋,李晶,I.L.Datti,张健,林志魁,林占熺
(1.福建农林大学园艺学院,福建福州350002;2.国家菌草工程技术研究中心,福建福州350002;3.福建农林大学生命科学学院,福建福州350002)
利用拮抗、ITS和RAPD技术对灵芝属菌株分类的研究
[KH-*3D]王锦锋1,2,李晶1,2,I.L.Datti2,3,张健2,3,林志魁2,3,林占熺2*
(1.福建农林大学园艺学院,福建福州350002;2.国家菌草工程技术研究中心,福建福州350002;3.福建农林大学生命科学学院,福建福州350002)
为了更科学的对灵芝菌株进行分类与鉴定,利用拮抗、ITS和RAPD技术对21株灵芝菌株进行分类鉴定和遗传多样性分析。拮抗试验结果表明,大部分菌株之间存在不同程度的拮抗作用,少数菌株之间没有拮抗或拮抗极不明显;ITS序列分析结果表明,21个菌株分为3个组,菌株间遗传距离的变化范围在0.000~0.072,平均遗传距离为0.039,菌株1号(赤68)与3号(Ga15)的遗传距离为0.000,证明其亲缘性较近;RAPD结果表明,各菌株间的相似系数在0.83以上,所有菌株分为两个组,4号(867)菌株单独为1组,其它20个菌株为1组;相似系数在0.932的相似水平上,所有菌株分为14个组;1号菌株与3号菌株的相似系数则超过了0.95,有可能是同种异名。3种方法其结论一致,且能准确鉴定21株灵芝亲缘关系。
拮抗试验;ITS序列分析;RAPD技术;遗传多样性;亲缘性
灵芝(Ganoderma lucidum Curtis P.Karst.)又称瑞芝、瑞草,早在《神农本草经》和《本草纲目》中就有记载其药用价值,在我国已有2000多年的栽培及应用历史[1]。1881年,灵芝首先被英国真菌学家Karsten命名,当时该属只有Polyporus lucidus一个种,因而这个种就成了单模式标本,但遗憾的是没有真正的灵芝标本材料保存下来[2]。随后各国的分类学家就根据认为重要的形态特征对各自收集到的菌株进行分类和定名,因此出现了不同地域有不同的灵芝菌种命名,导致了灵芝属的命名混乱,同名异种和同种异名的现象普遍存在[3]。随着灵芝产品的开发,建立一套灵芝菌种的保障体系尤为重要。拮抗性实验因其快速、便捷等优点,在真菌分类学上被广泛的应用,成为了初步研究菌株分类的一种方法[1-2]。White等[4-5]为真菌rRNA基因的ITS设计了3对特异引物可用于大多数担子菌和子囊菌。Williams和Welsh开创RAPD技术以来,该技术在食(药)用菌研究领域已得到广泛的应用[6-7]。本文应用拮抗试验、ITS序列分析和RAPD分子标记技术对收集的菌株进行亲缘关系研究,更好地对灵芝菌株进行有效的鉴别和分类,进一步开发利用灵芝资源,为灵芝产品进入国际市场提供科学的依据。
1 材料与方法
1.1 供试菌株的实验编号及相关信息
本实验所用菌株均由国家菌草工程技术研究中心提供,具体编号见表1。
表1 供试菌株实验编号Table 1Sources of strains and tested number
1.2 试剂与仪器
DNA提取试剂盒和胶回收盒购于OMEGA公司,2×Taq PCR MasterMix和6×Loading Buffer购于TIANGEN公司,Marker Plus(D2000 Plus)购于Gen-Star公司,核酸染料和RNaseA购于索莱宝公司。
S1000TMThermal Cycler型PCR仪和PowerPacTMBasic型电泳槽购于BIO-RAD公司,G:Box F3型高级凝胶成像系统购于SYNGENE公司。
1.3 供试培养基
PDA培养基:马铃薯200 g煮熟30 min后过滤,滤液中加入葡萄糖20 g,KH2PO41.0 g,MgSO4· 7H2O 0.5 g,定容至1 L,pH自然。
1.4 基因组DNA的提取
1.4.1 菌丝体培养将灵芝菌种接种到灭菌后的PDA液体培养基中,于28℃,150 r/min恒温摇床中培养8~10 d,收集新鲜菌丝体备用。
1.4.2 DNA提取与检测按OMEGA DNA提取试剂盒进行样品DNA提取,提取的样品DNA在1%琼脂凝胶电泳(120 V,30 min),核酸染料染色,在凝胶成像系统下观察DNA的电泳条带。
1.5 拮抗实验
担子菌体细胞的非亲和性(Samatic incompatibility)即高等担子菌在双核菌丝阶段发生的认识自我和排斥异己的过程。遗传背景明显不同的个体在交界区会形成明显的分界线,该现象称为拮抗反应[8],是体细胞非亲和性的具体体现。将不同菌株活化后,在同一平板上按“品”字形接种3个菌株,25℃恒温下培养,观察菌丝间的拮抗反应情况并做记录。
1.6 ITS实验
选用两条特异引物ITS1和ITS4对21个菌株的基因组DNA进行特异性扩增(表2)。25 μl的反应体系为:2×Taq PCR MasterMix 12.5 μl、引物ITS1 1 μl、引物ITS4 1 μl、DNA 1 μl、ddH2O 9.5 μl。ITS扩增反应程序为:94℃预变性5 min,然后94℃变性45 s,55℃退火45 s,72℃延伸1 min,进行30次循环后72℃延伸10 min。反应结束后,取10 μl PCR产物于1.5%琼脂糖凝胶上电泳后拍照记录,纯化后送专业机构测序。
1.7 RAPD实验
选用10个随机引物对21个菌株进行扩增(表3)。25 μl的反应体系为:2×Taq PCR MasterMix 12.5 μl、Primer 1 μl、DNA 1 μl、ddH2O 9.5 μl。RAPD扩增反应程序为:92℃预变性5 min,然后92℃变性1 min,37℃退火1 min,72℃延伸2 min,进行35次循环后72℃延伸10 min。反应结束后,取10 μl PCR产物于1.5%的琼脂凝胶上电泳后拍照记录。
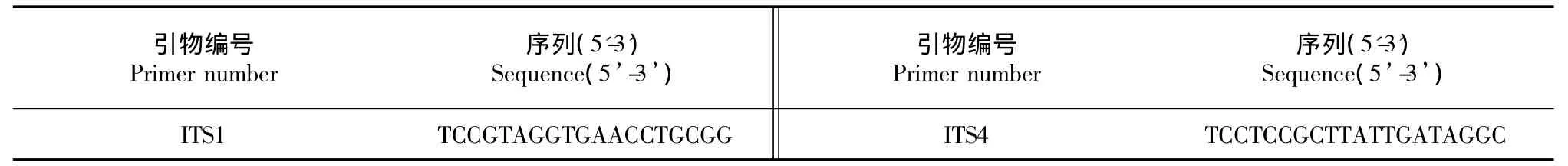
表2 ITS特异引物及其序列Table 2ITS specific primers and sequences

表3 RAPD随机引物及其序列Table 3RAPD random primers and sequences
1.8 数据处理分析
使用MEGA5.0和Clustal X软件进行ITS序列分析,并构建聚类分析树状图。RAPD分子标记分析通过在琼脂凝胶上DNA条带出现与否分别记为1和0。用Gel-Pro analyzer和UPGMA(NTSYS2.1)软件进行聚类分析,构建树状图[8]。
2 结果与分析
2.1 拮抗实验
图1 显示,2、18和19号菌株之间有拮抗性;9与3号菌株以及8号菌株之间有拮抗性,而3和8号菌株之间没有拮抗性。
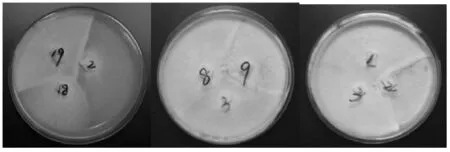
图1 部分供试菌株间的拮抗现象Fig.1Antagonistic phenomena of section strains
表4 表明,1号菌株与3、8、10、21号菌株;2号菌株与6、9号菌株;3号菌株与5、7、8、10、11、20号菌株;5号菌株与19号菌株;6号菌株与9号菌株;8号菌株与10、20、21号菌株;10号菌株与18号菌株;14号菌株与16号菌株;17号菌株与19号菌株; 20号菌株与21号菌株间没有拮抗反应或拮抗性反应极不明显,说明其亲缘关系很近。而其它各菌株之间都有着不同程度的拮抗反应,这表明菌株之间的亲缘关系较为疏远。
2.2 ITS序列分析
采用特异性引物ITS1和ITS4对供试菌株DNA进行PCR扩增,均获得约650 bp的单一条带(图2),经切胶回收后进行序列分析。
21株供试菌株的ITS序列依次与GenBank中已登录的11条ITS序列(其中10条为灵芝菌株ITS序列,1条为香菇ITS序列)进行相似性比对,采用MEGA5.0软件对比对结果以Neighbor-Joining(邻接法)构建系统发育进化树(图3)。分支上的数字为利用Bootstrap分析(重复1000次)获得的可信度。由图3可看出,21株菌株大致分为3个群,GroupⅠ、GroupⅡ和GroupⅢ(图中的AB286065.1为香菇序列号)。GroupⅠ包括17、19、5、11、1315号和16号;GroupⅡ包括2、6、9、4和12号;GroupⅢ包括8、10、14、1、3、7、18、20和21号。根据Liao等[9]报道G.sichanense,G.weberianum和G.tenue的相似性很大(94.3%)。Renske等[10]报道的观点,即当序列的相似度达到99%以上,即可以判定为同一个种。因此,21株菌株的亲缘性很近,种内多样性程度低。

表4 不同供试菌株间的拮抗反应情况Table 4Antagonistic situation of different Ganoderma strains
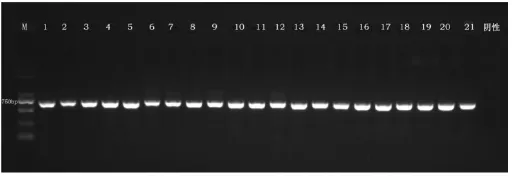
图2 21株供试菌株的ITS-PCR凝胶电泳图Fig.2ITS-PCR gel electrophoresis of 21 tested strains
经MEGA5.0软件的分析得出21株灵芝菌株测得的序列之间的遗传距离(表5)。21株菌株间遗传距离的变化范围在0.000~0.072,平均遗传距离为0.039,菌株间的遗传距离都比较小,表明它们之间的遗传关系都比较近。
2.3 RAPD分析
通过选取的10条随机引物(表3)对供试样品DNA进行扩增,采用Gel-Pro analyzer软件对条带进行分析,共产生200条条带,其中引物S32和S53的扩增效果较好(图4)。
采用UPGMA软件对RAPD分子标记结果进行聚类分析,建立遗传相似距离矩阵(表6)以及聚类图(图5)。结果表明,21株菌株相互间的遗传相似距离在0.807~0.950,其中1和3号菌株的相似距离最大达到0.950,而最低相似距离为0.807,分别为4与8、10、12、14、15、16、20和21号菌株。相似距离矩阵结果表明,各菌株之间的亲缘性都很近,亲缘性最近的是1与3号,其结果与拮抗性实验结果和ITS序列分析结果一致。
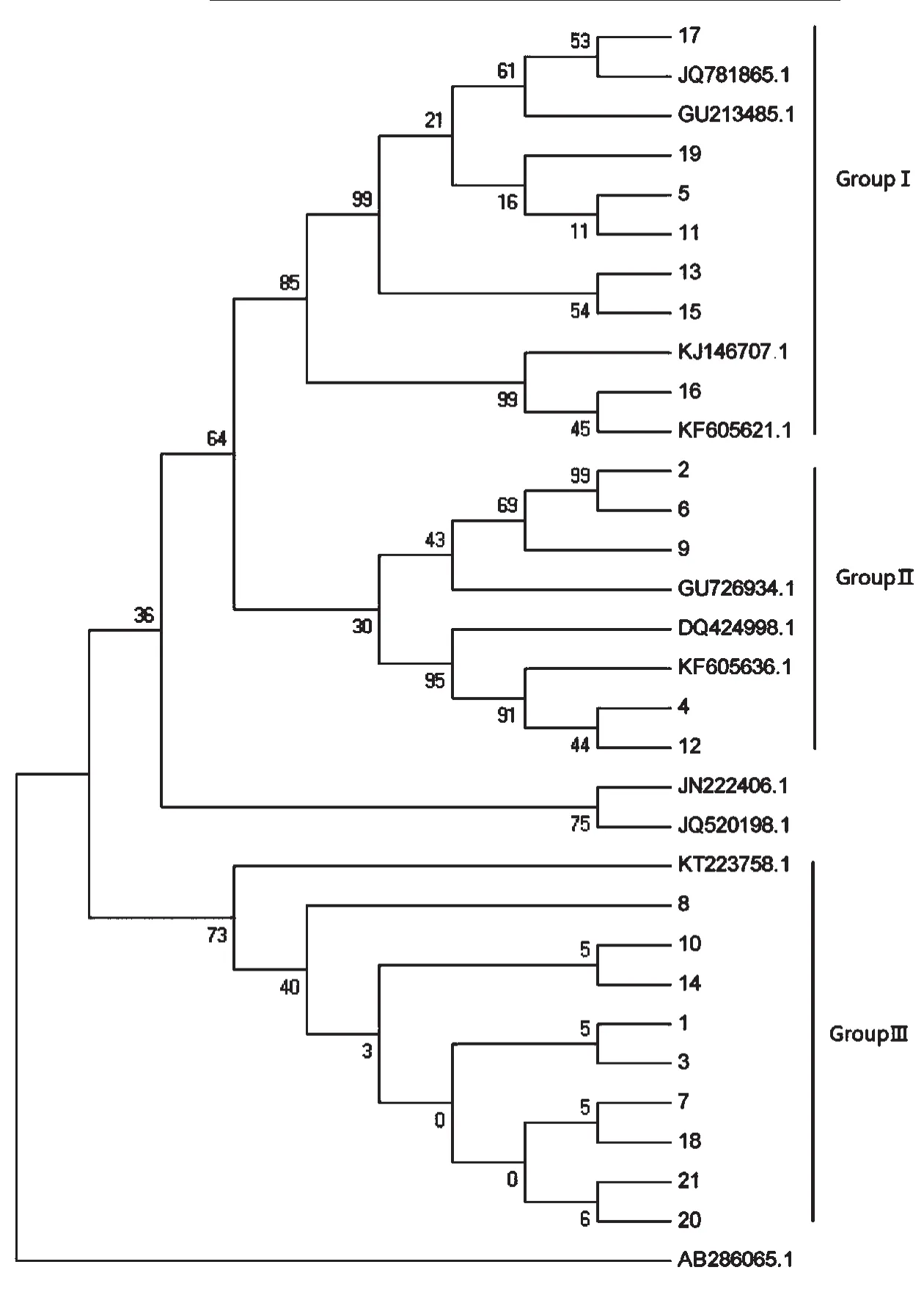
图3 21株供试菌株的ITS序列分析树状图Fig.3ITS sequence analysis dendrogram of 21 tested strains
图4 引物S32和S53的RAPD电泳图谱Fig.4RAPD electrophoresis map of S32 and S53
图5 表明,当相似系数在0.83时,所有菌株分为两个群,4号菌株单独为1组,其余20个菌株为1组。当相似系数达到0.932时,所有菌株将分成14个组,1与3号菌株为1组,20与21号菌株为1组,14与16号菌株为1组,8与10号菌株为1组,2和 19号菌株为1组,5和7号菌株为1组,13和15号菌株为1组,其余的各单独为1组。1与3号菌株的相似系数超过了95%。说明两者的亲缘性非常近,有可能来源于同一个菌株。

表5 供试菌株ITS序列间的遗传距离矩阵Table 5Genetic distance matrix between the ITS sequences of test strain
3 讨论

图5 供试菌株的RAPD聚类图Fig.5RAPD cluster diagram of the tested strains
我国作为食(药)用菌生产大国,有种植超过一半品种的食(药)用菌[11]。灵芝是最重要的药用菌之一,其中赤芝是最主要的栽培品种,在中国已有上千年的历史。但目前因为地域、标准、来源等原因,造成灵芝分类及命名混乱。在灵芝属建立之后,分类学家们根据灵芝的形态特征对灵芝进行分类,然而灵芝子实体的形态可能会随外界因素及地域的影响而变化,根据形态来对灵芝进行分类并不能完全建立一个稳定的分类系统[12]。因此,为了保证菌株的可持续栽培,建立一个可靠的灵芝遗传多样性评价方法显得尤为重要。

表6 供试菌株的遗传相似距离矩阵Table 6Genetic similarity matrix of tested strains
拮抗反应是细胞非亲和性的具体表现,因其直观、快速和易实现的优点而在真菌的亲缘性关系的初步研究中得到广泛的应用[13-14]。拮抗反应在一定时期内作为真菌菌种鉴定的主要方法,但其也有一定的局限性,其往往很难区分出两个在遗传上有相似背景或者亲缘关系很近的菌株[15-16]。因此目前在真菌的遗传多样性研究中只是用拮抗性实验作为菌株鉴定的初步研究,更进一步的研究就需要通过现代分子生物学的方法[8]。
自从White等[4-5]人在1990年首次设计ITS引物对真菌核内核糖体RNA基因进行扩增以来,ITS技术在真菌分类、鉴定的研究中应用的越来越广泛。由于ITS技术能实质性地反映出属间、种间以及菌株间的碱基对差异,且ITS的序列片段较小,研究人员可以从中获得所需要的重要信息,易于分析。该技术目前已广泛应用于真菌属内不同种间或者近缘属间的系统发育的研究[17-18],已被证明在种间的水平上鉴定效果显著[19]。Liao等[9]应用ITS序列分析成功鉴定了来自亚洲和欧洲的灵芝并进行了分类。Yan等[20]利用ITS技术和比较生态学研究了被一些人认为是同种异名的Phialophora verrucose和Phialophora americana,得出了它们是不同种的结论。本文研究结果表明大部分灵芝是可以利用ITS序列分析成功进行鉴定与分类,本研究还发现G.carnosum、G.pfeifferi、G.tsugae、G.weberianum、G.resinaceum、G.sessile跟赤芝(Ganoderma lucidum)的亲缘性很近。
由于不同的物种的总DNA序列有所不同,因此与引物结合的位点及位点之间的距离也会有所差异应用PCR扩增后的具有多态性的DNA片段就可以比较全面的反映出物种的遗传多样性[8],从而将不同种进行区分。Yan等[21]应用RAPD技术成功的区分了Auricularia auricular和Auricularia polytrichap。Kelly Y.C.Lam等[14]应用RAPD技术成功的对Cordyceps sinensis进行了鉴定。Khush等[22]人应用RAPD技术对Agaricus bisporus进行了分类。Hseu等[23]应用RAPD技术区分了Ganoderma lucidum。Chiu等[24]人的实验对Lentinula edodes进行了分类与鉴定。本研究应用拮抗、ITS和RAPD技术对21株灵芝菌株进行分类,结果表面1和3号菌株在拮抗实验中没有表现拮抗反应,ITS序列分析其处在GroupⅢ中且相似距离为0.000,在RAPD聚类分析是相似系数达到了95%,3种方法结果一致,两者有可能源于同一菌株,即同种异名。本实验对21株灵芝菌株进行了初步的分类与鉴定,对它们之间更详细具体的分类与鉴定还有待进一步的研究。
[1]唐传红,苏春丽,张劲松,等.灵芝属分类学研究进展[J].食用菌学报,2007,14(3):86-90.
[2]MISHRAAK,PANDEYAK,SINGHP,et al.Mycoparasites of Ganoderma lucidum(Leyss:Fr)Karst and their Botanical Management[J].Proceedings of the National Academy of Sciences,India Section B:Archives of Biological Sciences,2013,83(1):119-123.
[3]赵继鼎,徐连旺,张小青.中国灵芝的分类研究[J].真菌学报,1983,2(3):159-167.
[4]陈剑山,郑服丛.ITS序列分析在真菌分类鉴定中的应用[J].安徽农业科学,2007,35(13):3785-3786.
[5]白树猛,田黎.ITS序列分析在真菌分类鉴定和分子检测中的应用[J].畜牧与饲料科学,2009,30(1):52-53.
[6]施庆利,罗信昌.RAPD技术在木耳杂交种及原生质体融合子鉴定分析中的应用[J].中国食用菌,1993,12(6):3-4.
[7]ZHANGYF,MOLINAFI.Studies on the Differentiation of Lentinula edodes strains by RAPD Assay[J].Journal of Edible Fungi,1994,1 (1):22-27.
[8]唐传红,张劲松,陈明杰,等.利用拮抗试验和RAPD对灵芝属菌株进行分类研究[J].微生物学通报,2005,32(5):72-76.
[9]LIAOBS,CHENXC,HANJP,et al.Identification of Commercial Ganoderma(Lingzhi)Species by ITS2 Sequences[J].Chinese Medicine Journal,2015,56(2):1186-1195.
[10]张肖雅.17个灵芝菌株的表型分析及分子身份证的构建[D].东北农业大学,2013:26-29.
[11]YANPS,LUOXC,ZHOUQ.RAPD Molecular Differentiation of the Cultivated Strains of the Jelly Mushrooms,Auricularia auricula and A.polytricha[J].World Journal of Microbiology&Biotechnology,2004,20:795-799.
[12]CHRISTIANR,KATHRINW,PAULMK.An Assessment of the Taxonomy and Chemotaxonomy of Ganoderma[J].Fungal Diversity,2014,313(6):1007-1022.
[13]WANGXC,XIRJ,WANGDM,et al.The Species Identify of the Widely Cultivated Ganoderma[J].PLoS One,2012,7(7):40857-40863.
[14]CHENS,XUJ,ZHUY,et al.Genome Sequence of the Model Medicinal Mushroom Ganoderma lucidum[J].Nature Communications,2012(3):913-919.
[15]ZHANGY,GUOLD.Progress of Fungal DNA Barcode[J].Mycosystema,2012,31(6):809-820.
[16]PARKYJ,KWONOC,SONES,et al.Genetic Diversity Analysis of Ganoderma Species and Development of A Specific Marker for Identification of Medicinal Mushroom Ganoderma lucidum[J].African Journal of Microbiology Research,2012,6(25):5417-5425.
[17]CHENSL,YAOH,HANJP,et al.Validation of the ITS2 Region as a Novel DNA Barcode for Identifying Medicinal Plan Species[J].PLoS ONE,2010,5(1):8613-8621.
[18]HANJ,ZHUY,CHENX,et al.The Short ITS2 Sequence Serves As A Efficient Taxonomic Sequence Tag in Comparison with the Fulllength ITS[J].BioMed Research International,2013,2013:1155-1165.
[19]LAMKYC,CHENGKL,XINGZ,et al.Authentication of Cordyceps sinensis by DNA Analyses:Comparison of ITS Sequence Analysis and RAPD-Derived Molecular Markers[J].Molecules,2015,20:22454-22462.
[20]YANPS,JIANGJH,LIGF,et al.Mating System and DNA Polymorphism of Monokaryons with Different Mating Type of Strophariaannulata[J].World Journal of Microbiology and Biotechnology,2003,19:737-740.
[21]YANPE,JIANGJH,WANGDC,et al.Molecular Taxonomic Relationships of Auricularia Species Inferred From RAPD Marker[J].Fungi System,2002,21(1):47-52.
[22]KHUSHRS,BECKERE,WACHM.DNA Amplification Polymorphisms of the Cultivated Mushroom Agaricus bisporus[J].Applied and Environmental Microbiology,1992,58:2971-2977.
[23]HSEURS,WANGHH,WANGHF,et al.Differentiation and Grouping of Isolates of the Ganoderma lucidum Complex by Random Amplified Polymorphic DNA-PCR Compared with Grouping on the Basis of Internal Transcribed Spacer Sequence[J].Applied and Environmental Microbiology,1996,62:1354-1363.
[24]CHIUSW,MAALF,MOORED.Genetic Homogeneity of Cultivated Strains of Shiitake(Lentinula edodes)Used in China as Revealed by the Polymerase Chain Reaction[J].Mycological Research,1996,100:1393-1399.
(责任编辑 李山云)
Study on Classification of 21 Strains of Ganoderma by Antagonistic Effect,ITS and RAPD Technology
WANG Jin-feng1,2,LI Jing1,2,I.T.Datti2,3,ZHANG Jian2,3,LIN Zhi-kui2,3,LIN Zhan-xi2*
(1.College of Horticulture,Fujian Agriculture and Forestry University,Fujian Fuzhou 350002,China;2.China National Engineering Research Center of JUNCAO Technology,Fujian Fuzhou 350002,China;3.College of Life Sciences,Fujian Agriculture and Forestry University,Fujian Fuzhou 350002,China)
To more scientifically classify and identify Ganoderma Strain,genetic diversity analysis were carried out in 21 Ganoderma strains by using antagonistic experiment,ITS sequence analysis and RAPD molecular marker.The result of antagonistic test showed that,most of the strains had different degrees of antagonism,and there was no antagonistic or antagonistic is not very obvious between a few strains.In ITS sequence analysis,21 Ganoderma strains were divided into 3 groups.The results indicated that the genetic distance ranged from 0.000-0.072 and the average distance is 0.039.The genetic distance between No.1(G.68)and No.3(Ga15)was 0.000,which indicated that they have close genetic relationship.The RAPD data indicated that the similarity coefficient between each strain was over 0.83,all the strains were divided into 2 groups,the No.4(867)strain was selected as one group,the other 20 strains were another group.At the level of 0.932,all strains were divided into14 group.The similarity coefficient of No.1(G.68)and No.3(Ga15)exceeded 0.95,which indicated that the two strains maybe synonyms.The results of these three methods are consistent.Therefore,these three methods can be used to identify relative relationships among 21 Ganoderma lucidum strains.
Antagonistic effect;ITS sequence analysis;RAPD technology;Genetic diversity;Affinity
S567.3+1
A
1001-4829(2017)1-0026-08
10.16213/j.cnki.scjas.2017.1.006
2016-02-03
菌草食用菌产业化关键技术研究与示范(2014NZ0 002-1);福建省菌草生态产业协同创新中心(K80ND8002)
王锦锋(1990-),男,福建寿宁人,硕士研究生,主要研究生方向为菌草栽培食药用菌,E-mail:535108335@qq.com,*为通讯作者:林占熺,E-mail:lzxjuncao@163.com。

