8种经济鲍COⅠ基因全序列比较及系统发育分析
王礼闪, 薛艳洁, 房沙沙, 刘 晓
(宁波大学 海洋学院, 宁波 315211)
8种经济鲍COⅠ基因全序列比较及系统发育分析
王礼闪, 薛艳洁, 房沙沙, 刘 晓
(宁波大学 海洋学院, 宁波 315211)
为了研究不同地理种群的8种鲍的系统发育关系,以期为鲍属的种间鉴定与鲍的遗传育种提供分子生物学依据,以8种不同的鲍为实验材料。利用PCR扩增技术得到8条COⅠ基因全序列,10条其他鲍种的COⅠ基因全序列来源于GenBank。对18条的COⅠ基因全序列的比对分析表明,COⅠ基因的全序列共1542 bp,编码513个氨基酸;A+T的含量大于G+C的含量;在1542个碱基位点中存在526个变异位点,变异率为34.11%,氨基酸的变异率为7.8%。从遗传距离的分析中发现,同种鲍的遗传距离小于0.010;不同的鲍种之间的平均遗传距离在0.159~0.196之间;构建的遗传进化树分析得出:鲍的遗传进化受到地理分布的影响,但是不同地理种群的鲍之间并不存在明显的地理分布差异,其中美洲分布的红鲍和绿鲍与亚洲分布的皱纹盘鲍亲缘关系较近。
鲍;COⅠ基因;系统发育树
鲍是隶属于软体动物门(Mollusca)腹足纲(Gastropoda)原始腹足目(Archaeogastropoda)鲍科(Haliotidae)鲍属(Haliotis)的一种具有极高食用和药用价值的贝类。全世界的鲍有100种左右,主要经济种类有20种,我国沿海自然分布有8种鲍,其中主要经济种有皱纹盘鲍、杂色鲍和九孔鲍[1]。我国的鲍养殖业排名世界第一,但随着养殖规模的不断扩大,种质资源的衰退严重影响了我国鲍的养殖业。为丰富我国鲍的种质资源和养殖品种,研究者相继从美国、日本和澳大利亚引进了红鲍、绿鲍、盘鲍、西氏鲍和绿唇鲍等[2-4],其中从日本引进的盘鲍成为了我国的重要养殖种,而从美国和澳洲的引种不理想[1]。
线粒体DNA作为分子标记的一种,在软体动物门的系统发生和物种鉴定中已得到广泛应用[5-6]。COⅠ(细胞色素C氧化酶亚基Ⅰ)为线粒体DNA的13个编码基因之一,因其适中的遗传进化速度和较强的种间辨析能力成了研究鲍系统进化及种间鉴定的重要选择,并得到证明[7]。以往的研究主要以通用引物扩增COⅠ基因部分片段来分析鲍的遗传进化关系和进行种间鉴定[8-9]。本研究通过COⅠ基因完整序列研究了分布于澳洲、美洲和亚洲不同地区的8种重要经济鲍的序列特征和遗传进化关系,以期为鲍属物种的鉴定及主要养殖鲍的遗传背景提供可靠的分子生物学依据,以促进我国鲍养殖业的发展。
1 材料与方法
1.1 实验材料
本实验所用材料共8种鲍(由中国科学院海洋研究所刘晓老师提供):羊鲍(Haliotis.ovina)、绿唇鲍(Haliotis.laevigata)、黑唇鲍(Haliotis.rubra)、黑足鲍(Haliotis.iris)、红鲍(Haliotis.rufescens)、绿鲍(Haliotis.fulgens)、耳鲍(Haliotis.asinina)和西氏鲍(Haliotis.sieboldii),用75%的酒精固定保存样本。
1.2 试验方法
1.2.1 DNA提取
DNA的提取使用OMEGA的DNA提取试剂盒(E.Z.N.A Tissue DNA Kit)。并按说明书进行操作。
1.2.2 PCR扩增
根据本实验室获得的相关序列设计引物,信息见表1。
PCR反应体系60 μL:DNA模板3 μL、正反向引物各1.5 μL、10×PCR BufferⅡ 6 μL、dNTP 9.6 μL、TaKaRa LA Taq酶0.6 μL、ddH2O 37.8 μL。过程:94℃预变性5 min; 94℃变性30 s,50℃~57℃(根据引物设定)退火1 min,72℃延伸3 min(根据扩增的片段长度设定),35个循环; 72℃10 min。
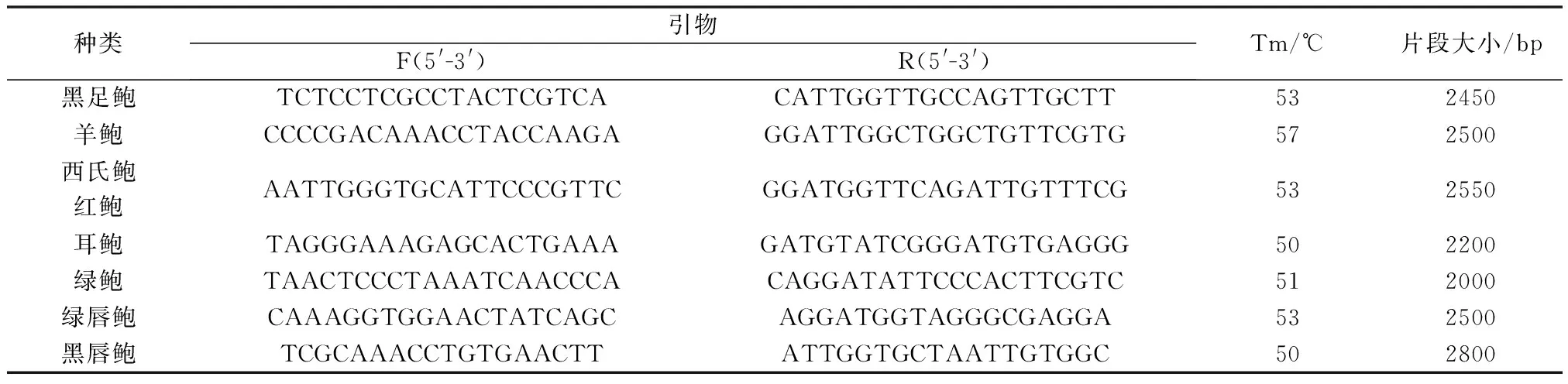
表1 引物信息及样品采集地
1.2.3 PCR产物测序
PCR产物经1%的琼脂糖凝胶电泳(图1)检测并切胶回收(选用OMEGA的试剂盒:E.Z.N.A. Gel Extraction Kit),然后送上海华大基因有限公司测序。
1.2.4 数据处理与分析
用SeqMan和Chromas软件对测序结果进行核对、剪切和拼接,通过ORF finder找出拼接结果中的COⅠ基因全序列,然后用得到的COⅠ基因全序列通过BLAST比对、查找并下载10条其他鲍种的COⅠ基因全序列。应用MEGA 5.05比对18条COⅠ全序列,并分析其碱基组成、变异位点、编码氨基酸,然后计算遗传距离和构建遗传进化树。
2 结果分析
2.1 碱基组成及变异分析
用扩增得到的COⅠ基因全序列通过BLAST比对,在GenBank中下载相关鲍种的10条COⅠ基因全序列,共18条,每条COⅠ基因全序列所对应的种类、注册号和各碱基含量如表2所示。

图1 PCR电泳图
分析发现,COⅠ基因全长为1542 bp,其中碱基A/T/C/G在各种间的总体含量浮动不大,分别在26.72%~28.60%、27.43%~29.70%、25.23%~27.24%和16.47%~18.03%之间,A+T的含量在54.80%~57.98%之间,A+T含量大于C+G含量。图2为各种鲍A+T含量的条形图,从图中可以看出分布于澳洲的黑足鲍、绿唇鲍和黑唇鲍中的A+T含量明显小于其他地理群体中鲍的A+T含量。COⅠ全基因共有514个密码子,编码513个氨基酸,第514个密码子为终止密码子,起始和终止密码子分别为ATG和TAA。在514个密码子的每个密码子中第2个碱基的T/C/A/G的含量较稳定,而第3个碱基G的含量在10%以下。在1542 bp的序列中,存在变异位点526个,总变异率为34.11%;简约性信息位点469个,单变异信息位点57个。包含转换147个,颠换78个,转换/颠换比率(R)为1.9,转换与颠换多发生在每个密码子的第3个碱基上。而其编码的513个氨基酸中存在变异位点为40个,氨基酸的变异率为7.80%,远低于核苷酸的变异率。

图2各鲍A+T含量
2.2 遗传距离的分析
基于COⅠ基因全序列计算各种鲍两两之间的遗传距离,结果见表3。同种鲍即黑唇鲍(7、10)、绿唇鲍(8、9)、皱纹盘鲍(11、12、13)和杂色鲍(17、18)的遗传距离较小,分别为0.005、0.003、0.002~0.010和0.008。羊鲍、耳鲍、杂色鲍、黑足鲍、疣鲍(除去其3种之间)、西氏鲍(除去与皱纹盘鲍之间)、皱纹盘鲍(除去与西氏鲍之间)、红鲍、绿鲍、绿唇鲍和黑唇鲍分别与其他鲍种的平均遗传距离为:0.186、0.183、0.196、0.183、0,184、0.181、0.181、0.159、0.166、0.172和0.169,范围在0.159~0.196之间。

图3 基于COⅠ基因序列构建的鲍的NJ树
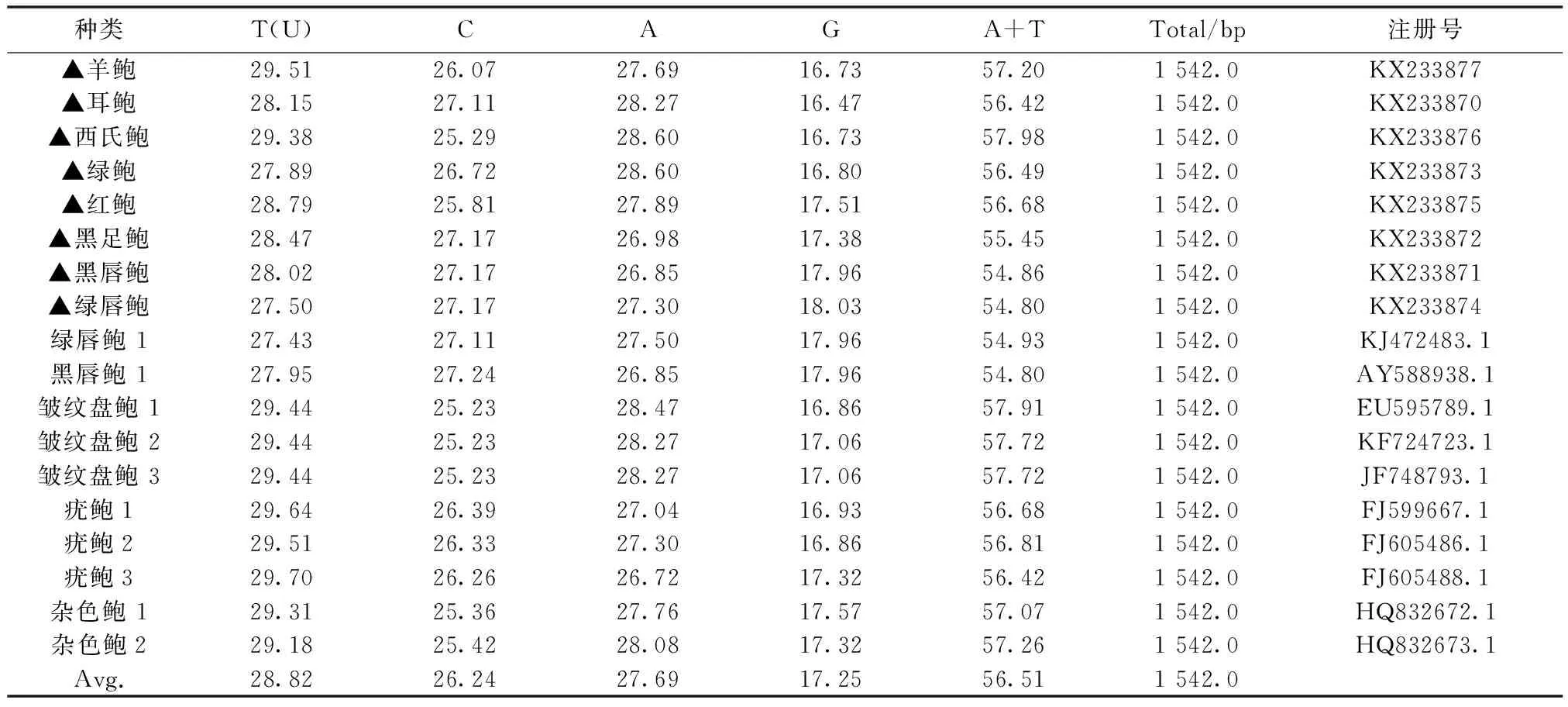
种类T(U)CAGA+TTotal/bp注册号▲羊鲍29.5126.0727.6916.7357.201542.0KX233877▲耳鲍28.1527.1128.2716.4756.421542.0KX233870▲西氏鲍29.3825.2928.6016.7357.981542.0KX233876▲绿鲍27.8926.7228.6016.8056.491542.0KX233873▲红鲍28.7925.8127.8917.5156.681542.0KX233875▲黑足鲍28.4727.1726.9817.3855.451542.0KX233872▲黑唇鲍28.0227.1726.8517.9654.861542.0KX233871▲绿唇鲍27.5027.1727.3018.0354.801542.0KX233874绿唇鲍127.4327.1127.5017.9654.931542.0KJ472483.1黑唇鲍127.9527.2426.8517.9654.801542.0AY588938.1皱纹盘鲍129.4425.2328.4716.8657.911542.0EU595789.1皱纹盘鲍229.4425.2328.2717.0657.721542.0KF724723.1皱纹盘鲍329.4425.2328.2717.0657.721542.0JF748793.1疣鲍129.6426.3927.0416.9356.681542.0FJ599667.1疣鲍229.5126.3327.3016.8656.811542.0FJ605486.1疣鲍329.7026.2626.7217.3256.421542.0FJ605488.1杂色鲍129.3125.3627.7617.5757.071542.0HQ832672.1杂色鲍229.1825.4228.0817.3257.261542.0HQ832673.1Avg.28.8226.2427.6917.2556.511542.0

表3 各鲍间的遗传距离(K2-P模型)
2.3 系统演化分析
以三角帆蚌(Hyriopsiscumingii)为外群,根据COⅠ全序列构建NJ遗传进化树(图3)。从图3可以看出,各鲍与外群分开并聚为一支。羊鲍和耳鲍分别单独一支;而美洲的红鲍、绿鲍与亚洲的西氏鲍、皱纹盘鲍遗传关系较近,先聚在一起,后再与新西兰的黑足鲍聚为一支;澳洲的绿唇鲍和黑唇鲍遗传关系比较近;杂色鲍与其他鲍种的遗传关系都比较远;欧洲的疣鲍聚为一支。
3 讨论
鲍属的种类较多,形态较为接近,以传统的形态学分析来鉴定鲍属种类较难。辛一[7]通过测定皱纹盘鲍线粒体COⅠ、COⅡ和CYTB的完整序列,并结合杂色鲍、疣鲍和黑唇鲍的线粒体基因信息,分析了COⅠ、COⅡ和CYTB在鲍属物种鉴定中的适用性,得出这3个基因都适用于鲍属物种的鉴定,因COⅠ信息相对较多,故用COⅠ进行鲍属物种的鉴定更为方便。本研究通过对18条COⅠ基因全序列进行比对分析,发现鲍的COⅠ基因全长为1542 bp,其长度在不同鲍中非常保守,共编码513个氨基酸,其核苷酸的变异率远高于氨基酸的变异率,这和简并密码子的存在有关,另外核苷酸的突变主要发生在每个密码子的第3位上并且突变位点的分布较分散。从图2可得到分布于澳洲的鲍种的A+T含量较其他地理种群要小。从遗传距离上看,同种间的遗传距离极小,小于0.010,不同种的遗传距离在0.159~0.196之间。其中欧洲的3种疣鲍被认为同一物种的不同亚种[10],其遗传距离在0.021~0.028之间;西氏鲍与皱纹盘鲍的遗传距离在0.022~0.025之间,与欧洲的3种疣鲍间遗传距离接近,达不到种间的分化要求。应用同工酶[11-14]、微卫星[15]、RAPD[16-17]、18S[18]、16S[19-21]和COⅠ[8-9]部分序列分析鲍的遗传多样性和遗传进化关系也越来越多,这些技术在研究遗传距离及遗传进化关系方面与本研究有一定差别。
鲍的遗传背景离不开其系统发育关系,从本研究的结果可以看出分布于美洲的红鲍、绿鲍和分布于亚洲的皱纹盘鲍、杂色鲍、耳鲍、羊鲍之间并没有表现出地理隔离所造成的遗传差异特征,另外红鲍与皱纹盘鲍间的遗传进化关系比红鲍和绿鲍间更近,皱纹盘鲍与红鲍、绿鲍的遗传差异比皱纹盘鲍与同分布于亚洲的杂色鲍、耳鲍、羊鲍的遗传差异小。由此可看出:鲍的进化虽然受到地理条件的影响,但不同鲍种的物种分化并不存在明显的地理分布差异,且美洲分布的红鲍和绿鲍与在亚洲分布的皱纹盘鲍遗传进化关系较近。
由于不同的技术分析鲍的遗传进化关系的结果之间存在一定的差别,故其相关研究需要加深并综合起来分析。随着几种鲍的线粒体DNA 编码区序列的公布[8,22-24],为鲍的遗传背景研究提供了方便。我国在世界上虽然是排名第一的养鲍大国,但我国的鲍养殖业同样面临大量的问题和严峻的挑战。本研究通过COⅠ全序列研究不同地理种群鲍种间的DNA序列差异和系统发育关系,旨在为鲍属物种的鉴定和我国鲍的养殖提供可靠的分子生物学依据,以促进我国鲍的养殖业的发展。
[1]赖龙玉, 严正凛. 鲍遗传育种研究进展[J]. 福建农业学报, 2013, 28(12): 1303-1309.
[2]聂宗庆, 王素平, 李木彬, 等. 盘鲍引进养殖与人工育苗试验[J]. 福建水产, 1995(1): 9-16.
[3]蔡明夷, 柯才焕, 周时强, 等. 鲍遗传育种研究进展[J]. 水产学报, 2004, 28(2): 201-208.
[4]庄世鹏, 蔡敬东, 曾海强, 等. 澳洲鲍(绿唇鲍)引种养殖试验观察[J]. 水产科技, 2002(3): 14-16.
[5]程汉良, 夏德全, 吴婷婷, 等. 6种帘蛤科贝类及4个地理种群文蛤线粒体COI基因片段序列分析[J]. 海洋学报, 2007, 29(5): 109-116.
[6]CHEN A H, LI Z X, FENG G N, et al. Phylogenetic relationships of the genus Meretrix (Mollusca:Veneridae) based on mitochondrial COI gene sequences [J]. Zoological Research, 2009, 30(3): 233-239.
[7]辛 一. 线粒体COI、COII和CYTB基因在鲍属物种鉴定中的适用性分析[J]. 海洋科学, 2011, 35(11): 58-62.
[8]陈 丽, 申 欣, 孟学平, 等. 皱纹盘鲍和九孔鲍遗传差异nad2-cox1分析及鲍属系统发育[J].广东海洋大学学报, 2015, 35(1): 28-34.
[9]高海霞,侯旭光,祝 茜.黑足鲍COⅠ基因序列在遗传分析中的初步研究[J].海洋科学,2010,34(11):14-17.
[10]WORMHOUDT A V, BRAS Y L, HUCHETTE S, et al.Haliotismarmoratafrom Senegal; a sister species ofHaliotistuberculata: morphological and molecular evidence [J]. Biochemical Systematics and Ecology, 2009, 37(6): 747-755.
[11]黎中宝, 邓书林, 许秀芹, 等. 盘鲍和皱纹盘鲍等位酶的生化遗传分析[J]. 海洋科学, 2004, 38(4): 43-47.
[12]柯才焕, 陈晓佳, 周时强, 等. 几种养殖鲍同工酶生化遗传的比较研究[J]. 台湾海峡, 2003, 22(2): 173-180.
[13]黎中宝, 田 柱, 朱冬蕊, 等. 九孔鲍和杂色鲍等位酶的生化遗传分析[J]. 海洋科学, 2004, 28(2): 27-31.
[14]黎中宝, 刘文彪, 韩 芳, 等. 4种经济鲍遗传多样性与分化的研究[J]. 中国生态农业学报, 2005, 13(4): 15-19.
[15]李 莉, 孙振兴, 杨树德, 等. 用微卫星标记分析皱纹盘鲍群体的遗传变异[J]. 遗传, 2006, 28(12): 1549-1554.
[16]张桂玲, 黎中宝, 谢德华. 羊鲍野生群体遗传多样性与分化的研究[J]. 海洋科学, 2011, 35(3): 12-16.
[17]李太武, 杨文新, 宋林生, 等. 皱纹盘鲍(HaliotisdiscushannaiIno)和杂色鲍(HaliotisdiversicolorReeve)遗传多样性的RAPD研究[J]. 海洋与湖沼, 2003, 34(4): 444-449.
[18]黄 勃, 方再光, 刘均玲, 等. 耳鲍(Haliotisasinina)核糖体小亚基(18S rRNA)编码基因的克隆与序列分析[J]. 海洋与湖沼, 2007, 38(3): 212-216.
[19]杨建敏, 郑小东, 王如才, 等. 3种鲍16SrRNA基因片段序列的初步研究[J]. 青岛海洋大学学报(自然科学版), 2003, 33(1): 37-40.
[20]陈 丽, 申 欣, 向治楚, 等. 皱纹盘鲍4个群体与九孔鲍16S rRNA基因遗传差异分析[J]. 水产科学, 2014, 33(11): 702-707.
[21]王鹭骁, 柯才焕, 王志勇, 等. 中国沿岸几种鲍线粒体16S rRNA基因片段序列比较及鲍属系统发育[J]. 中国水产科学, 2006, 13(2): 167-173.
[22]VAN WORMHOUDT A, ROUSSEL V, COURTOIS G, et al. Mitochondrial DNA introgression in the European abaloneHaliotistuberculatatuberculata: evidence for experimental mtDNA paternal inheritance and a natural hybrid sequence[J]. Mar Biotechnol 2011, 13(3): 563-574.
[23]XIN Y, REN J, LIU X, et al. Mitogenome of the small abaloneHaliotisdiversicolorReeve and phylogenetic analysis withinGastropoda[J]. Marine Genomics, 2011, 4(4): 253-262.
[24]MAYNARD B T, KERR L J, MCKIERNAN J M, et al. Mitochondrial DNA sequence and gene organization in the Australian blacklip abaloneHaliotisrubra(Leach)[J]. Marine Biotechnology, 2005, 7(6): 645-658.
Comparison of CO I gene complete sequence and phylogenetic analysis in eight kinds of economic abalone
WANG Li-shan, XUE Yan-jie, FANG Sha-sha, LIU Xiao
(College of Marine, Ningbo University, Ningbo 315211, China)
In order to study the phylogenetic relationships of eight species of abalone in different geographical populations, and to provide molecular biological basis for the identification of the species and the genetic breeding of abalone, this study used 8 kinds of abalone as the experimental materials. The whole sequences of 8 CO I genes were obtained by PCR amplification technique, the other 10 were found from GenBank. Through comparative analysis, it was found that the total length of CO I gene was 1542 bp, which encoded 513 amino acids, and the content of A + T was more than G + C. There were 526 mutation sites, and the mutation rate was 34.11%, while the variation rate of amino acid was 7.8%. From the analysis of genetic distance, it was found that the genetic distance of the same species was less than 0.010. The average genetic distance among different species of abalone was between 0.159-0.196. The phylogenetic tree was constructed to get the following conclusions. Abalone genetic evolution was affected by the influence of geographical distribution, but there was no obvious geographical distribution characteristic between different abalone; andHaliotisrufescensandHaliotisfulgens, which were distributed in the Americas, had a close relationship withHaliotisdiscushannaidistributing in Asia.
abalone; CO I gene; phylogenetic tree
2016-05-23;
2016-06-08
贝类遗传育种研究(D01304134900)
王礼闪,硕士研究生,专业方向为水产养殖,E-mail: wlsyanjiusheng@163.com
刘 晓,研究员,专业方向为水产养殖,E-mail: liuxiao@nbu.edu.cn
Q78;S917.4
A
2095-1736(2017)02-0036-04
doi∶10.3969/j.issn.2095-1736.2017.02.036

