人类miR-425靶基因预测及生物信息学特征分析
许宇彪 杨建荣 李碧锦 何二松 林昌荣 莫凯迪
·论著·
人类miR-425靶基因预测及生物信息学特征分析
许宇彪 杨建荣 李碧锦 何二松 林昌荣 莫凯迪
目的 采用生物信息学工具分析人类miR-425(has-miR-425)的转录调控、靶基因功能和蛋白-蛋白互作,为研究其在细胞调控过程中的作用提供理论依据。方法 应用miRBase、UCSC Genome Browser等数据库,分析has-miR-425在不同物种的序列情况,以及在基因组上下游10kb以内的CpG岛分布情况、转录起始位置(TSS)、转录因子结合位置(TFBS)等;选择TargetScan、PicTar、MiRanda三种计算方法预测has-miR-425的靶基因,取三个预测结果的交集靶基因分别进行注释描述和富集分析。采用STRING软件对交集靶基因进行蛋白与蛋白互作网络分析。结果 has-miR-425序列在人、大鼠、猕猴、黑猩猩之间呈现高度保守性;has-miR-425可能具有独立的启动子。共预测到319个潜在的靶基因,这些靶基因主要参与蛋白去磷酸化、细胞氮化合物反应、氮化合物反应等生物学过程;主要分子功能为酶结合、转移酶活性、金属离子结合等;主要细胞组成为:膜结合细胞器、细胞内膜有界细胞器、细胞核等。蛋白-蛋白互作分析显示靶基因中PAFAH1B1与DYNC1I2、MAP2K6与ZAK之间的关联度最高。结论 通过对has-miR-425转录调控元件、靶基因的分析,为has-miR-425在细胞调控中的功能与调控机制提供了重要的理论依据。
miR-425;转录调控;靶基因预测;生物信息学
miRNA是一类存在于真核生物体内长度为19~25个核苷酸的内源性非编码RNA,通过与靶基因的mRNA3’端非编码区完全或不完全互补配对,进而降解靶基因mRNA或抑制mRNA翻译,实现在转录后水平的基因调控作用[1,2]。miRNA参与调节细胞分化、增殖、凋亡、致癌、抑癌、激素分泌等多种生物学过程,具有广泛的生物学活性[3]。人类miR-425(has-miR-425)定位于染色体上的1p13.3,广泛表达于人体多个组织,在肝脏、肺、免疫系统、胃肠道、心脏等组织表达量相对较高,参与多种生物学过程并与多种疾病的发生密切相关[4]。has-miR-425部分靶基因已鉴定,但这些靶基因调控机制及生物学功能尚不完全清楚。本文应用生物信息学分析方法对has-miR-425的功能和靶基因进行系统分析整理,为后续深入研究提供理论指导和实验基础。
1 资料与方法
1.1 miR-425转录情况分析 应用NCBI Mapviewer、UCSC Genome Browser等在线工具,分析miR-425在基因组上下游10 kb以内的CpG岛分布情况;应用UCSC Genome Browser、Promoterscan等在线工具,分析miR-425在基因组上游10 kb以内可能的转录起始位置(TSS)及可能的polyA信号位置;应用PromoterScan、CBRC的TFSEARCH数据库等在线工具,分析miR-425在基因组上游10 kb以内可能的转录因子结合位置(TFBS);应用NCBI Mapviewer、miRbase等在线分析工具,分析miRNA所在区域的EST数据。
1.2 miR-425潜在靶基因预测 选择miRbase(http://www.mirbase.org/)查找miRNA序列,选择miRWalk (http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/genepub.html)网址,该网站整合了TargetScan、PicTarl、miRanda等数据库,选中TargetScan、PicTarl、miRanda三种数据库分别预测miR-425的潜在靶基因,得出的结果即为3种数据库得出的交集。
1.3 靶基因功能分析 将miR-425的交集靶基因提交Gene Ontology (http://geneontology.org/)数据库进行基因功能富集分类和GO注释描述,分别提取基因的GO生物学过程(Biological Process)、分子功能(Molecular Function)和细胞组成(Cellular Component)。
1.4 蛋白-蛋白互作分析 将miR-425的交集靶基因提交输入在线分析工具STRING 10.0 (http://www.string-db.org/)进行蛋白-蛋白相互作用(protein-protein interaction,PPI) 网络分析,分析基因对应蛋白之间的相互作用关系,并计算蛋白间的连接系数(r)和绘制网络图。使用Cytoscape 3.2软件进行交集靶基因的分析,采用BINGO插件进行GO注释的显著性分析,并绘制基因间作用图。通过Fisher精确检验计算P值,以P<0.05为显著性阈值,得到基因集合相对于背景具有统计意义。
2 结果
2.1miR-425在不同物种序列的保守性比较 经miRBase数据库分别检索人、大鼠、猕猴、黑猩猩和马不同物种成熟microRNA序列,发现人、大鼠、猕猴、黑猩猩的保守序列相同,而马的保守序列与上述物种不同,说明miR-425物种间有一定的差异。见表1。

表1 不同物种miR-425序列比较
2.2hsa-miR-425转录规律预测 利用NCBIMapviewer、UCSCGenomeBrowser等在线工具,分析hsa-miR-425上下游10kb内序列特征,结果显示,其有可能的TSS及众多TFBS,其上游存在约2 900bp的CpG岛,因此推测hsa-miR-425有可能拥有独立的转录单元。见表2。

表2 hsa-miR-425启动子预测结果(-10 kb~+10 kb)
注: -无; +有
2.3 靶基因功能注释TargetScan、PicTarl、miRanda三个数据库预测hsa-miR-425的潜在靶基因,取这些靶基因的交集,共得到319个靶基因,将这些交集靶基因放入在GeneOntology网站进行功能注释分析。GO功能注释显示这些潜在靶基因主要参与蛋白去磷酸化、细胞氮化合物反应、氮化合物反应、神经元分化信号转导负调控等生物学过程;主要分子功能为酶结合、转移酶活性、金属离子结合等;细胞组成为:膜结合细胞器、细胞内膜有界细胞器、细胞核。见表3。
2.4 蛋白-蛋白互作分析 将319个hsa-miR-425交集靶基因放入STRING网站进行蛋白与蛋白互作网络分析,共得到153个节点及77条相互关系连接线,其中PAFAH1B1与DYNC1I2、MAP2K6与ZAK之间的关联度最高。见图1、2,表4。
3 讨论
miRNA是近年来分子医学领域中最重要的发现之一,它在机体发育和疾病状态下功能也越来越引人关注。研究已证实,miRNA调控着人类近1/3基因的功能,几乎参与一切重要生命活动[5]。但由于miRNA种类和数量较多,且其表达具有显著的时序性和组织特异性,即在发育疾病的不同阶段或在不同组织细胞中具有特异性表达的特征,仅有少数miRNA的生物学功能得到阐明,因而通过生物信息学对miRNA进行分析快速准确地预测靶基因,正确认识靶基因之间的相互作用对研究miRNA的生物学功能有十分重要的意义。
miR-425已被报道与多种基因的转录后调控相关[6]。有研究发现IL-1β可以通过激活NF-κB诱导miR-425表达上调,负性调控PTEN进而促进胃癌细胞的增殖和抗凋亡作用,且miR-425启动子上B结合位点是IL-1β诱导miR-425转录激活必需的[7]。在采用芯片技术比较4对高转移肝细胞患者的癌组织样本与其相对应的正常组织之间的差异miRNAs时,发现与正常组织相比,高转移肝细胞癌中发现miR-425表达上调[8]。还有研究使用芯片技术筛选与喉鳞状细胞癌生物学特征密切相关的差异表达miRNA,结果从喉癌的miRNA表达谱中筛选出的转移相关差异表达miRNA,发现miR-425是差异表达倍数较高的miRNA,提示miR-425有可能成为评估喉癌转移风险的新型分子标志[9]。He等[10]在裸鼠肝癌细胞移植瘤模型中研究证实,CTNNA3能通过抑制Akt信号通路,进而降低细胞增殖核抗原(PCNA)和基质金属蛋白酶MMP-9,增加细胞周期抑制剂p21(Cip1/Waf1)的表达而达到抑制肝癌细胞增殖、迁移与侵袭而起到抑癌作用,而miR-425z则可与CTNNA3 3'非翻译区直接结合并抑制其表达而起到促癌作用,因而miR-425/CTNNA3轴对肝癌的病理机制的阐明和肝细胞癌的治疗策略的制定具有潜在的价值。

表3 基因功能注释和信号通路分析
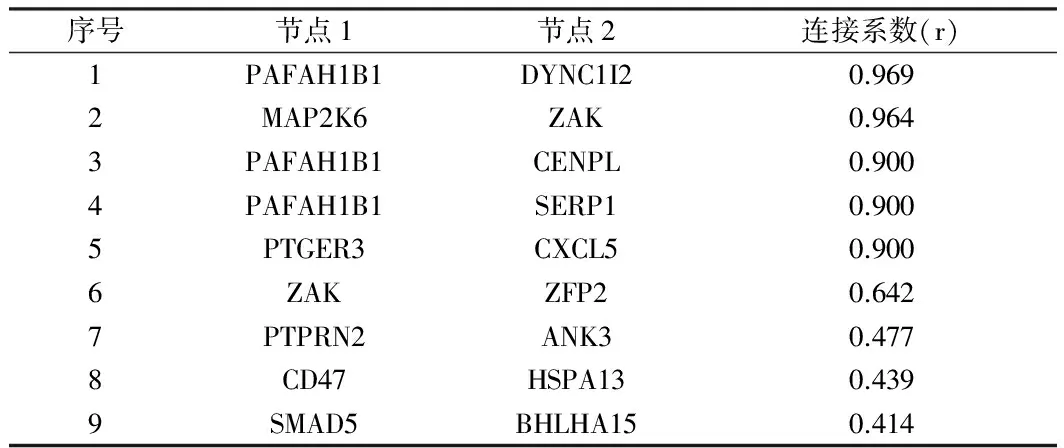
表4 hsa-miR-425靶基因的蛋白-蛋白互作分析
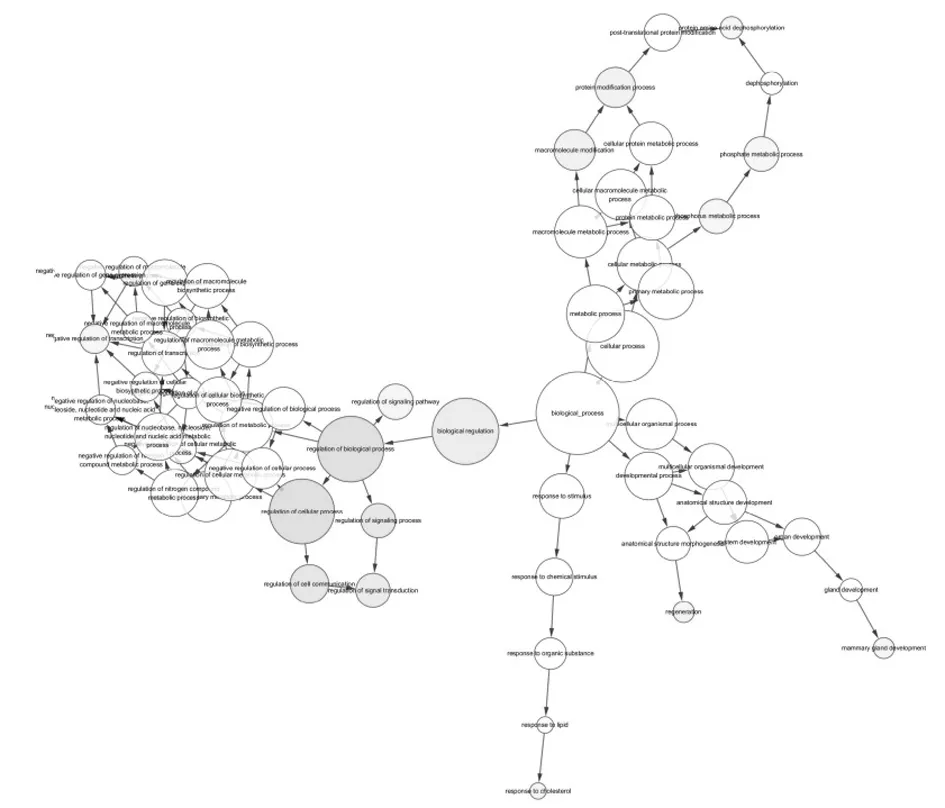
图1miR-425预测靶基因功能的层次网络(节点大小代表基因数量多少,颜色深浅代表P值大小,颜色越深,P值越小)
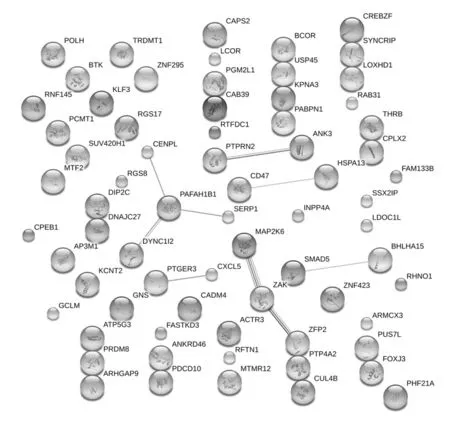
图2miR-425预测靶基因蛋白-蛋白互作网络图(每个点代表一个蛋白,连接线代表蛋白间有互作联系,连接线越多,互作关系越强)
本研究发现人类miR-425序列在人、大鼠、猕猴、黑猩猩之间具有高度保守性,提示其可能具有较为强大的生物信息学功能。进一步分析hsa-miR-425上下游10kb内序列特征,结果显示,其有可能的TSS及众多TFBS,其上游存在约2 900bp的CpG岛,这些结果有助于理解miR-425在调控细胞功能的可能机制。本研究采用TargetScan、PicTarl、miRanda三种计算方法预测miRNA作用的靶基因,并取靶基因的交集进行GO功能注释,从多层次多角度对hsa-miR-425的预测基因进行描述,揭示了hsa-miR-425靶基因参与的细胞的主要生物学过程、分子功能和在细胞组成的作用,为了解这些靶基因的作用提供了参考依据。最后我们采用蛋白-蛋白互作分析探索hsa-miR-425靶基因对应蛋白的作用关系,发现PAFAH1B1与DYNC1I2、MAP2K6与ZAK之间的关联度最高,提示这些靶基因在hsa-miR-425的细胞调控过程中可能发挥较大的作用。
总之,本研究结果揭示hsa-miR-425的序列保守性,生物学功能和靶基因的功能,为全面了解hsa-miR-425的生物学作用提供了重要的生物信息学基础。然而,由于各种在线工具、数据库等具有一定的局限性,本研究结果还需要对这些生物学信息预测结果进行进一步的实验设计和验证,以期更准确的阐明hsa-miR-425的功能,为其在疾病的诊断、治疗和预后提供理论依据和实验基础。
1BartelDP.MicroRNAs:genomics,biogenesis,mechanism,andfunction.Cell,2004,116:281-297.
2Alvarez-GarciaI,MiskaEA.MicroRNAfunctionsinanimaldevelopmentandhumandisease.JDevelopment2005,132:4653-4662.
3ChristodoulouF,RaibleF,TomerR,etal.AncientanimalmicroRNAsandtheevolutionoftissueidentity.Nature,2010,463:1084-1088.
4LiL,ChenHZ,ChenFF,etal.GlobalmicroRNAexpressionprofilingrevealsdifferentialexpressionoftargetgenesin6-hydroxydopamine-injuredMN9Dcells.NeuromolecularMed,2013,15:593-604.
5LiX,NieJ,MeiQ,etal.MicroRNAs:Novelimmunotherapeutictargetsincolorectalcarcinoma.WorldJGastroenterol,2016,22:5317-5331.
6AvciCB,HarmanE,DodurgaY,etal.Therapeuticpotentialofananti-diabeticdrug,metformin:alterationofmiRNAexpressioninprostatecancercells.AsianPacJCancerPrev,2013,14:765-768.
7 刘军,厉天瑜,张宁,等.IL-1β诱导的miR-425表达上调对胃癌细胞生长的影响.中华医学杂志,2014,99:1889-1893.
8 宋晓,蔡振旭.MicroRNAs的失调在高转移肝细胞癌中的作用.中华临床医师杂志电子版,2013,7:130-133.
9 李琳,张宗敏,刘宇,等.应用基因芯片检测喉鳞状细胞癌甲醛固定石蜡包埋组织microRNA表达谱.中华病理学杂志,2010,39:391-395.
10HeB,LiT,GuanL,etal.CTNNA3isatumorsuppressorinhepatocellularcarcinomasandisinhibitedbymiR-425.Oncotarget,2016,7:8078-8089.
Target gene prediction and bioinformatics characteristics analysis of human miR-425
XUYubiao,YANGJianrong,LIBijin,etal.
DepartmentofGeneralSurgery,JiangbinHospitalofGuangxiZhuangNationalityAutonomousRegion,Nanning530021,China
Objective To investigate the transcriptional regulation,target gene function and protein-protein interaction of the human miR-425 (has-miR-425) by bioinformatics tools in order to provide theoretical basis for researching its role in cell regulation and control.Methods The has-miR-425 sequences in different species,the distribution of CpG island within 10 KB upstream and downstream in genome,the transcription start sites (TSS) and transcription factor binding position (TFBS) were analyzed by using miRBase,UCSC Genome Browser database.The three calculation methods of targetscan,pictar and miRanda were adopted to predict the target gene of has-miR-425.The intersectional target genes of the three prediction results were analyzed by annotation description and enrichment analysis.The STRING software was used to analyze the intersectional target genes,protein-protein interaction.Results The has-miR-425 sequences were highly conserved among human,rat,rhesus monkey and chimpanzee,and has-miR-425 might have independent promoter.The 319 potential target genes were forecasted,and these target genes were mainly involved in the biological processes such as protein dephosphorylation,cellular response to nitrogen compound and response to nitrogen compound.The molecular functions mainly included enzyme binding,transferase activity,metal ion binding and so on,however, the main cell components were membrane-bounded organelle,intracellular membrane-bounded organelle and nucleus. The protein-protein interaction analysis showed that the correlation degree between PAFAH1B1 and DYNC1I2 and between MAP2K6 and ZAK was the highest.Conclusion The research resullts provide an important theoretical basis for the researches about function and regulation mechanism of has-miR-425 in cell regulation and control through the analysis of has-miR-425 transcriptional regulatory elements and target genes.
miR-425; transcriptional control; target gene prediction; bioinformatics
10.3969/j.issn.1002-7386.2017.07.004
项目来源:广西医药卫生自筹经费计划课题(编号:Z2015076);广西科学研究与技术开发计划项目(编号:桂科攻12239015)
530021 南宁市,广西壮族自治区江滨医院普通外科
杨建荣,530021 南宁市,广西壮族自治区江滨医院普通外科;
E-mail:1637340358@qq.com
R 349.83
A
1002-7386(2017)07-0977-04
2016-10-17)

