侵染广东番茄的番茄褪绿病毒分子鉴定
汤亚飞, 何自福*, 佘小漫, 蓝国兵
(1. 广东省农业科学院植物保护研究所, 广州 510640; 2. 广东省植物保护新技术重点实验室, 广州 510640)
研究简报
侵染广东番茄的番茄褪绿病毒分子鉴定
汤亚飞1,2, 何自福1,2*, 佘小漫1, 蓝国兵1
(1. 广东省农业科学院植物保护研究所, 广州 510640; 2. 广东省植物保护新技术重点实验室, 广州 510640)
在广东番茄上发现一种新病害,病株表现为叶片褪绿,叶脉颜色变深及叶片增厚等症状。利用番茄褪绿病毒Tomatochlorosisvirus(ToCV)HSP70基因的两对特异引物对番茄病样进行RT-PCR检测,结果表明,从所采集的6份病样中均扩增到预期大小的DNA特异片段。对其中1份样品的扩增片段进行克隆与序列分析,结果表明,扩增片段包括1个长度为1 665个核苷酸的完整病毒基因,其核苷酸序列与已报道的ToCVHSP70基因有较高的同源性,表明广东番茄受到了ToCV的侵染。但ToCV广东番茄分离物的HSP70序列与国内外已报道的各分离物的同源性均低于82%,存在较大差异,其中与塞浦路斯tomato、约旦JU_20分离物的同源性最高,为81.8%。这是ToCV在广东发生的首次报道,也是该病毒HSP70基因序列存在显著差异分离物的首次发现。
番茄病毒病; 番茄褪绿病毒;HSP70基因
番茄褪绿病毒Tomatochlorosisvirus(ToCV)属于长线形病毒科Closteroviridae毛形病毒属Crinvirus,其基因组为二分体正义单链RNA(+ssRNA),RNA1和RNA2分别包装在2种不同的病毒粒子中,RNA1含4个ORFs,编码与病毒复制相关蛋白等,RNA2含9个ORFs,编码外壳蛋白(CP)和热激蛋白(HSP70)等[1-2]。该病毒可由烟粉虱BemisiatabaciGennadius、温室白粉虱TrialeurodesvaporariorumWestwood、纹翅粉虱T.abutiloneaHaldeman 、银叶粉虱B.argentifoliiBellows and Perring以半持久方式传播[1-2],但不能经机械摩擦传播。该病毒可侵染茄科、番杏科、苋科、夹竹桃科、藜科、菊科和蓝雪科等科的多种植物[3]。番茄感染ToCV后,植株下部叶片黄化,叶脉浓绿而脉间褪绿,叶片变脆易折,严重时叶片黄褐色,甚至出现坏死等症状,番茄品质与产量严重下降[4]。
ToCV最早于1998年在美国佛罗里达州被发现[5],随后在欧洲[6-8]、非洲[9]、亚洲[10-15]、南美洲[16]、北美洲[17-19]等多个国家和地区相继被报道。我国2004年首次在台湾番茄上发现该病毒[13],尔后直到2012年一直未见该病毒的相关报道;但自2013年开始,该病毒陆续在江苏[20]、北京[14,21]、山东[4,22-23]、河南[24]、河北[25]、天津[26]等蔬菜产区被发现。2015年初,我们在广东省惠州市番茄产区发现了疑似被ToCV侵染的番茄病株,且植株上烟粉虱数量较多。为了弄清该病害的病原种类,笔者对病样进行了分子检测与鉴定。
1 材料与方法
1.1 番茄病样
番茄病样叶片于2015年1月采自广东省惠州市番茄产区,共6份,编号为GD01~GD06。田间病株表现为下部叶片首先发病,叶片脉间变黄,叶脉颜色加深变成深绿色,叶片增厚,发病严重时植株死亡。
1.2 番茄叶组织总RNA提取
应用RNA提取试剂盒(北京全式金生物技术有限公司)抽提番茄病样叶片的总RNA。分别取6个番茄病样和1个健康番茄样品的叶组织100 mg,按照试剂盒说明书的步骤抽提总RNA,最终将RNA沉淀溶解于40 μL DEPC处理的ddH2O中。
1.3 RT-PCR检测
分别以抽提的番茄叶片总RNA作为模板,用随机引物(TaKaRa公司)逆转录合成cDNA。反应体系和具体步骤为:模板RNA 4 μL,50 μmol/L random 6 mers引物 1 μL,10 mmol/L dNTPs 1 μL,RNase-free dH2O 4 μL;65℃变性5 min后,冰上迅速冷却,然后再加入5×PrimeScript Ⅱ Buffer(TaKaRa)4 μL、RNase Inhibitor (40 U/μL)0.5 μL、PrimeScript Ⅱ RTase(200 U/μL)1 μL和RNase-free dH2O 4.5 μL,反应体系总体积为 20 μL,30℃孵育10 min后,42℃孵育50 min,最后70℃反应15 min使酶失活。
进一步利用ToCVHSP70 基因的2对特异引物HSP1-F/R(HSP1-F:GTTAACTCAGTTTAACTTGATTCCG;HSP1-R:CACTTGACCAAATCGTCAAACG)和HSP2-F/R(HSP2-F:CATACAATATAATAAGTGATGATGGGAG; HSP2-R:CAAGACAGTTAGTTCTCAGTACTAAAC)[12]分别进行PCR扩增。反应体系为:反转录产物 1 μL,灭菌水 9.5 μL,PCR Mixer 12.5 μL(TaKaRa)10 μmol/L上下游引物各 1 μL,总体积为 25 μL。反应程序:94℃ 4 min;94℃ 45 s,50℃ 45 s,72℃ 60 s,35 个循环;72℃ 10 min。PCR产物经1% 的琼脂糖凝胶电泳检测。
1.4 PCR产物克隆和测序
采用琼脂糖凝胶回收试剂盒(北京全式金生物技术有限公司) 回收病样GD01的PCR特异条带,纯化后克隆到 pMD20-T 载体(TaKaRa)上,并转化大肠杆菌菌株 DH5α,随机挑取3个阳性克隆送上海生工生物技术有限公司测序。
1.5 序列分析
利用DNAStar软件(DNASTAR Inc,Madison,USA)对所获得的基因序列进行拼接,利用BLAST程序进行序列相似性搜索,进一步用DNAStar的MegAlign进行序列比较分析,进化树构建采用MEGA 5.05的邻接法(neighbor joining,NJ)。
2 结果与分析
2.1 病样RT-PCR扩增结果
利用ToCVHSP70 基因的2对特异引物 HSP1-F/R 和 HSP2-F/R 分别进行RT-PCR扩增,从6份番茄病样总RNA中均能扩增到1条与预期大小一致的特异片段,长度分别为943 bp和913 bp,而健康番茄样品中未扩增出任何片段(图1)。
2.2HSP70 基因序列分析
PCR产物测序结果表明,引物HSP1-F/R扩增获得的943 bp片段和引物HSP2-F/R扩增获得的913 bp片段与设计预期相符;两片段3′端和5′端有70 bp序列相同,采用软件SeqMan对这两个片段序列进行拼接获得1 785 bp序列,其中包含有一个长度为1 665 bp的完整ORF。由此得出,广东番茄分离物GD01的HSP70全长为1 665个核苷酸(GenBank登录号:KT962275),编码554个氨基酸。BLAST结果显示,与该基因序列有较高同源性的序列均为ToCV分离物的HSP70基因,其氨基酸序列与ToCV各分离物的HSP70氨基酸序列同源性均在75%以上。进一步比较(表1)显示,该基因序列与来自塞浦路斯、约旦、乌拉圭、古巴、土耳其、留尼旺岛、韩国、西班牙、美国、希腊、日本、巴西及中国等的22个ToCV分离物的序列同源性均在79%以上,其中与塞浦路斯tomato和约旦JU_20 分离物的同源性最高,为81.8%:与中国河北、山东、江苏、河南等分离物的同源性为80.2%~80.5%。这些结果表明,广东番茄受到了ToCV侵染,但所研究的GD01分离物HSP70基因与已报道分离物存在一定的差异。
为了分析分离物GD01与已报道各ToCV分离物的HSP70亲缘关系,选取了来自我国不同地区的7个分离物以及12个国家的15个ToCV分离物,以同属的番茄侵染性褪绿病毒Tomatoinfectiouschlorosisvirus西班牙TICV-SP5131分离物(FJ542305)为外组构建系统进化树(图2)。可以看出,分离物GD01与来自中国不同地区的7个分离物及国外的15个ToCV分离物聚集在一个大的分支上,但独立在一个小分支上,说明广东番茄分离物GD01与已报道的ToCV各分离物亲缘关系相对较远。
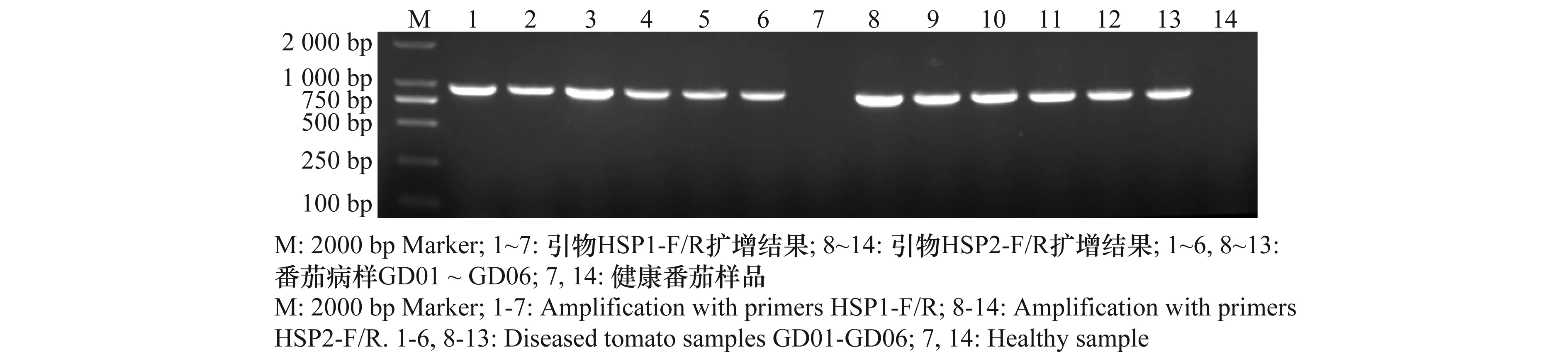
图1 番茄病样RT-PCR检测结果Fig.1 Detection results of the diseased tomato samples using RT-PCR

病毒分离物Virusisolate地理来源GeographicoriginGenBank登录号Accessionno.同源性/%IdentityToCV-tomato塞浦路斯CyprusAM15895881.8ToCV-JU_20约旦JordanKP71334981.8ToCV-CRS07乌拉圭UruguayKC62601981.7ToCV-CCu古巴CubaFJ36085381.6ToCV-Fethiye土耳其TurkeyEU06936381.5ToCV-Reunion1留尼旺岛ReunionAJ96839481.2ToCV-JJ韩国SouthKoreaKP13710180.7ToCV-AT80/99西班牙SpainDQ13614680.5ToCV-SDTADP中国ChinaKC81262480.5ToCV-SDLC中国ChinaKC81262680.4ToCV-SDSG中国ChinaKC70951080.4ToCV-Handan中国ChinaKM65582980.4ToCV-HBLF中国ChinaKP21720280.4ToCV-Florida美国USAAY90344880.4ToCV-Gr-535希腊GreeceEU28474480.4ToCV-HP韩国SouthKoreaKP11453780.4ToCV-IS29韩国SouthKoreaKP11452980.4ToCV-Tochigi日本JapanAB51344280.4ToCV-Nanjing中国ChinaKJ81504580.3ToCV-HNZZZM2中国ChinaKP26498880.2ToCV-ToC-Br2巴西BrazilJQ95260180.2ToCV-FL47美国USAHQ87984579.5

图2 基于HSP70基因核苷酸序列构建的GD01与其他22个ToCV分离物的系统进化树Fig.2 Phylogenetic tree of GD01 and other 22 isolates of ToCV based on the nucleotide sequences of HSP70 gene
3 讨论
本研究采用扩增长线形病毒科病毒最保守蛋白之一HSP70的2对特异引物 HSP1-F/R 和 HSP2-F/R 对广东省疑似ToCV侵染的番茄病样进行RT-PCR检测,检测结果显示该批病样中确实存在ToCV,并获得了广东番茄分离物GD01的HSP70的全基因序列。序列分析显示,分离物GD01的HSP70氨基酸序列与GenBank登录的ToCV的HSP70氨基酸序列同源性均在75%以上。根据目前国际病毒分类委员会第九次报告公布的分类方案[27],确定侵染广东番茄的病毒分离物GD01属于ToCV的一个分离物,这是首次在广东省检测到该病毒。
ToCV广东番茄分离物GD01的HSP70基因序列与塞浦路斯ToCV-tomato、约旦ToCV-JU_20 分离物同源性最高,但仅为81.8%,而与中国其他地区分离物同源性最高为80.5%;中国已报道的ToCV各分离物的HSP70基因序列同源性均在99%以上,系统进化树分析显示ToCV所有分离物聚集在一个大的分支上,但分离物GD01独立在一个小分支上。由此可见,ToCV广东番茄分离物GD01与国内外已报道的分离物存在差异较大。推测造成这种差异的原因可能与病毒的地理分化有关,也有可能与传毒介体不同有关。无论是哪种原因占主导,本研究结果一方面说明危害我国的ToCV种群存在遗传多样性,另一方面揭示了我国粉虱传病毒的复杂性,这些都将为粉虱传病毒的重组、变异与流行提供条件,需要加强对其进行监测。
由ToCV侵染引起的番茄褪绿病毒病是我国近年暴发的一种新病害,目前已在河北、山东等黄河中下游番茄产区暴发和流行。例如,2013年山东省大面积暴发番茄褪绿病毒病,部分温室发病株率20%~100%,造成番茄减产10%~40%[22]。该病毒病也在江苏和台湾发生,但在华南地区还未见报道。ToCV可以侵染多种植物,我国目前已报道的寄主有番茄[4,23-26]、甜椒[21]和茄子[28-29]3种作物。这3种作物在广东省种植非常广泛,几乎遍及各个蔬菜产区;此外,传播介体粉虱在广东省常年普遍发生,一旦发生 ToCV,极有可能通过粉虱或种苗调运造成大面积暴发和流行。本研究首次在广东番茄产区发现了番茄褪绿病毒病,目前仅零星发生,应引起有关部门和生产者的重视。
[1] Wintermantel W M, Wisler G C, Anchieta A G, et al. The complete nucleotide sequence and genome organization ofTomatochlorosisvirus[J]. Archives of Virology, 2005, 150(11): 2287-2298.
[2] Kataya A R A, Stavridou E, Farhan K, et al. Nucleotide sequence analysis and detection of a Greek isolate ofTomatochlorosisvirus[J]. Plant Pathology, 2008, 57(5): 819-824.
[3] Wintermantel W M, Wisler G C.Vector specificity, host range, and genetic diversity ofTomatochlorosisvirus[J]. Plant Disease, 2006, 90(6): 814-819.
[4] 赵黎明, 李刚, 刘永杰, 等. 侵染番茄的番茄褪绿病毒山东泰安分离物的分子鉴定和序列分析[J]. 植物保护, 2014, 40(5): 34-39.
[5] Wisler G C, Li R H,Liu H Y, et a1.Tomatochlorosisvirus: a new whitefly-transmitted, phloem-limited, bipartite closterovirus of tomato [J].Phytopathology, 1998, 88(5): 402-409.
[6] Accotto G P, Vaira A M, Vecchiati M, et al. First report ofTomatochlorosisvirusin Italy [J]. Plant Disease, 2001, 85(11): 1208.
[7] Dalmon A, Bouyer S, Cailly M, et al. First report ofTomatochlorosisvirusandTomatoinfectiouschlorosisvirusin tomato crops in France [J]. Plant Disease, 2005, 89(11): 1243.
[8] Navas-Castillo J, Camero R, Bueno M, et al. Severe yellowing outbreaks in tomato in Spain associated with infections ofTomatochlorosisvirus[J]. Plant Disease, 2000, 84(8): 835-837.
[9] Fiallo-OlivéE, Hamed A A, Moriones E, et al. First report ofTomatochlorosisvirusinfecting tomato in Sudan[J]. Plant Disease, 2011, 95(12): 1592.
[10]Segev L, Wintermantel W M, Polston J E, et al. First report ofTomatochlorosisvirusin Israel [J]. Plant Disease, 2004, 88(10): 1160.
[11]Çevik B, Erklß G. First report ofTomatochlorosisvirusin Turkey [J]. Plant Pathology, 2008, 57(4): 767.
[12]Hirota T, Natsuaki T, Murai T, et al. Yellowing disease of tomato caused byTomatochlorosisvirusnewly recognized in Japan [J]. Journal of General Plant Pathology, 2010, 76(2): 168-171.
[13]Tsai W S, Shih S L, Green S K, et al. First report of the occurrence ofTomatochlorosisvirusandTomatoinfectiouschlorosisvirusin Taiwan [J]. Plant Disease, 2004, 88(3): 311.
[14]Zhao R N, Wang R, Wang N, et al. First report ofTomatochlorosisvirusin China [J]. Plant Disease, 2013, 97(8): 1123.
[15]Abou-Jawdah Y, El Mohtar C, Atamian H, et al. First report ofTomatochlorosisvirusin Lebanon [J]. Plant Disease, 2006, 90(3): 378.
[16]Barbosa J C, Teixeira A P M, Moreira A G, et al. First report ofTomatochlorosisvirusinfecting tomato crops in Brazil [J]. Plant Disease, 2008, 92(12): 1709.
[17]Alvarez-Ruiz P, Jimenez C G, Leyva-López N E, et al. First report ofTomatochlorosisvirusinfecting tomato crops in Sinaloa Mexico [J]. Plant Pathology, 2007, 56(6): 1043.
[18]Martínez-Zubiaur Y, Fiallo-Olivé E, Carrillo-Tripp J, et al. First report of Tomatochlorosisvirusinfecting tomato in single and mixed infections withTomatoyellowleafcurlvirusin Cuba [J]. Plant Disease, 2008, 92(5): 836.
[19]Wintermantel W M, Polston J E, Escudero J, et al. First report ofTomatochlorosisvirusin Puerto Rico [J]. Plant Disease, 2001, 85(2): 228.
[20]Karwitha M, Feng Z K, Yao M, et al. The complete nucleotide sequence of the RNA1 of a Chinese isolate ofTomatochlorosisvirus[J]. Journal of Phytopathology, 2014, 162(6): 411-415.
[21]赵汝娜, 王蓉, 师迎春, 等. 侵染甜椒的番茄褪绿病毒的分子鉴定[J]. 植物保护, 2014, 40(1): 128-130.
[22]刘永光, 魏家鹏, 乔宁, 等. 番茄褪绿病毒在山东暴发及其防治措施[J]. 中国蔬菜, 2014(5): 67-69.
[23]Zhao L M, Li G, Gao Y, et al. Molecular detection and complete genome sequences ofTomatochlorosisvirusisolates from infectious outbreaks in China [J]. Journal of Phytopathology, 2014, 162(10): 627-634.
[24]胡京昂, 万秀娟, 李自娟, 等. 河南番茄褪绿病毒的分子鉴定[J]. 中国蔬菜, 2015(12): 25-28.
[25]孙国珍, 高利利, 陆文利, 等. 河北省设施番茄褪绿病毒分子检测和鉴定研究[J]. 北方园艺, 2015(9): 95-98.
[26]高利利, 孙国珍, 王勇, 等. 天津地区番茄褪绿病毒的分子检测和鉴定[J]. 华北农学报, 2015, 30(3): 211-215.
[27]King A M Q, Adams M J, Carstens E B, et al. Virus taxonomy: ninth report of the international committee on taxonomy of viruses [M]. Amsterdam: Elsevier Academic Press, 2011.
[28]Zhou Y, Yan J Y, Qiao G H, et al. First report ofTomatochlorosisvirusinfecting eggplant (Solanummelongena) in China [J]. Plant Disease, 2015, 99(11): 1657.
[29]周莹, 黄金宝, 乔广行, 等. 北京地区茄子感染番茄褪绿病毒的分子鉴定[J]. 植物保护学报,2016, 43(1): 168-172.
(责任编辑:杨明丽)
Molecular identification ofTomatochlorosisvirusinfecting tomato in Guangdong Province
Tang Yafei1,2, He Zifu1,2, She Xiaoman1, Lan Guobing1
(1.PlantProtectionResearchInstitute,GuangdongAcademyofAgriculturalSciences,Guangzhou510640,China;2.GuangdongProvincialKeyLaboratoryofHighTechnologyforPlantProtection,Guangzhou510640,China)
A new disease on tomato recently occurs in Guangdong Province. The symptoms of the diseased tomato plants include leaf chlorotic, vein dark-greening and leaf thickening. The expected DNA fragments were amplified from all 6 diseased samples with two specific primer pairs forHSP70 gene ofTomatochlorosisvirus(ToCV) by RT-PCR. And a full length ofHSP70 (1 665 bp) was obtained by sequencing, which shared high nucleotides identity with those from other ToCV isolates. The result indicated that the collected tomato samples have been infected by ToCV in Guangdong. But there exists great difference in nucleotide sequence ofHSP70 between the isolate from Guangdong (GD01) and other ToCV isolates from all over the world. The nucleotide sequence ofHSP70 from isolate GD01 shared the highest identity (81.8%) with isolate tomato from Cyprus and JU_20 from Jordan. This paper was the first report on the occurrence of ToCV in Guangdong, and also the first report of significant difference in nucleotide sequence ofHSP70 between ToCV GD01 and other isolates.
tomato viral disease;Tomatochlorosisvirus;HSP70 gene
2016-04-20
2016-06-27
公益性行业(农业)科研专项(201303028);广东省科技计划项目(2014B070706017,2015A020209070)
S 432.41
A
10.3969/j.issn.0529-1542.2017.02.022
Research Notes
* 通信作者 E-mail:hezf@gdppri.com

