肝癌中CpG甲基化与microRNA-34沉默的研究
林 兰,刘继斌
(南通市肿瘤医院,江苏 南通 226361)
肝癌中CpG甲基化与microRNA-34沉默的研究
林 兰,刘继斌
(南通市肿瘤医院,江苏 南通 226361)
目的 探讨肝癌中miR-34家族甲基化的价值。方法 入组经病理组织学确诊的43例原发性肝癌手术病例以及临近癌旁组织,使用甲基化特异性PCR(MSP)方法分析miR-34在肝癌组织和癌旁组织中的甲基化状态,使用逆转录PCR法验证MSP结果。结果 肝癌组织中miR-34a和miR-34b/c甲基化频率启动分别为72.1%和79.1%,均高于癌旁组织(P<0.05);miR-34a和miR-34b/c在肝癌组织中明显比癌旁组织表达下调(P<0.05);肝癌组织中miR-34b表达与CpG甲基化呈负相关(P<0.05)。结论 肝癌组织中DNA甲基化可能参与了miR-34b的失活。
微小RNA-34;甲基化;肝癌
原发性肝癌是全球最为常见的恶性肿瘤之一,新发病例居我国所有恶性肿瘤的第4位;死亡病例居全部癌症死亡病例第2位[1]。某些微小RNA(microRNA,miR)的表达异常可促进肝癌的发生。与编码基因相同,miR启动子区域CpG岛甲基化异常,也可以引起miR表达缺失,影响肿瘤恶性表型的发展。本文初步探讨了肝癌中CpG甲基化与miR-34沉默的关系。
1 资料与方法
1.1 研究对象
1.1.1 肝癌病例组 收集2010年4月至2011年10月南通市肿瘤医院收治的经病理组织学确诊的43例原发性肝癌手术病例以及临近癌旁组织,无年龄、性别限制,患者的人口统计数据包括性别、年龄、乙型肝炎病毒感染情况、丙型肝炎病毒感染情况、乙型肝炎肝硬化情况、具有完备的流行病学和临床资料(如肿瘤的大小、单个或多发、有无包膜及有无门静脉癌栓、临床分期和Child分级等)、已经随访和采集外周血生物样本;手术前未接受过放疗、生物治疗或抗肿瘤药物化疗。该研究所需标本均经过南通市伦理委员会同意通过。
1.1.2 资料和标本的收集 调查问卷主要包括一般情况、既往史、家族史、临床诊断和治疗情况等。为保证调查和随访资料的质量,课题组统一制订问卷调查和随访工作手册,培训调查和随访人员,统一方法和标准。对所有资料由专人进行编码,并在复核无误后双轨录入计算机。对所有病例定期随访,每3个月一次直至死亡。
每个研究对象均采用真空抗凝采血管采集手术前静脉血5 mL,4 h内离心分别分离血浆、白细胞和红细胞至1.5 mL离心管中(均各保存2份),-80 ℃冷冻保存备用。同时,手术切除的组织标本离体10 min内取材,经焦碳酸二乙酯处理的冷PBS冲洗后,液氮冷冻保存备用。
1.2 方法
1.2.1 组织DNA和RNA的提取 分别采用QIAamp DNA mini kit、QIAamp Circulating Nucleic Acid Kit以及Trizol提取冰冻组织血液中DNA和RNA,其DNA和RNA浓度和纯度均经NanoDrop 2000分光光度计检测符合研究需要。
1.2.2 甲基化特异性PCR(MSP) 基因组DNA(1 μg)严格按照EZ DNAMethylation-GoldTMKit甲基化试剂盒说明书操作。甲基化和非甲基化引物使用有关文献报道过的[2-5]。阳性对照采用Qiagen甲基化和非甲基化人类DNA质控品,阴性对照采用蒸馏水。miR-34a扩增程序为[4]:95 ℃ 5 min,95个循环; 2 ℃,20 s;68 ℃,30 s; 72 ℃,30 s; 66 ℃,2个循环和65 ℃,34个循环;最后72 ℃为4 min。miR-34b/c扩增程序为[4],37 ℃,20 s,95个循环; 61 ℃,30 s和72 ℃,30 s,4 min。PCR产物在质量分数2.5%琼脂糖凝胶和溴化乙锭染色。
1.2.3 定量逆转录PCR(RT-PCR) miR-34a、miR-34b和miR-34c的表达使用定量RT-PCR进行检测。总RNA(1 μg)使用miR特异性茎环逆转录引物和逆转录酶XL进茎环逆转录(RT)。cDNA在进行Real-time PCR前首先用水进行20倍稀释,Real-time PCR 采用Power SYBR Green PCR Master Mix进行扩增。Real-time PCR是在ABI 7900 Real-Time PCR 系统里以一式三份的形式进行操作的。使用小核RNA U6进行了归一化处理[6-7]。相关表达通过公式2-ΔCt计算,ΔCt=ctmirna-ctu6。
1.3 统计学处理 应用EpiData对流行病学资料和实验室数据进行录入和管理,采用SAS软件对数据进行分析,分析方法包括计数资料的χ2检验、非参数检验,单因素和多因素Logistic回归分析,COX回归模型分析等,检验水准α=0.05。
2 结果
2.1 肝癌患者的临床病理特征 43例肝癌患者中,男31例,女12例;中位年龄53岁;乙型肝炎病毒感染者28例,占65.1%;饮酒者13例,占30.2;吸烟者15例,占34.9%。见表1。
2.2 miR-34在肝癌组织和癌旁组织中的甲基化状态 miR-34a启动子在43例肝癌组织中有31例(72.1%)发生了甲基化,高于癌旁组织22例(51.2%)(P=0.046)。79.1%(34/43)的肝癌组织和53.5%(23/43)的癌旁组织显示miR-34b/c启动子发生甲基化(P=0.012)。见图1。
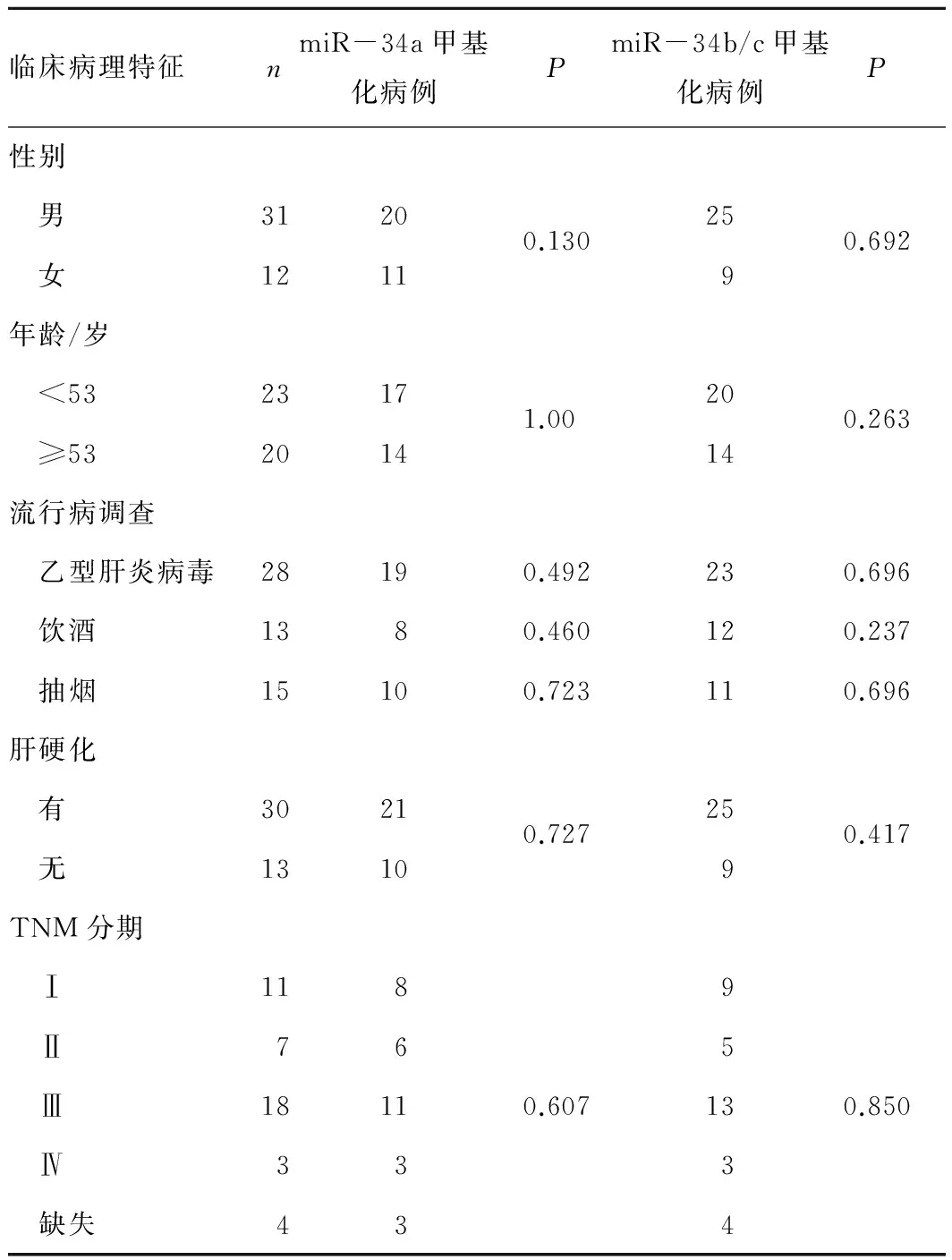
表1 肝癌患者miR-34甲基化与临床病理特征的关系
2.3 miR表达和miR甲基化之间的关系 为了进一步调查在肝癌中是否DNA甲基化沉默了miR-34家族的表达,我们评估了在甲基化和非甲基化组miR-34a、miR-34b和miR-34c表达水平,结果显示,miR-34b表达水平在甲基化组明显低于非甲基化组(P=0.026)。2组miR-34a和miR-34c没有观察到明显变化。见图2。
3 讨论
miR作为一类重要的癌基因和抑癌基因,参与肿瘤维持生长信号、逃避生长抑制、抵抗凋亡、无限复制能力、引起血管生成、激活侵袭和转移、调控能量代谢、逃避免疫摧毁等生物学行为[8]。以肝癌为例,miR-7可以通过调控PI3K/Akt/mTOR信号通路抑制肝癌的生长[9];miR-26a可以通过调控cMet信号通路抑制肝癌血管生成,影响肝癌患者的预后转归[10]。尽管miR可以靶向调控多个mRNA,发挥其重要生物学功能,但是miR异常表达的调控机制尚不明确。
DNA甲基化和组蛋白修饰也一直是基因表达调控方面的研究热点[11]。已有研究[12]证据提示,miR异常表达可能受到DNA甲基化的影响。miR200b/miR200a/miR-429簇上游4个异常低甲基化的CpG位点可以在肝癌起始细胞和早期肝癌组织中上调miR-429的表达,进而通过一些信号通路促进肝细胞自我更新、恶性分化、化疗抵抗以及致瘤等[13]。
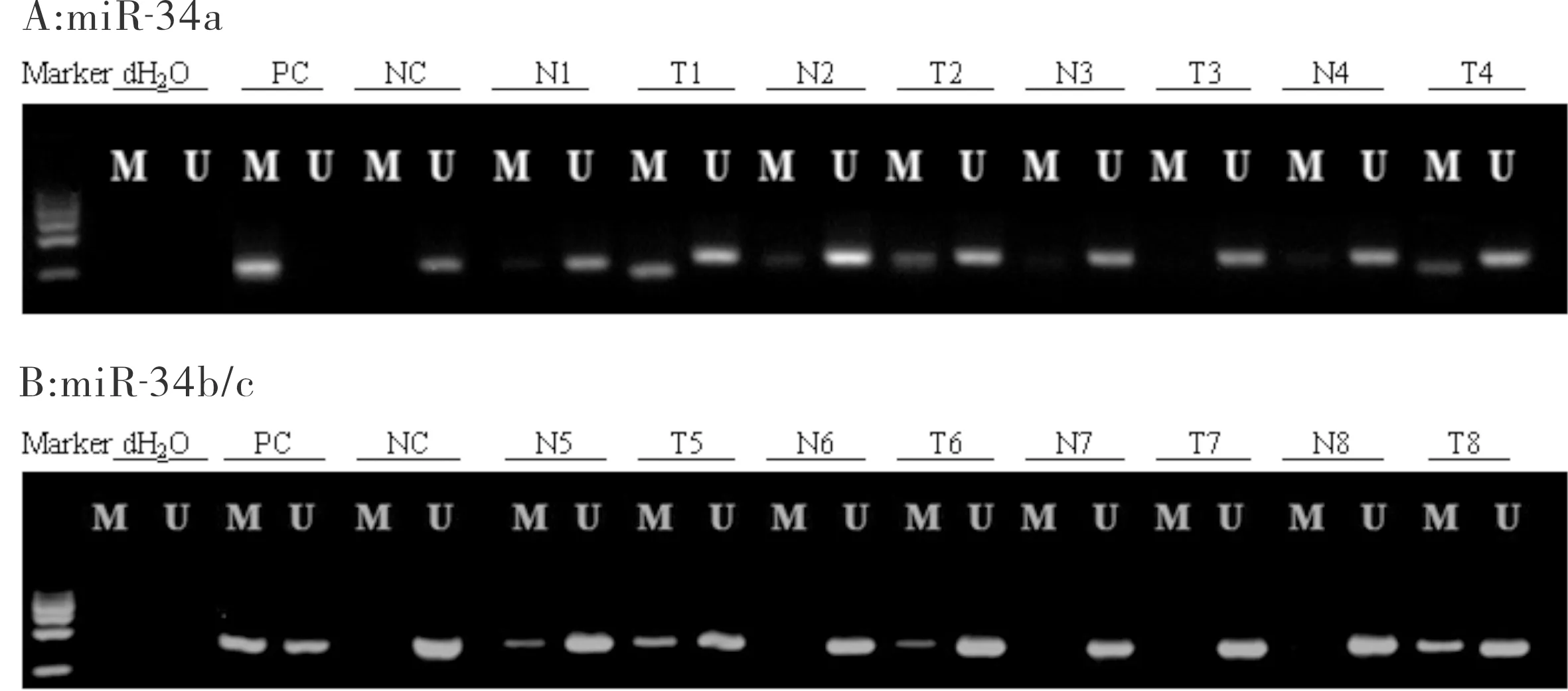
图1 肝癌组织和癌旁组织中miR-34a(A)和miR-34b/c(B)的MSP检测结果

图2 肝癌组织中miR-34b的甲基化状态与表达的关系
miR-34家族是P53的转录目标,气可以抑制肝癌细胞的增殖、转移和侵袭。miR-34家族DNA甲基化改变已经在多种恶性肿瘤中被发现。在我们的研究中,我们首先探讨了肝癌中miR-34家族CpG岛甲基化,并且通过甲基化特异性PCR分析发现miR-34a和miR-34b/c甲基化的频率分别为72.1%和79.1%。异常的DNA甲基化可抑制miR的表达,我们发现在肝癌组织中miR-34家族的表达水平被下调,此结果提示我们在肝癌组织中miR-34家族具有抑制肿瘤的特性。此外,进一步的分析表明,对于本研究的肝癌患者,甲基化的肿瘤组织中miR-34b的表达水平明显低于未甲基化组织,提示我们miR-34b启动子区域CpG岛异常甲基化可能沉默了其的表达。总之,肝癌组织中DNA甲基化可能参与了miR-34b的失活。
[1]陈建国,张思维,陈万青.中国2004-2005年全国死因回顾抽样调查肝癌死亡率分析[J].中华预防医学杂志,2010,44(5):383-389.
[2]LODYGIN D,TARASOV V,EPANCHINTSEV A,et al.Inactivation of miR-34a by aberrant CpG methylation in multiple types of cancer[J].Cell Cycle,2008 ,7(16):2591-2600.
[3]TOYOTA M,SUZUKI H,SASAKI Y,et al.Epigenetic silencing of microRNA-34b/c and B-cell translocation gene 4 is associated with CpG island methylation in colorectal cancer[J].Cancer Res,2008,68(11):4123-4132.
[4]VOGT M,MUNDING J,GRÜNER M,et al.Frequent concomitant inactivation of miR-34a and miR-34b/c by CpG methylation in colorectal,pancreatic,mammary,ovarian,urothelial,and renal cell carcinomas and soft tissue sarcomas[J].Virchows Arch,2011,458(3):313-322.
[5]LUJAMBIO A,CALIN GA,VILLANUEVA A,et al.A microRNA DNA methylation signature for human cancer metastasis[J].Proc Natl Acad Sci U S A,2008,105(36):13556-13561.
[6]HU Z,CHEN J,TIAN T,et al.Genetic variants of miRNA sequences and non-small cell lung cancer survival[J].J Clin Invest,2008,118(7):2600-2608.
[7]HU Z,SHU Y,CHEN Y,et al.Genetic polymorphisms in the precursor MicroRNA flanking region and non-small cell lung cancer survival[J].Am J Respir Crit Care Med,2011,183(5):641-648.
[8]KONG YW,FERLAND-MCCOLLOUGH D,JACKSON TJ,et al.microRNAs in cancer management[J].Lancet Oncol,2012,13(6):e249-e258.
[9]PASQUINELLI AE.MicroRNAs and their targets: recognition,regulation and an emerging reciprocal relationship[J].Nat Rev Genet,2012,13(4):271-282.
[10]JIANG J,GUSEV Y,ADERCA I,et al.Association of MicroRNA expression in hepatocellular carcinomas with hepatitis infection,cirrhosis,and patient survival[J].Clin Cancer Res,2008,14(2):419-427.
[11]FURUTA M,KOZAKI KI,TANAKA S,et al.miR-124 and miR-203 are epigenetically silenced tumor-suppressive microRNAs in hepatocellular carcinoma[J].Carcinogenesis,2010,31(5):766-776.
[12]DATTA J,KUTAY H,NASSER MW,et al.Methylation mediated silencing of MicroRNA-1 gene and its role in hepatocellular carcinogenesis[J].Cancer Res,2008,68(13):5049-5058.
[13]KERKEL K,SPADOLA A,YUAN E,et al.Genomic surveys by methylation-sensitive SNP analysis identify sequence-dependent allele-specific DNA methylation[J].Nat Genet,2008,40(7):904-908.
Study on Methylation of MicroRNA-34 in the Hepatocellular Carcinoma
LIN Lan,LIU Jibin
(NantongTumourHospital,Nantong226361,China)
Objective To explore the value of methylation of microRNA-34 (miR-34) family in the hepatocellular carcinoma.Methods Tumor and adjacent non-tumor tissues were collected from 43 hepatocellular carcinoma patients who underwent surgery.Methylation specific PCR (MSP) method was used to analyze the methylation status of miR-34 in the hepatocellular carcinoma tissues and the adjacent non-tumor tissues,and the results were verified by reverse transcription PCR method.Results The methylation frequencies of miR-34a and miR-34b/c were 72.1% and 79.1% in the hepatocellular carcinoma tissues,which were significantly higher than that in the adjacent non-tumor tissues (P<0.05),respectively.The expressions of miR-34a and miR-34b/c were significantly down-regulated in the hepatocellular carcinoma tissues compared with adjacent non-tumor tissues (P<0.05).The expression of miR-34b were negatively related with DNA methylation in the hepatocellular carcinoma tissues.Conclusion DNA methylation may be involved in the inactivation of miR-34b in HCC.
microRNA-34;methylation;hepatocellular carcinoma
中国博士后科学基金资助项目(编号:2013M541563);江苏省六大人才高峰基金资助项目(编号:wsw-009)
林兰(1968-),女,副主任技师,主要从事肿瘤免疫学检验以及肿瘤预后研究工作。E-mail :kindrabbit75@163.com
刘继斌(1974-),男,博士,主任医师,主要从事肿瘤生物治疗相关研究。E-mail :tians2008@163.com
10.3969/j.issn.1673-5412.2017.01.005
R735.7;R730.23
A
1673-5412(2017)01-0019-04
2016-12-20)

