烟草基因组中AGO蛋白的生物信息学分析
蔡健宇,武晓云,郭玉双,李翔宇
(1.浙江农林大学 农业与食品科学学院,浙江 临安 311300;2.贵州省烟草科学研究院 烟草行业分子遗传重点实验室,贵州 贵阳 550081;3.黑龙江省农业科学院 作物研究所,黑龙江 哈尔滨 150086)
烟草基因组中AGO蛋白的生物信息学分析
蔡健宇1,武晓云1,郭玉双2*,李翔宇3
(1.浙江农林大学 农业与食品科学学院,浙江 临安 311300;2.贵州省烟草科学研究院 烟草行业分子遗传重点实验室,贵州 贵阳 550081;3.黑龙江省农业科学院 作物研究所,黑龙江 哈尔滨 150086)
对在NCBI上登录的烟草AGO蛋白(argonaute proteins)的信息进行了整理,并对其组成成分、系统发育树、信号肽、跨膜结构域、细胞定位、亲水性/疏水性、蛋白质的二/三级结构等进行了预测和分析。结果表明:在烟草基因组中共有22个AGO蛋白,可分为8类;烟草的AGO蛋白与拟南芥的AGO一样在进化上分为3个分支,其编码的蛋白是一个无跨膜结构域、无信号肽/导肽的亲水性蛋白,α-螺旋散布于整个蛋白质中,具有PAZ和PIWI两个特征结构域。此外,烟草的AGO2/4/7/16/PNH1定位在细胞核中,AGO10定位在细胞质中,AGO5定位在线粒体中。
生物信息学;烟草;Argonaute (AGO);分子结构;功能预测
AGO蛋白(argonaute proteins)作为RNA诱导的沉默复合物(RISC)的核心成分,是一类广泛存在于生物界中、高度保守的一类蛋白。AGO蛋白最初由Bohmert等[1]在拟南芥中发现,由于该拟南芥AGO突变体的外观与船蛸属(Argonauta)一种小章鱼的触角相似,因此被命名为argonaute,对应的蛋白即为AGO1。AGO蛋白家族最初被确定为3种蛋白质:果蝇的PIWI蛋白、拟南芥的AGO1蛋白和ZWILLE蛋白。Yigit等根据AGO蛋白的系统进化关系,对来自植物、动物和真菌中代表物种的AGO蛋白家族成员做了进一步分类,即能够结合miRNA和siRNA的AGO-like蛋白、能结合piRNA的PIWI-like蛋白和仅在蠕虫中发现且能结合次生siRNA的GROUP3[2-4]。在不同物种中AGO家族成员的数量也存在一定的差异,例如在线虫(Caenorhabditiselegans)基因组中存在27个AGO基因[5],在拟南芥(Arabidopsisthaliana) 基因组中有10个AGO基因[6],在果绳(Drosophilamelanogaster)基因组中含有5个AGO基因[7],在水稻(Oryzasativa)基因组中包含19个AGO基因[8],在人类(Homosapiens)基因组中编码8个AGO蛋白[9]。
AGO蛋白由700~1200个氨基酸组成(约90 ~100 kDa),由N末端、PAZ、MID和PIWI四个结构域组成,但在一些植物AGO蛋白中也发现了一些新的结构域[3,10]。PAZ区能非序列特异性识别结合双链小RNA 3′末端悬垂的2个核苷酸;MID与PIWI界面处的“保守口袋”识别结合小RNA 5′端第1位核苷酸,其中PIWI 结构域是AGO蛋白行使切割功能的活性中心,具有类似于RNA酶H催化中心的三级结构;具有切割功能的AGO蛋白能对靶mRNA进行切割并降解,但并不是所有含PIWI结构域的AGO蛋白都具有切割功能[2,11-13]。
AGO蛋白在生物界中的作用十分广泛而又极其重要。目前已知的有以下3个方面:在动、植物中,AGO蛋白与小RNA共同参与维持基因组的稳定性,调控个体的生长发育水平以及对生物和非生物胁迫的反应等重要的生理活动,对靶mRNA进行切割或翻译水平上的抑制以及在转录和转录后水平上控制转座元件的移动;在原生动物中,AGO蛋白能够指导拟核形成过程中DNA的去除;在真菌中,它们能指导异染色质的组装等[14-16]。不过,现今仍有许多AGO蛋白的未知功能有待发掘。
烟草相对于马铃薯、番茄等茄科作物而言,已知信息多,易培养,针对它的各种技术成熟,转基因技术稳定,是植物学研究中的模式生物,已被广泛地用于研究植物细胞周期、氧化胁迫以及尼古丁的合成等基础生物学问题。同时,烟草也是研究植物与病毒相互作用的理想材料,被用于研究病毒的复制与运动、致病机制、植物的抗病反应等。本文利用生物信息学方法,通过National Center for Biotechnology Information (NCBI)核酸和蛋白质数据库,对普通烟草的AGO蛋白进行了整理,并对其基因序列和氨基酸序列的理化性质、结构特征、功能及系统演化关系等进行了预测和分析,以期为进一步研究烟草AGO蛋白的功能提供一定的理论依据。
1 材料与方法
1.1 序列下载
数据资料来源于NCBI核酸及蛋白数据库中已登录的烟草AGO蛋白的核酸序列及其对应的氨基酸序列: NtAGO1(Accession NO: XM_016590746.1)、NtAGO2(Accession NO:XM_016629769.1)、NtAGO4(Accession NO: XM_016618801.1)、NtAGO5(Accession NO:XM_016651704.1)、NtAGO7(Accession NO: XM_016605722.1)、NtAGO10(Accession NO:XM_016625398.1)、NtAGO16(Accession NO: XM_016645858.1)、NtAGO PNH1(Accession NO: XM_016646861.1),以及The Arabidopsis Information Resource (TAIR)数据库中拟南芥AGO蛋白的核酸序列及其对应的氨基酸序列: AtAGO1(Locus: AT1G48410)、AtAGO2(Locus: AT1G31280)、AtAGO3(Locus: AT1G31290)、AtAGO4(Locus: AT2G27040)、AtAGO5(Locus: AT2G27880)、AtAGO6(Locus: AT2G32940)、AtAGO7(Locus: AT1G69440)、AtAGO9(Locus: AT5G21150)、AtAGO10(Locus: AT5G43810)。
1.2 生物信息学分析软件
以CBS(http://www.cbs.dtu.dk)、EBI(http://www.ebi.ac.uk)、ExPASy(http://cn.expasy.org)、NCBI(http://www.ncbi.nlm.nih.gov)等网站提供的在线生物信息学软件为基础,结合DNAMAN6.0软件对烟草和拟南芥中的AGO蛋白进行相关分析[17]。其中,核酸及氨基酸序列的组成成分和理化性质分析使用DNAMAN6.0和ProtParam;开放阅读框(Open reading frame, ORF)的查找和翻译利用NCBI的ORF Finder;核酸及对应氨基酸序列的同源性比对和系统发育树构建则使用在线的Blast、Clustal Omega、MEGA5.05和Tree-V32软件完成;特征结构域及信号肽的预测利用在线工具ScanProsite、TargetP、ChloroP、SignalP完成;用TMHMM、WoLF PSORT、Protscale在线软件进行跨膜结构域、亚细胞定位及亲/疏水性的分析;蛋白质二/三级结构的预测利用SOPMA和SWISS-MODEL在线工具完成。
2 结果与分析
2.1 烟草AGO蛋白的核酸序列及其对应氨基酸序列的理化性质分析
对NCBI基因库中登入的烟草AGO蛋白进行整理,共有22个,发现烟草中的AGO蛋白分为8类,如表1所示。其中AGO1-3、AGO1-4、AGO4A、AGO10-1和AGO10-2含有3个剪切突变体,AGO4-3、AGO16-1和AGO16-2含有2个剪切突变体。此外,以AGO1-1、AGO2-1、AGO4-1、AGO5、AGO7-1、AGO10-1、AGO16-1和AGO PNH1-1为烟草各类AGO蛋白的代表进行后续的分析。
通过NCBI的ORF Finder、ExPASy的ProtParam和DNAMAN 6.0软件对这8类AGO蛋白:NtAGO1(XM_016590746.1)、NtAGO2(XM_016629769.1)、NtAGO4(XM_016618801.1)、NtAGO5(XM_016651704.1)、NtAGO7(XM_016605722.1)、NtAGO10(XM_016625398.1)、NtAGO16(XM_016645858.1)、NtAGO PNH1(XM_016646861.1)的核酸序列及对应氨基酸序列进行分析[18],结果见表2。从表2可以看出:烟草中不同AGO基因ORF的碱基数及其编码的氨基酸残基数虽然都略有差异,但都符合AGO蛋白的标准[19];不同AGO蛋白的分子量、理论等电点、酸性与碱性氨基酸的比例基本一致。蛋白质的不稳定系数与其结构有关,当一个蛋白质的不稳定系数大于40%时,则表示该蛋白质不稳定;当蛋白质的不稳定系数小于40%时,则表示该蛋白质稳定。由表2可知,烟草中的各类AGO蛋白结构都不稳定。除AGO2和AGO7外,其他AGO基因都含有较多的外显子。除AGO5和AGO10外,其他AGO基因都含有1~2个同源基因。

表1 烟草中AGO蛋白分类统计结果
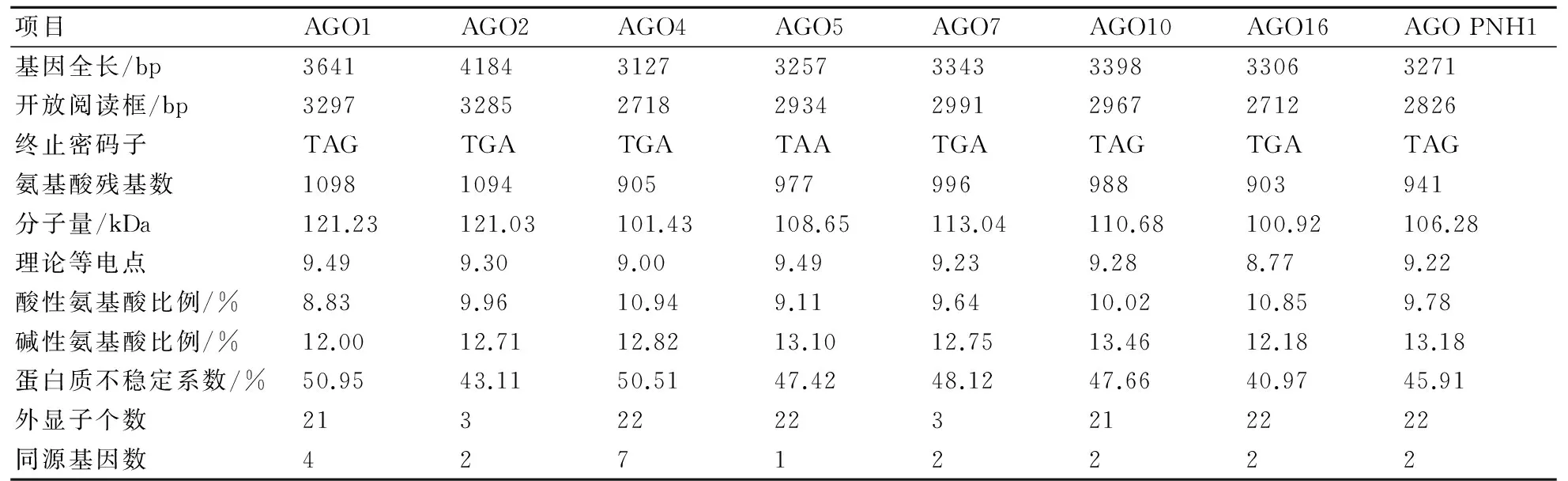
表2 烟草AGO蛋白的核酸序列及其对应氨基酸序列的理化性质
2.2 烟草AGO基因及与拟南芥AGO基因的氨基酸序列比对分析
用EBI的Clustal Omega程序对烟草中不同AGO蛋白的氨基酸序列进行多序列比对,结果如图1所示。烟草不同AGO蛋白之间的氨基酸序列同源性并不高,其中只有134个氨基酸是完全一致的,141个氨基酸为保守氨基酸。同时,利用ExPASy的ScanProsite对烟草AGO的结构域进行分析,发现所有AGO蛋白都含有该家族的两个特征结构区,即PAZ和PIWI。其中AGO4和AGO16的结构域相似度很高;AGO2和AGO7的结构域相似度很高;AGO1、AGO5、AGO10和AGO PNH1的结构域相似度很高。

符号“*”、“:”、“.”分别表示氨基酸完全相同、保守和半保守3种情况。保守结构域motif用方框表示。
用MEGA5.05和Tree-V32对烟草和拟南芥的AGO蛋白的氨基酸序列进行系统发育树分析,算法为泊布校正,重复检测1000次,进化树中的数字代表计算频率值,结果如图2所示。烟草中的AGO蛋白与拟南芥中的AGO蛋白一一对应,其中烟草中的AGO PNH1与拟南芥的AGO10有较近的亲缘关系,烟草中的AGO16和拟南芥的AGO6有较近的亲缘关系。此外,烟草的AGO蛋白也与拟南芥一样在进化上可分为3类: AGO1、AGO5、AGO10和AGO PNH1为1个进化类; AGO4和AGO16为1个进化类; AGO2和AGO7为1个进化类。
2.3 烟草AGO蛋白信号肽及导肽的预测与分析
信号肽(signal peptide)是新合成多肽链中用于指导蛋白质的跨膜转移(定位)的N-末端的氨基酸序列。导肽又称转运肽(Transit peptides)或导向序列(targeting sequence),它是游离核糖体上合成的蛋白质的N-端信号。信号肽及导肽的预测和分析,对正确认识和理解蛋白质的细胞定位及结构域的分选有着重要的作用。分别用CBS的TargetP1.1 Server、ChloroP 1.1 Server和SignalP 4.1 Server对烟草AGO蛋白氨基酸序列的信号肽及导肽进行预测,结果如表3所示。结果表明,除AGO1、AGO5、AGO7外的其他AGO蛋白含有叶绿体转运肽、线粒体目标肽及信号肽的分值均较低,且无对应氨基酸切割位点,由此可推测AGO2、AGO4、AGO10、AGO16、AGO PNH1不具有导肽和信号肽。AGO1虽然线粒体目标肽的分值较高,但由于没有相应切割位点,其具有导肽的可能性较低。AGO5在ChloroP1.1 Server中预测有叶绿体转运肽,但在TargetP1.1 Server中预测分值较低,有待进一步检验。AGO7在ChloroP 1.1 Server和TargetP 1.1 Server中都预测出有叶绿体转运肽,但由于没有预测到切割位点,其具有导肽的可能性需要进一步检验。综上所述,烟草AGO蛋白可能都为胞浆蛋白。
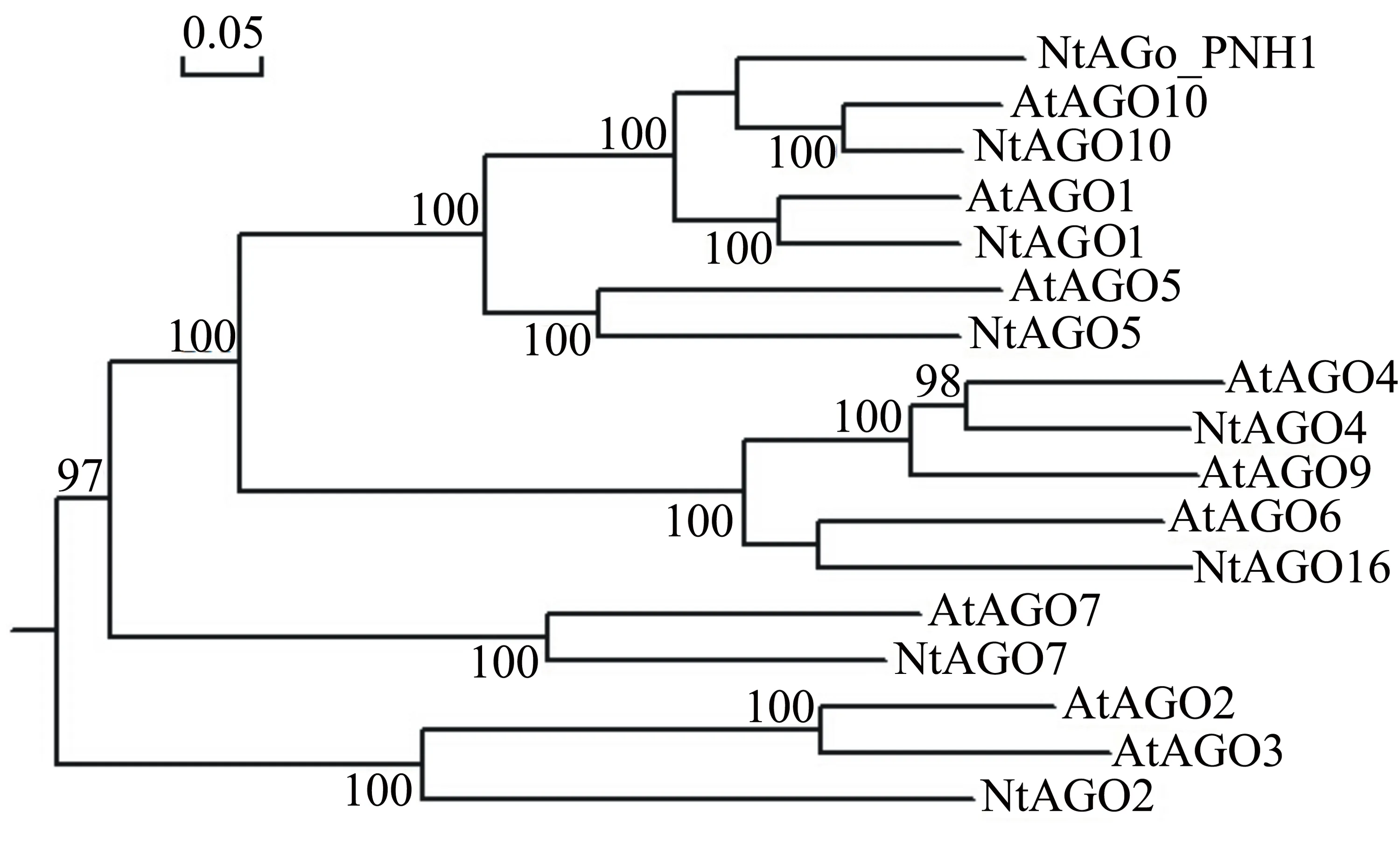
图2 烟草与拟南芥中AGO蛋白的系统发育树

AGO蛋白叶绿体转运肽(cTP)线粒体目标肽(mTP)信号肽(sP)AGO10.442/0.0640.5920.037AGO20.442/0.0600.3300.020AGO40.432/0.4400.0300.016AGO50.552/0.3040.4340.000AGO70.515/0.7490.0240.012AGO100.484/0.2930.0640.005AGO160.438/0.2240.0820.171AGOPNH10.447/0.1310.1960.013
2.4 烟草AGO蛋白跨膜结构域的预测与分析
跨膜结构域是膜内在蛋白与膜脂相结合的部位,一般由20个左右的疏水氨基酸残基组成,形成α-螺旋,固着于细胞膜上起“锚定”作用。跨膜结构域的预测和分析,对正确认识和理解蛋白质的结构、功能、分类及在细胞中的作用部位均有着重要的指导意义。使用CBS的TMHMM 2.0对烟草AGO蛋白氨基酸序列的跨膜结构域进行预测,结果如图3所示。预测结果表明,8个烟草AGO蛋白的跨膜结构域可能性均小于1,且都位于膜外,即不存在跨膜结构域。结合上述导肽/信号肽的预测结果,可以推断烟草AGO蛋白在膜内合成前体蛋白后,不进入其他细胞器,而是留在细胞质内加工成成熟的蛋白发挥功能,并不与膜脂结合。
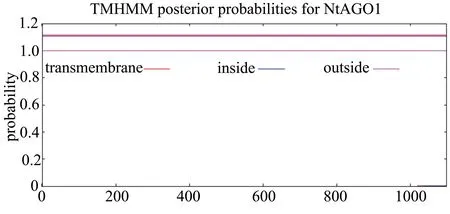
图3 烟草AGO蛋白跨膜结构域预测结果(以AGO1为例)
2.5 烟草AGO蛋白的亚细胞定位分析
蛋白质在细胞中合成后会被转运到特定的细胞器中,只有被转运到正确的部位才能参与细胞的各种生命活动。蛋白质的亚细胞定位是蛋白质功能研究的一个重要方面,蛋白在细胞中的定位与蛋白执行的功能密切相关[20]。因此,对烟草AGO蛋白在细胞中的定位作出预测将有助于对其功能的预测。利用WoLF PSORT在线工具对烟草AGO蛋白的氨基酸序列进行亚细胞定位分析,结果见表4。从表4中可知,烟草中不同AGO蛋白的亚细胞定位差异很大,AGO2、AGO4、AGO7、AGO16和AGO PNH1定位在细胞核中,AGO5定位在线粒体中,AGO1可能在细胞核中也可能在细胞质基质中,AGO10定位在细胞质中的可能性最大。
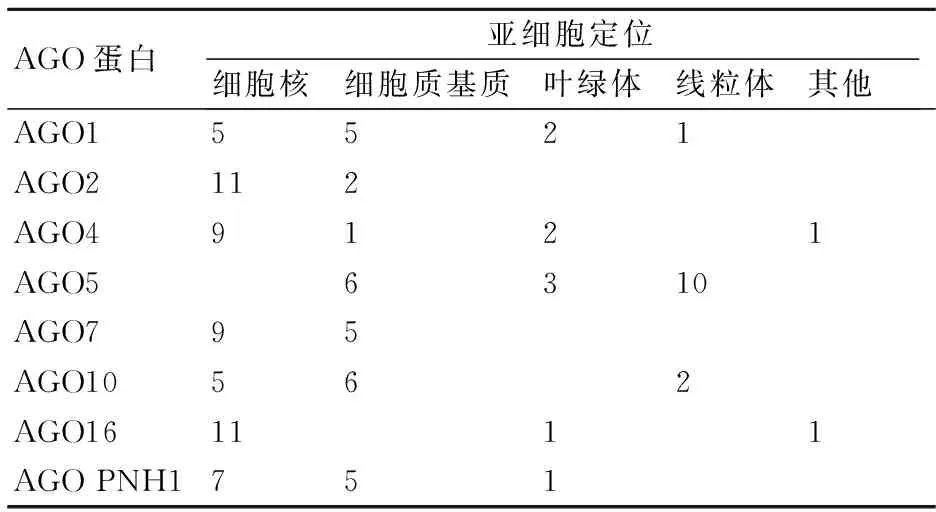
表4 烟草AGO蛋白的亚细胞定位预测结果(可信度)
2.6 烟草AGO蛋白亲水性/疏水性的预测与分析
构成蛋白质的20种氨基酸都具有疏水性,即氨基酸具有远离周围的水分子,将自己埋进蛋白质核心的相对趋势。这一趋势加上空间立体条件和其它一些因素,决定了一个蛋白质最终折叠成的三维空间构象。因此,对亲水性/疏水性的预测和分析结果可为烟草AGO蛋白次级结构的预测提供重要的理论依据。用ExPASy的ProtScale软件对烟草AGO蛋白的亲水性/疏水性进行预测,结果如图4所示。AGO1多肽链的第573位的谷氨酸Glu具有最低的分值-3.622,亲水性最强;第725位的缬氨酸Val具有最高的分值2.556,疏水性最强。整条多肽链表现为亲水性,没有明显的疏水区域。对烟草中其他AGO蛋白的亲水性/疏水性进行分析,与AGO1的结果类似,即整条多肽链没有明显的疏水区域,表现为亲水性。这与上述的跨膜结构域的预测分析结果一致。
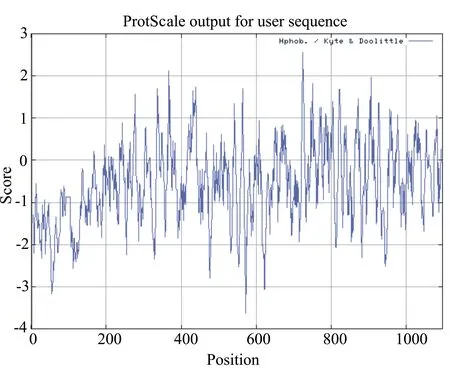
图4 烟草AGO蛋白亲水性/疏水性的预测结果(以AGO1为例)
2.7 烟草AGO蛋白二级/三级结构的预测分析
蛋白质的生物学功能是由其高级结构所决定的,因此,对烟草AGO蛋白高级结构的预测和分析对理解其结构和功能之间的相关性具有重要意义。用SOPMA在线工具对烟草AGO蛋白的二级结构进行预测,结果如表5和图5所示。结果表明:α-螺旋是烟草AGO蛋白二级结构中最主要的结构元件,不同AGO蛋白的α-螺旋比例有一定的差异,其中AGO2的α-螺旋比例明显少于其他AGO蛋白;β-转角在烟草AGO蛋白二级结构中所占的比例较少,且各个AGO蛋白的β-转角比例趋于一致;烟草AGO蛋白二级结构中的各个原件都均匀地分布在整个蛋白上,没有明显的密集区域。
用ExPASy的SWISS-MODEL对烟草AGO蛋白的氨基酸序列进行蛋白质三维结构同源性建模[21],预测结果如图6所示。从图6中可以看出,烟草不同AGO蛋白的三维结构不尽相同。其中AGO1/2/4/5/7/16的功能结构域在空间上折叠成锤子状,锤身主要由α-螺旋和延伸链构成,包含了PIWI特征结构区;柄部主要由不规则卷曲组成,包含了PAZ特征结构区。AGO5/7/16的柄部还有少量的α-螺旋。AGO10和AGO PNH1的空间结构为哑铃状,主要由α-螺旋和延伸链构成的一端包含了PIWI特征结构区,主要由不规则卷曲组成的一端包含了PIWI特征结构区。
3 讨论
AGO蛋白在植物界中普遍存在,在RNAi及相关途径中起着非常重要的作用。Vaucheret[3]对拟南芥AGO蛋白进行系统进化分析时发现,AGO1、AGO5和AGO10有较近的亲缘关系,AGO4、AGO6和AGO9有较近的亲缘关系,AGO7、AGO2和AGO3有较近的亲缘关系。其中具有切割活性的AGO蛋白包括介导mRNA裂解的AGO1、指导DNA甲基化的AGO4、调控植株发育的AGO7,其余成员还未发现切割活性[5,22-24]。此外,在近几年对其他几种AGO蛋白的功能研究中发现:AGO2具有DNA双链断裂修复的功能[25],且在抗病毒上对AGO1有非冗余的互补[26-28];AGO6在DNA甲基化上与AGO4存在部分冗余,而且对某些与异染色质相关的siRNA的积累有着重要作用[29];AGO7和AOG1在病毒诱导的基因沉默(virus induced gene silencing, VIGS)上既互补又部分冗余,其中AGO1占主导作用而AGO7起辅助作用[30];AGO9的缺失会导致胚珠内产生多个有功能的雌配子体[31];AGO10和AGO1也存在功能上的冗余,不过由于它们在组织表达上的差异,AGO10对AGO1功能的互补并不是很强[32]。

图6 烟草AGO蛋白的三维结构预测结果
尽管对植物AGO蛋白有了一定的认知,但是主要集中在AGO1、AGO4和AGO7,对其余AGO蛋白的研究并不多,有待于更加深入的研究。此外,对植物AGO蛋白的研究主要集中在拟南芥中,对其他植物材料的研究很少,研究成果不具有普适性。目前烟草AGO蛋白的研究还处于起步阶段,对于其在植物中确切的细胞定位及具体的生物学功能还知之甚少。本研究对目前已发现的烟草AGO蛋白进行了整理,并进行了生物信息学的预测和分析,可为后期AGO蛋白氨基酸序列的结构、功能域、蛋白质空间结构及性质的研究提供一定的理论依据。
[1] Bohmert K, Camus I, Bellini C, et al. AGO1 defines a novel locus ofArabidopsiscontrolling leaf development[J]. The EMBO Journal, 1998, 17(1): 170-180.
[2] Tolia N H, Joshua-Tor L. Slicer and the argonautes[J]. Nature Chemical Biology, 2007, 3(1): 36-43.
[3] Vaucheret H. Plant argonautes[J]. Trends in Plant Science, 2008, 13(7): 350-358.
[4] Yigit E, Batista P J, Bei Y, et al. Analysis of theC.elegansArgonaute family reveals that distinct Argonautes act sequentially during RNAi[J]. Cell, 2006, 127(4): 747-757.
[5] Mallory A C, Elmayan T, Vaucheret H. MicroRNA maturation and action: the expanding roles of ARGONAUTEs[J]. Current Opinion in Plant Biology, 2008, 11(5): 560-566.
[6] Kapoor M, Arora R, Lama T, et al. Genome-wide identification, organization and phylogenetic analysis of Dicer-like, Argonaute and RNA-dependent RNA polymerase gene families and their expression analysis during reproductive development and stress in rice[J]. BMC Genomics, 2008, 9(1): 451.
[7] Song J J, Joshua T L. Argonaute and RNA: getting into the groove[J]. Current Opinion in Structural Biology, 2006, 16(1): 5-11.
[8] Wu J, Yang Z, Wang Y, et al. Viral-inducible Argonaute18 confers broad-spectrum virus resistance in rice by sequestering a host microRNA[J]. Elife, 2015(4): e05733.
[9] Song J J, Liu J, Tolia N H, et al. The crystal structure of the Argonaute2 PAZ domain reveals an RNA binding motif in RNAi effector complexes[J]. Nature Structural & Molecular Biology, 2003, 10(12): 1026-1032.
[10] Mirzaei K, Bahramnejad B, Shamsifard M H, et al. In silico identification, phylogenetic and bioinformatic analysis of argonaute genes in plants[J]. International Journal of Genomics, 2014, 2014.
[11] 苏雪婷,李洁,邵宁生. Argonaute蛋白的结构与功能[J].医学分子生物学杂志,2014,11(1):37-42.
[12] Höck J, Meister G. The Argonaute protein family[J]. Genome Biol, 2008, 9(2): 210.
[13] Thomson T, Lin H. The biogenesis and function PIWI proteins and piRNAs: progress and prospect[J]. Annual Review of Cell and Developmental Biology, 2009, 25: 355.
[14] Madrid C, Novais J A. On the role of some ARGONAUTE proteins in meiosis and DNA repair inArabidopsisthaliana[J]. 2014.
[15] Zhai J, Zhao Y, Simon S A, et al. Plant microRNAs display differential 3′ truncation and tailing modifications that are ARGONAUTE1 dependent and conserved across species[J]. The Plant Cell, 2013, 25(7): 2417-2428.
[16] Vaucheret H, Mallory A C, Bartel D P. AGO1 homeostasis entails coexpression of MIR168 and AGO1 and preferential stabilization of miR168 by AGO1[J]. Molecular Cell, 2006, 22(1): 129-136.
[17] Mount D W, Mount D W. Bioinformatics: sequence and genome analysis[M]. New York: Cold Spring Harbor Laboratory Press, 2001.
[18] Gasteiger E, Hoogland C, Gattiker A, et al. Protein identification and analysis tools on the ExPASy server[M]. Springer, 2005.
[19] 李超,杜志游,陈集双.解读AGO蛋白结构及其功能[J].中国生物化学与分子生物学报,2009,25(11):969-976.
[20] 生物化学与生物物理进展[J].生物化学与生物物理进展,2016(2).
[21] Schwede T, Kopp J, Guex N, et al. SWISS-MODEL: an automated protein homology-modeling server[J]. Nucleic Acids Research, 2003, 31(13): 3381-3385.
[22] Mallory A, Vaucheret H. Form, function, and regulation of ARGONAUTE proteins[J]. The Plant Cell, 2010, 22(12): 3879-3889.
[23] Ye R, Wang W, Iki T, et al. Cytoplasmic assembly and selective nuclear import ofArabidopsisArgonaute4/siRNA complexes[J]. Molecular Cell, 2012, 46(6): 859-870.
[24] Baumberger N, Baulcombe D.ArabidopsisARGONAUTE1 is an RNA slicer that selectively recruits microRNAs and short interfering RNAs[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(33): 11928-11933.
[25] Wei W, Ba Z, Gao M, et al. A role for small RNAs in DNA double-strand break repair[J]. Cell, 2012, 149(1): 101-112.
[26] Wang X B, Jovel J, Udomporn P, et al. The 21-nucleotide, but not 22-nucleotide, viral secondary small interfering RNAs direct potent antiviral defense by two cooperative argonautes inArabidopsisthaliana[J]. The Plant Cell, 2011, 23(4): 1625-1638.
[27] Harvey J J, Lewsey M G, Patel K, et al. An antiviral defense role of AGO2 in plants[J]. PloS One, 2011, 6(1): e14639.
[28] Jaubert M, Bhattacharjee S, Mello A F, et al. ARGONAUTE2 mediates RNA-silencing antiviral defenses against potato virus X inArabidopsis[J]. Plant Physiology, 2011, 156(3): 1556-1564.
[29] Zheng X, Zhu J, Kapoor A, et al. Role ofArabidopsisAGO6 in siRNA accumulation, DNA methylation and transcriptional gene silencing[J]. The EMBO Journal, 2007, 26(6): 1691-1701.
[30] Qu F, Ye X, Morris T J.ArabidopsisDRB4, AGO1, AGO7, and RDR6 participate in a DCL4-initiated antiviral RNA silencing pathway negatively regulated by DCL1[J]. Proceedings of the National Academy of Sciences, 2008, 105(38): 14732-14737.
[31] Durán-Figueroa N, Vielle-Calzada J P. ARGONAUTE9-dependent silencing of transposable elements in pericentromeric regions ofArabidopsis[J]. Plant Signaling & Behavior, 2010, 5(11): 1476-1479.
[32] Zhu H, Hu F, Wang R, et al.ArabidopsisArgonaute10 specifically sequesters miR166/165 to regulate shoot apical meristem development[J]. Cell, 2011, 145(2): 242-256.
(责任编辑:黄荣华)
Bioinformatics Analysis of Argonaute Proteins in Genome of Tobacco
CAI Jian-yu1, WU Xiao-yun1, GUO Yu-shuang2*, LI Xiang-yu3
(1. School of Agriculture and Food Science, Zhejiang Agricultural and Forestry University, Lin’an 311300, China; 2. Key Laboratory of Molecular Genetics of Tobacco Industry, Guizhou Academy of Tobacco Science, Guiyang 550081, China; 3. Crop Research Institute, Heilongjiang Academy of Agricultural Science, Harbin 150086, China)
This study summarized the information of tobacco argonaute (AGO) proteins which were registered in NCBI, and predicted and analyzed their compositions, phylogenetic tree, signal peptides, transmembrane domain, subcellular localization, hydrophobicity, hydrophilicity, and secondary / tertiary protein structure. The results showed that 22 AGO proteins existed in the genome of tobacco, and they could be divided into 8 categories. Like the AGO proteins inArabidopsisthaliana, the AGO proteins in tobacco had 3 branches in phylogenetic tree; these AGO proteins were hydrophilic proteins without both transmembrane domain and signal peptides, and they possessed two characteristic domains (PAZ and PIWI) with α-helix spreading in the secondary structure of the protein. In addition, the AGO2/4/7/16/PNH1 of tobacco was positioned on nucleus, AGO10 was positioned on cytoplasm, and AGO5 was positioned on mitochondrion.
Bioinformatics; Tobacco; Argonaute protein; Molecular structure; Functional prediction
2016-09-13
国家自然科学基金项目“烟草抗马铃薯Y病毒相关miRNA的筛选与功能分析”(31360431)。
蔡健宇(1992─),男,硕士研究生,研究方向为植物分子生物学。*通讯作者:郭玉双。
S572
A
1001-8581(2017)02-0001-08

