肺腺癌和肺鳞癌中mRNA可变剪接特征的比较
陈梦竹,王秀杰,夏立新
1) 深圳大学医学部,呼吸疾病国家重点实验室深圳大学变态反应分室,广东深圳518060;2) 中国科学院遗传与发育生物学研究所,北京100101
【生物工程/Bioengineering】

肺腺癌和肺鳞癌中mRNA可变剪接特征的比较
陈梦竹1,王秀杰2,夏立新1
1) 深圳大学医学部,呼吸疾病国家重点实验室深圳大学变态反应分室,广东深圳518060;2) 中国科学院遗传与发育生物学研究所,北京100101
利用SUPPA(a super-fast pipeline for alternative splicing)软件分析癌症基因组数据库癌基因组图谱中肺腺癌和肺鳞癌样本的RNA-Seq数据,发现在鉴定的7种主要可变剪接转录本中外显子跳跃转录本发生频率最高.通过癌组织与癌旁组织的比较,系统地鉴定了肺腺癌和肺鳞癌中差异表达的可变剪接转录本,发现约60%的差异可变剪接转录本是两种肺癌亚型所共有的.功能富集分析表明,癌组织特异的可变剪接转录本主要富集在细胞周期调控、脱氧核糖核酸代谢和核糖核酸代谢等生物过程中,而癌旁组织特异的可变剪接转录本则主要富集在囊泡介导转运、多肽转运和脂质转运等生物过程中.通过肺癌驱动基因可变剪接转录本的比较分析,鉴定了两种肺癌亚型共有或特有的可能具有癌驱动功能的可变剪接转录本.
生物信息学;肺腺癌;肺鳞癌;可变剪接;外显子跳跃;可变起始外显子
肺癌不仅是全球死亡率最高的癌症,且发病率在男性和女性中分别仅次于发病率最高的前列腺癌和乳腺癌[1-2].临床诊断的肺癌大多为恶性上皮肿瘤,根据肿瘤细胞的显微形态分为小细胞肺癌(small cell lung cancer, SCLC)和非小细胞肺癌(non-small cell lung cancer, NSCLC).非小细胞肺癌发病率约占肺癌总发病率的85%~90%,包括肺腺癌(lung adenocarcinoma, LUAD)和肺鳞癌(lung squamous carcinoma, LUSC)两种主要亚型[3].深入研究两种肺癌亚型的基因组变异,有助于更好地理解肺腺癌和肺鳞癌在发病机理、临床特征及诊疗方案等方面存在的差异[4-9].
可变剪接是由一种信使核糖核酸(messenger ribonucleic acid, mRNA)前体通过选择性剪接产生多种不同成熟mRNA的过程,约95%的人多外显子基因发生可变剪接[10].根据可变剪接发生的位置,mRNA的可变剪接类型转录本主要分为可变5′剪接位点(alternative 5′ splice-site, A5)、可变3′剪接位点(alternative 3′ splice-site, A3)、可变起始外显子(alternative first exon, AF)、可变终止外显子(alternative last exon, AL)、内含子滞留(retained intron, RI)、外显子跳跃(skipping exon, SE)和互斥外显子(mutually exclusive exons, MX)[11-12].全基因组分析表明特异发生在肿瘤细胞中的某些可变剪接对癌症的发生具有重要作用[13].例如,癌细胞中原癌基因和抑癌基因异常的剪接转录本可致原癌基因的持续激活和抑癌基因部分功能的丧失,从而诱发癌症.癌症相关基因的可变剪接与肺癌的进展关系密切[14].已有研究结果表明,细胞凋亡调节因子Bcl-x基因前体mRNA通过可变剪接形成Bcl-xL和Bcl-xS两种成熟的mRNA,分别行使抑制凋亡和促进凋亡的功能.肺癌细胞中产生Bcl-xL转录本的比例较高,打破Bcl-xL和Bcl-xS在控制细胞凋亡中的平衡,从而倾向于抑制细胞凋亡,促进肺癌的发生[15].
随着高通量测序技术的广泛应用,全基因组水平对肺癌可变剪接变异特征的研究也逐步深入,但仍缺乏较为系统地对于肺腺癌和肺鳞癌两种主要肺癌亚型的比较分析.本研究利用已有癌症基因组数据库癌基因组图谱(the cancer genome atlas, TCGA),对LUAD和LUSC癌组织与其对应的癌旁组织在mRNA可变剪接方面的变异特征进行了系统的比较分析,发现两者共有和各自特有的可变剪接变异,探讨了可变剪接变异在两种癌症进展中的潜在作用机制的异同,为肺癌的靶向治疗提供新思路.
1 研究方法
1.1 肺腺癌和肺鳞癌数据下载
从癌症基因组数据库(the cancer genome atlas,TCGA.https://tcga-data.nci.nih.gov/tcga/)下载571例LUAD和547例LUSC患者癌组织及相应癌旁组织的转录本表达数据、基因组注释和临床信息,用于后续的分析.
1.2 可变剪接转录本的产生与PSI (percentage of spliced in per event)值计算
利用SUPPA(a super-fast pipeline for alternative splicing analysis)软件[16],并结合TCGA提供的基因组注释信息(https://tcga-data.nci.nih.gov/tcgafiles/ftp_auth/distro_ftpusers/anonymous/other/GAF/GAF3.0/transcript.genome.v3_0.gaf.gz),获得LUAD和LUSC中存在的A5、 A3、 AF、 AL、 RI、 SE和MX可变剪接转录本, 并计算可变剪接转录本的PSI值.
1.3 差异可变剪接转录本的鉴定
根据PSI值,如果某个可变剪接转录本在癌组织中的PSI值(PSI癌组织)与正常组织中的PSI值(PSI癌旁组织)的差值满足PSI癌组织-PSI癌旁组织> 0.1 或PSI癌组织-PSI癌旁组织< -0.1,该可变剪接转录本即为差异的可变剪接转录本.挑选在研究群体中出现频率大于10%的可变剪接转录本做后续分析.若具有该差异可变剪接转录本的患者中PSI癌组织- PSI癌旁组织> 0.1的患者数T, 是PSI癌组织-PSI癌旁组织<-0.1的患者数N的2.0倍以上(T/N > 2.0), 该差异的可变剪接转录本倾向于在癌组织中出现,则定义该差异的可变剪接转录本为癌特异的差异可变剪接转录本;若T/N< 0.5, 该差异的可变剪接转录本倾向于在癌旁组织中出现,则定义该差异的可变剪接转录本为癌旁特异的差异可变剪接转录本.
1.4 差异可变剪接转录本的比较
比较LUAD和LUSC中差异可变剪接转录本,获得两者共有和特有的差异可变剪接转录本,利用DAVID[17]和RVIGO(reduce visualize gene ontology)[17-18]软件对其相应基因进行功能富集分析.利用Pfam数据库[19]评估差异的可变剪接转录本对蛋白结构域的影响,筛选与LUAD和(或)LUSC进展相关的可变剪接转录本.
1.5 肺癌驱动基因的筛选
在癌驱动基因数据库(a database for human cancer driver gene research, DriverDB;http://driverdb.tms.cmu.edu.tw/driverdbv2/index.php)网站上,选择在15个已知的癌驱动基因鉴定工具中(ActiveDriver、Dendrix、MDPFinder、Simon、Netbox、OncodriveFM、MutSigCV、MEMo、CoMDP、DawnRank、DriverNet、e-Driver、iPAC、MSEA和OncodriveCLUST)任意3个(至少3个)都可以筛查到的驱动基因作为最终癌症驱动基因的集合.
2 结果分析
2.1 LUAD和LUSC中差异可变剪接转录本的鉴定
通过TCGA数据库下载LUAD和LUSC中转录本的表达数据.在571例LUAD患者中,有57例患者同时具有癌组织和对应癌旁组织转录本表达数据,在547例LUSC患者中,有50例患者同时具有癌组织和对应癌旁组织转录本表达数据,选取57例LUAD和50例LUSC患者的转录本表达数据进行后续分析.
利用SUPPA软件,基因组注释信息以及转录本表达数据,获得LUAD和LUSC中7种主要可变剪接类型(A5、A3、AF、AL、RI、SE和MX)的所有可变剪接转录本(图1),并计算每个可变剪接转录本在57对LUAD和50对LUSC样本中相应的PSI值,筛选差异的可变剪接转录本.将LUAD或LUSC群体中发生频率在10%以上的差异可变剪接转录本,即在LUAD样本中至少6人(57×10%)或在LUSC样本中至少5人(50×10%)同时具有该差异可变剪接转录本,挑选出来进行后续分析,并鉴定了LUAD和LUSC样本中癌特异和癌旁特异的差异可变剪接转录本,用于后续分析(表1).

图1 七种主要可变剪接示意图Fig.1 The schematic diagram of seven main alternative splicing (AS) types
对每种类型差异可变剪接转录本占总差异可变剪接转录本的百分比进行统计发现,在LUAD和LUSC中,SE类型可变剪接转录本所占比例都最高,分别为43.62%和42.66%(图2).其中, MRE11A、 IRAK1、 FHL1、 TCF4、 NUMB和TERT基因的可变剪接转录本在肺癌可变剪接转录本的研究中已有报道[20-26],部分证明了该分析结果的准确性.
2.2 LUAD与LUSC样本中差异可变剪接转录本的比较
通过比较LUAD和LUSC中差异可变剪接转录本,获得了两种肺癌亚型共有以及各自特有的差异可变剪接转录本,其中,共有的差异可变剪接转录本约为60%(图3).

表1 LUAD和LUSC差异可变剪接转录本中7种可变剪接类型统计结果

图2 肺腺癌和肺鳞癌中7种类型可变剪接转录本的百分比统计Fig.2 Percentage of each AS type in LUAD or LUSC

图3 肺腺癌和肺鳞癌中共有可变剪接转录本统计Fig.3 The percentage of the common differentially expressed AS transcripts of LUAD and LUSC out of the total differentially expressed AS transcripts
LUAD和LUSC样本中共有和特有的差异可变剪接转录本及其对应基因数目(括号内标注)的统计如图4.其中,红色数字表示肺腺癌和肺鳞癌癌组织共有的特异剪接转录本(基因);蓝色数字表示癌旁组织共有的特异剪接转录本(基因).将癌组织和癌旁组织特异的可变剪接转录本所对应的基因进行后续的功能分析.
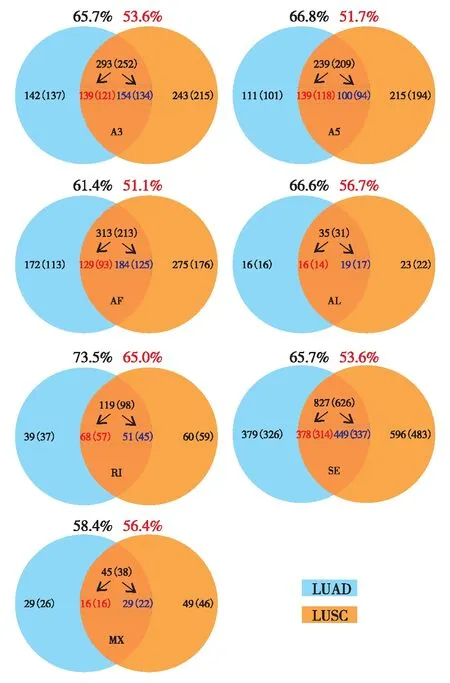
图4 LUAD和LUSC样本中共有和特有的差异可变剪接转录本(基因)的统计Fig.4 The number of total and common differentially expressed AS transcripts (genes) of each AS type in LUAD and LUSC
2.3 LUAD和LUSC样本中共有差异可变剪接转录本对应基因的功能分析
功能富集分析的结果表明,两种肺癌亚型共有的癌组织特异的差异可变剪基因主要富集在细胞周期调控、脱氧核糖核酸(deoxyribonucleic acid,DNA)代谢和RNA代谢等生物过程中见图5.其中,点的大小代表该GO term在GO注释(gene ontology annotation, GOA)中的代表频率;点的颜色代表每个GO term的lgP.

图5 LUAD和LUSC样本中共有的癌组织特异的差异可变剪接转录本对应基因的功能富集分析(P< 0.05, Fisher精确检验)Fig.5 GO analyses of the common cancer-specific AS transcripts of LUAD and LUSC (P< 0.05, Fisher’s exact test)
而两种肺癌亚型共有的癌旁组织特异的差异可变剪接转录本对应基因主要富集在囊泡介导转运、多肽转运、脂质转运和细胞凋亡等生物过程(图6),癌组织与癌组织特异的差异可变剪接基因的功能富集结果存在明显差异.

图6 LUAD和LUSC样本中共有的癌旁组织特异的差异可变剪接转录本对应基因的功能富集分析(P< 0.05, Fisher精确检验)Fig.6 GO analyses of the common normal-specific AS transcripts (P< 0.05, Fisher’s exact test)
2.4 癌驱动基因的可变剪接分析
癌驱动基因是致癌的关键基因.在LUAD和LUSC中分别筛选出359和254个癌驱动基因.结果表明,LUAD和LUSC中共有的差异可变剪接转录本所对应的基因为1 097个,分别包含了21个LUAD和14个LUSC的驱动基因(表2和表3).研究结果表明, MET基因产生的可变剪接转录本在第10个外显子上存在跳跃后终止的现象,且可能影响其编码蛋白的激酶结构域,如图7(a).LUAD中VCAN基因产生的外显子跳跃转录本可能破坏其编码蛋白的EGF蛋白结构域,如图7(b),LUSC中外显子跳跃转录本可能使ITGA8缺失一个Integrin_alpha2结构域,如图7(c).
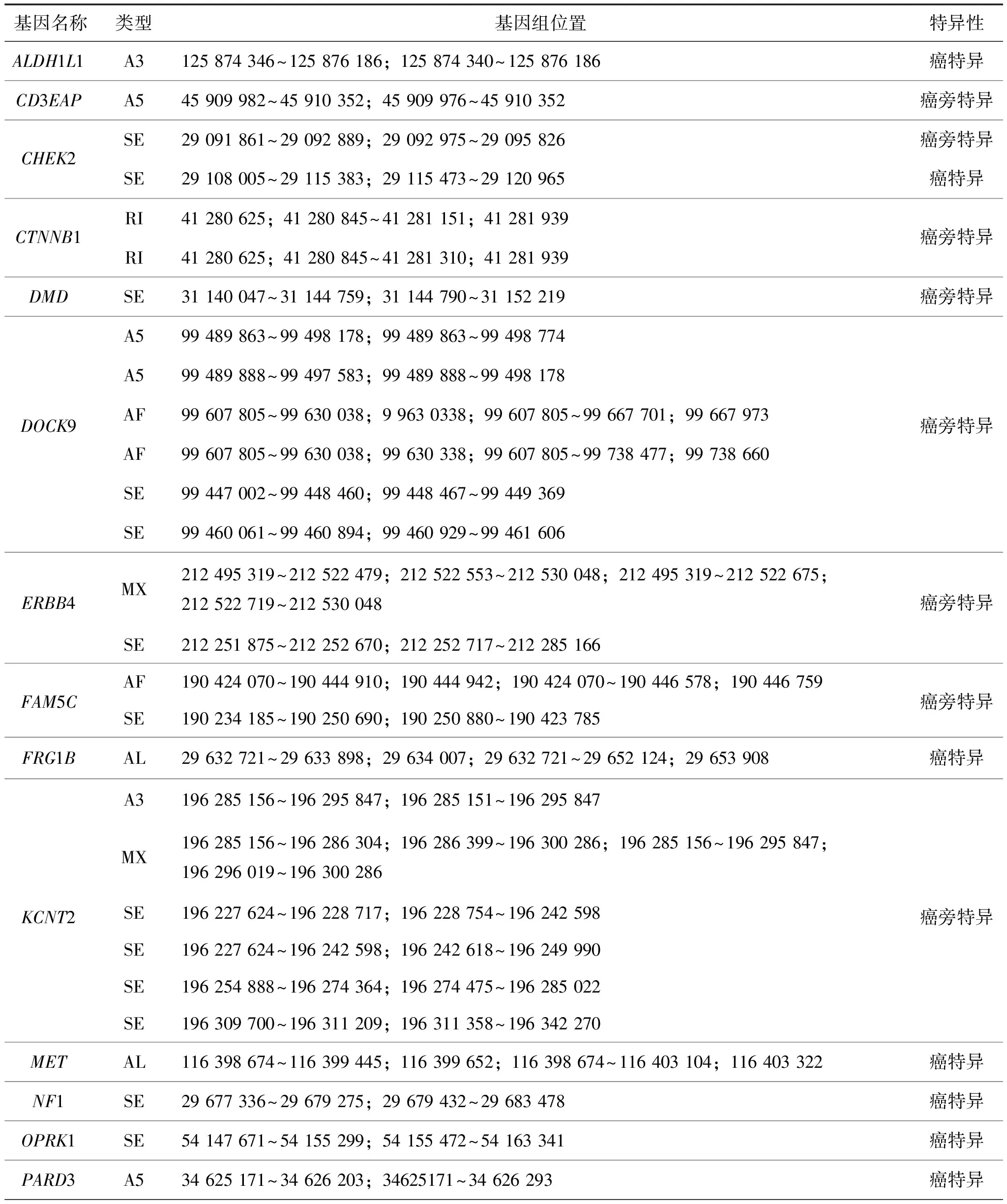
表2 LUAD癌驱动基因中共有的差异可变剪接转录本

(续表2)

表3 LUSC癌驱动基因中共有的差异可变剪接转录本

(续表3)

图7 LUAD和LUSC样本中癌驱动基因的共有差异可变剪接转录本举例Fig.7 Examples of the common differentially expressed AS transcripts of cancer driven genes in LUAD and LUSC
结果表明,LUAD和LUSC中亚型特异的差异可变剪接转录本对应的基因数目分别为632和941,其中,包括16个LUAD和17个LUSC的驱动基因(表4和表5).本研究发现ALDH1L1基因的LUAD亚型特异转录本包括两个外显子跳跃,如图8(a). PLCB1基因存在可变起始外显子产生的可变剪接转录本,且该转录本特异地在LUSC中表达,如图8(b).

表4 驱动基因中LUAD亚型特异的差异可变剪接转录本
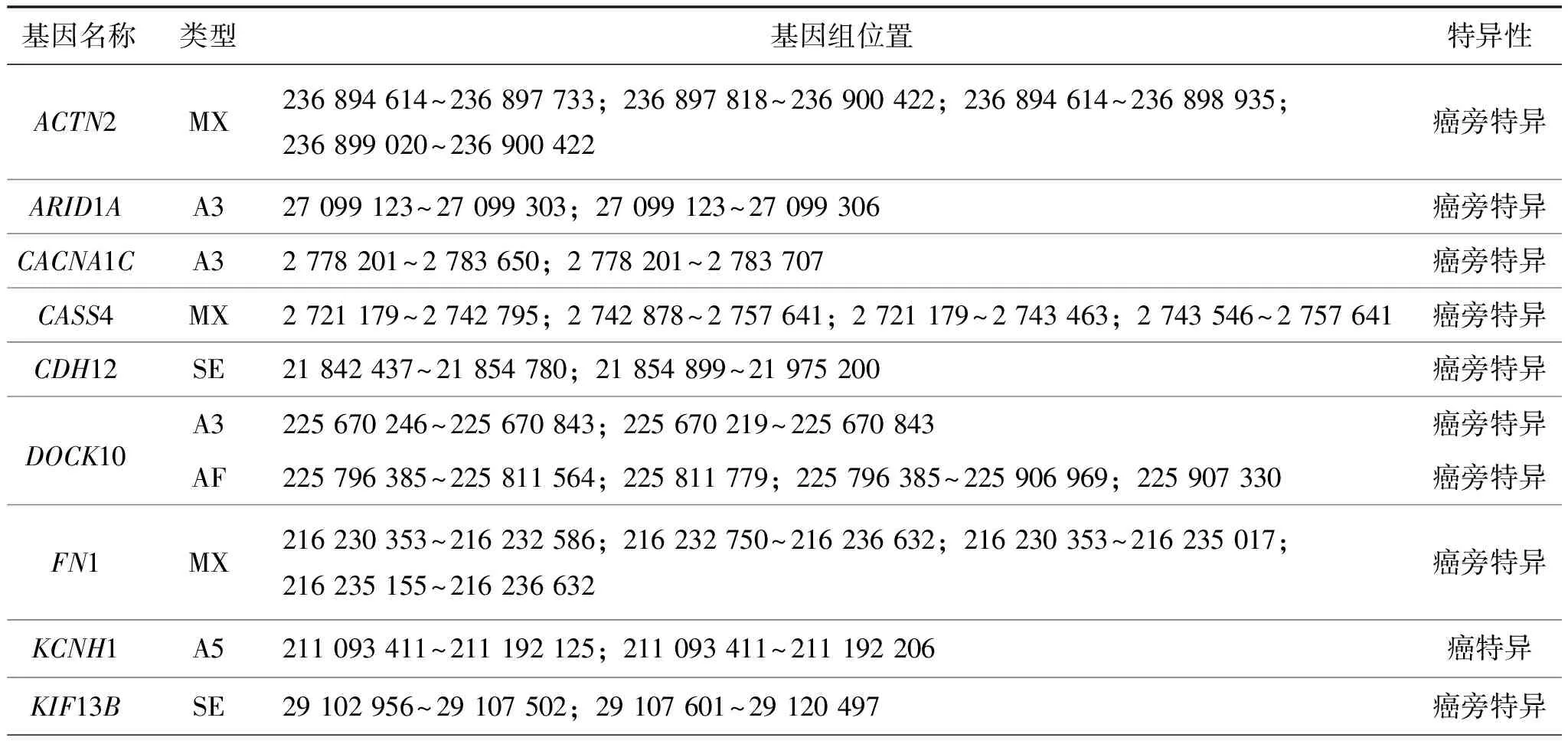
表5 驱动基因中LUSC亚型特异的差异可变剪接转录本Table 5 Subtype-specific differentially expressed AS transcripts of cancer driven genes in LUSC

(续表5)

图8 癌驱动基因中LUAD或LUSC亚型特异的差异可变剪接举例Fig.8 Examples of the subtype-specific differentially expressed AS transcripts of cancer driven genes in LUAD or LUSC
3 讨 论
TCGA癌症基因组数据库为研究者提供了丰富的癌症数据资源,以便深入地挖掘数据的生物意义.本研究利用TCGA数据库中肺腺癌和肺鳞癌样本的转录本表达数据,获得肺腺癌和肺鳞癌样本中所有的可变剪接转录本及对应的PSI值,通过比较癌组织和癌旁组织中PSI值,获得肺腺癌和肺鳞癌样本与相应癌旁组织相比存在表达差异的可变剪接转录本,以及肺腺癌和肺鳞癌样本中共有和特有的差异可变剪接转录本及对应基因.并对两种亚型共有的癌特异和癌旁特异的差异可变剪接转录本对应的基因进行了功能富集.系统地分析和比较两种肺癌亚型驱动基因的可变剪接差异,为理解肺腺癌和肺鳞癌发生机制的异同提供新线索.
两种肺癌亚型共有的差异可变剪接基因功能富集的结果发现癌特异的差异可变剪接基因富集在细胞周期调控、DNA代谢和RNA代谢等生物过程中,而癌旁特异的差异可变剪接基因富集在囊泡介导转运、多肽转运、脂质转运和细胞凋亡等生物过程中,表明癌特异和癌旁特异的差异可变剪接基因的功能富集存在明显的差异.该结果一方面体现了这些基因在癌症发生和发展过程中功能的不同,另一方面由于数据类型和SUPPA软件对可变剪接鉴定方法的局限,不能排除转录本表达水平对癌特异和癌旁特异的差异可变剪接转录本鉴定结果的影响.
文献[6]报道,肺腺癌样本中MET基因产生第14个外显子跳跃的可变剪接转录本,且与基因的功能和癌症的进展相关.通过鉴定,本研究发现在肺腺癌样本中MET基因产生第10个外显子跳跃导致翻译提前终止的转录本,并且通过结构域分析发现这一可变剪接可能会影响其编码蛋白的激酶结构域.在分析的57例肺腺癌患者和50例肺鳞癌患者中,分别有38.6%和16.0%的患者具有该可变剪接转录本.MET基因中该可变剪接转录本的功能则需进一步研究.
癌驱动基因是癌症发生和发展的关键调控基因,本研究重点分析和比较了驱动基因在肺腺癌和肺鳞癌中可变剪接转录本的异同.ALDH1L1基因产生存在两个外显子跳跃的可变剪接转录本,并且该转录本仅特异地在肺腺癌中表达.PLCB1在肺鳞癌中存在一个由可变外显子起始产生的转录本,结构域分析表明该肺鳞癌亚型特异的转录本可能影响其编码蛋白的结构域.该研究为肺腺癌和肺鳞癌的治疗提供新的线索以上分析的结果是基于有限数据量进行的比较,随着未来数据量的不断增长,肺腺癌和肺鳞癌样本中可变剪接差异的比较会更加细致准确.
结 语
肺癌是高发病率和高死亡率的癌症.通过分析TCGA数据库中两种主要的非小细胞肺癌亚型(肺腺癌和肺鳞癌)样本中癌组织和相应癌旁组织转录本的表达数据,获得了肺腺癌和肺鳞癌样本在癌组织与其对应癌旁组织中存在表达差异的7种主要类型的可变剪接转录本,并对其中癌驱动基因的差异可变剪接模式进行了细致的比较分析.该研究将有助于深入理解两种肺癌亚型的发病机理,为肺腺癌和肺鳞癌的靶向治疗提供新的参考.
/ References:
[1] Siegel R L, Miller K D, Jemal A. Cancer statistics, 2015[J]. CA: A Cancer Journal for Clinicians, 2015, 65(1): 5-29.
[2] Chen Wanqing, Zheng Rongshou, Baade P D, et al. Cancer statistics in China, 2015[J]. CA: A Cancer Journal for Clinicians, 2016, 66(2): 115-132.
[3] Chang J T H, Lee Y M, Huang R S. The impact of the Cancer Genome Atlas on lung cancer[J]. Translational Research, 2015, 166(6): 568-585.
[4] Subramanian J, Govindan R. Lung cancer in never smokers: a review[J]. Journal of Clinical Oncology, 2007, 25(5): 561-570.
[5] Hammerman P S, Lawrence M S, Voet D, et al. Comprehensive genomic characterization of squamous cell lung cancers[J]. Nature, 2012, 489(7417): 519-525.
[6] Collisson E A, Campbell J D, Brooks A N, et al. Comprehensive molecular profiling of lung adenocarcinoma[J]. Nature, 2014, 511(7511): 543-550.
[7] Paez J G, Jänne P A, Lee J C, et al. EGFR mutations in lung cancer: correlation with clinical response to gefitinib therapy[J]. Science, 2004, 304(5676): 1497-1500.
[8] Antonicelli A, Cafarotti S, Indini A, et al. EGFR-targeted therapy for non-small cell lung cancer: focus on EGFR oncogenic mutation[J]. International Journal of Medical Sciences, 2013, 10(3): 320-330.
[9] Rekhtman N, Paik P K, Arcila M E, et al. Clarifying the spectrum of driver oncogene mutations in biomarker-verified squamous carcinoma of lung: lack of EGFR/KRAS and presence of PIK3CA/AKT1 mutations[J]. Clinical Cancer Research, 2012, 18(4): 1167-1176.
[10] Pan Qun, Shai O, Lee L J, et al. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing[J]. Nature Genetics, 2008, 40(12): 1413-1415.
[11] Chen Mo, Manley J L. Mechanisms of alternative splicing regulation: insights from molecular and genomics approaches[J]. Nature Reviews Molecular Cell Biology, 2009, 10(11): 741-754.
[12] Wahl M C, Will C L, Lührmann R. The spliceosome: design principles of a dynamic RNP machine[J]. Cell, 2009, 136(4): 701-718.
[13] Venables J P, Klinck R, Koh C, et al. Cancer-associated regulation of alternative splicing[J]. Nature Structural & Molecular Biology, 2009, 16(6): 670-676.
[14] Chen J, Weiss W A. Alternative splicing in cancer: implications for biology and therapy[J]. Oncogene, 2015, 34(1): 1-14.
[15] Takehara T, Liu Xiaolong, Fujimoto J, et al. Expression and role of Bcl-xL in human hepatocellular carcinomas[J]. Hepatology, 2001, 34(1): 55-61.
[16] Alamancos GP, Pagès A, Trincado JL, et al. Leveraging transcript quantification for fast computation of alternative splicing profiles[J]. RNA, 2015, 21(9): 1521-1531.
[17] Huang Dawei, Sherman B T, Lempicki R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nature Protocols, 2009, 4(1): 44-57.
[18] Supek F, Bošnjak M,kunca N, et al. REVIGO summarizes and visualizes long lists of gene ontology terms[J]. PLoS One, 2011, 6(7): e21800.
[19] Finn R D, Coggill P, Eberhardt R Y, et al. The Pfam protein families database: towards a more sustainable future[J]. Nucleic Acids Research, 2016, 44(D1): D279-D285.
[20] Wamg Z N, Lo H S, Yang H, et al. Computational analysis and experimental validation of tumor-associated alternative RNA splicing in human cancer[J]. Cancer Research, 2003, 63(3): 655-657.
[21] Pio R, Blanco D, Pajares M J, et al. Development of a novel splice array platform and its application in the identification of alternative splice variants in lung cancer[J]. BMC Genomics, 2010, 11: 352.
[22] Planque C, Choi Y H, Guyetant S, et al. Alternative splicing variant of kallikrein-related peptidase 8 as an independent predictor of unfavorable prognosis in lung cancer[J]. Clinical Chemistry, 2010, 56(6): 987-997.
[23] Fan Y C, Min L, Chen H, et al. Alternative splicing isoform of T cell factor 4 K suppresses the proliferation and metastasis of non-small cell lung cancer cells[J]. Genetics and Molecular Research : GMR, 2015, 14(4): 14009-14018.
[24] Misquitta-Ali CM, Cheng E, O’Hanlon D, et al. Global profiling and molecular characterization of alternative splicing events misregulated in lung cancer[J]. Molecular and Cellular Biology, 2011, 31(1): 138-150.
[25] Pelosi G, Schianchi E, Dell’orto P, et al. Detecting cell-free circulating hTERT mRNA in the plasma may identify a subset of nonsmall cell lung cancer patients[J]. Laboratory Investigation, 2006, 448(1): 7-15.
[26] Fujiwara M, Kamma H, Wu W, et al. Expression and alternative splicing pattern of human telomerase reverse transcriptase in human lung cancer cells[J]. International Journal of Oncology, 2004, 24(4): 925-930.
【中文责编:晨 兮;英文责编:艾 琳】
Comparison of alternative splicing in lung adenocarcinoma and lung squamous carcinoma
Chen Mengzhu1, Wang Xiujie2†, and Xia Lixin1†
1) State Key Laboratory of Respiratory Disease for Allergy at Shenzhen University, Health Science Center,Shenzhen University, Shenzhen 518060, Guangdong Province, P.R.China 2) Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101, P.R.China
Lung cancer is a common disease with high morbidity and mortality. Studies have shown that alternative splicing (AS) is closely associated with lung carcinogenesis, but the difference of alternative splicing between lung adenocarcinoma (LUAD) and lung squamous carcinoma (LUSC) remains unclear. Here, we analyze the RNA-Seq data of LUAD and LUSC from TCGA using SUPPA software, and find that skipping exon (SE) is the most frequently occurred AS transcript among seven main AS transcripts identified. By comparing tumor tissues with normal ones, we systematically identify differentially expressed AS transcripts in both LUAD and LUSC, of which around 60% are identical between these two subtypes. Functional analysis reveals that tumor-specific AS transcripts are mainly enriched among genes associated with cell cycle regulation, deoxyribonucleic acid metabolism and RNA metabolism, whereas normal-specific AS transcripts are enriched among genes mainly involved in vesicle-mediated transport, peptide transport and lipid transport. By comparing AS transcripts of cancer-driven genes between LUAD and LUSC, we identify some common and subtype-specific AS transcripts with cancer-driven potentiality.
bioinformatics; lung adenocarcinoma (LUAD); lung squamous carcinoma (LUSC); alternative splicing (AS); skipping exon (SE); alternative first exon (AF)
Received:2016-06-18;Revised:2016-11-14;Accepted:2016-11-20
Foundation:National High-Tech Research and Development Program (2012AA020409); Shenzhen Science and Technology Basic Research Foundation(JCYJ20150525092941055)
† Corresponding author:Professor Wang Xiujie. E-mail: xjwang@genetics.ac.cn; Professor Xia Lixin. E-mail:xialixin@126.com
:Chen Mengzhu, Wang Xiujie, Xia Lixin. Comparison of alternative splicing in lung adenocarcinoma and lung squamous carcinoma[J]. Journal of Shenzhen University Science and Engineering, 2017, 34(1): 33-45.(in Chinese)
R 734.2;Q 811.4
A
10.3724/SP.J.1249.2017.01033
国家高技术研究发展计划资助项目(2012AA020409);深圳市知识创新计划基础研究资助项目(JCYJ20150525092941055)
陈梦竹(1982—),女,深圳大学博士后研究人员.研究方向:生物信息学.E-mail:mzchen@szu.edu.cn
引 文:陈梦竹,王秀杰,夏立新.肺腺癌和肺鳞癌中mRNA可变剪接特征的比较[J]. 深圳大学学报理工版,2017,34(1):33-45.

