LPS刺激诱导大鼠原代枯否细胞基因表达谱变化分析①
谈志丽 朱 彤 涂文娟 刘亮明
(南京医科大学附属上海松江中心医院感染科,上海交通大学附属第一人民医院松江分院感染科,上海201600)
LPS刺激诱导大鼠原代枯否细胞基因表达谱变化分析①
谈志丽 朱 彤 涂文娟 刘亮明
(南京医科大学附属上海松江中心医院感染科,上海交通大学附属第一人民医院松江分院感染科,上海201600)
目的:研究脂多糖(Lipopolysaccharide,LPS)刺激原代大鼠枯否细胞(Kupffer cell,KC)基因表达谱的变化情况。方法:胶原酶灌注消化和不连续密度梯度离心分离培养大鼠原代肝脏KC,采用LPS刺激细胞。OneArray基因表达谱芯片检测LPS刺激后,细胞内基因表达谱的变化。采用实时荧光定量PCR法对表达上调最显著的基因进行验证。结果:基因芯片结果显示LPS刺激后,原代KC内基因表达谱发生了明显变化。与正常对照组相比,LPS刺激组基因表达上调27个(包括Ces1f、Slc17a3、Slc21a4、Hsd17b2、Sorbs2、Ccdc116、Mgam、Myo5b、Etl4、Fabp1、Kif4b、Fosl1、Cyp4a1、Penk、Tmem221、Rpl5、Nr2f1、Hoxb1、Gpr165、Fam90a13p、Kpna6、Irak1bp1、Kcnh1和4个尚未命名基因),表达下调4个(包括Oc90、Tagln、Arxes2和Olr830)。其中Ces1f为上调最显著的基因。实时荧光定量PCR验证结果显示,LPS刺激诱导KC对Ces1f基因表达水平上调达23.88倍。结论:LPS刺激可诱导大鼠原代KC基因表达谱发生变化。其中,Ces1f基因的上调表达最为显著。
原代枯否细胞;脂多糖;基因表达谱;芯片分析;大鼠
枯否细胞(Kupffer cell, KC)是位于肝内的组织巨噬细胞,具有变形和吞噬的功能,是肝脏固有免疫系统的重要成分[1-3]。脂多糖(Lipopolysaccharide,LPS)是革兰氏阴性细菌细胞壁中的重要化学组分,又称为细菌内毒素,由O-侧链、核心寡糖和脂质A组成,能够通过CD14分子和Toll样受体(Toll-like receptor,TLR)激活机体固有免疫系统并引发强效的促炎症反应[4]。在LPS的刺激下,KC激活并表达CD14分子和TLR4,刺激信号进一步通过激活接头蛋白MyD88启动下游p38 丝裂原活化蛋白激酶(p38 mitogen-activated protein kinase,p38 MAPK)和核因子-кB(Nuclear factor-κB,NF-κB),从而启动炎性信号通路,促进前炎细胞因子的释放[5]。但是LPS刺激诱导KC活化的机制和活化后基因的表达情况尚不清楚。为此,在本实验中,我们采用LPS刺激大鼠原代KC,利用OneArray芯片分析了其基因表达谱的变化,并对上调最明显的基因,采用实时荧光定量PCR加以验证,以进一步了解LPS刺激对KC基因表达的影响。
1 材料与方法
1.1 材料
1.1.1 实验动物 健康雄性SD大鼠,清洁级,体重220~250 g,由上海交通大学附属第一人民医院实验动物中心提供[SYXK(沪)2009-0086]。实验前禁食12 h,实验动物的使用符合国家动物保护法。
1.1.2 主要试剂和耗材 Ⅳ型胶原酶、1640培养液和胎牛血清(美国Gibco公司);Percoll细胞分离液(瑞典GE Healthcare公司);青链霉素混合液(德国Hyclone公司);脂多糖(美国Sigma公司);Trizol试剂(美国Invitrogen公司);逆转录试剂盒和实时荧光定量PCR相关试剂(美国Thermo公司);RNA扩增试剂盒、OneArray芯片、前杂交液、荧光标记杂交试剂盒、清洗液(华联生物科技有限公司)。
1.2 方法
1.2.1 大鼠原代肝脏KC的分离培养 大鼠原代KC的分离采用Ⅳ型胶原酶灌注及Percoll密度梯度离心法,具体步骤参照文献[6]。细胞获得后加入1640培养液(10% 胎牛血清,链霉素1 000 U/L,青霉素1 000 U/L)、置于37℃、5%CO2培养箱中培养3~4 h,用1×PBS洗去未贴壁细胞,即可得到纯化的KC。细胞活力采用台盼蓝染色,细胞鉴定采用墨汁吞噬和CD163染色分析(细胞活力达95%、纯度达90%时用于下一步实验)。
1.2.2 细胞的分组与处理 细胞接种于6孔板,每孔细胞数4×106个。培养24 h待其稳定贴壁后,将细胞随机分成两组,即正常对照组和LPS刺激组。纯1640培养液500 μl分别加入每孔细胞中,LPS刺激组予以终浓度20 μg/ml的LPS刺激[7,8],正常对照组加入同等体积的1×PBS。置于37℃、5%CO2培养箱中培养,6 h后收集细胞用于后续实验。
1.2.3 总RNA的提取 采用Trizol试剂,具体操作按照说明书进行。抽提得到的RNA采用OD260/280吸光度比值及琼脂糖凝胶电泳鉴定其纯度及完整性,并测定所得RNA的浓度。
1.2.4 RNA样品荧光标记反应 RNA质量检测合格后,将样品RNA逆转录为第一链cDNA,接着再加入RNase和DNA Polymerase以合成第二链cDNA;随后,以T7启动子完成反义链RNA合成(antisense RNA, aRNA),过程中加入Amino Allyl UTP [ 5-(3-aminoallyl)-UTP,aa-UTP],形成 Amino Allyl-aRNA (aa-aRNA),再加入NHS-CyeDye。NHS 与 Amino Allyl结合后,使aa-aRNA 变成 CyeDye-aRNA 完成荧光标记。
1.2.5 Phalanx OneArray芯片预杂交 芯片预加热至60℃,置入100 %乙醇中,孵育约15 s,震荡20 s。用去离子水彻底冲洗,去除残留的乙醇。将芯片完全浸入预杂交溶液并静置2 h。纯水清洗后,置于干燥黑暗处,完成预杂交处理。
1.2.6 OneArray芯片杂交 预杂交的芯片与空白芯片对夹,套上热缩膜置于热水中完成热缩后,置于50℃烘箱中。将预热溶解的NewOA Hyb Buffer缓慢加入已完成荧光标记的RNA样品中,配置成aRNA混合液。将混合液加入芯片切口处,套第二层热缩膜并完成第二次热缩,置于50℃杂交烘箱旋转架上(2 r/min、16 h)。于42℃清洗液中小心拆开芯片,多次清洗后甩干,置于黑色芯片盒中,准备扫描。
1.2.7 影像扫描和数据分析 Agilent Microarray Scanner (G2505C)扫描仪进行扫描,藉由扫描仪XDR功能使用PMT 100% 与 10% 以 10 μm的分辨率进行两次扫描。再利用GenePixTM4进行影像数据获取,获得两份初级数据,最后利用华联芯片内置控制探针对两份数据进行整合,整合完成的数据采用Rosetta Resolver®System (Rosetta Biosoftw-are)进行分析处理,按照log2(对照组/刺激组)绝对值≥1且P值<0.05选取差异基因。并在R 3.03版本环境下,进行差异基因分析。
1.2.8 实时荧光定量PCR 引物的设计与合成由上海生工生物技术有限公司完成。目的基因和内参引物序列见表 1。RNA作为模板用于第一链cDNA的合成,具体流程按照逆转录试剂盒说明书进行。实时荧光定量PCR反应体系(50 μl)的建立:Maxima SYBR Green qPCR Master mix(×2) 25 μl、PCR Sense Primer(10 μmol/L)1.0 μl、PCR Antisense Primer(10 μmol/L) 1.0 μl、cDNA模板4.0 μl、ROX solution 0.1 μl,加无核酸酶水至50 μl。两步法进行扩增,第一步:50℃,2 min,1个循环;第二步:95℃,10 min,1个循环;95℃,15 s,40个循环;60℃,60 s,40个循环。内参基因为GAPDH。反应结束后,保存PCR扩增曲线、熔解曲线和相对应的CT值,采用2-ΔΔCT方法对数据进行相对定量分析。
表1 基因扩增引物序列和产物长度
Tab.1 Primer sequences and product length of gene amplification

GenePrimersequences5′→3′Productlength(bp)Ces1fSenseprimerTTTGCACTGCCTACCAGAGTC128AntisenseprimerGGTGGTGAAGAGGGGTTTCCGAPDHSenseprimerGACATGCCGCCTGGAGAAAC244AntisenseprimerGTCCACCACCCTGTTGCTGTAG
1.3 统计学分析 基因芯片分析每组采用5个样本,其余所有实验重复操作6次,实验数据的处理和分析采用SPSS16.0,计算各组2-ΔΔCT均值和标准差,数据分析采用成组t检验,检验水准α=0.05。
2 结果
2.1 大鼠原代肝脏KC显微镜下表现 KC初分离时镜下呈折光性很强的圆球形(图1A);培养3 h后KC呈扁圆形,并已牢固贴壁;24 h后KC充分舒展,大小一致,但形态不规则,可呈对角形、星形等(图1B)。
2.2 RNA质检结果 采用 Agilent 2100 Bioanalyzer分析仪分析,正常对照组与LPS刺激组 RNA OD260/280均大于1.8,RIN 值均大于6.0,凝胶电泳显示 28S 和 18S 两条主带(图 2),符合基因表达谱芯片试验要求。
2.3 基因芯片分析结果 运用OneArray基因芯片对KC基因表达情况进行分析,根据正常对照组与LPS刺激组两组间基因表达水平t检验的结果,按照log2(对照组/刺激组)绝对值≥1且P值<0.05的条件筛选差异表达的基因。结果显示,LPS刺激组与正常对照组细胞相比,存在差异性表达的基因共31个。基因芯片差异性表达火山图见图3。其中,表达上调的基因数共27个,表达下调的基因有4个(见表2)。将以上差异表达基因运用Cluster 3.0进行聚类分析,数据导入Treeview 1.1.6.0得到分层聚类图(图4)。

图1 大鼠原代KC显微镜下表现Fig.1 Performance of rat primary Kupffer cells und-er microscopeNote:A.Performance of primary Kupffer cells at the beginning of separation (×100);B.Performance of primary Kupffer cells at 24 h after culture(×200).
2.4 实时荧光定量PCR熔解和扩增曲线 对表达上调最显著的Ces1f基因采用实时荧光定量PCR加以验证。Ces1f和GAPDH熔解曲线呈单一明显的高峰(图5A、B),说明在荧光定量PCR过程中没有产生引物二聚体和非特异性扩增,引物特异性良好。扩增曲线示Ces1f扩增时Tm值为81.4, GAPDH扩增时Tm值为82.2(图5C、D)。曲线平行性好,间距均一,说明目的基因和内参基因扩增效率基本一致,可以采用2-ΔΔCT方法进行相对定量分析。

图2 总RNA 凝胶电泳图Fig.2 Gel electrophoresis of total RNANote:1.Control;2.LPS.

图3 两组KC基因差异性表达火山图Fig.3 Volcano plot of differential expression profile of KC genes between two groupsNote:A.Control;B.LPS.The horizontal axis represents the fold difference of gene expression between the two groups, the vertical axis represents the P values between the two groups.The left and right upper quadrant were up-regulated and down-regulated genes respectively.
表2 两组细胞间存在差异性表达的基因和其GO生物学功能分类
Tab.2 Differentially expression genes and GO functions
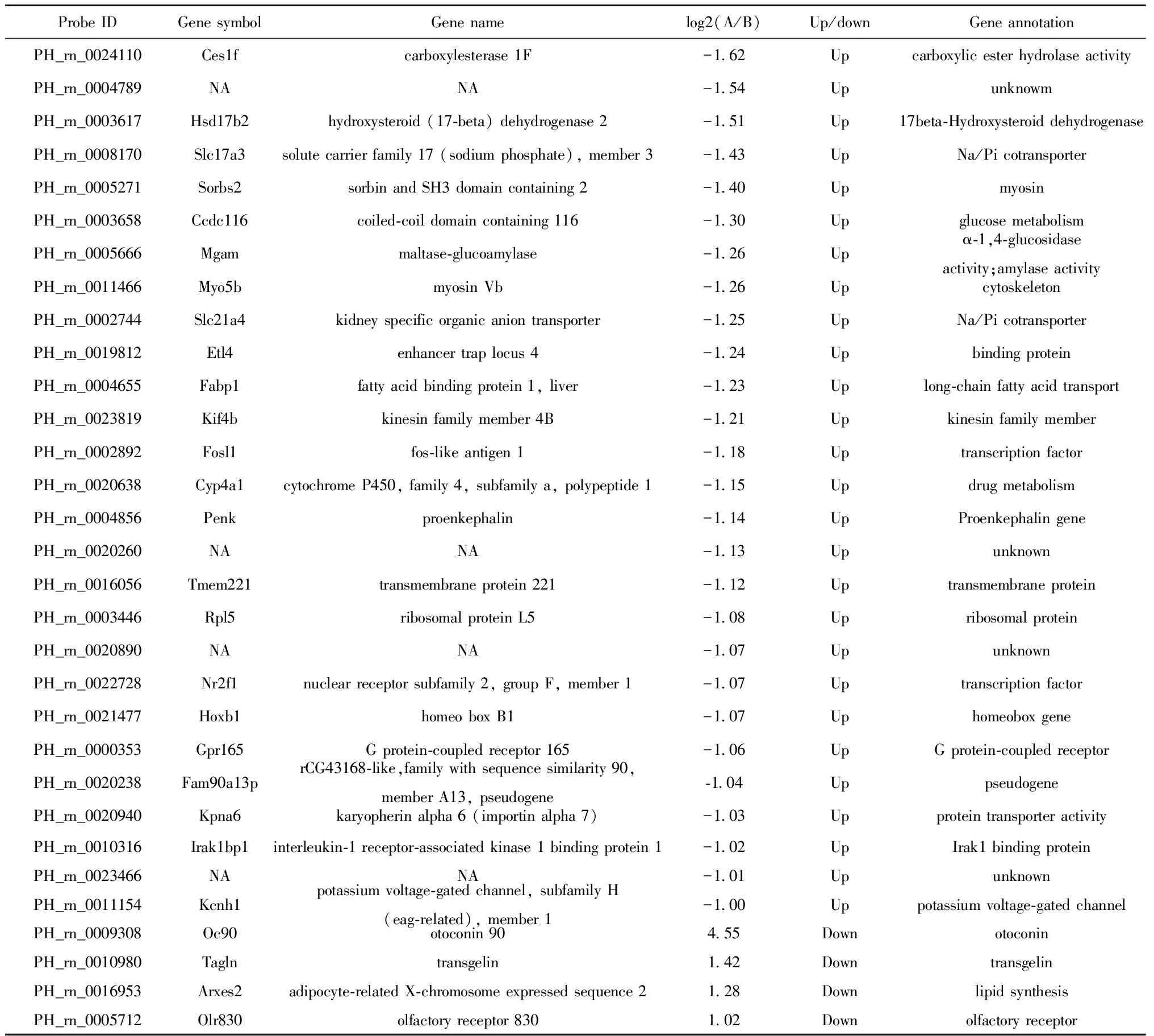
ProbeIDGenesymbolGenenamelog2(A/B)Up/downGeneannotationPH_rn_0024110Ces1fcarboxylesterase1F-162UpcarboxylicesterhydrolaseactivityPH_rn_0004789NANA-154UpunknowmPH_rn_0003617Hsd17b2hydroxysteroid(17⁃beta)dehydrogenase2-151Up17beta⁃HydroxysteroiddehydrogenasePH_rn_0008170Slc17a3solutecarrierfamily17(sodiumphosphate),member3-143UpNa/PicotransporterPH_rn_0005271Sorbs2sorbinandSH3domaincontaining2-140UpmyosinPH_rn_0003658Ccdc116coiled⁃coildomaincontaining116-130UpglucosemetabolismPH_rn_0005666Mgammaltase⁃glucoamylase-126Upα⁃1,4⁃glucosidaseactivity;amylaseactivityPH_rn_0011466Myo5bmyosinVb-126UpcytoskeletonPH_rn_0002744Slc21a4kidneyspecificorganicaniontransporter-125UpNa/PicotransporterPH_rn_0019812Etl4enhancertraplocus4-124UpbindingproteinPH_rn_0004655Fabp1fattyacidbindingprotein1,liver-123Uplong⁃chainfattyacidtransportPH_rn_0023819Kif4bkinesinfamilymember4B-121UpkinesinfamilymemberPH_rn_0002892Fosl1fos⁃likeantigen1-118UptranscriptionfactorPH_rn_0020638Cyp4a1cytochromeP450,family4,subfamilya,polypeptide1-115UpdrugmetabolismPH_rn_0004856Penkproenkephalin-114UpProenkephalingenePH_rn_0020260NANA-113UpunknownPH_rn_0016056Tmem221transmembraneprotein221-112UptransmembraneproteinPH_rn_0003446Rpl5ribosomalproteinL5-108UpribosomalproteinPH_rn_0020890NANA-107UpunknownPH_rn_0022728Nr2f1nuclearreceptorsubfamily2,groupF,member1-107UptranscriptionfactorPH_rn_0021477Hoxb1homeoboxB1-107UphomeoboxgenePH_rn_0000353Gpr165Gprotein⁃coupledreceptor165-106UpGprotein⁃coupledreceptorPH_rn_0020238Fam90a13prCG43168⁃like,familywithsequencesimilarity90,memberA13,pseudogene⁃104UppseudogenePH_rn_0020940Kpna6karyopherinalpha6(importinalpha7)-103UpproteintransporteractivityPH_rn_0010316Irak1bp1interleukin⁃1receptor⁃associatedkinase1bindingprotein1-102UpIrak1bindingproteinPH_rn_0023466NANA-101UpunknownPH_rn_0011154Kcnh1potassiumvoltage⁃gatedchannel,subfamilyH(eag⁃related),member1-100Uppotassiumvoltage⁃gatedchannelPH_rn_0009308Oc90otoconin90455DownotoconinPH_rn_0010980Taglntransgelin142DowntransgelinPH_rn_0016953Arxes2adipocyte⁃relatedX⁃chromosomeexpressedsequence2128DownlipidsynthesisPH_rn_0005712Olr830olfactoryreceptor830102Downolfactoryreceptor
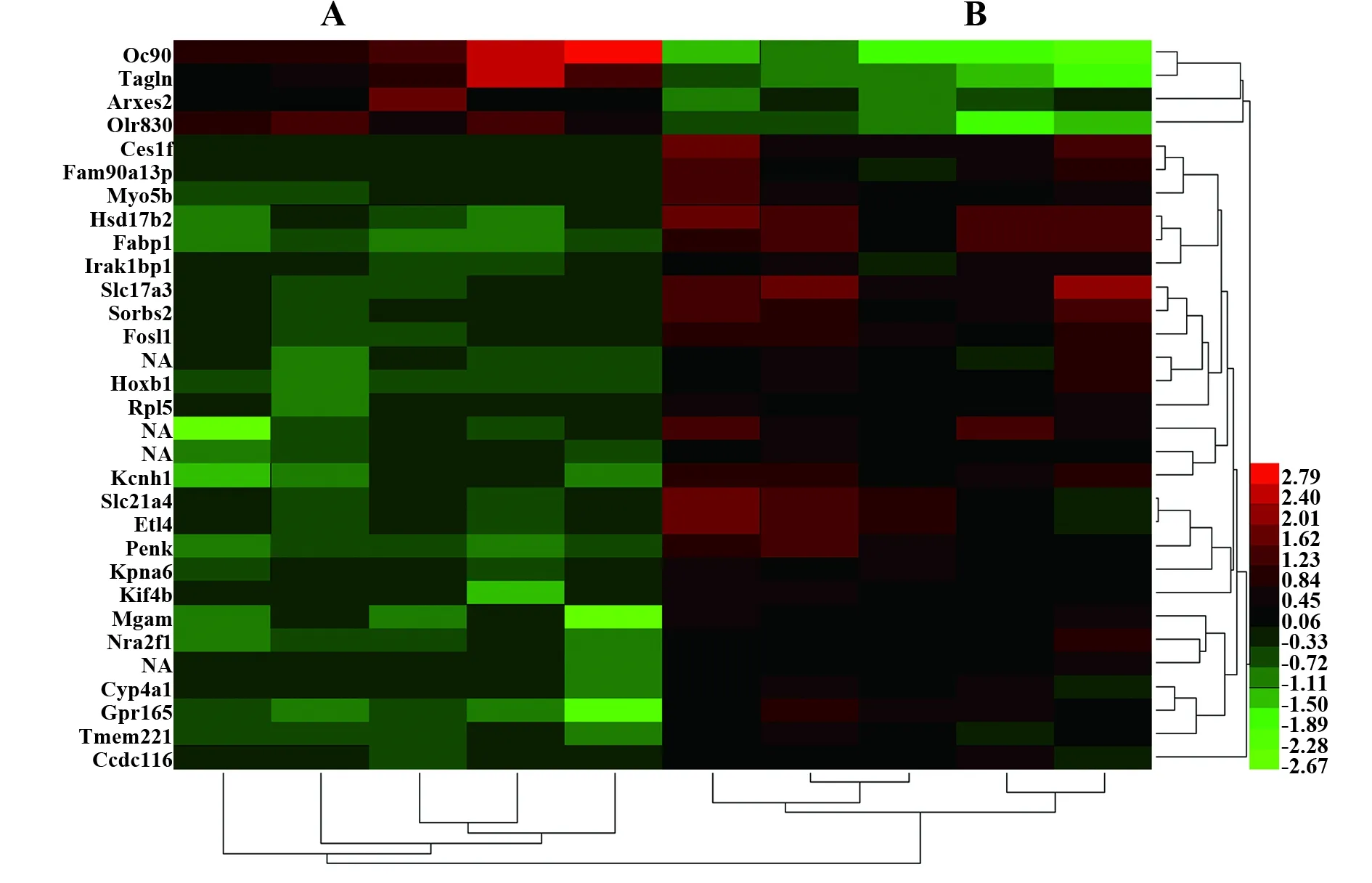
图4 基因差异性表达的分层聚类图Fig.4 Hierarchical cluster diagram of differential expression profile of genesNote:A.Control;B.LPS.

图5 实时荧光定量PCR熔解和扩增曲线Fig.5 Melting curve and amplification curve of genes Note:A.Melting curve of Ces1f;B.Melting curve of GAPDH; C.Amplification curve of Ces1f;D.Amplification curve of GAPDH.

图6 原代KC Ces1f基因表达情况Fig.6 Expression of Ces1f at mRNA between normal and LPS-stimulated Kuffer cellsNote:*.P<0.01 vs control.
2.5 两组细胞Ces1f基因表达情况 实时荧光定量PCR结果显示,Ces1f基因的表达水平分别为正常对照组细胞1.00±0.00、LPS刺激组细胞23.88±6.04。经统计学处理,两组间存在显著性差异(P<0.01),见图6。
3 讨论
KC是肝内重要的固有免疫细胞,能够有效清除外来病原体和其毒性产物,发挥免疫防御的功能。同时KC的激活,常伴随着大量促(或前)炎因子的产生和释放,造成肝组织的炎症损伤[9]。KC激活和其前炎因子释放所介导的肝脏损伤是酒精性肝炎、非酒精性脂肪肝和急性肝衰竭发生发展过程中的重要事件,并且在这些过程中,LPS都是关键的始动因素[10,11]。LPS通过与KC表面的TLR4受体结合,启动MyD88依赖和TRIF依赖的信号通路[12],诱导促炎因子释放,从而发挥免疫防御作用和炎性损伤效应。
为进一步了解LPS刺激对KC的影响,在本研究中,我们采用基因芯片分析了LPS刺激诱导大鼠原代KC内基因表达谱的变化。我们的芯片分析结果显示,LPS刺激诱导了KC内基因表达谱的明显变化。与正常细胞相比较,LPS刺激KC共有27个基因表达明显上调,主要有Ces1f、Slc17a3、Slc21a4、Hsd17b2、Sorbs2和Ccdc116等,其中尚有4个未命名且功能不详的基因;另外,LPS刺激后,表达下调的基因有4个,包括Arxes2、Olr830、Oc90和Tagln。通过基因GO生物学功能分析,我们发现,LPS刺激诱导表达上调的基因主要有以下几类:
3.1 外来化学物处理基因(Xenobiotic-processing gene, XPG) Ces1f、Slc17a3、Slc21a4、Cyp4a1、Kcnh1、Nr2f1和Kpna6。外来化学物(Xenobiotic)是指药物和来自环境中的化学物如杀虫剂和致癌物等。在机体内,这些物质主要由肝脏XPG的表达产物进行解毒和清除。肝脏的XPG有吸收(uptake)和排出(efflux)转运体(transporters)、Ⅰ期酶和Ⅱ期酶等。吸收转运体负责将外来化学物从门脉血流中转运至肝内以备代谢清除。在肝内,这些化学物可被Ⅰ期酶氧化、还原或水解,并进一步在Ⅱ期酶的作用下发生接合反应。排出转运体则可将这些代谢产物排泌入胆道或血液中。其中,Ces1f和Cyp4a1属于Ⅰ期酶。Ces1f有羧酸酯酶活性,能分解多种含羧酸酯键的药物,如血管紧张素转换酶抑制剂、奥司他韦等[13-16]。近年也发现其可能存在去乙酰化酶活性,使药物去乙酰化而代谢失活[17];Cyp4a1则是细胞色素P450家族成员,属于单加氧酶系,主要参与机体大多数药物的氧化代谢[18]。Slc17a3和Slc21a4是有机阴离子转运体,均为钠依赖性的转运体,在尿酸、甲氨喋呤等代谢产物和药物代谢转运过程中有重要的作用[19,20]。Kcnh1为钾门控性离子通道,可调节细胞内外钾离子的浓度差[21],可能参与钠依赖性阴离子转运体的调控。转录因子Nr2f1的功能主要在于调控XPG活性包括激活Ⅰ期酶和促进转运体蛋白质的翻译,而Nr2f1的表达水平又受到Kpna6的调控[22]。
3.2 脂肪分解代谢相关基因 Fabp1和Hsd17b2。Fabp1主要位于肝细胞胞浆内,是肝细胞内脂肪酸的结合蛋白,其功能在于通过将脂质配体(长链脂肪酸/脂酰辅酶A)或葡萄糖转运入细胞核内,来激活核受体PPARα,诱导脂肪酸β氧化相关的基因转录,进而促进脂肪氧化分解和产能[23]。Hsd17b2是17β-羟基类固醇脱氢酶的异构体,主要参与类固醇激素的生物合成[24]。研究显示,肥胖者体内类固醇激素代谢多异常,且脂肪组织中Hsd17b2表达下调[25]。因此,Hsd17b2的上调表达可能有助于促进脂肪的分解代谢。
3.3 糖类代谢相关基因 Mgam和Ccdc116。其中,Mgam基因编码麦芽糖酶,具有α糖苷酶活性,参与半乳糖、淀粉和蔗糖的分解代谢,生成葡萄糖[26]。Ccdc116的功能目前还不清楚,但近年发现其在胰岛内分泌细胞和胰腺内分泌肿瘤细胞中表达增高[27],提示其可能与糖类代谢有一定的关系。
3.4 细胞骨架和运动相关基因 Sorbs2、Fosl1、Myo5b和Kif4b。Sorbs2是接头蛋白家族成员,其功能在于调控细胞骨架组织(Cytoskeletal organization)和协助信号传导。研究显示,该蛋白水平增高有助于抑制细胞迁移和促进细胞黏附[28,29];Fosl1 是一个亮氨酸拉链蛋白,可通过抑制整合素αV、β3的表达,促进内皮细胞迁移[30];Myo5b属于肌球蛋白家族成员,主要参与极化上皮细胞顶膜的囊泡运输,能选择性转运Na/H交换蛋白3(Sodium/hydrogen exchanger 3,NHE3)和囊性纤维化跨膜转导调节因子(Cystic fibrosis transmembrane conductance regulator,CFTR)[31];Kif4b是一驱动蛋白家族成员,主要位于胞浆和细胞核中,与囊泡的顺向运输有关[32],可介导细胞内多种功能蛋白的胞内运输[33]。
3.5 免疫炎症相关基因 Irak1bp1、Etl4和Penk。Irak1bp1是抑炎基因,可抑制机体的固有免疫炎症反应[34,35]。已证实,Irak1bp1可促进NF-κB p50亚基核转位。在核内,p50相互结合形成抑制性同源二聚体p50/p50 。p50/p50一方面与NF-κB p65亚基竞争炎症基因的DNA结合位点,进而抑制这些炎症基因的转录激活,另一方面还可以结合IL-10基因的DNA结合位点,促进抑制性细胞因子IL-10的转录表达;Etl4可能参与了巨噬细胞的固有免疫反应[36];Penk是一种神经肽类激素,对多种免疫细胞的活性有影响。例如,Penk可影响单核细胞的趋化功能、增强NK细胞和CTL 细胞的细胞毒作用等,在内毒素性休克发生发展的病理生理机制中起重要作用[37]。
3.6 其他基因 转录因子Hoxb1可调控组织细胞增殖分化[38],Rpl5是核糖核蛋白的组成成分[39],Fam90a13p为假基因,G蛋白偶联受体家族蛋白Gpr165和跨膜蛋白Tmem221的功能尚未明确。本次芯片分析还发现了4个未命名的基因表达上调。
此外,LPS刺激KC内表达下调的基因共有4个,包括Arxes2、Olr830、Oc90和Tagln。Arxes2主要位于细胞的内质网,为脂类合成必需的反转录转座子基因。已证实其在细胞脂肪生成过程中表达水平明显升高[40];Olr830为G蛋白偶联受体家族成员,与嗅觉感知相关[41],但在巨噬细胞中的功能尚不清楚;Oc90与分泌型磷脂酶A2二级结构有25%~34%的氨基酸相似,含有两个PLA2L样结构域,存在钙离子结合功能[42];Tagln又称为SM22, 其主要功能在于调控血管平滑肌细胞骨架重构[43]。
因此,肝内KC受LPS刺激激活后,增强了对外来化学物包括药物的代谢和清除,促进了脂肪的分解和糖代谢,增加了细胞骨架蛋白水平和细胞黏附能力,并对细胞的迁移能力和免疫炎症效应有调控作用。实验研究的完成,有助于加深对KC生物学功能的理解,加深对KC作为肝内固有免疫细胞针对外来刺激特别是伤害性刺激产生反应的了解,这为将来更加深入地阐明肝组织炎症损伤的病理生理学机制奠定研究基础。
[1] Crispe IN.The liver as a lymphoid organ[J].Annu Rev Immunol,2009,27:147-163.
[2] Parker GA,Picut CA.Liver immunobiology[J].Toxicol Pathol,2005,33(1):52-62.
[3] Jenne CN,Kubes P.Immune surveillance by the liver[J].Nat Immunol,2013,14(10):996-1006.
[4] Triantafilou M,Triantafilou K.Lipopolysaccharide recognition:CD14,TLRs and the LPS-activation cluster[J].Trends Immunol,2002,23(6):301-304.
[5] Li XH,Gong JP,Shi YJ,etal.In vitro expression of CD14 protein and its gene in Kupffer cells induced by lipopolysaccharide[J].Hepatobiliary Pancreat Dis Int,2003,2(4):571-575.
[6] 叶长根,梁冬雨,赵 亮,等.大鼠枯否细胞的分离、鉴定以及LPS刺激诱导TNF-α的表达[J].世界华人消化杂志,2013,21(4):307-312.
[7] Liu LM,Zhao L,Liang DY,etal.Effects of urotensin-II on cytokines in early acute liver failure in mice[J].World J Gastroenterol,2015,21(11):3239-3244.
[8] Liu LM,Liang DY,Ye CG,etal.The UII/UT system mediates upregulation of proinflammatory cytokines through p38 MAPK and NF-κB pathways in LPS-stimulated Kupffer cells[J].PLoS One,2015,10(3):e0121383.
[9] Su GL.Lipopolysaccharides in liver injury:molecular mechanisms of Kupffer cell activation[J].Am J Physiol Gastrointest Liver Physiol,2002,283(2):G256-265.
[10] Yang Q,Shi Y,He J,etal.The evolving story of macrophages in acute liver failure[J].Immunol Lett,2012,147(1-2):1-9.
[11] Arguello G,Balboa E,Arrese M,etal.Recent insights on the role of cholesterol in non-alcoholic fatty liver disease[J].Biochim Biophys Acta,2015,1852(9):1765-1778.
[12] Szabo G,Bala S,Petrasek J,etal.Gut-liver axis and sensing microbes[J].Dig Dis Basel Switz,2010,28(6):737-744.
[13] Thomsen R,Rasmussen HB,Linnet K,etal.In vitro drug metabolism by human carboxylesterase 1:focus on angiotensin-converting enzyme inhibitors[J].Drug Metab Dispos Biol Fate Chem,2014,42(1):126-133.
[14] Zhu HJ,Markowitz JS.Activation of the antiviral prodrug oseltamivir is impaired by two newly identified carboxylesterase 1 variants[J].Drug Metab Dispos Biol Fate Chem,2009,37(2):264-267.
[15] Redinbo MR,Bencharit S,Potter PM.Human carboxylesterase 1:from drug metabolism to drug discovery[J].Biochem Soc Trans,2003,31(Pt 3):620-624.
[16] Schreiber R,Taschler U,Wolinski H,etal.Esterase 22 and beta-glucuronidase hydrolyze retinoids in mouse liver[J].J Lipid Res,2009,50(12):2514-2523.
[17] Kurokawa T,Fukami T,Nakajima M.Characterization of species differences in tissue diltiazem deacetylation identifies Ces2a as a rat-specific diltiazem deacetylase[J].Drug Metab Dispos Biol Fate Chem,2015,43(8):1218-1225.
[18] Guengerich FP.Cytochrome p450 and chemical toxicology[J].Chem Res Toxicol,2008,21(1):70-83.
[19] Jutabha P,Anzai N,Kitamura K,etal.Human sodium phosphate transporter 4 (hNPT4/SLC17A3) as a common renal secretory pathway for drugs and urate[J].J Biol Chem,2010,285(45):35123-35132.
[20] Saito H,Masuda S,Inui Ki.Cloning and functional characterization of a novel rat organic anion transporter mediating basolateral uptake of methotrexate in the kidney[J].J Biol Chem,1996,271(34):20719-20725.
[21] Haitin Y,Carlson AE,Zagotta WN.The structural mechanism of KCNH-channel regulation by the eag domain[J].Nature,2013,501(7467):444-448.
[22] Sun Z,Wu T,Zhao F,etal.KPNA6 (Importin {alpha}7)-mediated nuclear import of Keap1 represses the Nrf2-dependent antioxidant response[J].Mol Cell Biol,2011,31(9):1800-1811.
[23] Hostetler HA,Balanarasimha M,Huang H,etal.Glucose regulates fatty acid binding protein interaction with lipids and peroxisome proliferator-activated receptor α[J].J Lipid Res,2010,51(11):3103-3116.
[24] Payne AH,Hales DB.Overview of steroidogenic enzymes in the pathway from cholesterol to active steroid hormones[J].Endocr Rev,2004,25(6):947-970.
[25] 倪 鑫,唐晓露.17β羟基类固醇脱氢酶[J].生理科学进展,2002,33(2):173-175.
[26] Diaz-Sotomayor M,Quezada-Calvillo R,Avery SE,etal.Maltase-glucoamylase modulates gluconeogenesis and sucrase-isomaltase dominates starch digestion glucogenesis[J].J Pediatr Gastroenterol Nutr,2013,57(6):704-712.
[27] Tsolakis AV,Grimelius L,Islam MS.Expression of the coiled coil domain containing protein 116 in the pancreatic islets and endocrine pancreatic tumors[J].Islets,2012,4(5):349-353.
[28] Kioka N,Ueda K,Amachi T.Vinexin,CAP/ponsin,ArgBP2:a novel adaptor protein family regulating cytoskeletal organization and signal transduction[J].Cell Struct Funct,2002,27(1):1-7.
[29] Roignot J,Soubeyran P.ArgBP2 and the SoHo family of adapter proteins in oncogenic diseases[J].Cell Adhes Migr,2009,3(2):167-170.
[30] Galvagni F,Orlandini M,Oliviero S.Role of the AP-1 transcription factor FOSL1 in endothelial cells adhesion and migration[J].Cell Adhes Migr,2013,7(5):408-411.
[31] Vogel GF,Klee KMC,Janecke AR,etal.Cargo-selective apical exocytosis in epithelial cells is conducted by Myo5B,Slp4a,Vamp7,and Syntaxin 3[J].J Cell Biol,2015,211(3):587-604.
[32] Peretti D,Peris L,Rosso S,etal.Evidence for the involvement of KIF4 in the anterograde transport of L1-containing vesicles[J].J Cell Biol,2000,149(1):141-152.
[33] Martinez NW,Xue X,Berro RG,etal.Kinesin KIF4 regulates intracellular trafficking and stability of the human immunodeficiency virus type 1 Gag polyprotein[J].J Virol,2008,82(20):9937-9950.
[34] Conner JR,Smirnova II,Moseman AP,etal.IRAK1BP1 inhibits inflammation by promoting nuclear translocation of NF-B p50[J].Proc Natl Acad Sci,2010,107(25):11477-11482.
[35] Conner JR,Smirnova II,Poltorak A.Forward genetic analysis of Toll-like receptor responses in wild-derived mice reveals a novel antiinflammatory role for IRAK1BP1[J].J Exp Med,2008,205(2):305-314.
[36] Sima C,Cheng Q,Rautava J,etal.Identification of quantitative trait loci influencing inflammation-mediated alveolar bone loss:insights into polygenic inheritance of host-biofilm disequilibria in periodontitis[J].J Periodontal Res,2016,51(2):237-249.
[37] Ovadia H,Magenheim Y,Behar O,etal.Molecular characterization of immune derived proenkephalin mRNA and the involvement of the adrenergic system in its expression in rat lymphoid cells[J].J Neuroimmunol,1996,68(1-2):77-83.
[38] Zagozewski JL,Zhang Q,Pinto VI,etal.The role of homeobox genes in retinal development and disease[J].Dev Biol,2014,393(2):195-208.
[39] Lin E,Lin SW,Lin A.The participation of 5S rRNA in the co-translational formation of a eukaryotic 5S ribonucleoprotein complex[J].Nucleic Acids Res,2001,29(12):2510-2516.
[40] Prokesch A,Bogner-Strauss JG,Hackl H,etal.Arxes:retrotransposed genes required for adipogenesis[J].Nucleic Acids Res,2011,39(8):3224-3239.
[41] Gibbs RA,Weinstock GM,Metzker ML,etal.Genome sequence of the Brown Norway rat yields insights into mammalian evolution[J].Nature,2004,428(1):493-521.
[42] Zhao X,Yang H,Yamoah EN,etal.Gene targeting reveals the role of Oc90 as the essential organizer of the otoconial organic matrix[J].Dev Biol,2007,304(2):508-524.
[43] Shen J, Yang M, Ju D,etal.Disruption of SM22 promotes inflammation upon artery injury via NF-κB activation[J].Circ Res, 2010, 106(8):1351-1362.
[收稿2016-03-29 修回2016-05-20]
(编辑 张晓舟)
Microarray analysis of differentially gene expression profile in LPS-stimulated primary Kupffer cells
TAN Zhi-Li,ZHU Tong,TU Wen-Juan,LIU Liang-Ming.
Department of Infection,Shanghai Songjiang Central Hospital Affiliated to Nanjing Medical University,Department of Infection,Songjiang Hospital Affiliated to First People′s Hospital of Shanghai Jiaotong University,Shanghai 201600,China
Objective:To investigate the changes of gene expression profile in lipopolysaccharide (LPS)-stimulated primary Kupffer cells (KCs).Methods:Rat KCs were isolated and purified by means of in situ perfusion and density gradient centrifugation.After being identified by ink phagocytosis and ED2 staining test,KCs were stimulated with LPS.Gene expression profile were studied using gene microarrays,and the most significant upregulated gene was verified using real-time PCR.Results:27 genes were upregulated including Ces1f,Slc17a3,Slc21a4,Hsd17b2,Sorbs2,Ccdc116,Mgam,Myo5b,Etl4,Fabp1,Kif4b,Fosl1,Cyp4a1,Penk,Tmem221,Rpl5,Nr2f1,Hoxb1,Gpr165,Fam90a13p,Kpna6,Irak1bp1,Kcnh1 and 4 unnamed genes and 4 downregulated including Oc90,Tagln,Arxes2 and Olr830 in LPS-stimulated KCs.Among the upregulated genes,Ces1f was the most significant upregulatory gene.Real-time PCR confirmed that the levels of Ces1f were 23.88 times higher in LPS-stimulated than control cells.Conclusion:There is a significant difference between LPS-stimulated and normal control cells in gene expression profile by microarray analysis,and Ces1f is the most significantly upregulated gene.
Primary Kupffer cells;LPS;Gene expression profile;Microarray analysis;Rat
10.3969/j.issn.1000-484X.2016.12.002
①本文为国家自然科学基金项目(81070357)。
谈志丽(1990年-),女,主要从事重症肝病免疫机制研究,E-mail:lwfdtzl895@live.com。
及指导教师:刘亮明(1968年-),男,医学博士,教授,主任医师,硕士生导师,主要从事肝脏病基础和临床研究,E-mail:liuliangming@Hotmail.com。
R575.3
A
1000-484X(2016)12-1734-07

