番茄幼苗盐胁迫响应蛋白质组分析
王 强,王 娟,张慧君,李 宁,唐亚萍,王柏柯, 杨生保,杨 涛,帕提古丽,余庆辉*
(1 新疆农业科学院园艺作物研究所/新疆园艺作物生物技术与分子育种重点实验室,乌鲁木齐830091;2 新疆农业科学院经济作物研究所,乌鲁木齐830091;3 淮北师范大学 生命科学学院,安徽淮北235000)
番茄幼苗盐胁迫响应蛋白质组分析
王 强1,王 娟2,张慧君3,李 宁1,唐亚萍1,王柏柯1, 杨生保1,杨 涛1,帕提古丽1,余庆辉1*
(1 新疆农业科学院园艺作物研究所/新疆园艺作物生物技术与分子育种重点实验室,乌鲁木齐830091;2 新疆农业科学院经济作物研究所,乌鲁木齐830091;3 淮北师范大学 生命科学学院,安徽淮北235000)
采用营养液栽培,以盐敏感型番茄品种M82为试材,利用双向电泳(2-DE)研究盐胁迫处理下幼苗叶片蛋白质的表达谱,并采用基质辅助激光解析飞行时间串联质谱(MALDI-TOF/TOF-MS)技术进行差异蛋白质的分离及质谱鉴定。结果表明:(1)盐胁迫处理下,利用2-DE获得差异显著蛋白点20个,其中17个蛋白质点丰度上调表达,3个蛋白质点丰度下调表达。(2)通过质谱分析和蛋白质NCBInr数据库检索,共鉴定出19个差异蛋白,分别为果糖-二磷酸醛缩酶、S-腺苷甲硫氨酸合成酶、甘油醛-3-磷酸脱氢酶等及3个功能未知蛋白;这些鉴定出的差异蛋白质与能量代谢、光合作用、蛋白合成、氧化还原平衡等过程相关,暗示所分离鉴定的蛋白可能参与了番茄的盐胁迫响应,为进一步研究番茄抗逆机制奠定基础。
番茄;盐胁迫;蛋白质组;基质辅助激光解析飞行时间串联质谱
蛋白质作为重要的生命大分子,在参与及调节细胞新陈代谢的过程中发挥着关键性作用,植物适应逆境环境的过程中伴随着蛋白质组分的明显改变[1-2]。随着蛋白质组学成功用于植物对逆境胁迫应答的研究,为新的抗逆基因(或蛋白)的鉴定开辟了方向,基于蛋白质组学的研究对于揭示蛋白质与胁迫响应之间的关系具有重要意义。目前关于番茄盐胁迫逆境的研究大多集中在生长发育、生理生化、基因调控和转录表达等方面对非生物胁迫响应机制的研究[3-4]。但是从蛋白质组学水平进行番茄盐胁迫条件下蛋白质表达变化的研究较少。作者前期通过对试验条件的摸索,已经建立了一套适于番茄叶片蛋白质的双向电泳体系[5]。利用这一体系,通过对番茄叶片蛋白进行双向电泳分析,获得盐胁迫下番茄蛋白质差异表达谱,分析番茄幼苗叶片的蛋白质差异表达,结合串联质谱(MALDI-TOF/TOF-MS)和数据库搜索鉴定这些特异差异表达蛋白,推测其在植物盐胁迫过程中的功能,从蛋白质水平探讨番茄幼苗对盐胁迫的响应机制。
1 材料和方法
1.1 材 料
以番茄品种M82为供试材料,于2014年4月种植于新疆农业科学院安宁渠综合试验基地,幼苗4片真叶时处理。对照(CK),正常营养液水培;盐胁迫处理,在正常营养液中直接添加分析纯NaCl使营养液中的NaCl浓度达到200 mmol/L;每个处理10株幼苗,重复3次。处理24 h后选取待测材料同一部位的叶片,每处理和对照分别设置3次重复,进行蛋白含量测定及双向电泳凝胶分析。
1.2 方 法
1.2.1 蛋白质的提取 蛋白质提取采用改良的三氯乙酸/丙酮沉淀法参照Gallardo等[6]并略作修改;采用Bradford[7]的方法进行蛋白质定量;双向电泳时蛋白质上样量为100 mg,使用18 cm pH 4~7 IPG胶条,参照Bio-Rad胶条使用说明书设置等电聚焦程序,每根18 cm胶条限制电流为50 μA,上样总体积为500 μL,凝胶染色方法采用改良的考马斯亮蓝染色法[8]。
1.2.2 图像与数据分析 采用GE Healthcare凝胶扫描仪对凝胶进行图像扫描,凝胶图像用PDQuest8.0软件进行蛋白点检测及图像分析,获得凝胶之间蛋白质点匹配信息及相对表达量信息。表达量变化达到3.0倍以上且差异显著(P< 0.05)的点被认为是差异表达蛋白点。
1.2.3 蛋白质酶解与质谱鉴定 在胶上切取差异蛋白点,用MilliQ水清洗3次,50 μL碳酸氢铵(100 mmol/L NH4HCO3)进行脱色,用50 μL MilliQ水处理5 min。加入50 μL含50%乙腈的水溶液处理5 min,弃去多余溶液,加入100%乙腈处理5 min。加入5 μL含10 ng胰蛋白酶的碳酸氢铵溶液(25 mmol/L NH4HCO3),用20 μL碳酸氢铵溶液进行覆盖,37 ℃酶解16 h。MALDI-TOF-TOF质谱(基质辅助激光解吸/电离飞行时间质谱仪)分析用MALDI-TOF-TOF质谱仪。
1.2.4 数据搜索 合并一级质谱和二级质谱数据,产生peak list文件,利用MASCOT(http://www.Matrixscience.com)对NCBInr数据库进行蛋白质检索,酶解过程采用的酶为胰蛋白酶,最大允许错切位点为1,可变修饰设置为carbamidomethylation(半胱氨酸),固定修饰为oxidized(甲硫氨酸),一级质谱容差为0.15 Da,二级质谱为0.25 Da,MASCOT检索P≤0.05的结果被认为鉴定成功。
2 结果与分析
2.1 盐胁迫下番茄幼苗差异蛋白质表达谱的分析
盐胁迫下,番茄幼苗叶片蛋白质表达图谱如图1所示。其中, A图为对照叶片的双向电泳图谱, B图为盐胁迫处理后番茄幼苗叶片的双向电泳图谱, 图1,C为利用PDQuest8.0软件,对图1,A、B进行差异比对蛋白图谱。结果显示每块胶上大约有1 000个可重复的蛋白质点,获得的蛋白质点标记清晰、分离效果好。盐胁迫后,有20个蛋白点的表达丰度在处理与对照间存在显著差异表达。
2.2 盐胁迫下番茄幼苗差异蛋白质的丰度变化
PDQuest8.0软件分析结果表明,一些蛋白点在盐胁迫处理前后的表达丰度存在明显的差异,这些变化反映了番茄叶片蛋白表达对于盐胁迫的应答。以蛋白质丰度变化在3.0倍以上作为检测阈值,进一步确定了在盐胁迫处理后,番茄叶片中有20个蛋白点发生了显著差异的丰度变化(表1)。其中,丰度上调的有17个,下调的有3个。获得对照组与盐处理组蛋白质表达情况,图2箭头所指为试验中确定的部分差异显著表达的蛋白质。
2.3 盐胁迫下番茄差异表达蛋白质的质谱鉴定及功能分类
为进一步确认差异表达蛋白质的种类和功能,对20个差异蛋白点经挖点、酶解以及MALDI-TOF/TOF质谱鉴定,获得了这些蛋白点的肽质量指纹图谱(PMF)和二级质谱图谱(MS/MSS)。并结合MASCOT软件检索NCBInr蛋白数据库,20个差异蛋白点中有19个成功鉴定,蛋白质鉴定成功率约为95%。鉴定成功的19个蛋白点均来源于番茄的相关蛋白(表2)。从表中可以看出,在成功鉴定的19个蛋白质中,蛋白点0010和0107都为果糖-二磷酸醛缩酶,蛋白点8311和8312都被鉴定为甘油醛-3-磷酸脱氢酶,这些蛋白点在胶上的分布位置不同,但分别被鉴定为相同蛋白,有的分子量相近或等电点相近,可能是同一个家族蛋白的不同形式,也可能与蛋白质转录翻译后存在修饰有关,如蛋白酶降解、糖基化和磷酸化修饰等结果造成的。这一现象在大豆种子[9]、大豆叶片[10]中均有报道。
对鉴定成功的19个差异蛋白点进行生物学功能分类,有16个蛋白质分别参与能量代谢、光合作用、转录、蛋白合成、氧化还原平衡等代谢途径(表2),其中,与能量代谢相关蛋白质点最多(6个),占总差异表达蛋白质总数的31.6%,其次是参与光合作用、转录、蛋白合成、氧化还原平衡相关蛋白质。有3个蛋白点0801、0904、5716有质谱图但经搜索蛋白质数据库无功能,这很有可能是由于番茄的蛋白质组序列没有很好的注释。
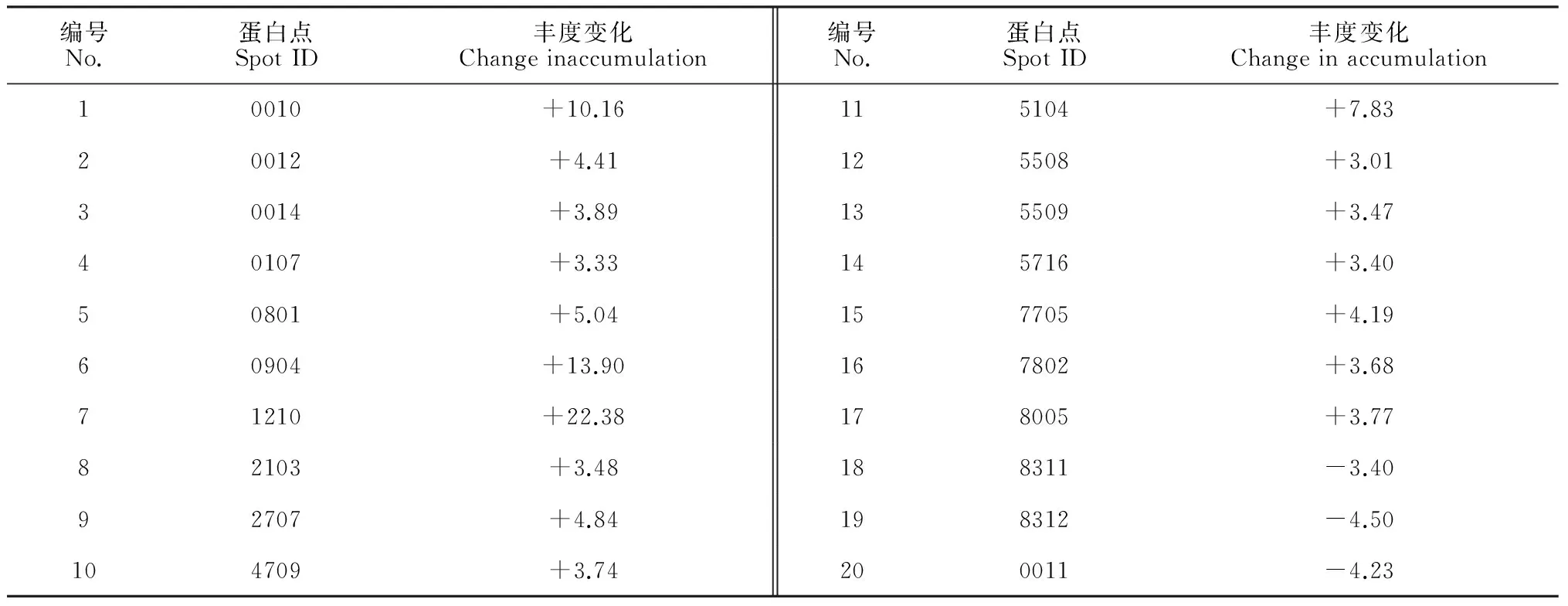
表1 盐胁迫下番茄幼苗差异表达蛋白质的丰度变化
注:+ 表示蛋白质表达量上调倍数,- 表示蛋白质表达量下调倍数
Note: +indicates the up regulated fold of protein expression, -indicates the down regulated fold of protein expression
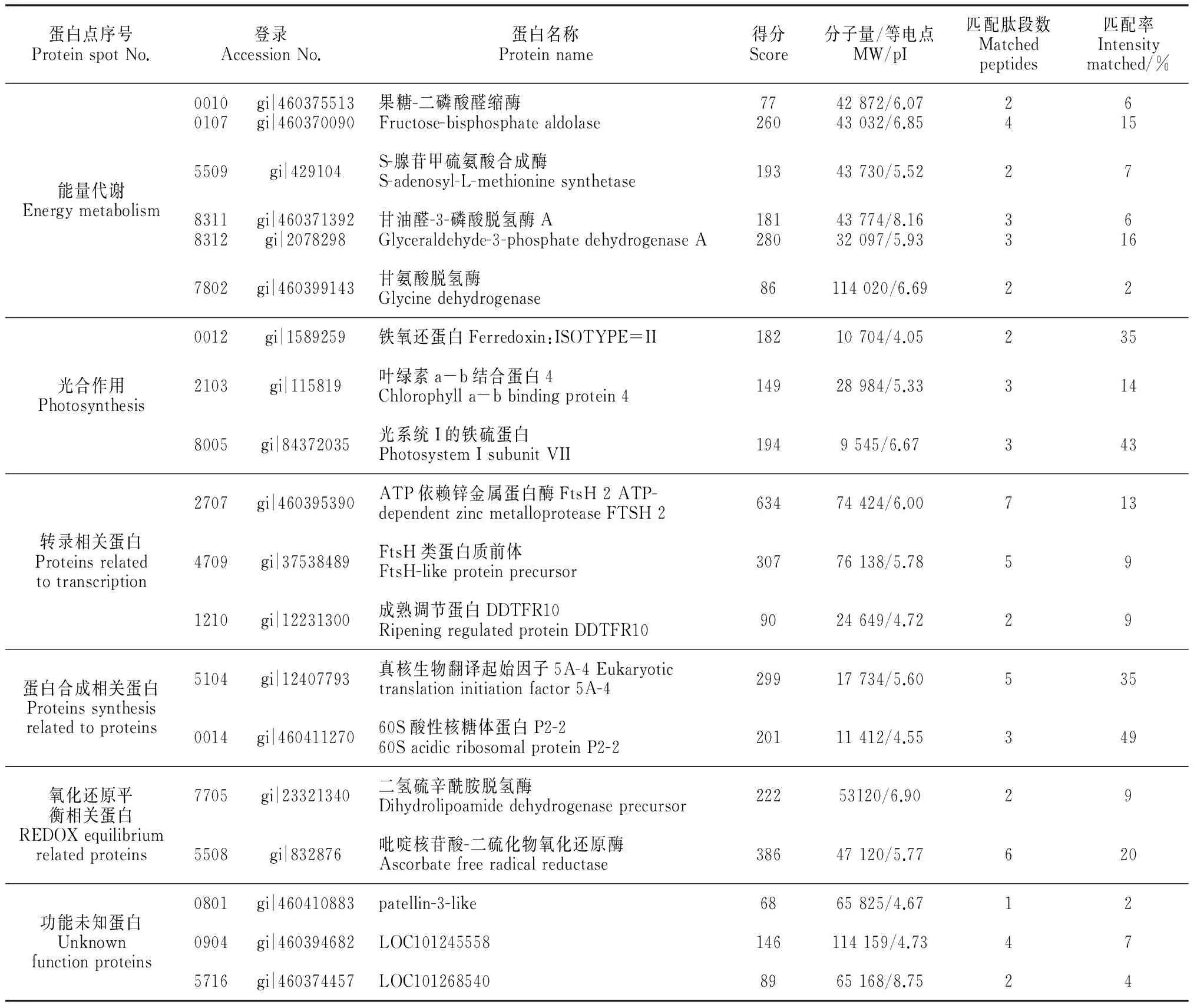
表2 盐胁迫下番茄幼苗差异表达蛋白质点的质谱鉴定
3 讨 论
本研究利用双向凝胶电泳技术对番茄幼苗盐胁迫下蛋白质的表达进行分析,通过差异蛋白质组比较、数据库检索后发现,20个差异表达蛋白点中有19个被成功鉴定,这些差异蛋白质主要参与能量代谢、光合作用、蛋白合成、氧化还原平衡等生物学过程,表明这些蛋白可能参与了番茄的盐胁迫响应。
3.1 能量代谢相关蛋白
已有研究表明植物受到盐胁迫后,一些基础代谢,如能量代谢受到影响,通过建立一个新的平衡以抵抗外界环境[11]。果糖-二磷酸醛缩酶在叶绿体中高效表达,催化卡尔文循环中果糖-1,6-二磷酸和景天庚酮糖-1,7-二磷酸的合成反应[12]。果糖-二磷酸醛缩酶存在于细胞质中,参与糖酵解过程。在各种非生物胁迫的逆境条件下,Konishi等[13]报道果糖-二磷酸醛缩酶可激活糖酵解途径,这一过程与赤霉素刺激水稻根系生长有关。也有研究表明,盐胁迫下,甜土植物与糖酵解和碳水化合物代谢有关的果糖-二磷酸醛缩酶正经历氧化胁迫,能量需求增加[14-15]。本实验结果显示果糖-二磷酸醛缩酶(0010和0107)受到盐胁迫刺激表达上调,可能与盐分产生的氧化应激有关。
甘油醛-3-磷酸脱氢酶是糖酵解途径中光合生物在逆境下物质和能量代谢途径的重要酶,不但释放能量,在机体缺氧条件下维持生命活动,而且也直接参与很多植物对环境胁迫的适应。本研究中甘油醛-3-磷酸脱氢酶(8311、8312)存在上调表达,当植物受到盐碱胁迫后,甘油醛-3-磷酸脱氢酶表达量增加,说明植物细胞内正进行着旺盛的呼吸代谢,将糖氧化分解成丙酮酸,并释放大量能量,用于维持植物生长发育以及抵抗外界胁迫[16-17]。
S-腺苷甲硫氨酸合成酶(SAMS)参与植物的硫代谢,催化甲硫氨酸和ATP反应生成S-腺苷甲硫氨酸(SAM),S-腺苷甲硫氨酸是植物体内转甲基反应的甲基供体及乙烯和多胺合成的前体[18]。作为辅助因子参与蛋白质、多糖、核酸和脂肪酸的转甲基反应。已有很多研究表明:乙烯[19]、多胺[20]等与植物的逆境调控都有密切关系,逆境胁迫下,这些大分子的必要甲基化修饰,可以减轻胁迫条件下的氧化损伤和氧化降解,进而维持自身的稳定。也有研究认为盐胁迫番茄悬浮细胞在转录水平上SAMS的调节作用,不同的SAMS基因在盐胁迫下的变化是不一样的,有些受诱导,有些受抑制[21]。本研究中,蛋白点5509被鉴定为S-腺苷甲硫氨酸合成酶,该蛋白点受到盐胁迫的诱导,上调作用更为显著,可能与盐胁迫下SAMS的诱导表达与植物需要更多的SAM有关。
甘氨酸脱氢酶参与植物的光呼吸,光呼吸被认为有利于植物在胁迫条件下维持电子传递,因此能阻止光抑制的产生[22]。有人用蛋白质组学的方法也发现低温胁迫促进豌豆线粒体中甘氨酸脱氢酶P亚基的降解[23]。甘氨酸脱氢酶(7802)在盐胁迫下被降解可能影响番茄的光呼吸,这可能是番茄对盐胁迫敏感的一个原因。
3.2 光合作用相关蛋白
在植物光合作用中,铁氧还原蛋白(Fd)从光系统Ⅰ(PSⅠ)中接受电子,再将电子传递到Fd:NADP+氧化还原酶,使NADP+还原,生成的还原力NADPH主要用于卡尔文循环中CO2的固定和叶绿体的其他代谢过程[24]。Fd是植物光合电子传递链中的一个关键传递体,在光合作用中与它所依赖的酶相互作用,调控着电子传递的方向,进而影响植物的光合作用[25]。有研究认为黄瓜叶片在低温弱光下光系统Ⅰ铁硫蛋白(PsaC)的抑制效应并不引起PSⅡ损伤,而是会引起PSⅠ的失活[26-27]。目前有研究发现PsaC在组装成PSⅠ的过程中需要PsaD和PsaE[28],但PsaC会在不依赖这两者的情况下独自丢失[29]。本实验成功鉴定到铁氧还原蛋白(0012)、光系统Ⅰ铁硫蛋白(8005),在盐胁迫下,番茄叶片表达量明显提高,可能通过光合电子传递链及PSⅠ在逆境中调节光合效率。叶绿素a/b结合蛋白(2103)是一种捕光复合物,在光合作用中作为光受体捕获或释放激发态光能,对促进光合作用十分重要。在本实验中叶绿素a/b结合蛋白质上调表达,可能这个光合作用蛋白参与了番茄的盐胁迫,在细胞遭受盐胁迫时起应激保护作用。
3.3 转录相关蛋白
前人研究认为FtsH类蛋白前体能够合成与FtsH蛋白序列和功能相似的蛋白质[30]。FtsH能够编码膜结合的金属蛋白酶而调控热激蛋白的转录,通过提高其表达量促进环境因子应激反应相关热激蛋白的形成。该基因在拟南芥中响应热胁迫的抗性发挥了关键作用[31]。本研究成功鉴定到FtsH类蛋白前体(4709)、ATP依赖锌金属蛋白酶FtsH2(2707),在盐胁迫下,表达量明显提高。说明在盐胁迫条件下FtsH类蛋白形成一种抵抗盐胁迫的机制,ATP依赖锌金属蛋白酶FtsH2可能通过提高其表达量促进盐胁迫因子应激反应相关热激蛋白的形成。
3.4 蛋白合成相关蛋白
本研究还发现一些与蛋白合成相关的差异表达蛋白,如真核生物翻译起始因子5A (5104)。eIF5A是公认的维持细胞活性必不可少的翻译起始因子,参与维持细胞活性、蛋白质合成、mRNA凋亡、植物的衰老和DNA复制等生物学过程[32],Rausell等[33]对植物的研究表明,盐胁迫下eIF1A能够增强蛋白质的翻译能力,对细胞的生存起重要作用。宋平等[34]研究表明,e1F4A是一种依赖于RNA的ATP酶,能促使mRNA与预起始复合物结合。本研究鉴定的eIF5A受盐胁迫的影响而上调表达,暗示着盐胁迫下,蛋白合成途径可能受到影响,番茄幼苗通过增强蛋白质的翻译能力,来抵御盐胁迫下可能受到的伤害。翻译调控蛋白60s酸性核糖体蛋白(0014),除参与蛋白质生物合成之外,还具有调控植物发育的功能。研究发现,许多核糖体蛋白既具有结构上的功能,同时又是翻译过程中必不可少的因子[35]。
3.5 氧化还原平衡相关蛋白
通过基因表达的调节,很多功能性蛋白的活性和丰度发生了改变,它们共同作用,越来越多的证据表明氧化还原平衡是连接胁迫感受和生理调节的一个桥梁[36]。二氢硫辛酰胺脱氢酶(7705)作为一种黄素蛋白氧化还原酶,属黄素蛋白氧化还原酶家族[37],以FAD为辅基接受质子与电子催化二硫键的形成。主要分布在线粒体中,参与甘氨酸代谢。吡啶核苷酸二硫化物氧化还原酶(5508),属于吡啶核苷酸-二硫化物氧化还原酶家族,催化依赖硫氧还蛋白NADPH的还原反应。
除以上具有已知功能的蛋白质外,2-DE检测到patellin-3-like(0801),LOC101245558(0904),LOC101268540(5716)的差异表达蛋白质经数据库检索后发现为功能未知蛋白质,可能是由于蛋白质信息在数据库中较少等原因造成,未来可结合串联质谱和数据库的增加等进行进一步功能分析,为探索番茄盐胁迫的机理提供更多线索。另外,植物的抗盐胁迫分子机制比较复杂,不仅包括翻译水平还涉及转录、转录后水平的调控。因此,下一步还要结合基因转录以及RNA转录后水平的调控等方面来进行更全面的研究。番茄在盐胁迫条件下,能量代谢、光合作用等多个生理过程协同响应盐胁迫,启动了防御反应,增强了基础代谢、能量转换、光合作用、转录、蛋白合成、氧化还原过程以适应环境的变化,这些综合的变化使得番茄能够适应盐胁迫环境。
[1] JORRIN N J,MALDONADO A M,ECHEVARRIA Z S,etal. Plant proteomics update (2007-2008):second-generation proteomic techniques,an appropriate experimental design,and data analysis to fulfill MIAPE standards,increase plant proteome coverage and expand biological knowledge[J].Journalofproteomics,2009,72(3):285-314.
[2] KOSOVA K,VITAMVAS P,PRASIL I T,etal. Plant proteome changes under abiotic stress contribution of proteomics studies to understanding plant stress response[J].JournalofProteomics,2011,74(8):1 301-1 322.
[3] 徐佳宁,徐艳群,严 振,等.番茄对非生物胁迫响应机制研究进展[J]. 山东农业科学,2015,47(12):120-124.
XU J N,XU Y Q,YAN Z,etal. Research Progress of Tomato Responses to Abiotic Stress[J].ShandongAgriculturalSciences,2015,47(12):120-124.
[4] 王利民,黄东风,林 琼,等.番茄作物抗盐性研究进展[J].福建农业学报,2015,30(10):1 019-1 026.
WANG L M,HUANG D F,LIN Q,etal. Recent advances in research on salt tolerance of tomato Plants[J].FujianJournalofAgriculturalSciences,2015,30(10):1 019-1 026.
[5] 王 强,王 娟,李 宁,等.适于番茄叶片蛋白质组分析的双向电泳优化体系的建立[J]. 新疆农业科学,2016,53(4):610-616.
WANG Q,WANG J,LI N,etal. Establishment of Two-dimensional Electrophoresis Optimization System for proteomic analysis of tomato leaves[J].XinjiangAgriculturalSciences,2016,53(4):610-616.
[6] GALLARDO K,JOB C,etal. Proteomic analysis ofArabidopsisseed germination and priming[J].PlantPhysiology, 2001,126:835-848.
[7] BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein dye binding[J].AnalyticalBiochemistry,1976,72:248-254.
[8] WANG X C,LI X F,LI Y X. A modified coomassie brilliant blue staining method at nanogram sensitivity compatible with proteomic analysis[J].BiotechnolLett,2007,29:1 599-1 603.
[9] WANG L Q,MA H,SONG L R,etal. Comparative proteomics analysis reveals the mechanism of pre-harvest seed deterioration of soybean under high temperature and humidity stress[J].JournalofProteomics,2012,75(7):2 109-2 127.
[10] MA H Y,SONG L R,SHU Y J,etal. Comparative proteomic analysis of seedling leaves of different salt tolerant soybean genotypes[J].JournalofProteomics.2012,75(6):1 529-1 546.
[11] YAN S P, ZHANG Q Y, TANG Z C,etal. Comparative proteomic analysis provides new insights into chilling stress responses in rice[J].MolCellProteomics. 2006,5:484-496.
[12] TAIZ L,ZEIGER E. Plant physiology translation[M]. Beijing:Science Press. 2009.
[13] KONISHI H,KITANO H,KOMATSU S. Identification of rice root proteins regulated by gibberellin using proteome analysis[J].Plant,Cell&Environment,2005,28(3):328-339.
[14] NDIMBA BK,CLUVASA S,Simon WJ,etal. Identification of Arabidopsis salt and osmotic stress responsive using two-dimensional difference gel electresis and mass spectrometry[J].Proteomics,2005,5(16):4 185-4 196.
[15] SOBHANIAN H,AGHAEI K,KOMATSU S. Changes in the plant proteome resulting from salt stress:the creation of salt-tolerant crops[J].JournalofProteomics,2011,74(8):1 323-1 337.
[16] PLAXTON W C. The organization and regulation of plant glycolysis[J].AnnualReviewofPlantPhysiologyandPlantMolecularBiology.1996,47(1):185-214.
[17] 郑 磊,刘关君,杨传平,等.盐胁迫下西伯利亚蓼蛋白质双向电泳分析及质谱鉴定[J]. 哈尔滨师范大学自然科学学报,2007,23(2):101-105.
ZHENG L,LIU G J,YANG CH P,etal. 2-DE analysis and mass spectrometry identification of proteins in polygumn sibiricum under salt stress[J],NaturalSciencesJournalofHarbinNormalUniversity,2007,23(2):101-105.
[18] Ma X L,WANG Z L,QI Y C,etal. Isolation of S-adenosylmethionine synthetase gene from Suaeda salsa and its differential expression under NaCl stress[J].ActaBotanicaSinicaChinese Edition, 2003,45(11):1 359-1 365.
[19] ZHANG Y, LYNCH J P,BROWN K. Ethylene and phosphorus availability have interacting yet distinct effects on root hair development[J].JournalExperimentalBotany, 2003,54(3):2 351-2 361.
[20] SHEN W,NADA K,TACHIBANA S. Involvement of polyamines in the chilling tolerance of cucumber cultivars[J].PlantPhysiology, 2000,124(3):431-439.
[21] VANBREUSEGEM F,DEKEYSER R,GIELEN J,etal. Characterization of a S-adenosylmethionine synthetase gene in rice[J].PlantPhysiol.,1994,105:1 463-1 464.
[22] WINGLER A,LEA PJ,QUICK WP,etal. Photorespiration:metabolic pathways and their role in stress protection[J].Philos.Traps.R.Soc.Lond.BBiol.Sci,2000,355,1 517-1 529.
[23] TAYLOR NL,HEAZLEWOOD JL,DAY DA,etal. Differential impact of environmental stresses on the pea mitochondrial proteome[J].Mol.Cell.Proteomics,2005,4:1 122-1 133.
[24] CECC ARELLI EA,ARAKAKI AK,CORTEZ N,etal. Functional plasticity and catalytic efficiency in plant and bacterial ferredoxin-NADP(H) reductases[J].Biochim.Biophys.Acta,2004,1698:155-165.
[25] 苟 萍,索菲娅,马东建,等.高等植物铁氧还蛋白的结构与功能[J]. 生命的化学,2007,27(1):51-53.
GOU P,SOPHIE,MA D J,etal. The structure and fuction of higher plant ferredoxin [J].ChemistryofLife,2007,27 (1):51-53.
[26] TERASHIMA I,FUNAYAMA S,SONOIKE K. The site of photo inhibition in leaves ofCucumissativusL.at low temperatures is photosystem I,not photosystem II[J].Planta,1994,193:300-306.
[27] SONOIKE K,TERASHIMA I,IWAKI M,etal. Destruction of photosystem I iron-sulfur centers in leaves ofCucumissativusL. by weak illumination at chilling temperatures[J].FEBSLett,1995,3(62):235-238.
[28] ANTONKINE M,JORDAN P,FROMME P,etal. Assembly of protein subunits within the stromal ridge of photosystem I. Structural changes between unbound and sequentially PSI-bound polypeptides and correlated changes of magnetic properties of the terminal iron sulfur clusters[J].J.Mol.Biol.,2003,327:671-697.
[29] RAJAGOPAL S,JOLY D,GAUTHIER A,etal. Protective effect of active oxygen scavengers on protein degradation and photochemical function in photosystem I sub membrane fractions during light stress[J].FEBSLett,2005,272(4):892-902.
[30] LINDAHL M,TABAK S,CSEKE L,etal. Identification,characterization,and molecular cloning of a homologue of the bacterial FtsH protease in chloroplasts of higher plants[J].JournalofBiologicalChemistry,1996,271(46):29 329-29 334.
[31] CHEN J,BURKE J J,VELTEN J,etal. FtsHl l protease plays a critical role in Arabidopsis thermotolerance[J].ThePlantJournal,2006,48(1):73-84.
[32] 袁金桥,颜贤忠.真核生物翻译起始因子5A的结构与功能研究进展[J]. 军事医学科学院院刊,2009,33(3):267-271.
YUAN J Q,YAN X ZH.Structure and function of eukaryotic initiation factor 5A:recent progress in research[J].BulletinoftheAcademyofMilitaryMedicalSciences,2009,33(3):267-271.
[33] RAUSELL A,KANHONOU R,YENUSH L,etal. The translation initiation factor eIF1A is an important determinant in the tolerance to NaCl stress in yeast and plants[J].ThePlantJournal,2003,34(3):257-267.
[34] 宋 平,张竞男,胡咖瑞,等.食蟹猴疟原虫中一个真核翻译起始因子4A (eIF4A)同源蛋白的cDNA克隆,表达和ATP酶活性测定[J]. 中国生物化学与分子生物学报,2004,20(3):294-304.
SONG P,ZHANG J N,HU J R,etal. cDNA cloning,expression and ATPase assay of a putative eIF-4A ofPlasmodiumcynomolgi[J].ChineseJournalofBiochemistryandMolecularBiology,2004,20(3):294-304.
[35] 顾志敏,王建飞,黄 骥,等.水稻胞质糖体蛋白基因OsRPS7的克隆与序列分析[J].遗传,2004,26(2):181-185.
GU Z M,WANG J F,HUANG J,etal. Molecular cloning and sequence analyzing of cytoplasmic ribosomal protein gene OsRPS7 from rice (OryzasativaL) [J].Hereditas,2004,26(2):181-185.
[36] FOYER CH,NOCTOR G .Redox homeostasis and antioxidant signaling:A metabolic interface between stress perception and physiological responses[J].PlantCell,2005,17,1 866-1 875.
[37] MORAN J F,SU Z H,Sarath G,etal. Molecular cloning,functional characterization,and subcellular localization of soybean nodile dihydrolipoamide reductase[J].PlantPhysiology,2002,128:300-313.
(编辑:宋亚珍)
Proteome Analysis of Tomato Seedlings in Response to Salt Stress
WANG Qiang1,WANG Juan2,ZHANG Huijun3,LI Ning1,TANG Yaping1,WANG Baike1,YANG Shengbao1,YANG Tao1,Patiguli1,YU Qinghui1*
(1 Institute of Horticulture,Xinjiang Academy of Agricultural Sciences/Key Laboratory of Horticultural Biotechnology & Molecular Breeding of Xinjiang,Urumqi 830091,China;2 Institute of Economic Crops,Xinjiang Academy of Agricultural Sciences,Urumqi 830091,China;3 School of Life Science,Huaibei Normal University,Huaibei,Anhui 235000,China)
Nutrient solution culture was used to M82 tomato salt sensitive variety as experimental materials. With two-dimensional electrophoresis (2-DE), we studied the salt conditions and processing tomato seedlings under salt stress protein expression profile,and identified the differences of protein separation and mass spectrometry trough tandem time-of-flight mass spectrometry (MALDI-TOF/TOF-MS) technology. The results showed that: (1) there were 20 protein spots presenting to express differentially after the total proteins were separated by 2-DE. The 17 proteins of all presenting spots were up-regulated,and 3 proteins were down-regulated. (2) Among the 20 protein spots,the annotations of the 19 differential expressed proteins were known by mass spectrometry and protein database retrieval,Identified differential proteins took part in fructose-bisphosphate aldolase,S-adenosyl-L-methionine synthetase,glyceraldehyde 3-phosphate dehydrogenase,etc,and three unknown functions of protein. These identified the differences of proteins involved in energy metabolism, photosynthesis,protein synthesis,redox balance and other related process,suggested that these proteins may be involved in the salt stress response of tomato,which lay a foundation for further research the mechanism to understand plant stress resistance.
tomato;salt stress;proteomics;MALDI-TOF/TOF-MS
1000-4025(2016)11-2226-07
10.7606/j.issn.1000-4025.2016.11.2226
2016-08-15;修改稿收到日期:2016-10-31
新疆维吾尔自治区自然科学基金(2014211B035);国家自然科学基金(31360482);农业部公益性行业科研专项(201303115)
王 强(1983-),男,硕士研究生, 副研究员,主要从事蔬菜栽培生理与逆境胁迫。E-mail:wangqiang201004@sina.com
*通信作者:余庆辉,博士研究生,研究员,主要从事加工番茄新品种选育和栽培。E-mail:yuqinghui98@sina.com
Q530;Q789
A

