黄牛GBP4基因拷贝数变异检测及其对5个生长性状的影响
熊业城,曹修凯,贺 花,2,宋林霏,黄 橙,党李苹,张晋媛,雷初朝,陈 宏,祁兴磊,黄永震*
(1.西北农林科技大学动物科技学院,陕西省农业分子生物学重点实验室,陕西 杨凌 712100;2.西北农林科技大学动物医学学院,陕西 杨凌 712100;3.河南泌阳县畜牧局,河南 泌阳 463700)
科学试验
黄牛GBP4基因拷贝数变异检测及其对5个生长性状的影响
熊业城1,曹修凯1,贺 花1,2,宋林霏1,黄 橙1,党李苹1,张晋媛1,雷初朝1,陈 宏1,祁兴磊2,3,黄永震1*
(1.西北农林科技大学动物科技学院,陕西省农业分子生物学重点实验室,陕西 杨凌 712100;2.西北农林科技大学动物医学学院,陕西 杨凌 712100;3.河南泌阳县畜牧局,河南 泌阳 463700)
[目的] 研究黄牛GBP4基因拷贝数变异检测及其对5个生长性状的影响。[方法] 本实验以200头秦川牛和100头夏南牛为研究对象,通过实时荧光定量PCR技术检测GBP4基因拷贝数变异。[结果] 分析发现,GBP4基因拷贝数变异的分布在2个品种之间存在显著差异(\%P\%<0.05)。秦川牛群体存在Gain和Normal两种类型;夏南牛群体存在Gain,Normal和Loss三种类型;其中Gain为拷贝数增加型,Normal为拷贝数不变型,Loss为拷贝数减少型。该基因拷贝数变异类型与秦川牛和夏南牛5个生长性状关联分析显示,GBP4基因拷贝数变异与秦川牛体高显著相关(Normal>Gain P=0.046)。[结论] 本研究结果表明,GBP4基因拷贝数变异可以作为秦川牛选育的候选分子标记。
GBP4基因;拷贝数变异;实时荧光定量PCR;效应分析
黄牛品种资源在我国十分丰富,并且黄牛对于畜牧业有着重要的影响力,是世界黄牛遗传资源的重要组成部分。有些品种(如秦川牛和夏南牛)具有适应性强、肉用性能良好的特点,且品种间差异显著,是研究基因组遗传变异和品种间进化关系的宝贵材料。
随着对基因组研究的逐渐深入,基因组上的一种亚显微水平的结构变异——拷贝数变异(Copy number variations,CNVs)被广泛报道[1-3],现已从“基因组变异”发展为“表型变化”的另一研究热点。由于CNV涉及到基因序列范围较广,甚至可能包含整个功能基因,因此,可以通过基因剂量效应的改变影响该基因控制的表型。在近期的《Genome Research》和 《BMC Genomics》杂志上,利用全基因组测序技术,检测到了包括GBP4基因在内的多个与牛生长、肉质相关的QTLs位于CNVs区域内,表明影响个体表型差异的CNVs广泛存在于基因组中[4]。
鸟苷酸结合蛋白4(GBP4)可以通过促进Caspase-II的转录,来推动Caspase-II介导的天然免疫[5]。本课题组前期研究结果表明,GBP4基因位于牛3号染色体的57554999到58293641的CNVs区域内(Btau 4.0)。已有的研究表明,该区域是牛的生长发育、肉质、胴体和泌乳和健康等性状的重要数量性状基因座(Quantitative trait loci,QTLs)[6]。
目前关于中国黄牛GBP4基因序列变异方面的分析尚未见报道。本研究采用实时荧光定量PCR技术,从DNA水平检测GBP4基因CNV在不同牛品种(秦川牛和夏南牛)中及同一品种内的分布差异基因组变异,确定群体中存在的不同CNV类型(如缺失、单拷贝、多拷贝),并将其与黄牛的重要生长性状进行关联分析。最终,期望找到决定牛生长发育的特异位点或者Marker基因,为我国黄牛的遗传改良和分子育种提供可靠的遗传学基础资料。
1 材料与方法
1.1 样品来源与数据收集
本研究所用300份黄牛血样包括:200份秦川牛基础母牛(陕西省秦川牛良种繁育中心,陕西扶风)和100份夏南牛基础母牛(夏南牛科技开发有限公司,河南泌阳),测定的主要性状包括:体重、体高、体长、胸围和坐骨端宽;测定时间为24月龄。
DNA样品采集时采用的是ACD抗凝,冰盒迅速带回实验室,-80℃保存备用。用苯酚-氯仿抽提法[7]提取基因组DNA,分光光度计测定浓度后,用灭菌蒸馏水稀释至标准浓度25 ng·μL-1,4℃保存待用。
1.2 引物的设计和PCR扩增
根据GenBank上公布的黄牛GBP4基因的DNA序列(登录号:NC_007301.6),再结合利用引物设计软件Primer5.0设计引物(F:5'-TGGGAATAACAACTGACACTG-3';R:5'-ATTGGGCTGAATCCTCTACTT-3');内参基因选择RPP30,引物序列(F:TGCTTCCATTGTTTCCTGATGA;R:TGGGACCAGGTTCCATGATC)。引物由南京金斯瑞生物科技有限公司合成。
Realtime-PCR反应体系:2×TaqTMII 5μL,Fp 0.5 μL,Rp 0.5μL,DNA 1μL,加ddH2O至12.5μL。
总之,党的十九大首提的乡村振兴战略下的“建立健全城乡融合发展体制机制和政策体系”,就是要通过改革、转型、创新去推动城乡地位平等、要素互动、共生共融。建构乡村振兴的“融”机制与敢为人先的小岗精神高度契合,乡村振兴战略“融”机制的建构既明确了乡村在中国特色社会主义现代化建设中的突出地位和在城乡关系中的平等地位,又为实现2018年中央一号文件提出的“让农业成为有奔头的产业,让农民成为有吸引力的职业,让农村成为安居乐业的美丽家园”的美好愿景奠定了坚实的基础。
Realtime-PCR反应条件为95℃预变性5 min;94℃变性30 s,60℃退火30 s,72℃延伸16 s,30个循环;72℃延伸10 min,4℃保存。
1.3 建立DNA池和琼脂糖凝胶电泳检测
在所有DNA样品中随机地提取的选出1/3的样品,各取5 μL,将所有提取的样品放入一根离心管中,充分混匀,调整终浓度为50~60 ng·μL-1,以建立黄牛牛混合DNA池。
将DNA池中的所提取浓度适宜的样品为模板进行扩增,并对PCR产物进行琼脂糖凝胶电泳检测:随机抽取10 μL PCR产物经限制性酶EaeI消化处理,选用2%(w/v)的琼脂糖凝胶,电泳50~60 min,采用EB染色,分析。[8]
1.4 统计分析
记录通过实时定量PCR技术所得黄牛GBP4基因拷贝数与引物拷贝数的值,并归纳统计出GBP4基因CNV在秦川牛和夏南牛中的分布情况。采用SPSS软件(Version 16.0)对GBP4基因CNV类型与黄牛生长性状进行关联分析。
2 结果
2.1 牛GBP4基因CNV在不同牛品种中的分布差异
受自然选择和人工选择的影响,同一物种的不同品种间基因组遗传变异存在差异。为了检测GBP4基因CNV在中国不同黄牛品种内的分布情况,我们分别选取了夏南牛和秦川牛进行检测,结果见图1。从图中可以看出,在秦川和夏南牛中,GBP4相对拷贝数分布较离散,都表现出拷贝数增加、正常和减少三种类型。夏南牛相对于秦川牛表现为拷贝数减少;同时,在夏南牛中,GBP4拷贝数相对于秦川牛分布较离散。
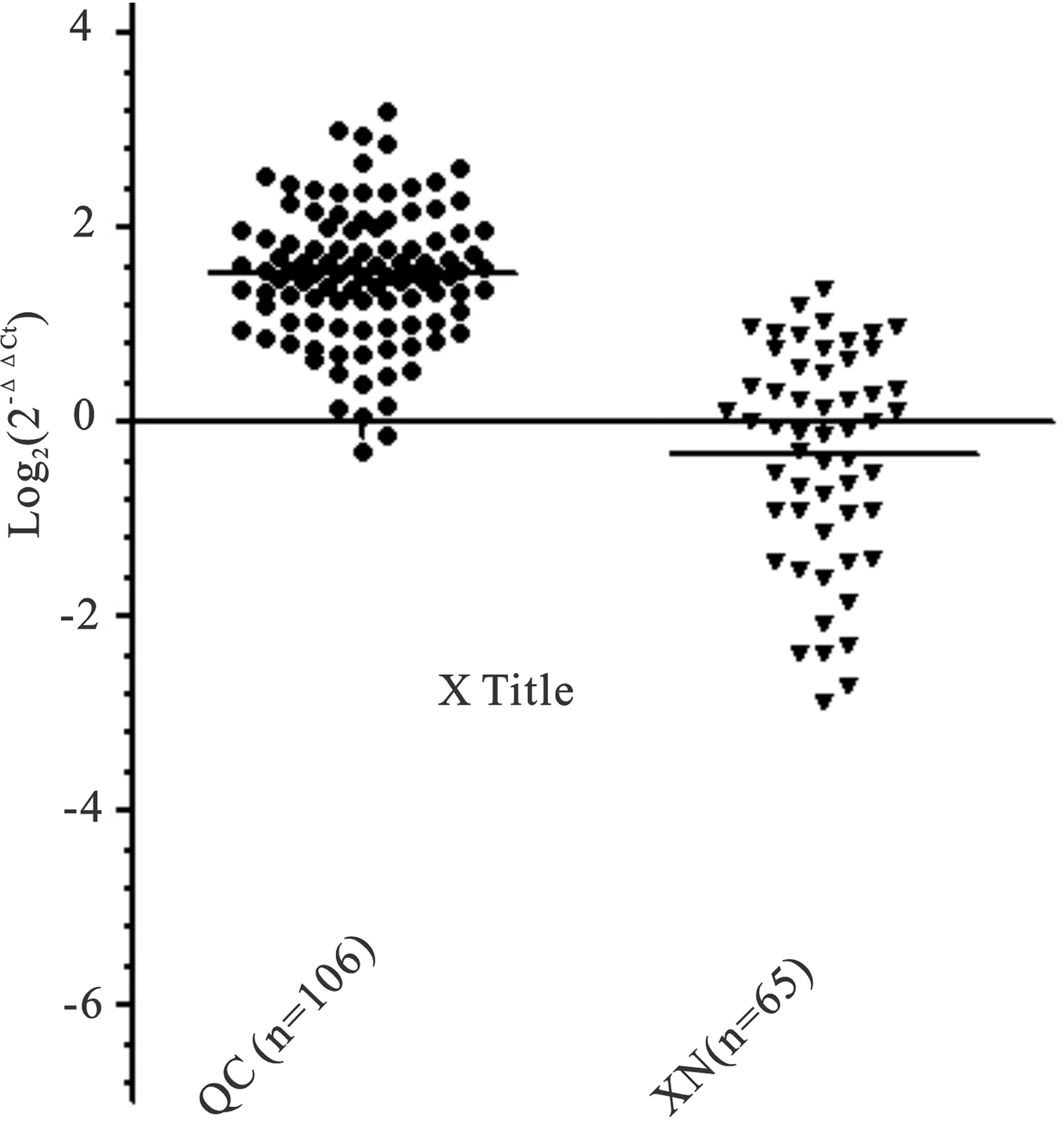
图1 GBP4基因在两个牛群中的拷贝数分布图
2.2 牛GBP4基因拷贝数变化对于牛的重要生长性状的关联分析
根据拷贝数目的差异,将CNV分为三种类型,分别是拷贝数增加(Log22-ΔΔCt> 0.5 Gain)、拷贝数减少(Log2 2-ΔΔCt< -0.5 Loss)和拷贝数不变(-0.5
3 讨论
3.1 牛GBP4基因CNV在不同牛品种中的分布差异
从图1中可以看出,GBP4基因CNV分布在2个品种中的差异较大,其中,在夏南牛中,该基因拷贝数变异分布比较离散,说明在夏南牛群体内不同个体间GBP4基因CNV也存在差异。总体来看,夏南牛与秦川牛之间存在显著差异(\%P\%<0.05)。
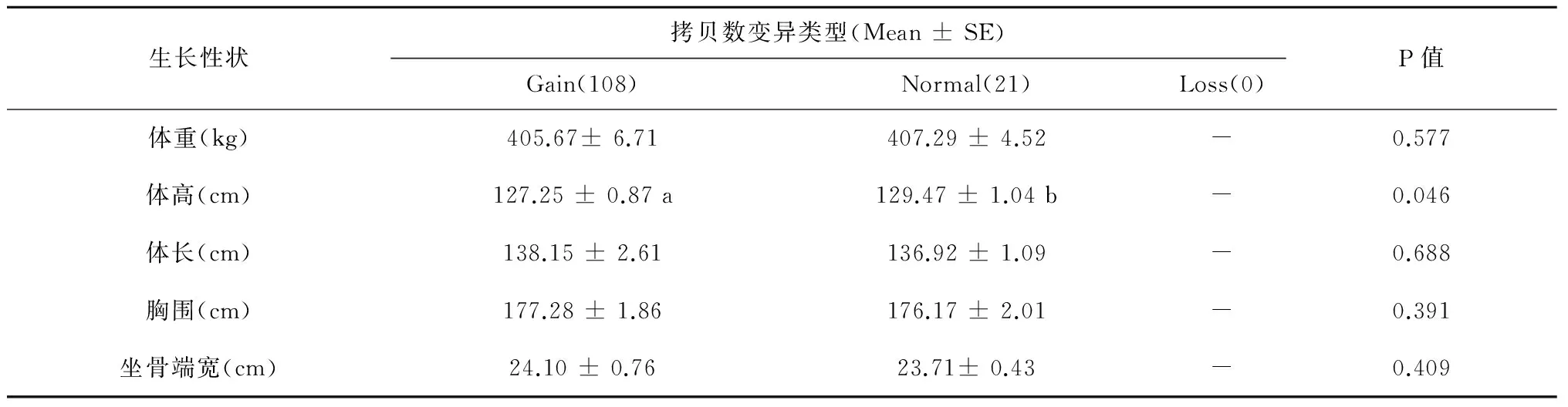
表1 牛GBP4基因CNV与秦川牛生长性状的关联分析
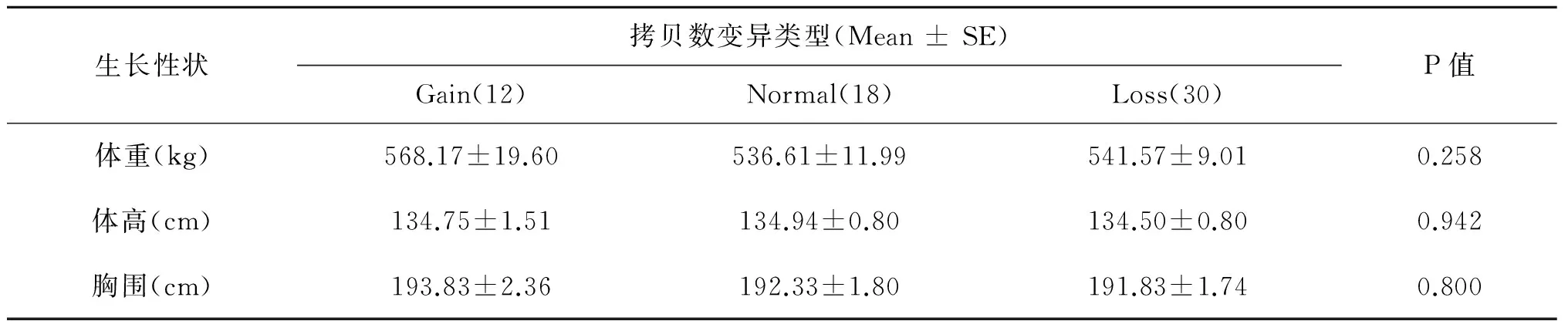
表2 牛GBP4基因CNV与夏南牛生长性状的关联分析
3.2 牛GBP4基因拷贝数变化对于牛的重要生长性状的关系
牛的健康、生长发育、泌乳、胴体和肉质等性状均由多基因座位控制。而通过实验确定影响生长性状的主基因对我国地方黄牛品种培育为肉用方向有重要意义。已有研究表明,在动物生长发育和泌乳过程中GBP4基因发挥着重要的作用。因此,我们能够将其作为候选基因之一与数量性状位点(QTL)来进行连锁分析,并作出数量性状的辅助标记选择。
[1] Khaja R,Zhang JJ,MacDonald JR,\%et al.\%Genome assembly comparison identifies structural variants in the human genome.Nature Genetics,2006,38(12):1413-1418.
[2] Redon R,Ishikawa S,Fitch KR,\%et al.\% Global variation in copy number in the human genome.Nature,2006,444(7118):444-454.
[3] Conrad DF,Bird C,Blackburne B, \%et al.\% Mutation spectrum revealed by breakpoint sequencing of human germline CNVs.Nature Genetics,2010,42(5):385-391.
[4] Bickhart DM,Hou Y,Schroeder SG,\%et al.\% Copy number variation of individual cattle genomes using next-generation sequencing[J].Genome Res.2012;22(4):778-790.
[5] Shi J,Zhao Y,Wang Y,\%et al.\% .Inflammatory caspases are innate immune receptors for intracellular LPS[J]. Nature.2014,514(7521):187-192.
[6] Zhang L,Jia S,Yang M,\%et al.\%.Detection of copy number variations and their effects in Chinese bulls.BMC Genomics.2014(17):15:480.
[7] [美]萨姆布鲁克,D w拉塞尔.分子克隆实指南[M].北京:科学出版社,2002.468-469.
[8] 娄佑武,吴志勇,王荣民等.吉安西杂母牛DGAT1基因K232A遗传多态性及其对乳成分影响的分析[J].中国牛业科学,2015,41(3):18-21.
The Detection of GBP4 Gene Copy Number Variation and Its Effect on Five Bovine Growth Traits
XIONG Ye-cheng1,CAO Xiu-kai1,HE Hua1,2,SONG Lin-fei1,HUANG Cheng1,DANG Li-ping1,ZHANG Jin-yuan1,LEI Chu-zhao1,CHEN Hong1,Qi Xing-lei3,HUANG Yong-zhen1*
(1.CollegeofAnimalScienceandTechnology,NorthwestA&FUniversity,ShaanxiKeyLaboratoryofMolecularBiologyforAgriculture,Yangling,Shaanxi, 712100,China;2.CollegeofVeterinaryMedicine,NorthwestA&FUniversity,Yangling,Shaanxi, 712100,China;3.BureauofAnimalHusbandryofBiyangCounty,Biyang,Henan, 463700,China)
【Objective】 This study investigated the detection of GBP4 gene copy number variation and its effect on five growth traits for cattle. 【Method】 A total of 300 individuals, including 200 Qinchuan cattle individuals and 100 Xianan cattle individuals, were selected as the experimental animals to detect the copy number variation of GBP4 gene by real-time fluorescent quantitative PCR ( RT-qPCR ).【Results】The result showed there were significant differences in the distribution of the copy number variation of GBP4 gene between the two varieties (\%P\%< 0.05 ). Qinchuan cattle group included two types: Gain and Normal, Xianan cattle group included three types: Gain, Normal and Loss. Gain is a copy number increased type; Normal is a copy number constant type; Loss is a copy number decreased type. According to the associated analysis between the copy number variation types and five growth character of Qinchuan cattle and Xianan cattle, it was found that the copy number variation had a significant relationship with body height of Qinchuan cattle (Normal > Gain P = 0.046). 【Conclusion】 These findings indicated that the copy number variation of GBP4 gene can be used as candidate molecular markers for Qinchuan cattle breeding.
GBP4 gene; copy number variation; real-time quantification PCR; effect analysis
2016-05-11
2016-05-13
本项目由国家肉牛牦牛产业技术体系专项(CARS-38)资助,中国博士后科学基金面上项目(2015M570857,2015M570856),陕西省科技统筹创新工程计划项目(2015KTCL02-08,2014KTZB02-02-02-02),国家发改委生物育种能力建设与产业化专项(2014-2573), 西北农林科技大学2015年大学生创新创业训练计划项目资助完成。
熊业城(1996-),男,江西省九江人,本科生,主要从事动物遗传与育种研究。
*通讯作者:黄永震(1982-),男,河南南阳人,博士,主要从事牛分子遗传与育种研究。
S823
A
1001-9111(2016)04-0009-04

