3种不同来源侧耳品种亲缘关系差异化分析
赵同心, 赵洪新, 王升厚
(1.沈阳师范大学 化学与生命科学学院,辽宁 沈阳 110034;2.浙江理工大学 生命科学学院,浙江 杭州 310018;3.沈阳师范大学 实验教学中心,辽宁 沈阳 110034)
3种不同来源侧耳品种亲缘关系差异化分析
赵同心1, 赵洪新2, 王升厚3*
(1.沈阳师范大学 化学与生命科学学院,辽宁 沈阳 110034;2.浙江理工大学 生命科学学院,浙江 杭州 310018;3.沈阳师范大学 实验教学中心,辽宁 沈阳 110034)
采取拮抗实验、酯酶同工酶、基因组进行多态性分析(RAPD)、内部转录间隔区序列(ITS)对来源不同的特大凤尾菇(Pleurotussajor-caju)、夏秀990(Pleurotusgeesteranus)和黑平A3三个侧耳品种的亲缘关系进行了对比分析。实验结果表明:供试的3个侧耳品种从形态学水平、蛋白质水平和分子水平上看,其品种间亲缘关系界定具有一致性。表现为特大凤尾菇与夏秀990的亲缘关系较近,与黑平A3的亲缘关系较远,夏秀990与黑平A3亲缘关系较远。本研究从不同的水平,为食用菌菌种属间亲缘关系的鉴定提供了综合分析方法,对区分同种异名、杂交亲本选择以及判定杂交结果具有指导意义。
侧耳;亲缘关系;酯酶同工酶;随机扩增多态性DNA(RAPD);转录间隔区序列(ITS)
侧耳属真菌常见的品种有糙皮侧耳(Pleurotusostreatus,俗称平菇),金顶侧耳(PleurotuscitrinopileatusSing,俗称榆黄蘑),漏斗状侧耳(Pleurotussajor-caju,俗称凤尾菇),刺芹侧耳(Pleurotuseryngi,俗称杏鲍菇),阿魏侧耳(PleurotusferulaeLanzi,俗称白灵菇)等,全世界约有50多种,我国现有36种[1],是目前人工栽培数量较大、范围较广的食用大型真菌。随着人工栽培规模的不断扩大,特别是选育新品种的种植与推广,新的商品名称或代号的菌种不断出现,出现了来源各异、同种异名或异种同名的混乱现象。由于不同种源对环境适应能力的差异,给引种菇农造成损失现象时有发生。为了对菌种做到科学管理、保护品种产权、杜绝生产领域的菌种乱引乱种现象,许多研究人员采取不同方法对侧耳属进行鉴别。黄奕存[2]提出了不同个体间的拮抗反应,从菌丝的形态学方面进行不同鉴定。李荣春等[3]采用酯酶同工酶方法对不同菌株间的相似程度进行鉴定分析。郑素月等[4]、张庆华等[5]、汤卫真等[6]分别分析了菌株之间酯酶同工酶的差异。随着分子生物学的发展,RAPD技术和ITS技术用于平菇分类。詹才新等[7]、陈明杰等[8]验证了RAPD可用于金针菇和香菇亲缘关系的鉴定,随后谭琦等[9]用RAPD技术分析并建立了遗传相关聚类图,分析出香菇杂交非对称杂交后代相似关系。White等[10]为真菌rRNA基因的ITS设计3对特异引物,即ITS1、ITS4、ITS5,可用于大多数担子菌和子囊菌。谭琦等[11]分析了中国6个白灵菇商业菌株的DNA图谱,证明6个菌株为同一个种。为提高品种间亲缘关系鉴定方法的有效性、可靠性和实用性,本研究选择不同来源侧耳属的3个品种为供试菌株,采取拮抗鉴定、酯酶同工酶分析、RAPD多态性分析和ITS间隔区序列比对,从表型、蛋白和分子3个水平进行综合运用,为菌种间亲缘关系鉴定提供一种更为科学有效的综合方法。
1 材料与方法
1.1 材料
1.1.1 供试菌株 夏秀990(秀珍菇,Pleurotusgeesteranus)、黑平A3(Pleurotusostreatus)、特大凤尾菇(Pleurotussajor-caju),由沈阳师范大学特种菌业研究所保存。
1.1.2 培养基 改良PDA培养基:葡萄糖20 g,蛋白胨5 g,磷酸二氢钾 2 g,硫酸镁1 g,水1 000 mL。
1.2 方法
1.2.1 培养方法 ①拮抗实验:取活化好的菌种,在改良PDA培养基上打孔,进行两两拮抗实验, 25 ℃培养箱中培养1周。②菌种培养:在液体PDA培养基中,200 r/min、25 ℃培养1周,取菌丝体提取蛋白质和DNA。酯酶同工酶的方法参考文献[12]。DNA提取用CTAB法。
1.2.2 RAPD 和ITS引物筛选与扩增 引物如表1所示。引物采取设计随机引物的方法,从引物库中进行筛选。引物合成和PCR产物测序,由上海生工生物公司完成。 RAPD PCR反应体系:10 ×TaqPCR Buffer 2.5 μL,随机引物2 μL(2 μmol/L),TaqDNA聚合酶 0.5 μL,DNA 0.5 μL,dNTP 0.5 μL(10 mmol/L),去离子无菌水 14 μL。PCR反应程序: 94 ℃预变性 5 min,94 ℃变性1 min,36 ℃退火1 min,72 ℃延伸80 s,30个循环,72 ℃保持10 min,4 ℃保存。样品取出置于冰箱-20 ℃中保藏。用0.8%琼脂糖凝胶电泳,切胶分离PCR产物,回收,送生工生物工程(上海)股份有限公司测序。
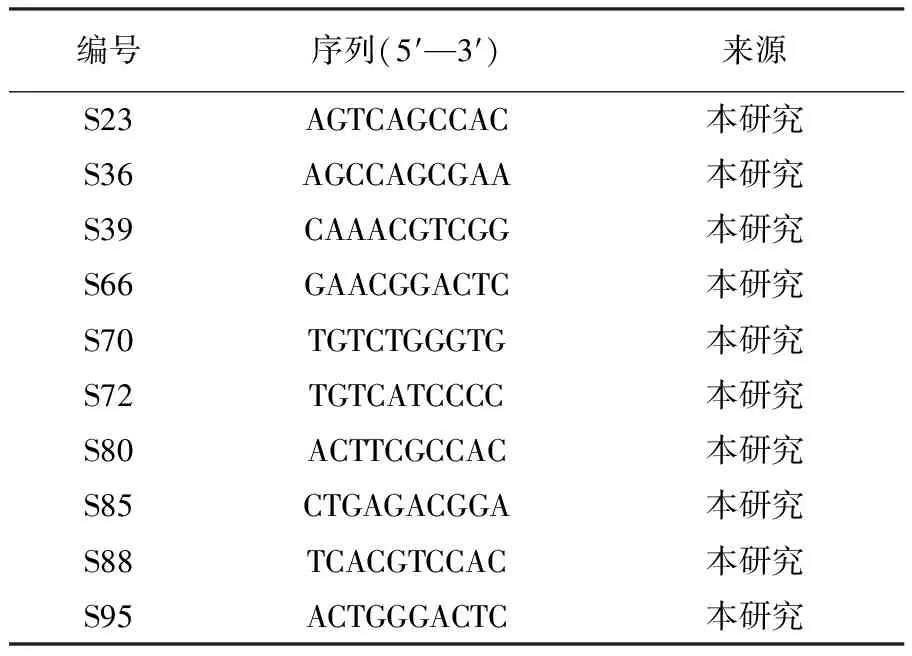
表1 RAPD 引物
ITS聚合酶链式反应PCR 扩增参考文献[7],用2个真菌引物ITS4和ITS5进行扩增。PCR反应体系 10 ×TaqPCR Buffer 2 μL,引物ITS4 0.5 μL,引物ITS5 0.5 μL,TaqDNA聚合酶 0.5 μL,DNA 2 μL,dNTP 0.5 μL(10 mmol/L),去离子无菌水 14 μL。
PCR反应条件:94 ℃ 预变性 5 min,94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸90 s,30个循环,72 ℃保持10 min,4 ℃保存,取出样品冰箱-20 ℃保藏。用0.8%琼脂糖凝胶电泳分离PCR产物。切胶回收PCR产物,将回收的产物连接到T载体上,转化到大肠埃希菌E.coli5α的感受态细胞中,Amp100抗性筛选,用菌落PCR筛选阳性的单菌落。用鼎国质粒提取试剂盒(NEP011-1)提取纯化质粒后,送生工生物工程(上海)股份有限公司进行测序。
1.2.3 数据分析 用SPASS进行数值分析,MAGE 6.0进行ITS序列分析亲缘关系。
2 结果与分析
2.1 拮抗实验
不同菌种之间均出现拮抗线(图1),说明3种平菇属于不同的种。
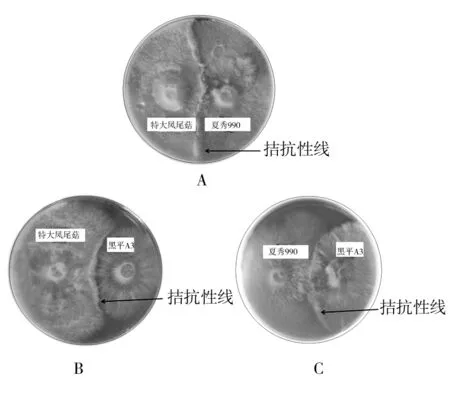
图1 3个平菇品种的拮抗反应
2.2 酯酶同工酶谱带分析
2.2.1 酶谱的类型 3个平菇品种共有18条酯酶同工酶条带(图2),分子量大约在70~170 kDa。 迁移率不同的为13条,其中黑平A3有4条,与其他两个品种相比,没有相同分子量的条带,说明与其他两个品种之间差异较大。夏秀990有5条酶带,与特大凤尾菇相同,说明二者亲缘关系较近;另外,特大凤尾菇有自己特异的4条带,与夏秀990不同。可能由于特大凤尾菇与夏秀990存在进化程度上的差异,或者特大凤尾菇进化的快些,导致了较多的特异性条带。

图2 酯酶同工酶电泳图
2.2.2 联合系数 根据sneath和sokal(1973年)提出的联合系数,可以清楚地反映品种间的亲缘关系。联合系数在0~1之间变化,其值越大两品种亲缘关系越近,越小亲缘关系越远。特大凤尾与夏秀990联合系数为0.556,说明二者亲缘关系较近,黑平菇的亲缘关系较其他两个关系较远,见表2。
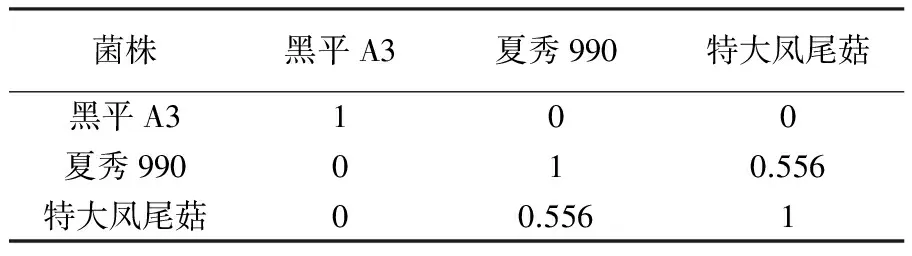
表2 3个平菇品种之间的联合系数
2.3 RAPD 条带分析
2.3.1 RAPD电泳条带分析 10个RAPD 引物对3种侧耳进行PCR扩增,筛选出S23、S88引物进行电泳分析。S23引物扩增结果,3个品种中有2条相同的条带,特大凤尾菇与夏秀990带型一致,其中夏秀990的3条带和特大凤尾菇相同,特大凤尾菇2条特异性条带,黑平A3特异性条带2条与另外两种明显不同。S88引物结果显示,特大凤尾菇5条带其中2条与夏秀990相同,特异性条带2条,夏秀990特异性条带1条。黑平A3有1条与夏秀990相同,2条特异性条带。结果见图3。总之,根据RAPD结果分析可知夏秀990和特大凤尾菇的条带较为一致,亲缘关系较近,在分子水平上可以看出它们有共同的特征条带,不同品种间存在着差异。
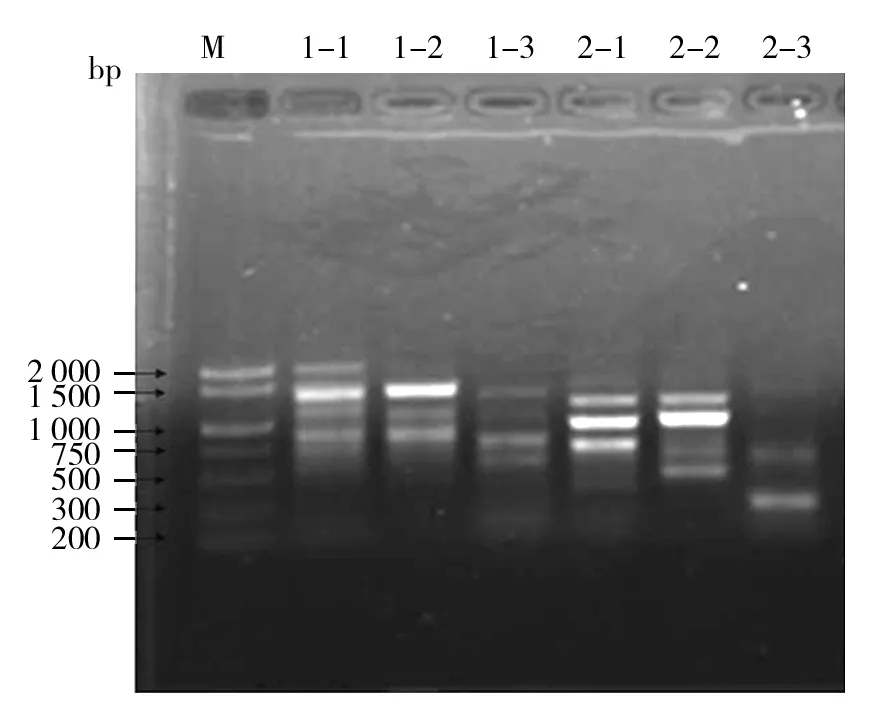
图3 3个平菇品种之间的RAPD条带
2.3.2 聚类分析 用SPASS进行数值分析,有条带的为1,无条带的为0,得到的聚类分析图见图4。由图4可以看出,特大凤尾菇和夏秀990亲缘关系较近,与黑平A3进化距离远。
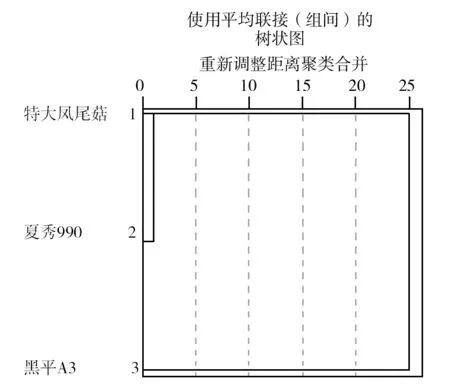
图4 3个平菇品种之间的聚类分析Fig.4 Cluster analysis among three kinds Pleurotus spp.
2.4 ITS序列分析
2.4.1 目的片段的扩增 用引物ITS4和ITS5扩增3个品种的ITS区,电泳图谱如图5所示,可以看出,PCR条带基本一致,分子量约为680 bp。
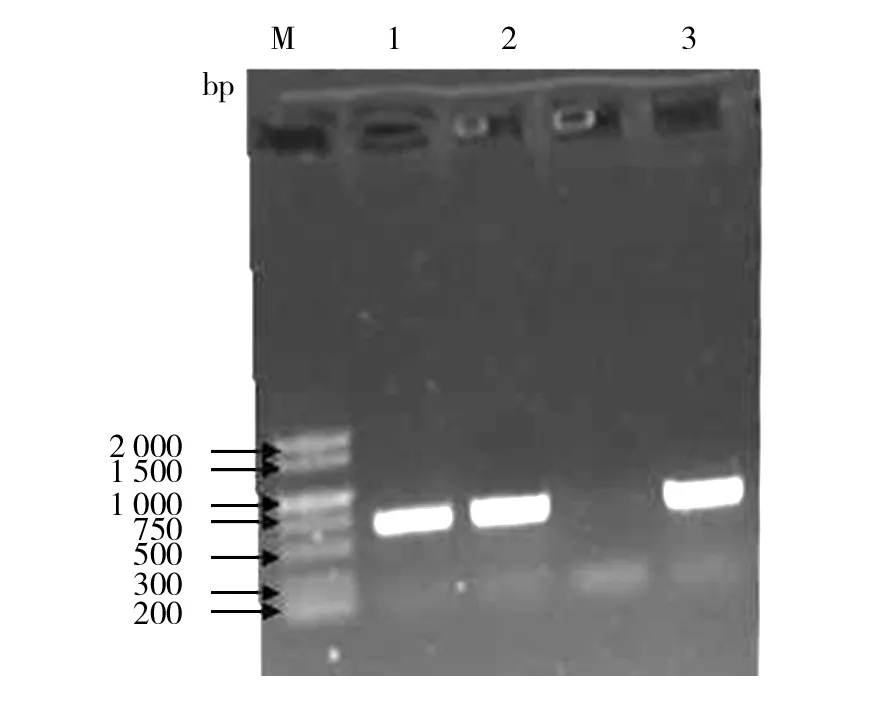
图5 3个平菇品种ITS PCR产物Fig.5 ITS PCR product among three kinds Pleurotus spp.1:特大凤尾菇;2:夏秀990;3:黑平A31:Phoenix mushroom;2:Xiaxiu 990;3:Black oyster mushroom A3
2.4.2 ITS分析 对3种平菇的ITS序列分析比对发现,夏秀990和特大凤尾菇的序列完全相同,无碱基差别,而黑平A3与夏秀990、特大凤尾菇碱基序列差别较大。1~54 bp为18S RNA,55~286 bp为转录间隔区1即ITS1,287~444 bp为5.8S RNA,445~642 bp为转录间隔区2即ITS2,643~703 bp为28S RNA。
由序列比对可以看出,在ITS1发生了碱基替换,第161 bp的C→T,第71 bp的T→C。在ITS2区序列出现碱基的替换和插入,在第466 bp的A→T,第 469 bp和第496 bp的T→C,第481 bp的T→A,第514 ~515 bp的TG→CT, 第517 bp和第521 bp A→G,在469 bp和470 bp之间插入了序列GGTTTTTTTCC。
2.4.3 平菇的进化树 用MAGE 6.0进行亲缘关系远近分析,结果显示:1号的特大凤尾菇和2号的夏秀990亲缘关系较近,3号黑平A3亲缘关系较远。本研究通过对ITS区域的扩增测序, 分析平菇不同品种间的亲缘关系并将平菇ITS区域序列登录到核苷酸序列数据库中,登录号分别为KP877605、KP877606、KP877607,见图6。

图6 3个平菇品种ITS聚类分析Fig.6 ITS Cluster Analysis among three kinds Pleurotus spp.
3 讨 论
本研究结合形态学的拮抗反应,蛋白质水平酯酶同工酶,分子水平的RAPD和ITS区序列分析得出了供试3种侧耳属品种间的亲缘关系。拮抗反应是真菌鉴定的常用手段,但往往不能有效区分出两个在遗传上有相似背景或亲缘关系很近的菌株,可作为初级分类的方法;同工酶是由不同等位基因或不同基因位点编码的[13],特别是在属内种间及品种之间的分类鉴定是非常有效的。同工酶技术也具有易受培养时间、培养基配方及培养温度的影响而表现出不一致性的缺点,并且不能直接反映 DNA 水平的情况[14]。因此做好分子水平的鉴定就成为完整实验方法的重要组成。本研究从ITS序列比对结果看,特大凤尾菇和夏秀990序列完全相同,说明它们的进化程度一致,同时也说明了ITS区序列长度有限,相对全基因组而言,有限的序列信息要想真正反映物种的进化规律,还需要其他的形态学等方面的数据。从序列比对可以看出的ITS区域的差异主要表现在ITS2区域,与司源等[15]的ITS1相对变化、ITS2相对保守的观点不同,进化的快慢可能是由于不同的生物ITS区,但是ITS2相对变化这一观点与Yao等[16]将ITS2区域作为动植物的DNA标记观点一致。
本研究从宏观水平、蛋白质水平和DNA水平等多个层面说明了平菇之间的进化程度与亲缘关系,同时也证明进行分类时应结合多种手段来说明它们之间的亲缘关系。
[1] 图力古尔,李玉.我国侧耳属真菌的种类资源及其生态地理分布[J].中国食用菌,2001,20(5):8-9.
[2] 黄亦存.高等担子菌的性非亲和性系统和体细胞非亲和性系统在遗传多样性保存中的作用[J].生物多样性,1996,4(1):41-44.
[3] 李荣春,陈严平.五种牛肝菌酯酶和过氧化物酶的同工酶研究[J].中国食用菌,1999,18(3)1:5-19.
[4] 郑素月,张金霞,黄晨阳,等.中国栽培平菇的酯酶同工酶分析[J].食用菌学报,2003,10(4):1-6.
[5] 张庆华,赵新海,钟丽娟,等.酯酶同工酶分析鉴别生产用平菇菌种真实性研究[J].山东农业科学,2009,26(6):10-12.
[6] 汤卫真,郑国扬,毕志树.利用同工酶酶谱法鉴别侧耳属的种和菌株[J].广西植物,1987,7(2):143-145.
[7] 詹才新,朱兰宝,杨新美.RAPD技术在金针菇杂交育种中的应用[J].食用菌学报,1995,2(1):1-7.
[8] 陈明杰,谭琪,汪昭月,等.应用RAPD技术对香菇融合菌株进行遗传鉴定[J].食用菌学报,1997,4(4):17-20.
[9] 谭琦,潘迎捷,陈明杰,等.香菇遗传研究和菌种选育的现状及发展[J].食用菌学报,1998,5(3):6-11.
[10]White T J,Bruns T D, Lee.S. ,et al.Analysis of phytogenetic relationships by amplification and direct sequencing of ribosomal RNA genes[C].INNISMA.PCR Protocols:A Guide to Methods and Applications.New York Academic,1990:315-322.
[11]谭琦,杨建明,陈明杰,等.用RAPD技术对香菇非对称杂交后代遗传性状的分析[J].食用菌学报,2001,8(2):10-14.
[12]胡能书,万国贤.同工酶技术及应用[M].长沙:湖南科学技术出版社,1985:49-181.
[13]熊全沫.同工酶电泳数据的分析及其种群遗传上的应用[J].遗传,1980,8(1):1-5.
[14]Gottlieb L D. Conservation and duplication of isozymes in plants[J].Science,1982,216(23):373-380.
[15]司源,郭亦琦,孔航辉.基于ORF Finder方法的植物ITS片段结构特点分析[J].华北农学报,2005, 20(5):54.
[16]Yao H,Song J,Liu C,et al.Use of ITS2 region as the universal DNA barcode for plants and animal[J].PLoS ONE,2010,5(10):13102.
Found projecd: National Natural Science Foundation of China (31160371, 30860165); National Key Technology Research and
Development Program of China (2012BAD19B02); National High Technology Research and Development Program of China (863
Program) ( 2011AA10A206)
Brief introduction to the writer: Cheng liang male, Ph D student. Research field: natural product.
Tel:0971-5313283, E-mail:liangcheng1979@163.com
Draft accepted date: 2015-04-03; Revision returned date: 2015-08-31
Alliance Variance Analysis of ThreePleurotusSpeciesfrom Different Sources
ZHAO Tong-xin1, ZHAO Hong-xin2, WANG Sheng-hou3
(1.Coll.ofChem. &LifeSci.ShenyangNormalUni.,Shenyang110034; 2.Coll.ofLifeSci.ZhejiangSci-Tech.Uni.,Hangzhou310018; 3.Exper’lTeach.Ctr.,ShenyangNormalUni.,Shenyang110034)
In this study the alliance among threePleurotusofphoenixtail mushroom,xiaxiu990 andblackoyster mushroom A3 from different sources with different morphology were carried out contrast analysis. The results showed that the tested threePleurotusspecies possessed the congruity among species alliance definition in morphology level, protein level and molecular level. The alliance betweenphoenixtail mushroom andxiaxiu990 displayed closer, but further fromblackoyster mushroom A3, and the alliance betweenxiaxiu990 andblackoyster mushroom A3 was further. This study provided comprehensive methods to analyze the characterization of the alliance of edible fungi of inter-genus in different levels, and had directive significance to separate different name of the same species, to select hybrid parents, and to judge the results of hybridization.
Pleurotusostreatus; alliance; esterase isozyme; random amplified polymorphic DNA (RAPD); transcribed spacer sequence (ITS)
辽宁省科学事业公益项目(2014003020)
赵同心 女,硕士研究生。研究方向为微生物制药。E-mail:txzhao@sina.com
* 通讯作者。男,教授,硕士生导师。主要从事药用真菌资源的开发与利用。E-mail:wshhwq2009@163.comn
2015-06-01;
2015-08-03
Q93-331
A
1005-7021(2016)02-0062-05
10.3969/j.issn.1005-7021.2016.02.011

