安徽东流水牛线粒体D-Loop区遗传多样性与系统进化分析
赵拴平,贾玉堂,徐 磊,阮永明
(安徽省农业科学院 畜牧兽医研究所,安徽 合肥230031)
安徽东流水牛线粒体D-Loop区遗传多样性与系统进化分析
赵拴平,贾玉堂,徐 磊,阮永明
(安徽省农业科学院 畜牧兽医研究所,安徽 合肥230031)
【目的】 分析安徽东流水牛群体的分子遗传特性,为今后开展我国地方水牛品种研究提供科学依据。【方法】 采用测序技术测定安徽东至县31头东流水牛线粒体DNA(mtDNA) D-Loop序列,结合GenBank数据库中已报道的22个群体的471条中国水牛D-Loop序列进行联合分析,利用DnaSP 5.1软件统计核苷酸多样性和单倍型多样性,利用MEGA 5.10软件构建N-J系统发生树,利用Network 4.60软件构建单倍型的media-joining网络。【结果】 在22个中国水牛群体中发现163个变异位点,180个单倍型,其核苷酸多样性为0.018 23±0.002 21,单倍型多样性为0.877 40±0.013 80,其中在东流水牛D-Loop序列中发现70个变异位点,构成35种单倍型,其核苷酸多样性为0.013 31±0.002 08,单倍型多样性为0.944 00±0.023 00,群体变异性水平与中国其他水牛群体接近。N-J系统发生树和media-joining进化网络显示,中国水牛分为沼泽型和江河型,前者又分为A和B支系,B支系包括b1和b2 2个亚支系,东流水牛分布于A和B支系中。【结论】 东流水牛群体遗传多样性丰富,且线粒体具有2个母系来源。
水牛;线粒体DNA;遗传多样性;单倍型
水牛是我国南方的传统家畜,主要分布于北纬36°以南,东经97°以东的广大地区[1],是我国热带、亚热带水稻区的主要役畜,在农业生产和畜牧业生产中具有重要的经济价值。据2013年联合国粮农组织FAO统计(http://faostat.fao.org),我国水牛存栏数2 300多万头,居世界第3位。在系统分类学中,水牛分为亚洲水牛和非洲水牛2个属,亚洲水牛根据其外貌、习性、生产性能、生物学特征以及染色体数目不同,进一步分为沼泽型水牛和江河型水牛。由于中国水牛长期以来以使役为主,肉、乳生产性能不高,随着近年来农业产业结构的调整,农村劳动力的转移,以及饲养水牛效益的下降,水牛的使用价值降低。东流水牛主要分布于安徽省沿江丘陵湖区的东至县、望江县、宿松县和贵池区等地,中心产区东至县位于长江中下游南岸,属于滨湖地区。东至县2002年以前东流水牛饲养量达到3.8万头,年出栏量达10 000头以上,2013年调查发现东流水牛存栏仅6 000多头,其中纯种东流水牛母牛存栏不足1 000头[2],亟需加以保护。
由于线粒体DNA(mtDNA)D-Loop序列变异速率为其他片段的5~10倍,在世代传递过程中没有重组,已被广泛应用于家畜起源进化、亲缘关系和群体遗传结构研究。在水牛研究方面,Lei等[3-4]通过对中国兴隆、福安、温州、汉中、信阳、滨湖和江汉7个地方水牛群体119个水牛个体线粒体D-Loop区序列研究发现,中国水牛遗传多样性丰富,遗传进化分析发现中国水牛分为A、B 2个支系,表明其有2个母系起源,并证明沼泽型水牛是在中国驯化而成的。亐开兴等[5]研究了滇东南水牛D-Loop区序列多态性,发现滇东南水牛遗传多样性较为丰富,属于沼泽型水牛。张自芳等[6]通过研究滇东南水牛D-Loop区,发现其存在2个母系血统来源,受河流型水牛基因渗入较小。张瑞光等[7]通过研究福安水牛线粒体D-Loop区,发现福安水牛遗传多样性丰富,系统发育揭示福安水牛存在2个母系血统来源。卢永芳等[8]研究了安徽江淮水牛D-Loop区序列多态性,发现江淮水牛具有丰富的遗传多样性,群体变异性水平与中国其他水牛群体接近。Yue等[9]通过研究我国22个沼泽型水牛群体线粒体DNA的遗传多样性,发现支系A水牛很可能在我国西南地区驯化而成。本研究通过测定31头安徽东流水牛线粒体DNA D-Loop序列,结合GenBank数据库中已报道的471条中国水牛D-Loop序列,研究分析东流水牛线粒体D-Loop区遗传多态性和系统进化关系,以期为地方水牛品种资源保护和开发利用提供科学依据。
1 材料与方法
1.1 材 料
东流水牛血样取自安徽省池州市东至县的水牛群体,样本间无亲缘关系,颈静脉采血法采集31头水牛血液10 mL/头,ACD抗凝,低温带回实验室,冻存于-80 ℃冰箱中。
为与东流水牛进行比较,从GenBank数据库中下载了来源于22个群体471条已报道的中国水牛D-Loop序列[4,9]。
1.2 方 法
1.2.1 DNA提取及mtDNA D-Loop区序列的PCR扩增和测序 利用DNA提取试剂盒(天根生化科技有限公司,北京)对31份血液样品进行基因组提取,1.0%琼脂糖凝胶电泳检测其完整性,并利用核酸分析仪测定DNA的浓度和纯度。利用引物CB1:5′-TAGTGCTAATACCAACGGCC-3′和CB2:5′-AGGCATTTTCAGTGCCTTGC-3′[10]扩增,获得915 bp的水牛线粒体D-Loop全序列。PCR反应体系:2 μL DNA,5 μL 10× PCR缓冲液, 4 μL 2.5 mmol/L dNTP混合物,正反向引物各1 μL (100 ng/μL),0.5 μL 5 U r-TaqDNA 聚合酶,去离子水补充至50 μL;扩增条件为:95 ℃预变性5 min;95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,32个循环;72 ℃总延伸5 min。扩增产物利用1.5%琼脂糖凝胶电泳检测,凝胶回收试剂盒进行产物纯化与回收, 委托上海生工生物工程有限公司进行测序。
1.2.2 数据处理 利用DNASTAR 5.0软件对水牛线粒体DNA D-Loop区测序序列进行校对,利用ClustalX进行序列比对,利用MEGA 5.10[11]查找序列的多态位点,利用DnaSP 5.1软件[12]统计核苷酸多样性(Nucleotide diversity)和单倍型多样性(Haplotype diversity),利用MEGA 5.10软件构建N-J(Neighbor-joining)系统发生树,分析不同单倍型间的进化关系,利用Network 4.60软件[13]构建单倍型的media-joining网络。
2 结果与分析
2.1 东流水牛mtDNA D-Loop区的核苷酸变异和单倍型多样性
通过分析安徽东流水牛mtDNA D-Loop区完整序列,发现其序列长度为911~ 915 bp, 核苷酸变异存在转换和颠换,转换中AG为 2.62% (24/915),TC为3.93%(36/915),颠换为1.42% (13/915) 。联合分析31条安徽东流水牛mtDNA D-Loop测序序列和23条GenBank数据库中已报道的东流水牛mtDNA D-Loop序列,结果(图1)发现,54个东流水牛群体中共存在70个变异位点,构成35种单倍型,其中6种单倍型(Hap10、Hap12、Hap14、Hap16、Hap18和Hap29)为共有单倍型,单倍型Hap16频率最大,为22.2%(12/54),其他单倍型均为不同个体所特有。东流水牛mtDNA D-Loop区核苷酸多样性为0.013 31±0.002 08,单倍型多样性为0.944 00±0.023 00,说明其具有丰富的遗传多样性。东流水牛突变位点和单倍型频率见图1。

N.单倍型频数 The frequency of haplotypes
图 1 安徽东流水牛35个线粒体DNA D-Loop单倍型的变异位点
Fig.1 Polymorphic sites of haplotype of 35 mitochondrial D-Loop region detected in 54 Dongliu buffaloes
对中国地方水牛22个群体共502条水牛mtDNA D-Loop序列进行联合分析,结果(表1)发现,共存在163个变异位点,构成180个单倍型,核苷酸多样性为0.018 23±0.002 21,单倍型多样性为0.877 40±0.013 80,其中19个是在安徽东流水牛中新发现的单倍型。广西水牛群体中共有29个个体,其中16个个体(9种单倍型)具有江河型水牛的血统,可能是引种引发的基因渗入。如表1所示,安徽东流水牛单倍型多样性和核苷酸多样性与其他水牛群体相差无几,说明其群体遗传多样性与其他水牛群体接近。各个水牛群体所对应的核苷酸多样性和单倍型多样性见表1。

表 1 中国水牛22个群体线粒体DNA D-Loop区的遗传多样性
注:n.样本数;k.单倍型数。
Note:n.Number of samples;k.Number of haplotypes.
2.2 中国地方水牛群体mtDNA D-Loop区的系统发生树和进化网络
利用东流水牛mtDNA D-Loop区35个单倍型构建N-J系统发生树,结果(图2)发现,35个单倍型明显分为A、B 2个大的支系,A支系包括28个单倍型,代表46个水牛个体,占总水牛群体的85.2%(46/54);B支系包含7个单倍型,代表8个水牛个体,说明东流水牛具有2个母系来源,即A和B支系,但B支系的单倍型变异程度(87.5%,7/8)比A支系(60.9%,28/46)高。
利用获得的180个中国水牛mtDNA D-Loop序列单倍型构建N-J系统发生树,以牛(Bostaurus)(登录号NC_006853)作为构建系统进化树的外群体参照,结果如图3所示。由图3可知,180个水牛单倍型分为沼泽型(Ⅰ)和江河型(Ⅱ)两个水牛群体,其中江河型水牛群体包含9个单倍型,代表广西水牛群体中16个个体,说明广西水牛在长期培育过程中,可能是引种引发的基因渗入;沼泽型水牛群体中有171个单倍型,代表486个个体,分为A、B 2个支系。
进化网络(Network)是一种可以客观描述物种内不同单倍型间遗传进化关系的方法,其中media-joining进化网络可以快速地进行大量单倍型数据分析[14]。本研究利用Network 4.60软件构建了沼泽型水牛171个mtDNA D-Loop单倍型间的media-joining进化网络,结果见图4。由图4可看出,A支系呈现以单倍型Hap55为中心,周围116个单倍型环绕的星状辐射结构,它们与中心单倍型有1~6个核苷酸的差异;B支系与A支系之间存在32个核苷酸的差异,包含b1和b2两个亚支系,其中b1与b2存在12个核苷酸差异;b1支系也呈现星状辐射结构,b2支系中部分单倍型与中心单倍型间变异性较大。
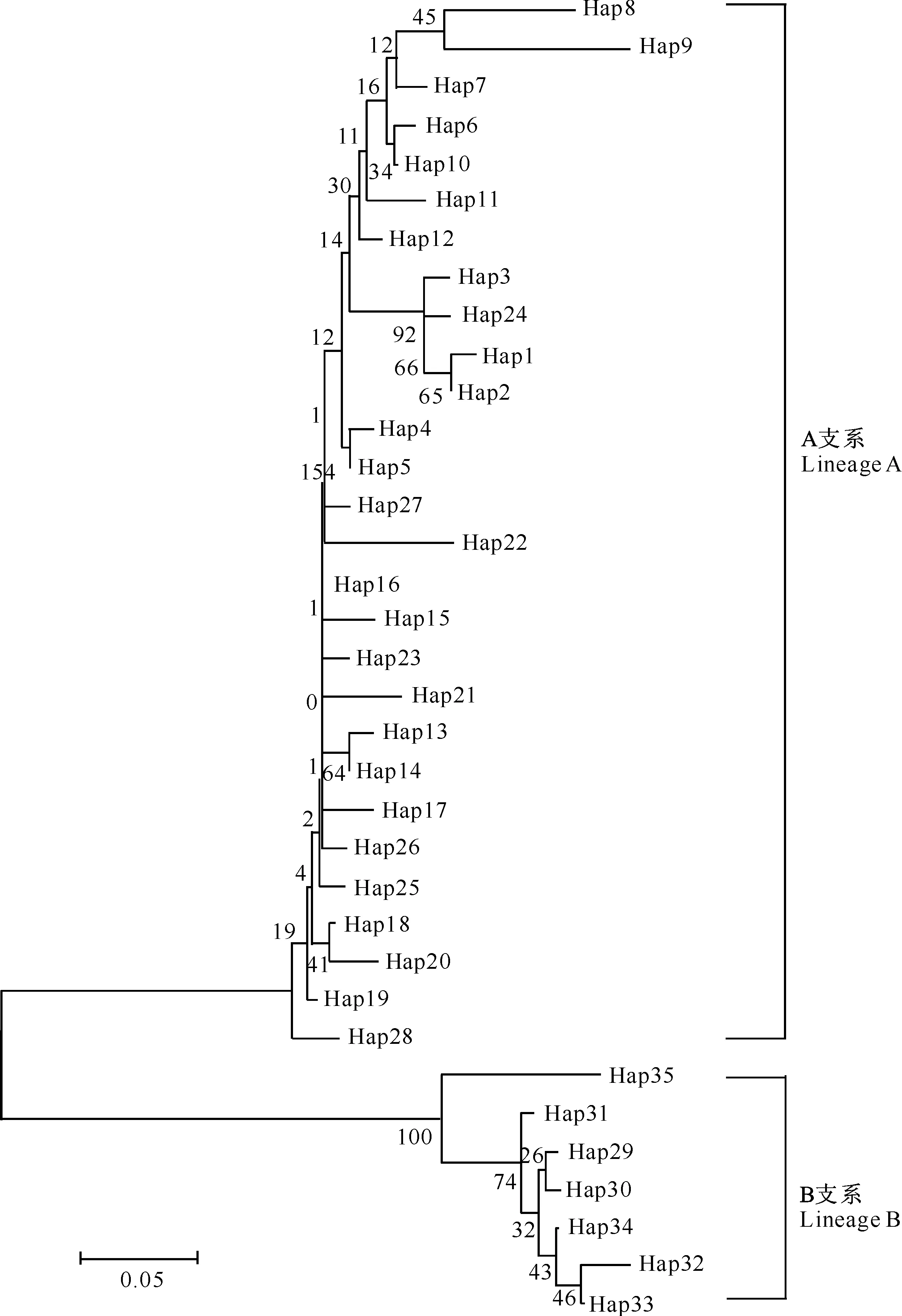
图 2 35个安徽东流水牛线粒体DNA D-Loop区单倍型的N-J系统发生树
3 讨 论
本研究通过分析安徽东流水牛mtDNA D-Loop区核苷酸变异,发现其转换中AG为 2.62% (24/915),TC为3.93%(36/915),颠换为1.42% (13/915),与Kierstein等[10]在沼泽型水牛和江河型水牛线粒体D-Loop区多态性分析中发现的3个颠换相比,东流水牛的遗传变异率较高。本研究发现,东流水牛具有2个母系来源(即A和B支系),研究结果与Lei等[3-4]所发现的中国水牛群体2个母系遗传起源的观点一致。对中国22个地方水牛群体进行遗传多样性分析,发现东流水牛的单倍型多样性(0.944 00±0.023 00)较西林水牛(0.963 00±0.028 00)和德昌水牛(0.970 00±0.044 00)稍低,比其他水牛群体(群体数量>10)均高,说明东流水牛的遗传多样性丰富。而且,尽管A支系为东流水牛的主要支系,B为次要支系,但B支系的单倍型变异程度(87.5%)比支系A(60.9%)高,且与其他地方品种牛研究结果一致,说明在水牛群体A支系中基因交流较为频繁。本研究通过对东流水牛mtDNA D-Loop区全序列的测定分析,发现其具有丰富的遗传多样性,且具有2个母系起源。同时,22个地方水牛群体进化网络显示,支系A呈现以单倍型Hap55为中心向外扩展的星状结构,b1和b2支系也具有星状结构的特征,预示着中国水牛经历了群体的扩张。

Ⅰ.沼泽型水牛群体 Swamp buffalo;Ⅱ.江河型水牛群体 River buffalo
图 3 180个中国地方水牛线粒体DNA D-Loop区单倍型的N-J系统发生树
Fig.3 Neighbor-joining phylogenetic tree of 180 Chinese buffaloes mtDNA D-Loop haplotypes

圆圈代表单倍型,其大小代表单倍型出现的相对频率,连线表示进化距离
图 4 中国沼泽型水牛线粒体DNA D-Loop单倍型间的media-joining进化网络图
Fig.4 Media-joining network showing genetic relationships among mtDNA D-Loop halotypes of Chinese swamp buffaloes
目前关于沼泽型水牛的起源问题,主要有2种观点,Lau等[15]通过分析研究亚洲水牛线粒体D-Loop区和细胞色素b基因(Cytb),发现亚洲水牛可能起源于一个类沼泽型野水牛,可能是现存的亚洲野水牛;提出沼泽型水牛和江河型水牛约在28 000到87 000年前分化形成;并假设亚洲野水牛起源于东南亚大陆,向北传播到中国,向西传播到印度次大陆,经过进化和驯化形成江河型水牛;沼泽型水牛在中国大陆驯化后,通过中国台湾和菲律宾传向婆罗洲和苏拉威西岛东部岛屿,或者通过东南亚大陆向南传向印度尼西亚西部群岛。Kierstein等[10]通过分析卡拉保水牛(Carabao)、摩拉水牛(Murrah)、贾法拉巴迪水牛(Jafarabadi)和地中海水牛(Mediterranean)线粒体DNA D-Loop区,提出沼泽型水牛和江河型水牛可能来自于印度次大陆的同一个驯化事件。然而,由于Kierstein等[10]和Lau等[15]在分析亚洲水牛的起源进化时缺乏中国水牛的数据资料,其结论难免有失偏颇。Lei等[4]通过分析中国7个水牛群体和东南亚水牛线粒体DNA D-Loop区序列,发现沼泽型水牛可能是在中国单独驯化而成。Yue等[9]通过对中国22个地方水牛品种线粒体DNA D-Loop区序列进行分析,证明中国西南地区是本地水牛的发源地,证明A支系水牛是在中国西南地区驯化而成的。Kumar等[16-17]通过对印度江河型水牛线粒体DNA的分析,推测江河型水牛可能是在印度西部驯化而成的。Mishra等[18]通过分析印度东北部、中国、地中海和东南亚水牛mtDNA,证明部分水牛在印度东北部地区驯化形成,而后传播到东南亚。总之,要彻底探明中国水牛的起源进化关系,尚需进一步深入研究。
[1]邱 怀.中国牛品种志 [M].上海:上海科技出版社,1986.
Qiu H.The Chinese cattle breed [M].Shanghai:Shanghai Scientific and Technical Publishers,1986.
[2]贾玉堂,赵拴平,徐 磊,等.东流水牛品种资源保护与开发利用对策 [J].中国草食动物科学,2014,34(1):66-69.
Jia Y T,Zhao S P,Xu L,et al.Preservation and utilization of Dongliu buffalo breed resources [J].China Herbivores Science,2014,34(1):66-69.
[3]Lei C Z,Zhang W,Chen H,et al.Two maternal lineages revealed by mtDNA D-Loop sequences in Chinese native water buffaloes (Bubalusbubalis) [J].Asian-Austral J Anim Sci,2007,20(4):471-476.
[4]Lei C Z,Zhang W,Chen H,et al.Independent maternal origin of Chinese swamp buffalo (Bubalusbubalis) [J].Anim Genet,2007,38(2):97-102.
[5]亐开兴,和占星,苗永旺,等.滇东南水牛mtDNA控制区遗传多样性及其系统地位分析 [J].西北农林科技大学学报(自然科学版),2011,39(9):73-80.
Qu K X,He Z X,Miao Y W,et al.Genetic diversity and phylogenetic status of mitochondrial DNA control region in Diandongnan buffalo [J].Journal of Northwest A&F University(Nat Sci Ed),2011,39(9):73-80.
[6]张自芳,刘丽仙,钱林东,等.滇东南水牛线粒体DNA 控制区遗传多样性分析 [J].云南农业大学学报, 2012,27(1):49-54.
Zhang Z F,Liu L X,Qian L D,et al.Characterizing genetic diversity of Diandongnan buffalo using mtDNA D-Loop sequences [J].Journal of Yunnan Agricultural University,2012,27(1):49-54.
[7]张瑞光,梁建平,陈 云,等.福安水牛线粒体DNAD-Loop区遗传多样性分析 [J].云南农业大学学报,2010,25(4):525-529.
Zhang R G,Liang J P,Chen Y,et al.Genetic diversity on mtDNA D-Loop region of Fu’an buffalo [J].Journal of Yunnan Agricultural University,2010,25(4):525-529.
[8]卢永芳,蔡治华,李 强,等.江淮水牛线粒体D-Loop区和Cytb基因遗传多态性及系统发育分析 [J].畜牧兽医学报,2012,43(5):701-707.
Lu Y F,Cai Z H,Li Q,et al.Genetic variation and phylogeney of Jianghuai buffalo based on mitochondrial DNA D-Loop andCytbgene [J].Acta Veterinaria et Zootechinica Sinica,2012,43(5):701-707.
[9]Yue X P,Li R,Xie W M,et al.Phylogeography and domestication of Chinese swamp buffalo [J].PLoS One,2013,8(2):e56552.
[10]Kierstein G,Vallinoto M,Silva A,et al.Analysis of mitochondrial D-Loop region casts new light on domestic water buffalo (Bubalusbubalis) phylogeny [J].Molecular Phylogenetics and Evolution,2004,30(2):308-324.
[11]Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods [J].Molecular Biology and Evolution,2011,28(10):2731-2739.
[12]Librado P,Rozas J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data [J].Bioinformatics,2009,25(11):1451-1452.
[13]Bandelt H J,Forster P,Rohl A.Median-joining networks for inferring intraspecific phylogenies [J].Molecular Biology and Evolution,1999,16(1):37-48.
[14]Posada D,Crandall K A.Intraspecific gene genealogies:trees grafting into networks [J].Trends Ecol Evol,2001,16(1):37-45.
[15]Lau C H,Drinkwater R D,Yusoff K,et al.Genetic diversity of Asian water buffalo (Bubalusbubalis):mitochondrial DNA D-Loop and cytochrome b sequence variation [J].Anim Genet,1998,29(4):253-264.
[16]Kumar S,Nagarajan M,Sandhu J S,et al.Phylogeography and domestication of Indian river buffalo [J].BMC Evolutionary Biology,2007,186:1471-2148.
[17]Kumar S,Nagarajan M,Sandhu J S,et al.Mitochondrial DNA analyses of Indian water buffalo support a disinct genetic origin of river and swamp buffalo [J].Animal Genetics,2007,38:227-232.
[18]Mishra B P,Dubey P K,Prakash B,et al.Genetic analysis of river,swamp and hybrid buffaloes of north-east India throw new light on phylogeography of water buffalo (Bubalusbubalis) [J].J Anim Breed Genet,2015,132(6):454-466.
Genetic variation and phylogeny of Dongliu Buffalo based on mitochondrial DNA D-Loop
ZHAO Shuanping,JIA Yutang,XU Lei,RUAN Yongming
(Institute of Animal Husbandry and Veterinary Research,Anhui Academy of Agricultural Sciences,Hefei,Anhui 230031,China)
【Objective】 This study investigated molecular genetic characteristics of Dongliu Buffalo from Anhui to provide scientific basis for further study of Chinese Buffalo.【Method】 A total of 31 Dongliu buffaloes from Anhui were sampled and their mtDNA D-Loop sequences were obtained,which were analyzed together with the published D-Loop sequences of 471 Chinese buffaloes in GenBank.The nucleotide diversity and haplotype diversity were found using DnaSP 5.1,the Neighbor-Joining tree were constructed with MEGA 5.10,and the media-joining network were constructed with Network 4.60.【Result】 A total of 163 nucleotide polymorphic sites and 180 haplotypes were identified in 22 Chinese buffaloes,and only 70 nucleotide polymorphic sites and 35 haplotypes were identified in Dongliu population.The estimated overall nucleotide diversity and haplotype diversity were (0.018 23±0.002 21) and (0.877 40±0.013 80),respectively.The genetic variability of Dongliu buffalo was similar with those of other Chinese buffalo populations,with nucleotide diversity of (0.013 31±0.002 08) and haplotype diversity of (0.944 00±0.023 00).The Neighbor-Joining tree and the media-joining network indicated that the mtDNA were divided into two lineages,the A and B lines of swamp buffaloes,and the B line could be further divided into sub-lineages of b1 and b2.The Dongliu buffaloes were distributed into A and B lines.【Conclusion】 The mitochondrial genetic diversity in Dongliu buffalo was rich,and originated from two maternal sources.
buffalo;mitochondrial DNA;genetic diversity;haplotype
时间:2016-10-20 16:36
10.13207/j.cnki.jnwafu.2016.12.004
2015-07-22
国家肉牛牦牛产业技术体系项目-合肥综合试验站(CARS-38);安徽省农业科学院院长青年创新基金项目(14B0428)
赵拴平(1984-),女,山西长治人,助理研究员,博士,主要从事肉牛分子育种工作。E-mail:zhaoshuanping@163.com
贾玉堂(1962-),男,安徽阜阳人,副研究员,主要从事草食动物遗传育种工作。E-mail:yutang2018@163.com
S823.8+32
A
1671-9387(2016)12-0021-07
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20161020.1636.008.html
——以云南墨江自治县为例

