香蕉枯萎病菌SIX2基因序列及特异性分析
杨腊英, 郭立佳, 汪 军, 刘 磊,梁昌聪, 王国芬, 陈平亚, 黄俊生
(中国热带农业科学院环境与植物保护研究所, 农业部热带农林有害生物入侵检测与控制重点实验室, 海口 571101)
香蕉枯萎病菌SIX2基因序列及特异性分析
杨腊英, 郭立佳, 汪 军, 刘 磊,梁昌聪, 王国芬, 陈平亚, 黄俊生*
(中国热带农业科学院环境与植物保护研究所, 农业部热带农林有害生物入侵检测与控制重点实验室, 海口 571101)
依据侵染的香蕉品种范围不同,引起香蕉枯萎病的尖孢镰刀菌古巴专化型(Fusariumoxysporumf.sp.cubense,Foc)分为4个生理小种。Foc在寄主植物木质部分泌的SIX(secreted in xylem)蛋白可能与不同生理小种侵染的香蕉品种范围不同存在密切关系,找到 Foc 4号生理小种(Foc4)特有的SIX蛋白编码基因将有利于进一步分析Foc4寄主范围更广的原因,从而开展抗病育种工作。采用PCR方法比较分析了国内不同地理区域及来源于澳大利亚与南非的Foc1、Foc2、Foc3、Foc4共56株菌株中的SIX2基因,并以8个以上其他专化型或其他种或属的热带作物病原菌共39株菌株分析了Foc4SIX2基因序列的特异性,分析了SIX2基因的灵敏度及利用其检测感病植株。仅从供试的Foc4菌株基因组DNA中扩增出SIX2基因序列,检测的DNA灵敏度达5 pg/25 μL,并可用于检测感病的球茎组织。Foc4中特有的SIX2基因序列为特异性鉴定感病植株病原菌种类提供了快速分子检测技术,为明确该基因是否决定性影响Foc4对寄主差异性的选择研究提供了基础。
尖孢镰刀菌古巴专化型;SIX2; 寄主选择性; 分子检测
香蕉枯萎病在世界主要香蕉产区先后发生并造成了极为严重的危害,是目前困扰世界香蕉产业发展的一大难题。该病由土壤真菌尖孢镰刀菌古巴专化型(Fusariumoxysporumf.sp.cubense,简称Foc)引起。依据Foc对不同品种香蕉的侵染特性将其划分为4个生理小种(race):其中1号生理小种(Foc 1)危害基因型为AAB的‘Gros Michel’及栽培种;2号生理小种危害‘Bluggoe’蕉及基因型为ABB 的相关煮食蕉类;3号生理小种危害不是香蕉类的‘Heliconia’种类,因而被Ploetz等认定其不为Foc[1-2];4号生理小种(Foc 4)则危害‘Cavendish’及其他对1号与2号生理小种敏感的蕉类。Foc4号生理小种更进一步被分为热带4号生理小种(TR4)与亚热带4号生理小种(ST4),其中,TR4在热带与亚热带区域均能侵染‘Cavendish’蕉类,而ST4则仅能在亚热带区域侵染‘Cavendish’蕉类[3-4],且主要在植物暴露于非生物胁迫下发生危害,例如低温和水涝。在我国广东、广西、云南、海南、福建局部地区有危害粉蕉(MusaABB ‘Pisang Awak’)的1号生理小种(Foc1)和危害巴西蕉(MusaAAA ‘Giant Cavendish cv. Bax’)和粉蕉的4号生理小种(Foc4)。国内外尚无有效的控制该病的手段,使用抗病品种是控制香蕉枯萎病最根本的出路[5],而对相关致病基因的认识最终可能有助于发展抗香蕉枯萎病的香蕉品种[6]。
多种植物病原菌,包括细菌、真菌、卵菌、线虫,分泌一些蛋白质和其他分子到其寄主植物的不同细胞区间以调节植物的防御体系及使其寄生性定殖[7-9]。在许多情况下,真菌感染特定植物种类的能力取决于其基因组中编码寄主选择性的“毒力因子”,这些因子可用于区分菌株的致病与不致病能力[10]。尖孢镰刀菌在与寄主的相互作用中会分泌几个特定的富含半胱氨酸的小分子量蛋白(15.8 ~29.9 kD)进入木质部中启动致病力,被称为SIX(secreted in xylem)蛋白。近来更多的对尖孢镰刀菌番茄专化型(F.oxysporumf.sp.lycopersici,简称Fol)的相关研究,揭示了SIX基因在Fol与番茄互作系统中的致病作用,到目前为止,通过反向遗传学等方法鉴定了Fol中的SIX1~SIX14共14个SIX基因[11-18]。
Foc侵入植株后,需定殖在寄主植物的木质部,不同生理小种侵染的香蕉品种存在的差异性与Foc在寄主植物木质部的成功定殖与否可能存在密切关系。因此Foc菌株亦可能启动相似基因表达分泌相关蛋白来决定Foc在寄主木质部的定殖。Meldrum等[19]从不同地理来源的Foc 4小种中分别克隆鉴定了SIX1、SIX7、SIX8基因,而测试的Foc1与Foc2中仅发现SIX1基因。郭立佳等[20]研究发现香蕉枯萎病菌4号生理小种(Foc4)中存在SIX1、SIX2、SIX6、SIX8基因,亚热带4号生理小种中存在SIX7基因。笔者所在实验室通过比较完成的Foc1菌株N2与Foc4菌株B2的全基因组测序发现Foc4菌株中存在特异性的SIX2基因[21],为证实SIX2基因为Foc4中特有,继而明确该基因是否决定Foc4对寄主的差异性选择,采用PCR方法比较分析了来自国内不同地理区域及来源于澳大利亚与南非的不同Foc1与Foc4菌株中的SIX2基因,并以多个其他专化型或其他病原菌属的菌株验证了该基因的序列特异性,同时为分析应用该基因开展田间病株的快速检测的可行性,分析了设计的基因引物序列的灵敏度,对病株不同染病部位开展了检测分析。
1 材料与方法
1.1 材料
1.1.1 试验材料
共93株测试菌株,其中来源于澳大利亚的BW1(Foc1)、Race2(Foc2)、Race3(Foc3)、BW4(ST4)以及2株非病原尖孢镰刀菌AB-11-5-2、AB-2-8的基因组DNA由广东省东莞市香蕉蔬菜研究所吕顺研究员赠送;来源于南非的race1(Foc1)、TR4(Foc4)、 STR4(Foc ST4)共3株由广东省农业科学院果树研究所李春雨研究员赠送;国内不同区域的2株Foc1(菌株编号:Foc1 c2、Foc1 e2)和6株Foc4(菌株编号:YF-10-12、YF-10-43、HBW 12、12、NX-27、PB3-15)菌株及5株尖孢镰刀菌其他专化型菌株苦瓜专化型F.oxysporumf.sp.momordicae(Fom)菌株FO kugua、冬瓜专化型F.oxysporumf.sp.benincasae(Fob)菌株FO donggua、黄瓜专化型F.oxysporumf.sp.cucumerinum(Fou)菌株FO huanggua、节瓜专化型F.oxysporumf.sp.chiehqua(Foh)菌株FO jiegua、甜瓜专化型F.oxysporumf.sp.melonis(Foe)菌株FO tiangua由华南农业大学王振中教授赠送;甘蓝专化型F.oxysporumf.sp.conglutinans(Foco)菌株Fo、FGL-12-48、FGL-13-1、FGL-13-8、FCH-12-9共5株由中国农业科学院蔬菜花卉研究所杨宇红副研究员惠赠;西瓜专化型F.oxysporumf.sp.niveum(Fon) 菌株22、75、624、Fon-01共4株由河北省农林科学院孔令晓研究员惠赠;番茄专化型F.oxysporumf.sp.lycopersici(Fol) oxy-1及3株未知F.oxysporum专化型菌株oxy-2、oxy-3、oxy-4由北京市农林科学院蔬菜研究中心赠送;2株串珠镰刀菌F.moniliforme(菌株编号:FJAT-829、FJAT-866)、2株茄病镰刀菌F.solani(菌株编号:FJAT-3752、FJAT-3756)及1株桃褐腐病菌Monilinialaxa(菌株编号:FJAT-772)由福建省农业科学院刘波研究员赠送;1株橡胶树流胶病病原菌F.solani(菌株编号:XJLJ4)及13株非镰刀菌菌株ND-12、ND-120(剑麻斑马纹病病菌Phytophthoranicotianae)、菌株PN006(橡胶树褐根病病菌Phellinusnoxius)、菌株CH008(柱花草炭疽病病菌Colletotrichumgloeosporioides)、菌株RC178(橡胶树炭疽病病菌C.acutatum)、菌株HJ-2、HJ-10(剑麻茎腐病病菌Aspergillusniger)、菌株Baifen(橡胶树白粉病病菌Oidiumheveae)、菌株HHNBT01、HHNTC01(橡胶棒孢霉落叶病病菌Corynesporacassiicola)、菌株mFQ-2(香蕉黑条叶斑病病菌Mycosphaerellafijiensis)、菌株Wei 1、MYD3(香蕉叶斑病菌Cercosporasp.)的基因组DNA分别由笔者所在单位的特色蔬菜花卉病害与线虫课题组、特色热作病害课题组、橡胶树病害课题组、热带果树病害课题组赠送;其他7株Foc1(菌株编号:N2、BXFZ-1 b-1、BXFZ-2 a-2、40#①、40#②、42#①、11#②)与32株Foc4菌株(菌株编号:B2、3#①、3#②、5#①、 5#②、6#①、6#②、7#①、7#②、9#①、9#②、34#①、 34#②、36#①、36#②、37#①、37#②、QWXZ b-2、NXXZ、NBXZ a、DTXZ-1 a-1、DTXZ-2W、DTXZ-3W、LDXZ a、LGXD-5W、LGXD-6W、ZJXD C、ZJB1 b1、ZJB2 b2、XNB2 a、XNB5 a、XNB6 a)由笔者所在实验室分离鉴定并保存。
1.1.2 试剂
rTaq酶、dNTPs、pMD18-T 载体购自TaKaRa公司,PCR Product Purification Kit购自上海博彩生物工程公司,引物由生工生物工程(上海)股份有限公司合成,其他常规化学生物试剂购自试剂销售公司。
1.2 方法
1.2.1 供试菌株的培养
分别将冷藏保存的供试菌种接种于PDA平板上活化,5 d后用打孔器在平板上打取菌饼(直径0.5 cm),挑取10块菌饼接入装有500 mL马铃薯葡萄糖培养液的1 L三角瓶中,于28℃、180 r/min振荡培养5 d,离心收集菌体用于提取DNA。
1.2.2 引物序列的设计
利用DNAStar version 5.0.221.0中的MegAlign比较分析测序的供试菌株的部分基因组序列和已在NCBI中登录的F.oxysporumf.sp.lycopersici(Fol)(AJ608703.3和GQ268949.1)与F.fujikuroi(HF679028.1)的SIX2基因序列,设计SIX2基因引物Foc-six2-f/Foc-six2-r。
各菌株内参基因的扩增应用rDNA核苷酸序列引物ITS1、ITS4;Foc4特异性引物Foc-1/Foc-2参照Lin等[22]的设计合成;香蕉组织内参基因Actin的扩增引物Actin2-F/Actin2-R参照Dita等[23]的设计合成。各个基因引物序列见表1。
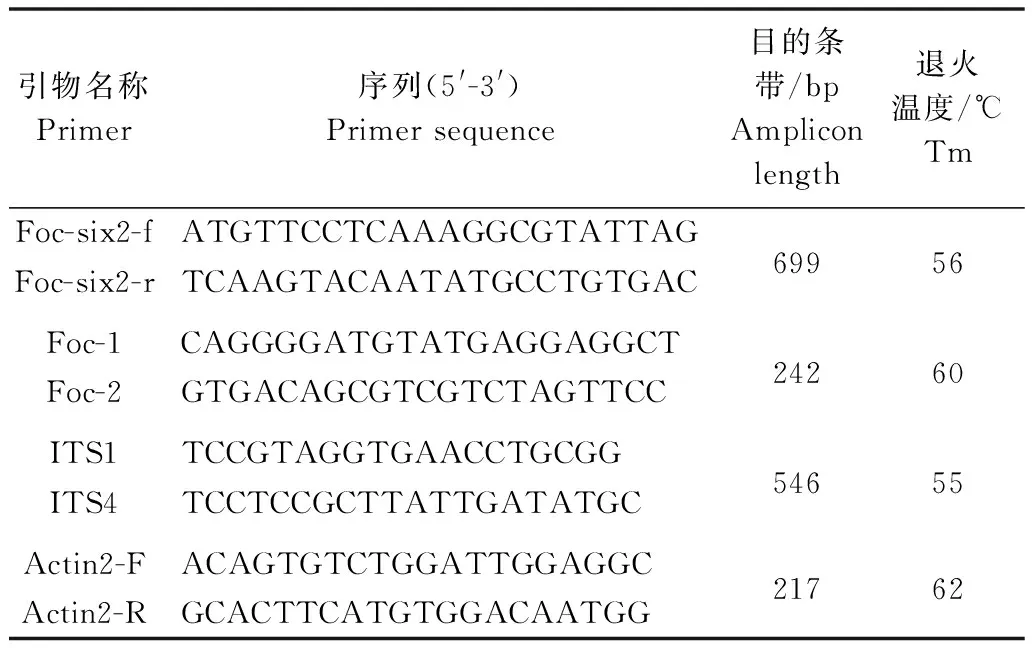
表1 本研究涉及的引物序列信息Table 1 Primer sequences used for PCR analysis in this study
1.2.3 菌株基因组DNA、RNA与感病植株基因组DNA的提取
以改进的SDS法提取各菌株基因组DNA[24];以接种B2菌株摇培过滤菌液后发病的香蕉植株为样本,分别抽提球茎组织与感病叶片叶柄总DNA,感病植株基因组DNA参照刘金福等[21]的方法提取。抽提所得DNA均用紫外分光光度计检测其纯度及浓度,以A260/A280的比值为1.8~2.0时最佳,未达到要求的均以酚氯仿再次抽提DNA,直至符合试验要求。调整DNA的浓度至约20 ng/μL,置于-20℃冰箱中保存备用。
菌体总RNA的提取采用异硫氰酸胍法。将经检测为完整的RNA反转录成完整的cDNA,具体步骤参照TaKaRa RNA PCR Kit说明书中反转录步骤进行。
1.2.4 SIX2基因对菌株BW4基因组DNA扩增的灵敏度分析
用无菌水对提取的BW4菌株的总DNA进行梯度稀释,得到浓度分别为50 ng/μL、5 ng/μL、500 pg/μL、50 pg/μL、5 pg/μL、500 fg/μL、50 fg/μL和5 fg/μL的BW4总DNA溶液。分别以各浓度的BW4总DNA溶液中的核酸为模板,以无菌水为对照,进行PCR扩增反应。
1.2.5 PCR反应体系及产物检测方法
反应体系为:TaKaRaTaqDNA聚合酶(5 U/μL) 0.2 μL,10×PCR buffer (Mg2+Plus) 2.5 μL,dNTPs (2.5 mmol/L) 2 μL,模板1 μL,20 μmol/L上下游引物各0.5 μL,加水补足至25 μL。
反应条件为: 94℃预变性5 min;94℃变性30 s,各引物序列对应退火温度退火30 s,72℃延伸45 s,35个循环;最后72℃延伸10 min。
反应产物经1%琼脂糖凝胶电泳后用凝胶成像系统(BIO-BAD Gel Doc XR)观察并拍照。
1.2.6 SIX2基因测序及序列分析
将PCR产物经1%的琼脂糖电泳检测后用PCR回收试剂盒回收,与pMD18-T载体连接,转化XL-1菌株挑选阳性克隆提取质粒酶切,送由生工生物工程(上海)股份有限公司测序。利用DNAStar v. 5.0.221.0预测SIX2蛋白序列,利用MegAlign比较分析本研究获得的B2菌株的SIX2基因序列与GenBank中登录的Fol分离物的SIX2基因序列(AJ608703.3和GQ268949.1)及F.fujikuroi(HF679028.1中3707948~3708646间碱基序列)所预测的SIX2蛋白序列差异性。
2 结果与分析
2.1 香蕉枯萎病菌SIX2基因扩增与序列比较分析
设计合成的Foc-six2-f/Foc-six2-r引物对仅能从测试的3株Foc4菌株基因组DNA与cDNA中PCR扩增出699 bp的目的条带,以Foc1 N2菌株基因组DNA与cDNA为模板则均不能扩增出目的条带(图1)。分别回收DNA与cDNA的PCR产物后连接至pMD18-T载体,比较分析SIX2基因的DNA与cDNA扩增序列,发现Foc4小种的SIX2基因无内含子。
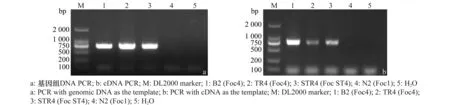
图1 以不同Foc生理小种的基因组DNA和cDNA扩增SIX2Fig.1 PCR products of SIX2 gene using genomic DNA and cDNA from four Foc isolates
DNAStar软件预测获得的B2SIX2基因编码的蛋白质序列由233个氨基酸组成,分子量约为25.5 kD,等电点为6.5左右。对该序列的信号肽预测结果表明Foc4菌株的SIX2蛋白质序列存在信号肽区域(信号肽预测网址:http:∥www.cbs.dtu.dk/services/SignalP/),剪切位置位于第20号与21号氨基酸之间。将B2菌株的 SIX2蛋白质序列与Fol及F.fujikuroi的SIX2蛋白质序列进行MegAlign比较分析,结果显示B2菌株的 SIX2与Fol及F.fujikuroi的SIX2氨基酸序列存在较大差别,但在第100、111、116、171、199、202、209、232这8个位置均存在保守的半胱氨酸残基。
2.2 尖孢镰刀菌古巴专化型不同生理小种及尖孢镰刀菌其他专化型SIX2基因扩增结果
PCR扩增结果表明(图2),供试菌株中,共40个Foc4菌株的基因组DNA中可扩增出目的条带,而从国内采集的粉蕉枯萎病病样上分离的Foc1菌株及来源于南非的Foc1菌株、澳大利亚的Foc1菌株、Foc2菌株、Foc3菌株共12个菌株的基因组DNA中无目的条带扩增出来,同时试验中供试的18株尖孢镰刀菌其他专化型菌株(苦瓜专化型、冬瓜专化型、黄瓜专化型、节瓜专化型、甜瓜专化型、番茄专化型、甘蓝专化型、西瓜专化型、3株未知专化型菌)的基因组DNA中亦没有扩增出目的条带。结果表明设计的引物对尖孢镰刀菌古巴专化型4号生理小种具有非常强的特异性,同时亦再次证实尖孢镰刀菌古巴专化型4号小种(Foc)中的SIX2基因序列与番茄专化型(Fol)中的SIX2基因序列存在较大的差异性。
利用Foc4的特异引物Foc-1/Foc-2对供试的71个菌株进行验证,结果表明,该引物对仅能从各Foc4菌株基因组DNA中扩增出217 bp的目的条带(图3),与SIX2基因引物对各菌株的扩增结果一致。

图2 尖孢镰刀菌古巴专化型不同生理小种及尖孢镰刀菌其他专化型SIX2基因产物Fig.2 Amplification products of SIX2 gene from Foc isolates belonging to different races and other formae specialis of Fusarium oxysporum
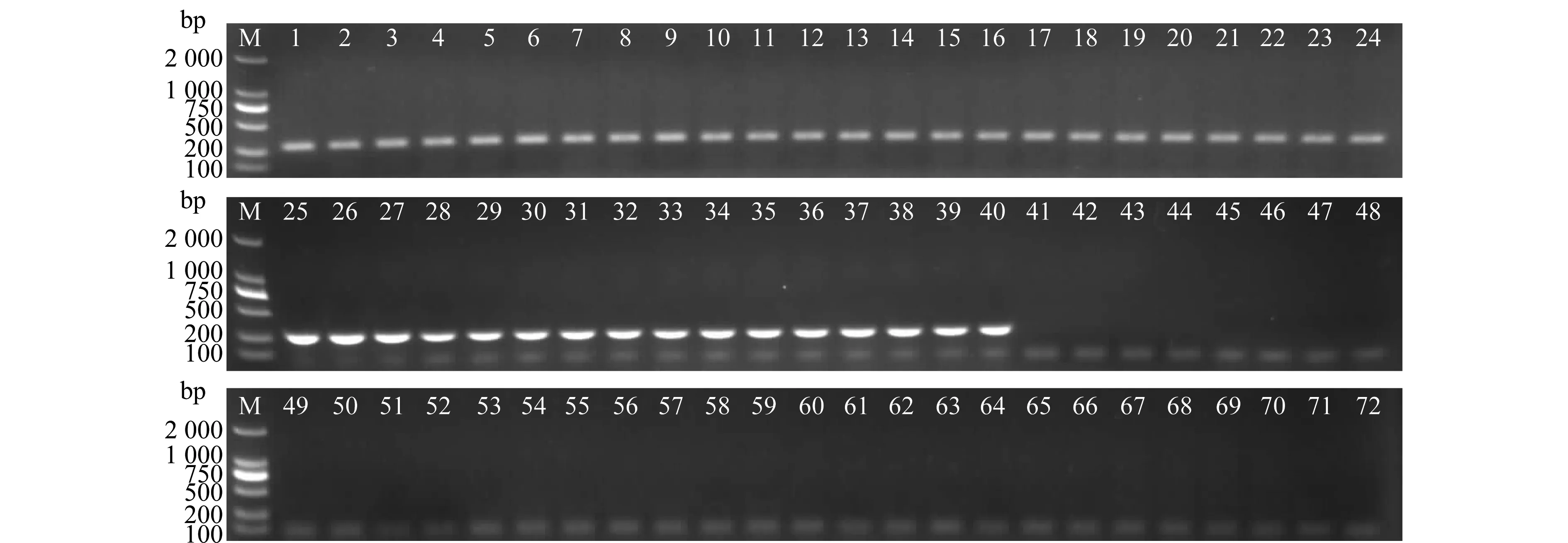
图3 引物Foc-1/Foc-2的特异性验证Fig.3 Specificity verification for primer Foc-1/Foc-2
2.3 镰刀菌属其他种及其他属菌株中SIX2基因扩增结果
验证分析了设计的Foc4SIX2基因引物Foc-1/Foc-2的特异性,结果(图4)表明,除阳性对照,属于Foc4小种的B2菌株基因组中扩增出目的条带外,阴性对照属于Foc1小种的N2菌株及供试的其他21个菌株基因组DNA中均没有扩增出目的条带。
2.4 SIX2基因引物Foc-six2-f/ Foc-six2-r的灵敏度
以菌株BW4基因组DNA 为模板测定SIX2基因引物Foc-six2-f/ Foc-six2-r的灵敏度,结果(图5)显示,50 ng/25μL(泳道1)、5 ng/25μL(泳道2)、500 pg/25μL(泳道3)、50 pg/25μL(泳道4)、5 pg/25μL(泳道5)均可以扩增到699 bp的目的条带,500 fg/25μL(泳道6)、50 fg/25μL(泳道7)、5 fg/25μL(泳道8)和阴性对照(泳道9)均不能得到任何条带。

图4 镰刀菌属不同种及其他属菌株的SIX2基因PCR扩增结果Fig.4 Amplification products of SIX2 gene from different species of Fusarium and other genus isolates
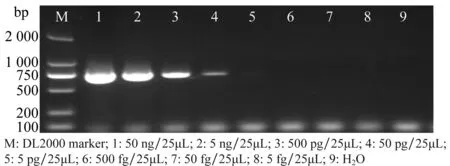
图5 SIX2基因引物Foc-six2-f/ Foc-six2-r的灵敏度Fig.5 Sensitivity of specific primers Foc-six2-f/ Foc-six2-r for Foc4 SIX2 gene
2.5 从发病植株检测香蕉枯萎病菌
分别以发病香蕉植株球茎与黄化叶片的叶柄总DNA为模板,以清水和4号生理小种菌株DNA做对照,利用设计的Foc4SIX2基因引物对香蕉组织内参引物Actin2-F/Actin2-R[20]进行多重PCR检测。结果(图6)显示,提取的香蕉球茎组织与黄化叶片叶柄的DNA均能扩增出217 bp的Actin2目的条带,香蕉感染B2的球茎组织DNA及4号生理小种阳性对照得到699 bp的SIX2基因条带,而黄化的香蕉叶片的叶柄DNA无SIX2基因条带扩增产物。由此可判断,Foc4SIX2基因引物对可用于田间所采集的感病香蕉的快速鉴定,也表明所建立的多重PCR检测体系适用于对罹病的香蕉球茎组织进行检测。
3 讨论与结论
Meldrum等[21]设计的引物不能从香蕉枯萎病菌中扩增出目的条带,笔者通过香蕉枯萎病菌全基因组序列比较分析后设计的扩增SIX2基因的引物可以从供试的各个Foc4菌株中扩增出699 bp的目条带,这是由于Meldrum等设计的引物序列处于差异性较大的碱基区域所致。B2 菌株SIX2氨基酸序列与Fol及F.fujikuroi的SIX2氨基酸序列存在较大差别,但在相同位置均存在8个保守的半胱氨酸残基, Chakrabarti等[26]推测尖镰孢西瓜专化型不同生理小种SIX6 蛋白之间存在的差异氨基酸可能决定了不同生理小种的致病力,笔者推测B2 SIX2蛋白序列中表现出差异的氨基酸亦有可能有致病因子的功能,待后期通过利用同源重组转化方法获得SIX2基因敲除的突变菌株后分析其致病力与生理生化指标变化情况等证实。

图6 利用SIX2基因对感病植株检测结果Fig.6 Detection of SIX2 gene from diseased plants
鉴于尖孢镰刀菌基因横向转移的证据[15],基于关键致病相关基因的病原菌分子诊断比基于系统发育相关基因更可靠。Fraser-Smith等[27]以SIX8基因序列为基础设计了区分Foc4小种及亚热带Foc4小种的特异性引物。本研究中利用SIX2基因序列引物对仅能从Foc4中扩增出特异性的目的条带,与已有的Lin等[22]的用于区分Foc4的引物Foc-1/Foc-2的扩增结果一致。同时本研究中设计的FocSIX2基因引物序列的灵敏度可达5 pg/25μL反应体系,可以应用于快速检测鉴定田间发病香蕉枯萎病植株病样及种植地土壤中病原菌的种类,为及时掌握香蕉枯萎病发生情况并采取有效的田间防控措施提供有力的技术支持,尤其可保障各组培苗公司出厂的香蕉种苗健康无菌,从而防止病原菌随种苗的远距离传播。
[1] Ploetz R C, Thomas J E, Slabaugh W R.Diseases of banana and plantain [M]∥Ploetz R C,ed. Diseases of tropical fruit crops. CABI Publishing, Wallingford, 2003: 73-134.
[2] Ploetz R C.Panama disease: an old nemesis rears its ugly head. Part 2.The Cavendish era and beyond [J].Plant Health Progress, 2006(206):1-17.
[3] Ploetz R C, Pegg K G.Fusarium wilt [M]∥Jones D R, ed. Diseases of banana, abacá and enset. CABI Publishing, Wallingford, 2000:143-159.
[4] Buddenhagen I W.Understanding strain diversity inFusariumoxysporumf.sp.cubenseand history of introduction of ‘tropical race 4’ to better manage banana production [M]∥Jones D, Van den Bergh I, eds. Proceedings of the International Symposium on Recent Advances in Banana Crop Protection for Sustainable Production and Improved Livelihoods, White River, South Africa. ISHS Acta Horticulturae, 2009, 828:193-204.
[5] Michielse C B, Rep M.Pathogen profile update:Fusariumoxysporum[J]. Molecular Plant Pathology, 2009, 10(3): 311-324.
[6] Fourie G, Steenkamp E T, Ploetz R C, et al. Current status of the taxonomic position ofFusariumoxysporumformaespecialiscubensewithin theFusariumoxysporumcomplex [J]. Infection, Genetics and Evolution, 2011, 11: 533-542.
[7] Block A, Li Guangyong, Fu Zhengqing, et al. Phytopathogen type III effector weaponry and their plant targets [J]. Current Opinion in Plant Biology, 2008, 11(4): 396-403.
[8] Davis E L, Hussey R S, Mitchum M G, et al. Parasitism proteins in nematode-plant interactions[J]. Current Opinion in Plant Biology, 2008, 11(4): 360-366.
[9] Misas-Villamil J C, van Der Hoorn R A.Enzyme-inhibitor interactions at the plant-pathogen interface [J]. Current Opinion in Plant Biology, 2008, 11(4): 380-388.
[10]Lievens B, Rep M, Thomma B P H J.Recent developments in the molecular discrimination of formae speciales ofFusariumoxysporum[J]. Pest Management Science, 2008, 64(8): 781-788.
[11]Rep M, van Der Does H C, Meijer M, et al. A small, cysteine-rich protein secreted byFusariumoxysporumduring colonization of xylem vessels is required for I-3-mediated resistance in tomato [J]. Molecular Microbiology, 2004, 53(5): 1373-1383.
[12]Houterman P M, Speijer D, Dekker H L, et al. The mixed xylem sap proteome ofFusariumoxysporum-infected tomato plants [J]. Molecular Plant Pathology, 2007, 8(2): 215-221.
[13]van Der Does H C, Rep M.Virulence genes and the evolution of host specificity in plant-pathogenic fungi[J]. Molecular Plant-Microbe Interactions, 2007, 20(10): 1175-1182.
[14]Lievens B, Houterman P M, Rep M.Effector gene screening allows unambiguous identification ofFusariumoxysporumf.sp.lycopersiciraces and discrimination from other formae speciales [J]. FEMS Microbiology Letters, 2009, 300(2): 201-215.
[15]Ma Lijun, van Der Does H C, Borkovich K A, et al. Comparative genomics reveals mobile pathogenicity chromosomes inFusarium[J]. Nature, 2010, 464(7287): 367-373.
[16]Rep M, Kistler H C.The genomic organization of plant pathogenicity inFusariumspecies [J]. Current Opinion in Plant Biology, 2010, 13(4): 420-426.
[17]Takken F, Rep M.The arms race between tomato andFusariumoxysporum[J]. Molecular Plant Pathology, 2010, 11(2): 309-314.
[18]Schmidt S M, Houterman P M, Schreiver I, et al. MITEs in the promoters of effector genes allow prediction of novel virulence genes inFusariumoxysporum[J]. BMC Genomics, 2013, 14: 119.
[19]Meldrum R A, Fraser-Smith S, Tran-Nguyen L T T, et al. Presence of putative pathogenicity genes in isolates ofFusariumoxysporumf.sp.cubensefrom Australia[J]. Australasian Plant Pathology, 2012, 41(5): 551-557.
[20]郭立佳, 杨腊英, 彭军, 等. 香蕉枯萎病病原菌Six同源基因的鉴定[J]. 热带作物学报, 2013, 34(12):2391-2396.
[21]Guo Lijia, Han Lijuan, Yang Laying, et al. Genome and transcriptome analysis of the fungal pathogenFusariumoxysporumf.sp.cubensecausing banana vascular wilt disease [J]. PLoS ONE, 2014, 9(4): e95543.
[22]Lin Yinghong, Chang Jingyi, Liu E T, et al. Development of a molecular marker for specific detection ofFusariumoxysporumf.sp.cubenserace 4 [J]. European Journal of Plant Pathology, 2009, 123(3): 353-365.
[23]Dita M A, Waalwijk C, Buddenhagen I W, et al. A molecular diagnostic for tropical race 4 of the banana fusarium wilt pathogen [J]. Plant Pathology, 2010, 59(2): 348-357.
[24]杨腊英, 黄华平, 唐复润, 等. 香蕉炭疽菌rDNA ITS区的分子鉴定与检测[J]. 植物病理学报. 2006, 36(3): 219-225.
[25]刘金福, 潘东明, 代立春, 等. 香蕉RAPD反应体系的建立[J]. 中国农学通报, 2009, 25(16): 51-55.
[26]Chakrabarti A, Rep M, Wang B, et al. Variation in potential effector genes distinguishing Australian and non-Australian isolates of the cotton wilt pathogenFusariumoxysporumf.sp.vasinfectum[J]. Plant Pathology, 2011, 60 (2): 232-243.
[27]Fraser-Smith S, Czislowski E, Meldrum R A, et al. Sequence variation in the putative effector geneSIX8 facilitates molecular differentiation ofFusariumoxysporumf.sp.cubense[J]. Plant Pathology, 2014, 63(5): 1044-1052.
(责任编辑:田 喆)
Sequence and specificity analysis ofSIX2 gene ofFusariumoxysporumf.sp.cubense
Yang Laying, Guo Lijia, Wang Jun, Liu Lei, Liang Changcong, Wang Guofen, Chen Pingya, Huang Junsheng
(Environment and Plant Protection Institute, Chinese Academy of Tropical Agricultural Sciences,Key Laboratory of Pests Comprehensive Governance for Tropical Crops, Ministry of Agriculture, Haikou 571101,China)
Fusariumoxysporumf.sp.cubense(Foc), causal agent ofFusariumwilt of banana, has been divided into four races according to cultivar specificity. There may be a close relationship between the races infested different banana varieties and the proteins secreted in xylem (SIX) by Foc when it invaded into the host plant xylem. The specificSIXgene will be conductive to further analyzing the reasons for broader host range of Foc4 and helpful for the disease-resistant cultivar breeding. Using PCR and sequencing approach,SIX2 gene were analyzed in 56 Foc isolates collected from different geographic areas of Southern China, Australia and South Africa. The specificity and sensitivity of the primers were determined by using other thirty-nine isolates including eight otherF.oxysporumformae specialis, two non-pathogenF.oxysporumand other genus or species of some tropical plant pathogens. With the designed primers, the target band was detected only in the DNA of Foc4 isolates, and the sensitivity of the primers were 5 pg/25 μL and could be used to detect diseased bulb section. Therefore,SIX2 gene specific in Foc4 could be applied for rapid molecular identification of the pathogens in susceptible plants, which will provide a basis for further research on whether the gene is clearly a decisive influence on the host selection of Foc4.
Fusariumoxysporumf.sp.cubense;SIX2; host selection; molecular diagnosis
2015-09-29
2015-12-18
国家自然科学基金(31301629)
S 436.681
A
10.3969/j.issn.0529-1542.2016.05.020
致 谢: 感谢华南农业大学王振中教授、广东省农业科学院果树研究所李春雨研究员、北京市农林科学院蔬菜研究中心、中国农业科学院蔬菜花卉研究所杨宇红研究员、河北省农林科学院植保所孔令晓研究员、福建省农业科学院刘波研究员、广东东莞香蕉研究所吕顺研究员及作者所在单位特色热作病害课题组与橡胶树病害课题组及热带果树病害课题组同事们赠送菌株,基于他们的支持才能较全面地验证本试验的相关研究结果,特此表示真诚的谢意。
* 通信作者 E-mail:H888111@126.com

