DNA条形码技术在鲻科鱼类鉴定中的应用*
刘 璐, 孙典荣, 李纯厚, 韩志强, 高天翔, 沈康宁, 宋 娜**
(1.中国海洋大学水产学院,山东 青岛 266003; 2.中国水产科学研究院南海水产研究所,广东 广州 510300;3. 浙江海洋大学水产学院,浙江 舟山 316022; 4. 台湾海洋大学海洋中心,台湾 基隆 20224)
DNA条形码技术在鲻科鱼类鉴定中的应用*
刘 璐1, 孙典荣2, 李纯厚2, 韩志强3, 高天翔3, 沈康宁4, 宋 娜1**
(1.中国海洋大学水产学院,山东 青岛 266003; 2.中国水产科学研究院南海水产研究所,广东 广州 510300;3. 浙江海洋大学水产学院,浙江 舟山 316022; 4. 台湾海洋大学海洋中心,台湾 基隆 20224)
本研究选用DNA条形码的通用基因片段,采用特异性扩增测序及与GenBank已有序列结合分析的方法,进行了鲻科(Mugilidae)6属17种鱼类的COI基因片段的序列比较、分子树构建和系统进化研究。研究表明,在所得的17种鲻科鱼类共有的555bp COI基因片段中平均GC含量为46.9%。其中,第二密码子位点含量最高(49.8%~56.2%),平均54.9%;第一密码子变化范围最大(31.9%~48.6%),平均42.9%;第三密码子差别不显著(42.5%~43.4%),平均42.7%。依据Kimura-2-parameter模型(K2P),17种鲻科鱼类种内遗传距离的平均值为0.004 5,种间遗传距离为0.191,是种内遗传距离的42倍。在分子系统树上,所有物种均呈单系,5属为独立分支,只有隶属于梭属(Liza)的尖头梭(Lizatade)与莫鲻属(Moolgarda)聚类,表现出与传统形态学分类不一致的结果。本研究结果验证了线粒体COI基因作为DNA条形码对鲻科鱼类进行物种鉴定的有效性,可用于辅助探讨鲻科科下属、种分类阶元系统发育问题。
鲻科;COI基因;DNA条形码;物种鉴定;系统发育
海洋生物分类和物种快速鉴定是开展渔业生物多样性和渔业资源管理的基础和根本前提,也是海洋生物多样性保护研究的重要内容[1][2]。形态学鉴定作为一种传统的分类方法已经成功的描述了地球上近百万种生物,为物种的分类和鉴定奠定了良好的基础。但同时传统形态学分类存在着很多不足[3-4],而DNA条形码(DNA Barcoding)技术作为近年来发展迅速的一种物种鉴定的手段已经引起越来越多的关注,尤其是在保护生物学和生物多样性调查等领域[5],主要应用于鉴定已有物种和发现新种[6]。相对于传统的形态学生物鉴定,DNA条形码优势在于可以揭示隐存种,鉴定缺少形态数据(比如未成熟的、部分或损坏样本、胃含物)或者从形态方面不易被区分的物种[7],目前DNA条形码已经成功的应用于多种海洋生物及其他多种生物类群的鉴定[10-13]。Hebert等发现线粒体细胞色素氧化酶亚基 1基因(Cytochrome oxidase subunit I,COI)能够对动物界的物种进行有效的鉴定,并且在多数动物类群中,基因能被通用引物所扩增,且都存在显著的序列变异,所选用的区域片段长度在580 bp左右[14]。
鲻科(Mugilidae)隶属硬骨鱼纲(Osteichthyes)鲻形目(Mugiliformes),在热带、亚热带、温带海域均有分布,是世界上分布最广泛的经济鱼类之一,亦是许多国家和地区的重要渔业和增养殖对象。由于大多数鲻科鱼类的外部形态都极为相似(尤其是在幼鱼阶段),因此一些鲻科鱼类的种类鉴定尚存在一定困难,导致鲻科鱼类的分类及系统演化关系存在争议[15-18]。鲻科鱼类曾归属于鲈形目(Perciformes),现在已单独划分到鲻形目[19]。Thomson等通过详细的调查取样并且综合近年来相关研究成果,将鲻科鱼类定为14个属,64个有效种[20],而2014年Eschmeyer[15]等将鲻形目又重新划分为10个属31个种。但目前研究表明仍有一些属被认为是无效属, 此外还可能存在有大量的隐存种(沈康宁等未发表*源于沈康宁待出版的书籍)。因此,本项研究采集了4属9种鲻科鱼类,同时结合已发表的5属8种鲻科鱼类的COI基因序列,开展了利用DNA条形码对鲻科鱼类分类鉴定的研究。本研究系统地分析了6属17种鲻科鱼类的线粒体COI基因,通过比较该基因序列在种内和种间的差异,构建了分子系统树,一方面,检验COI基因作为鲻科鱼类DNA条形码的标准基因进行物种鉴别的有效性;另一方面,剖析COI基因在鲻科鱼类辅助系统进化研究中的应用潜力。
1 材料与方法
1.1 样品采集及DNA提取
本研究采集了梭属(Liza)、鲻属(Mugil)、莫鲻属(Moolgarda)鲻科鱼类共计45个个体(见表1),其中采自中国近海的鲻科鱼类物种分类鉴定参照陈大刚等的分类方法[21],样品保存于中国海洋大学渔业生态学实验室。另有采自巴基斯坦近海鲻科鱼类,参照NCBI(http://www.ncbi.nlm.nih.gov/books/NBK2550 1/)以及Fishbase(http://fishbase.sinica.edu.tw/search.php)进行物种鉴定,样品保存于巴基斯坦卡拉奇大学渔业实验室。以上样品均取背部肌肉保存于酒精中,以备DNA基因组提取。
采用酚-氯仿[22]抽提方法提取基因组DNA, 将乙醇沉淀后的基因组DNA用50 μL超纯水溶解,储存在-20℃冰箱中备用。
1.2 引物设计、PCR及序列测定
扩增线粒体DNA COI片段的引物参照文献[9]中通用引物设计,分别为FishF1:5’—TCAACCAACCACAAAGACATTGGCAC—3’;FishR1: 5’— TAGACTTCTGGGTGGCCAAAGAATCA—3’。PCR反应体系总体积25μL,包括10 mmol/L Tris,pH=8.3,50 mmol/L KCl,1.5 mmol/L MgCl2,dNTP 200 μmol/L,上、下游引物各200 nmol/L,Taq DNA聚合酶1.25U,DNA模版1 μL,最后加灭菌蒸馏水至25 μL。反应条件如下:94 ℃预变性5 min,然后进行40个循环,每个循环包括95 ℃变性60 s,50 ℃退火45 s,72 ℃延伸60 s,最后72 ℃延伸10 min。为了避免DNA的污染,以上反应均设阴性对照组。将2 μL PCR反应产物取出,使用1.5%的琼脂糖凝胶电泳检测(U=5V/cm),经试剂盒对目的片段进行回收纯化,将阳性PCR产物送北京英潍捷基生物公司进行双向测序。
1.3 基因序列信息下载
从Genbank下载5属8种27个个体的鲻科鱼类的COI基因序列,与本研究所检测的9种鲻科鱼类,共计6属17种72个个体的COI基因同源序列进行比对,以鲻形目魣科的油魣(Sphyraenapinguis)、斑条魣(Sphyraenajello)的同源序列作为外群进行分析。物种的COI基因及其相关信息见表1。
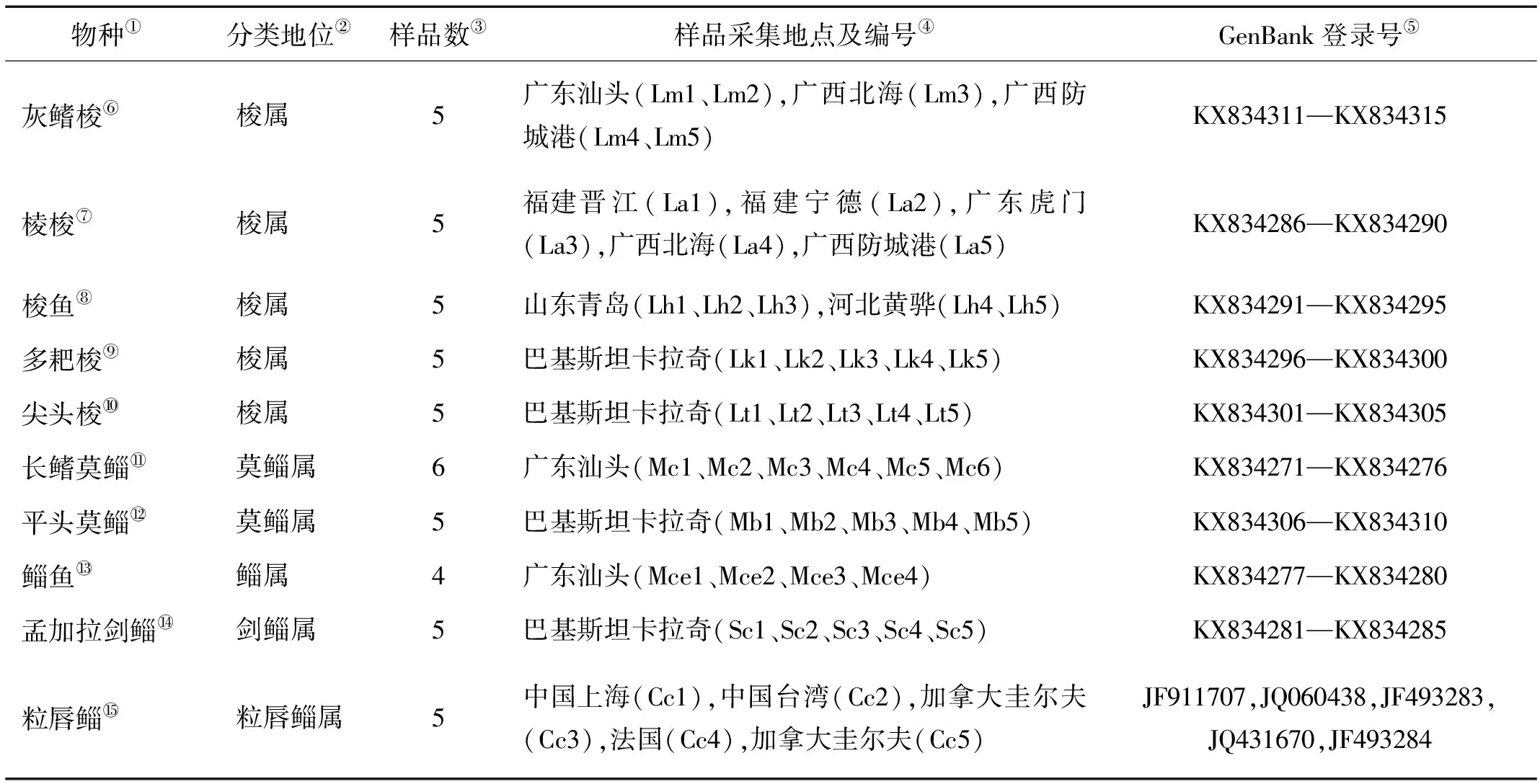
表1 所用的鲻科6属17种鱼类的COI基因以及外群相关信息
续表1
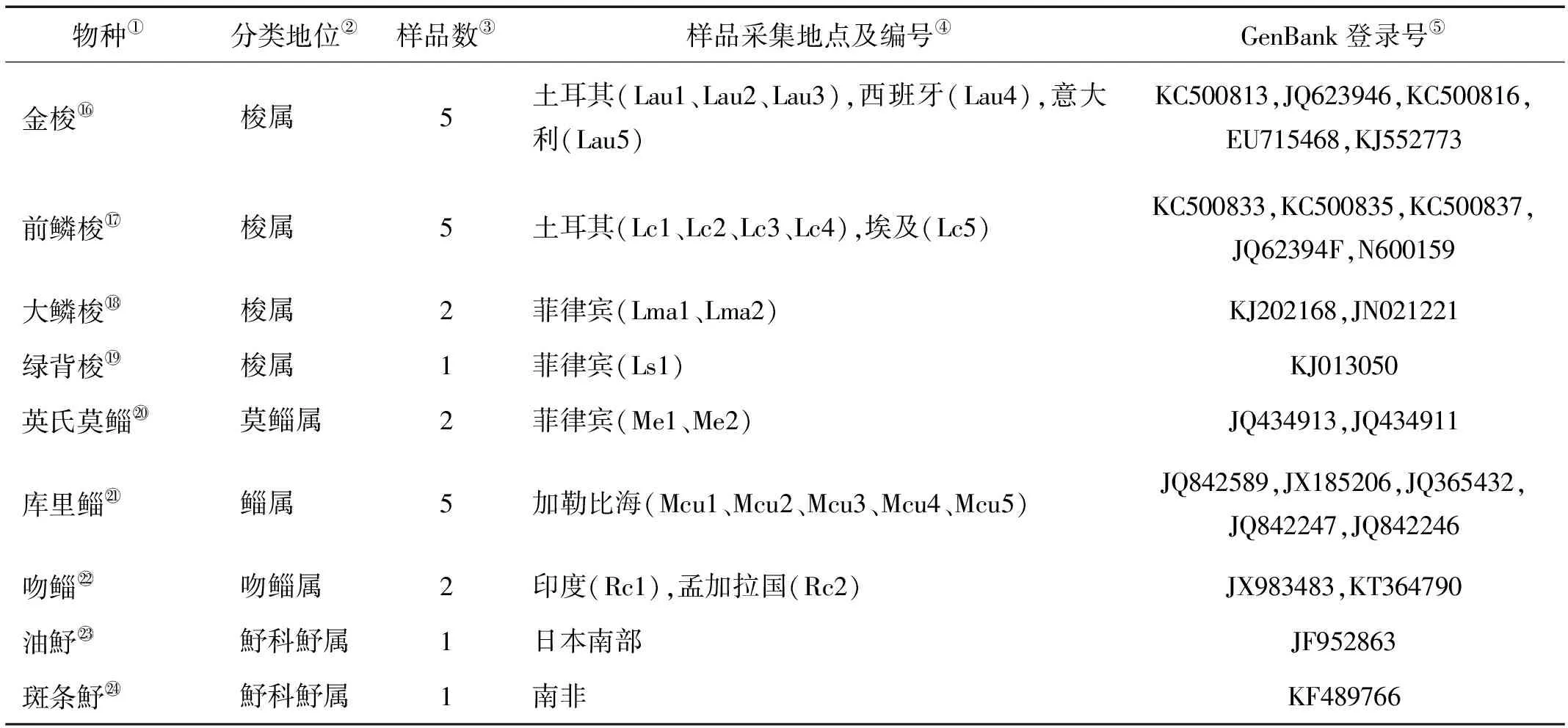
物种①分类地位②样品数③样品采集地点及编号④GenBank登录号⑤金梭梭属5土耳其(Lau1、Lau2、Lau3),西班牙(Lau4),意大利(Lau5)KC500813,JQ623946,KC500816,EU715468,KJ552773前鳞梭梭属5土耳其(Lc1、Lc2、Lc3、Lc4),埃及(Lc5)KC500833,KC500835,KC500837,JQ62394F,N600159大鳞梭梭属2菲律宾(Lma1、Lma2)KJ202168,JN021221绿背梭梭属1菲律宾(Ls1)KJ013050英氏莫鲻莫鲻属2菲律宾(Me1、Me2)JQ434913,JQ434911库里鲻鲻属5加勒比海(Mcu1、Mcu2、Mcu3、Mcu4、Mcu5)JQ842589,JX185206,JQ365432,JQ842247,JQ842246吻鲻吻鲻属2印度(Rc1),孟加拉国(Rc2)JX983483,KT364790油魣魣科魣属1日本南部JF952863斑条魣魣科魣属1南非KF489766
1.4 序列分析
所得测序结果通过NCBI(http://www.ncbi.nlm.nih.gov/)和BOLD(http://www.barcodinglife.org/)数据库进行相似性检索,确认实验所得序列的准确性。通过DNASTAR软件对测得的COI基因片段序列进行比对,并通过人工校正,结合GenBank数据库中下载的序列,使用MEGA 3.0计算所得序列的碱基组成、序列间的碱基变异频率和序列间的转换、颠换频率及其比率。通过PAUP v.4.0(Swofford, 2002)[23]软件基于邻接关系法(Neighbor-joining, NJ)、最大简约法(Maximum parsimony, MP)和最大似然法(Maximum likelihood, ML)构建系统发生树。通过MrBayes 3.0b4[24]软件基于贝叶斯推断法构建系统发生树。采用Modeltest v.3.06[25]根据AIC[26]准则对各序列片段的最适替换模型进行评估。NJ法采用1 000次自展分析检验各节点的置信度;MP法采用启发式搜索(Heuristic search)和TBR(Tree bisection reconnection)枝长交换法,重复1 000次;构建ML树时,在1 000次重复中选择似然率最大的拓扑结构作为最终树,采用1 000次无参数自展分析计算各节点的支持率[27]。
2 结果与分析
2.1 鲻科鱼类COI基因序列特征
本研究所用引物为FishF1和FishR1[14],共计扩增4属9种鲻科鱼类45条序列,以上序列均不存在序列长度上的变异,采用DNAStar系列软件对以上序列和已发表的鲻科鱼类COI同源序列进行比对分析,保留共有序列得到序列片段长度为555 bp,编码氨基酸185个。用MEGA 3.0软件计算72条序列(本研究得到扩增45条序列以及Genbank下载27条序列)的碱基组成,平均碱基组成为A:23.4%、T:29.7%、G:17.6%、C:29.3%,A+T含量(53.1%)高于G+C(46.9%)。针对鲻科鱼类COI片段的平均G+C含量为46.9%。其中,第二密码子位点含量最高54.9%,第一密码子为42.9%。第三密码子为42.7%(见表2)。
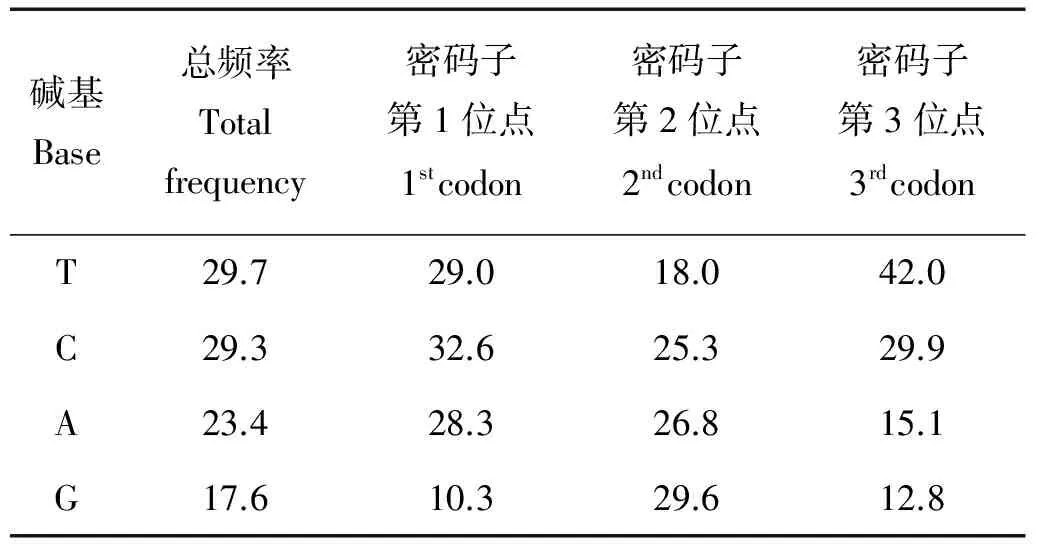
表2 鲻科17种鱼72个个体的COI基因部分序列中各碱基平均分布频率
所有个体序列核苷酸变异情况见表3。其中保守位点有343个,转换位点有59个,颠换位点有24个。其中第三密码子的保守位点数最多,有174个。第一密码子的转换位点和颠换位点数最多,分别为53和23个。
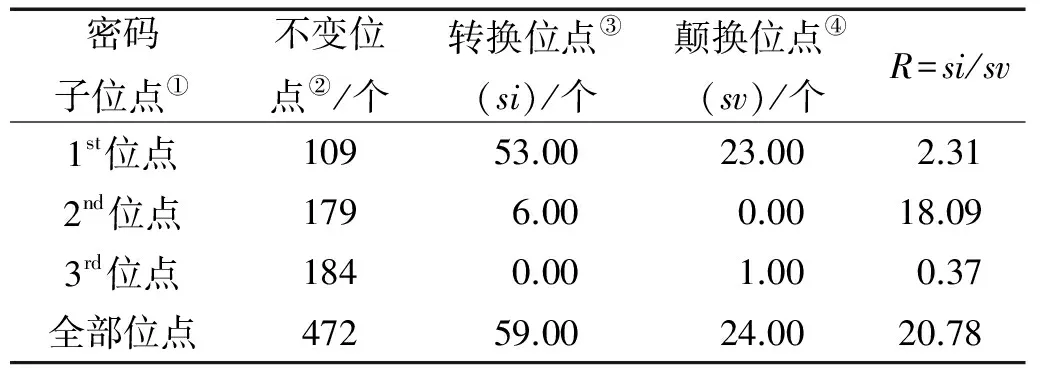
表3 鲻科17种鱼72个个体的COI基因部分序列3个密码子碱基变异情况
Note:①Codon sites;②Invariable sites;③Transition sites;④Transversion sites
如表4中所示,17种鲻科鱼类的COI基因片段平均GC含量为43.2%~49.0%,其中第二密码子位点的GC含量最高(49.8%~56.2%),第一密码子变化范围最大(31.9%~48.6%)。第三密码子GC含量差别不显著(42.5%~43.4%)。鲻鱼COI基因片段的GC含量最低(43.2%),多耙梭COI基因片段的GC含量最高(49.0%)。17种鲻科鱼类中吻鲻、长鳍莫鲻、鲻鱼等种内GC含量差异较大。
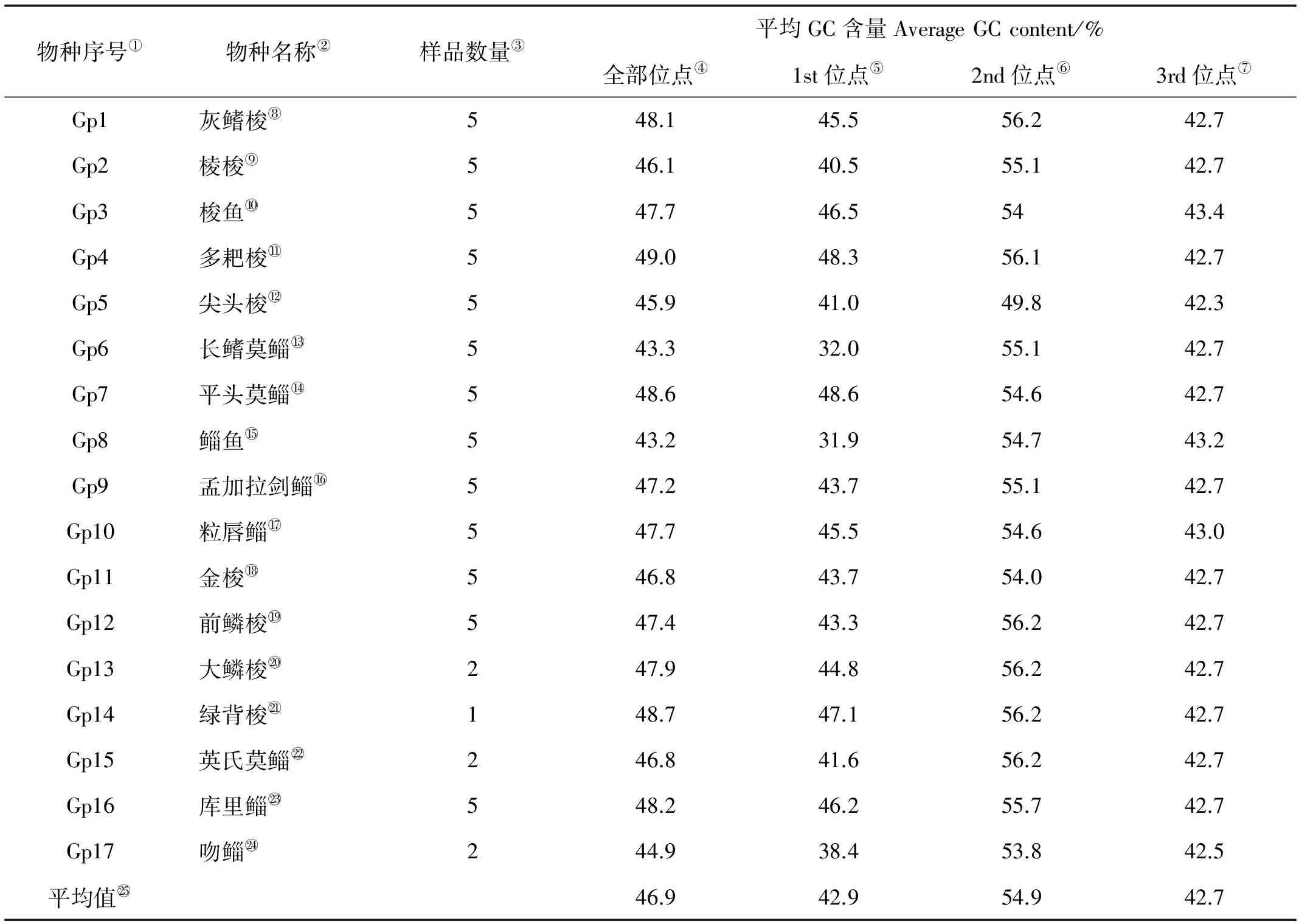
表4 鲻科17种鱼COI基因密码子位点GC含量
2.2 种间及种内的遗传距离
采用ModeltestV 3.06版本筛选得到Kimura-2-parameter模型,利用MEGA 3.0软件基于Kimura-2-parameter计算17种鲻科鱼类的种内和种间遗传距离(见表5)。鲻科鱼类种内遗传距离的平均值为0.0045,种间遗传距离的平均值为0.191,种间遗传距离是种内遗传距离的42倍。本研究中鲻鱼的种内遗传距离最大,为0.016。所有鲻科鱼类种内遗传距离均小于2%,符合种内遗传距离标准[14]。17种鲻科鱼类的种间遗传距离符合物种种间的遗传距离必须大于种内的遗传距离,二者之间的差异大约在10倍左右的标准。遗传距离最大的是库里鲻和平头莫鲻,遗传距离为0.262。前鳞梭和大鳞梭的遗传距离最小,为0.040。

表5 鲻科17种鱼两两比对的种间平均遗传距离和种内遗传距离
2.3 鲻科鱼类分类及分子系统树
本研究采用MP法、NJ法、ML法和贝叶斯法,对鲻科6属17种的72条COI基因序列构建分子系统树,结果如图1所示,4种分子系统树的结果具有相似的拓扑结构,并都具有较高的节点支持率。从图1和2中可知,MP、NJ、ML法构建的鲻科鱼类分子系统树显示:同一种鱼的不同个体均聚集在同一分支。所有物种均为单系,隶属于梭属的尖头梭与莫鲻属聚类,表现出与传统形态学分类不一致的结果。6属鲻科鱼类的聚类结果显示,它们的亲缘关系远近程度依次为鲻属、粒唇鲻属、莫鲻属、斧鲻属、梭属、剑鲻属。由贝叶斯法构建的系统发育树显示的结果与其他3种系统发育树的结果稍有不同,除隶属于梭属的尖头梭与莫鲻属聚类外,剑鲻属的孟加拉剑鲻与梭属聚类也表现出与传统形态学分类不一致的结果。此外,梭属、剑鲻属与鲻属、斧鲻属的遗传关系相较莫鲻属、粒唇鲻属更近。
3 讨论
本研究分析了17种鲻科鱼类的COI基因序列片段,其GC含量为43.2%~49.0%,平均46.9%;其中第二密码子位点的GC含量最高,第三密码子GC含量最低。以上研究的结果与柳淑芳等的研究结果一致[28]。2005年有ward等[9]对澳大利亚204种鱼(包括143种硬骨鱼和61种软骨鱼)的COI基因序列片段GC含量的分析结果显示,第三密码子位点的GC含量最低。2009年彭居俐等[7]针对鲤科鲌属4种鱼类的32条COI基因序列片段的GC含量研究同样显示第三密码子位点的GC含量最低。以上结果表明,线粒体基因组存在很强的GC→AT的进化选择压力。
利用DNA条形码技术进行物种分类鉴定必须满足以下条件:其一,在系统进化树上能够检测出物种的单系性。本研究中的系统进化树分析结果显示,17种鲻科鱼类均为单系,且分支支持率均较高,同一种鲻科鱼类的不同个体紧密的聚类在一起。其二,物种种间遗传距离应明显大于种内遗传距离。2003年Hebert等[29]通过研究DNA条形码确定了基于COI进行物种鉴定的遗传距离临界值,同时验证了DNA条形码在生物物种分类中的作用。该研究指出物种种内遗传距离小于1%,物种种间的遗传距离必须大于种内的遗传距离,二者之间的差异大约在10倍左右。本研究中17种鲻科鱼类的种内遗传距离的平均值为0.004 5,种间遗传距离平均值为0.191,种间遗传距离是种内遗传距离的42倍。由此说明COI基因序列能够对鲻科鱼类进行有效的物种鉴定。
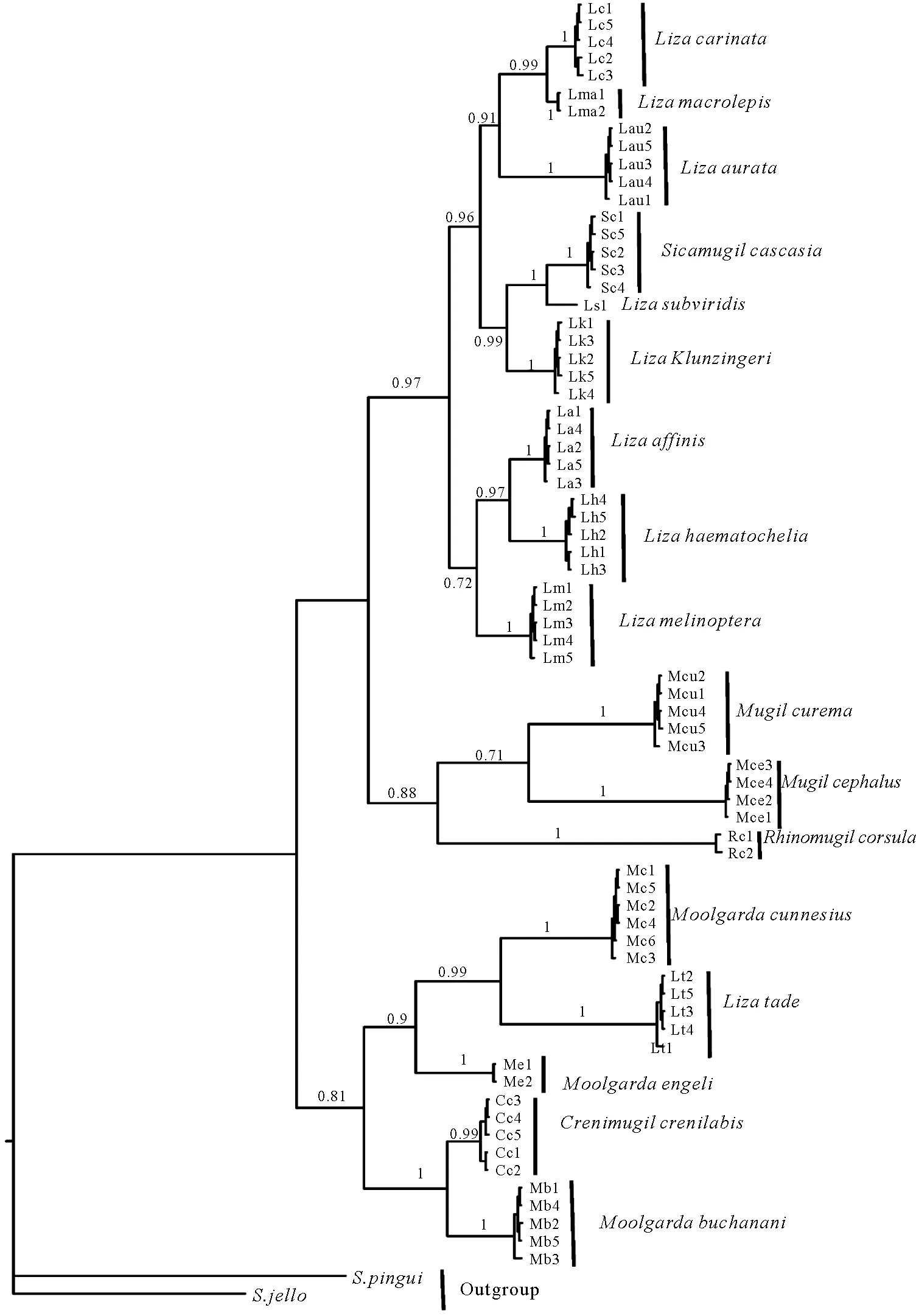
图1 贝叶斯法构建的鲻科鱼类分子系统树
分子系统树显示,隶属于梭属的尖头梭与莫鲻属聚类,剑鲻属的孟加拉剑鲻与梭属聚类,均表现出与传统形态学分类不一致的结果。2012年Durand等[30]通过线粒体序列片段分析发现采自缅甸和澳大利亚北部的Lizatade存在世系的分化,继而在2015年提出了隐存种存在的潜在可能(Durand JD等)[31]。由于样品采集自巴基斯坦,样品的形态学鉴定由卡拉奇大学研究人员协助完成,而分子生物学鉴定是通过与NCBI中序列比对得到的结论,所以Lizatade和Sicamugilcascasia也存在物种鉴定错误的可能性。关于梭属、剑鲻属和莫鲻属、粒唇鲻属的分化衍生的先后顺序由贝叶斯法构建的系统发育树显示的结果与其他三种系统发育树的结果有微弱差异存在。这可能是由于基于不同的构树方法,序列信息量的不同会导致在使用线粒体某个特定的片段进行独立分析时不能得到一致的结论[32-34]。在进行系统发育分析时,有时不同的方法不能得到一致的系统进化关系,基于信息量较少的序列构建的基因树可能会与物种树有所偏差,序列所含的信息量越多,越能够反映物种真实的进化情况。
综上所述, 本研究结果表明线粒体COI基因片段不但可以作为DNA条形码用于鲻科鱼类的物种鉴定,同时也可以用于鲻科鱼类种间系统发育关系的辅助探讨。
致谢:感谢中国海洋大学肖家光、周伟在数据分析中提供的帮助;集美大学黄良敏老师、国家海洋局第三研究所李渊博士、防城港市水产畜牧兽医局纪东平博士、大连市水产研究所闫龙、中国海洋大学单斌斌帮助采集样品。
[1] 杨俊杰. 鲽形目分类检索及分子生物学应用模式研究 [D]. 青岛: 中国海洋大学, 2009.
Yang J J. Taxonomic Identification and Application Mode in Molecular Biology of Pleuronectifotmes [D]. Qingdao: Ocean University of China, 2009.
[2] 王大海. 海水养殖业发展规模经济及规模效率研究 [D]. 青岛: 中国海洋大学, 2014.
Wang D H. A Study on the Problems on the Problems and Countermeasures of Scale Economy of Marine Aquaculture in China [D]. Qingdao: Ocean University of China, 2014.
[3] Knowlton N, Weight L A. New dates and new rates for divergence across the Isthmus of Panama [J]. Proceedings of the Royal Society B, 1998, 265: 2257-2263.
[4] Jarman S N, Elliott N G. DNA evidence for morphological and cryptic Cenozoic speciations in the Anaspididae, ‘living fossils’ from the Triassic [J]. Journal of Evolutionary Biology, 2000, 13: 624-633.
[5] Witt J D S, Therloff D L, Hebert P D N. DNA barcoding reveals extraordinary cryptic diversity in an amphipod genus: implications for desert spring conservation [J]. Molecular Ecology, 2006, 15: 3073-3082.
[6] Moritz C, Cicero C. DNA barcoding: promise and pitfalls [J]. PLoS Biology, 2004, 2: 279-354.
[7] 彭居俐, 王绪桢, 何舜平. DNA 条形码技术的研究进展及其应用 [J]. 水生生物学报, 2008, 32(6): 916-919.
Peng J L, Wang X Z, He S P. The process and application of DNA barcoding [J]. Acta Hydrobiologica Sinnica, 2008, 32(6): 916-919.
[8] Hebert P D N, Stoeckle M Y, Zemlak T S, et al. Identification of birds through DNA barcodes [J]. PLoS Biology, 2004, 2: 312.
[9] Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia’s fish species [J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360: 1847-1857.
[10] Smith M A, Poyarkov J R N A, Hebert P D N. CO1 DNA barcoding amphibians: take the chance, meet the challenge [J]. Molecular Ecology Resources, 2008, 8: 235-246.
[11] Burns J, Janzen D, Hajibabaei M, et al. DNA barcodes and cryptic species of skipper butterflies in the genusPericharesin Area de Conservación Guannacaste, Costa Rica [J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105: 6350-6355.
[12] Johnson S B, Warén A, Vrijenhoek R C. DNA barcoding ofLepetodriluslimpets reveals cryptic species [J]. Journal of Shellfish Research, 2008, 27(1): 43-51.
[13] Savolainen V, Cowan R S, Vogler A P, et al. Towards writing the encyclopedia of life: an introduction to DNA barcoding [J]. Philosophical Transactions of the Royal Society B, 2005, 360: 1805-1811.
[14] Hebert P D N, Ratnasingham S, deWaard J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species [J]. Proceeding of the Royal Society B Biological Sciences, 2003, 270: 96- 99.
[15] Eschmeyer W N. Catalog of Fishes[M].San Francisco: Special Publicution, California Academy of Sciences, 1998.
[16] Caldara F, Bargelloni L, Ostellari L, et al.Molecular phylogeny of grey mullets based On mitochondrial DNA sequence analysis: evidence of a differential rate of evolution at the intrafamily level [J].Molecular Phylogenetics and Evolution, 1996, 6: 416-424.
[17] Thomson J M. The Taxonomy of Grey Mullets. in Aquaculture of Greymullets[M]. Cambridge: Cambridge University Press, 1981: 1-16.
[18] Trewavas E, Ingham S E. A key to the species of Mugilidae (Pisces) in the northeastern Atlantic and Mediterranean, with explanatory notes[J]. Journal of Zoology, 1972, 167: 15-29.
[19] Nelson J S. Fishes of the World[M]. New York: John Wiley and Sons, 1994: 600.
[20] Thompson J D, Gibson T J, Plewniak F, et al. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools [J]. Nucleic Acids Research, 1997, 25: 4876-4882.
[21] 陈大刚, 张美昭. 中国海洋鱼类 [M]. 青岛: 中国海洋大学出版社, 2016: 600-605.
Chen D G, Zhang M Z. The Fish of Chinese Ocean[M]. Qingdao: China Ocean University Press, 2016: 600-605.
[22] Sambrook J, Fristch E F, Maniatis T. Molecular Cloning: A Laboratory Manual (Cold Spring Harbor Laboratory) [M]. New York: Cold Spring Harbor, 1989.
[23] Swofford D L. PAUP (version 3. 0): phylogenetic analysis using parsimony [M]. Champaign: Illinois Natural History Survey Ill, 1989.
[24] Ronquist F, Huelsenbeck J P. Mrbayes 3: bayesian phylogenetic inference under mixed models [J]. Bioinformatics, 2003, 19: 1572-1574.
[25] Posada D, Crandall K A. Modeltest: testing the model of DNA substitution [J]. Bioinformatics, 1998, 14: 817-818.
[26] Akaike H. Maximum likelihood identification of Gaussian autoregressive moving average models [J]. Biometrika, 1973, 60: 255-265.
[27] Felsenstein J. Confidence limits on phylogenies: an approach using the bootstrap [J]. Evolution, 1985, 39: 783-791.
[28] 柳淑芳, 陈亮亮, 戴芳群, 等. 基于线粒体COI基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用 [J]. 海洋与湖沼, 2010, 41(2): 223-232.
Liu S F, Chen L L, Dai F Q. Appolication of DNA barcoding gene COI for classifying family sciaenidae [J]. Oceanologia et Limnologia Sinica, 2010, 41(2): 223-232.
[29] Hebert P D N, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes [J]. Proceedings of the Royal Society Lond B, 2003, 270: 313-322.
[30] Durand J D, K N Shen, W J Chen, et al. Systematics of the grey mullets (Teleostei: Mugiliformes: Mugilidae): molecularphylogenetic evidence challenges two centuries of morphology-based taxonomy [J]. Molecular Phylogenetics and Evolution, 2012, 64: 73-92.
[31] Durand J D, Borsa P. Mitochondrial phylogeny of grey mullets (Acanthopterygii: Mugilidae) suggests high proportion of cryptic species [J]. Comptes Rendus Biologied, 2015, 338(4): 266-277.
[32] 程娇. 西北太平洋两种鲐属鱼类的分子系统地理学研究 [D]. 青岛: 中国海洋大学, 2013.
Cheng J. Molecular Phylogeography of TwoScomberSpecies in Northwestern Pacific [D]. Qingdao: Ocean University of China, 2013.
[33] Aoyama J, Nishida M, Tsukamoto K. Molecular phylogeny and evolution of the freshwater eel, genusAnguilla[J]. Molecular Phylogenetics and Evolution, 2001, 20: 450-459.
[34] Minegishi Y, Aoyama J, Inoue J G, et al. Molecular phylogeny and evolution of the freshwater eels genusAnguillabased on the whole mitochondrial genome sequences [J]. Molecular Phylogenetics and Evolution, 2005, 34: 134-146.
责任编辑 朱宝象
Application of DNA Barcoding in Classification of Mugilidae Fishes
LIU Lu1, SUN Dian-Rong2, LI Chun-Hou2, HAN Zhi-Qiang3, GAO Tian-Xiang3,SHEN Kang-Ning4, SONG Na1
(1.College of Fisheries, Ocean University of China, Qingdao 266003, China; 2.South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510300, China; 3.Fishery College, Zhejiang Ocean University, Zhoushan 316022, China; 4.Center of Excellence for the Oceans, National Taiwan Ocean University, Keelung 20224, China)
A total of 72 individuals from 17 species pertaining to 6 genera of Mugilidae were barcoded with COI gene which were sequenced and compared with each other. Our results indicated that the average GC content of the gene accounted for 46.9%. The GC content of codons was 42.9% (31.9%~48.6%) at the firstnucleotide position, 54.9% (49.8%~56.2%) at the second, and 42.7% (42.5%~ 43.4%) at the third. The average Kimura-2-parameter (K2P) distances within-species and pairwise-species were 0.45% and 19.1%, respectively. The K2P distance of pairwise-species was 42 times of that within-species. According to the maximum parsimony and neighbor-joining trees for all 72 sequences, 17 species, all individuals of each species formed a strong monophyletic group. However,Lizatadebelonging toLizagenera clustered withMologardagenera, which was different from the morphological characters. Our research demonstrated that the COI barcoding was a useful tool for fish species identification, which may also afford auxiliary discussions about the phylogeny of Family Mugilidae
Mugilidae; COI genes; DNA barcoding; species classification; phylogeny
国家自然科学基金项目(41506158);农业部南海渔业资源开发利用重点实验室开放基金课题(LSF2014-02);海洋公益性行业科研专项(201305043,201405010,201303050)资助
2016-07-28;
2016-09-13
刘 璐(1988-),女,博士生,从事渔业生态学研究。E-mail:liulouc@163.com
** 通讯作者:E-mail:songna624@163.com
S931
A
1672-5174(2016)11-178-09
10.16441/j.cnki.hdxb.20160270
刘璐, 孙典荣, 李纯厚, 等. DNA条形码技术在鲻科鱼类鉴定中的应用[J]. 中国海洋大学学报(自然科学版), 2016, 46(11): 178-186.
LIU Lu, SUN Dian-Rong, LI Chun-Hou, et al. Application of DNA barcoding in classification of mugilidae fishes[J]. Periodical of Ocean University of China, 2016, 46(11): 178-186.
Supported by National Natural Science Foundation of China(41506158);Key Laboratory for Exploitation & Utilization of Marine Fisheries Resource in South China Sea, Ministry of Agriculture(LSF2014-02);the Public Science and Technology Research Funds Projects of Ocean(201305043,201405010,201303050)

