Sanger法测定季节性H3N2亚型流感病毒全基因组序列
李希妍 赵翔 谭敏菊 蓝雨 成艳辉 黄维娟 王大燕 舒跃龙
102206 北京, 中国疾病预防控制中心病毒病预防控制所 国家流感中心 卫生部医学病毒和病毒病重点实验室
·短篇论著·
Sanger法测定季节性H3N2亚型流感病毒全基因组序列
李希妍 赵翔 谭敏菊 蓝雨 成艳辉 黄维娟 王大燕 舒跃龙
102206 北京, 中国疾病预防控制中心病毒病预防控制所 国家流感中心 卫生部医学病毒和病毒病重点实验室
目的 对流行的季节性甲型H3N2亚型流感病毒进行全基因组测序。方法 本文对甲型H3N2亚型流感病毒测序过程中的PCR扩增引物进行优化及精简,同时对PCR产物纯化方法进行了改进。使用优化后的34对PCR引物和优化后的纯化方法,对194株甲型H3N2亚型病毒进行了测序。结果 利用优化后的测序方法,可保证获得甲型H3N2亚型流感病毒的8个片段全基因组序列,而且大大缩短了实验时间,节约了实验成本和人力。结论 我们应大力开展A(H3N2)亚型流感病毒的序列测定工作,及时发现病毒变异情况,以推选出与人群中的流行株更为匹配的疫苗株。
Fundprogram:NationalScienceandTechnologyMajorProjectofChina(2014ZX10004002-001-003)
甲型H3N2[A(H3N2)]亚型流感病毒于1968年引起了全球范围的流感大流行,此后一直在人群中呈季节性流行。对流感病毒表面蛋白HA基因的分析表明,与人群中流行的其他季节性流感病毒相比,A(H3N2)亚型流感病毒的进化和变异最为迅速[1]。流感病毒是分节段的RNA病毒,对流感病毒全基因组8个基因片段的综合分析,可以进一步全面揭示流感病毒的重配和变异规律。既往有研究者曾公布A(H3N2)亚型流感病毒测序引物,本实验室对此方法进行了优化,可在10h左右快速获得A(H3N2)亚型流感病毒的全基因组序列,可用于流感病毒的日常监测和研究工作,及时进行病毒进化分析。
1 材料与方法
1.1 毒株来源 本研究的A(H3N2)亚型流感毒株均为国家流感中心保存,病毒分离时间自2010年1月至2015年10月。
1.2 病毒RNA的提取及基因扩增 利用Qiagen公司RNeasyMiniKit试剂盒进行RNA提取。从网站http://gsc.jcvi.org/projects/msc/influenza选取人甲型H3N2亚型扩增引物,再根据后续测序反应结果进行引物优化。所有测序引物由大连宝生物公司合成。
使用Qiagen公司OneStepRT-PCRKit进行基因扩增,反应体系为10μl。具体操作方法参见试剂盒说明书。PCR反应条件为60 ℃ 1min;42 ℃ 20min;50 ℃ 20min;95 ℃ 15min;94 ℃ 30s,55 ℃ 30s,72 ℃ 1min,循环35次;72 ℃ 10min;4 ℃保存。
1.3PCR产物纯化及序列测定PCR产物纯化分别使用Qiagen公司QIAquick96PCRPurificationKit和USB公司的ExoSAP-IT,两种方法进行比较。测序反应使用美国ABI公司的BigDyeTerminatorv3.1CycleSequencingKit。由于PCR引物已包含M13接头,因此用于测序反应的引物为通用引物M13。具体方法参见文献[2]。
1.4 序列分析 序列拼接采用DNAStar软件中的Seqman程序。序列比对使用MEGA5.0软件的ClustalW方法。
2 结果
2.1 纯化方法优化 最初使用Qiagen公司的QIAquick96PCRPurificationKit滤膜法进行PCR产物纯化,每纯化96个反应约40min,如果样品量增多,所需的纯化时间将加倍。
改进实验方法后,使用USB公司的ExoSAP-IT虾碱酶纯化,此纯化方法在加入虾碱酶后可用PCR仪进行热反应,无论样品量多少,只需30min即可完成纯化。因此更换试剂后,更加节省时间及人力。
2.2 引物匹配性优化 使用89对PCR引物对A(H3N2)亚型流感病毒进行全基因组测序,共测序毒株320株,采样日期为2010年1月至2014年5月。
在实验过程中,发现部分病毒未能通过一次测序实验而获得全基因组序列,个别引物在对这些病毒进行序列测定时不工作。为了确保所有病毒序列能够一次测通,对引物序列进行了优化,使用PrimerPremier5.0软件,根据已知病毒序列,设计并更换了4对引物,分别为PB1-5、PB1-7、PB1-8和PA-11。2.3 引物数量优化 用89对引物对A(H3N2)亚型流感病毒进行PCR反应,再使用通用引物M13分别进行正反向测序反应,最终获得178条序列,拼接后发现,每一核苷酸位点有4至9个测序反应。为了节约成本,能够在最短时间内完成全基因组测序工作,对测序所使用的PCR引物进行精简。最终确定了34对引物,扩增的目的片段之间相互重叠,能够覆盖整个流感病毒全基因组。
2.4 引物验证 使用筛选出的34对引物对采样日期在2014年3月至2015年10月之间的194株A(H3N2)亚型流感病毒进行测序,结果表明仅使用此34对引物,可以完成流感病毒所有8个片段的全基因组测序,显著减少了实验成本和实验时间。
3 结论
近年来,下一代测序 (Next-generationsequen-cing,NGS) 技术相继出现并迅速发展成熟。NGS以高通量、低成本为主要特点[3],不过,由于NGS技术相关的仪器费用较高,不利于广泛推广。而1977年Sanger发明的双脱氧末端终止法,即第一代测序技术,拥有较长读长(平均读长700bp)和极高准确率(99.9%)[4],对于少量测序仍是最佳选择。
目前有些实验室已有第一代测序平台,但是缺乏有效的测序引物,不能得以充分利用,所以我们希望共享我们优化后的测序方法。使用经过我们优化的测序引物对A(H3N2)流感病毒进行测序,其匹配性更高,而且大大减少了PCR扩增的数量,可以节约成本。
流感病毒抗原性易变,传播迅速,每年可引起季节性流行,并且在人群聚集的场所可发生暴发疫情。对孕妇、婴幼儿、老年人和慢性病患者等高危人群的危害尤为严重,接种流感疫苗是目前预防流感、降低流感疾病负担最有效的手段[5]。由于流感病毒易发生抗原性漂移和抗原性转变,而目前全球已上市的流感疫苗都不能克服流感病毒易于变异的特点,影响疫苗免疫效果的主要因素为疫苗株和流行株的匹配性,这就需要我们大力开展序列测定和分析工作,及时发现病毒变异情况,推选出与人群中的流行病毒更为匹配的疫苗株,为人群提供有效的保护。
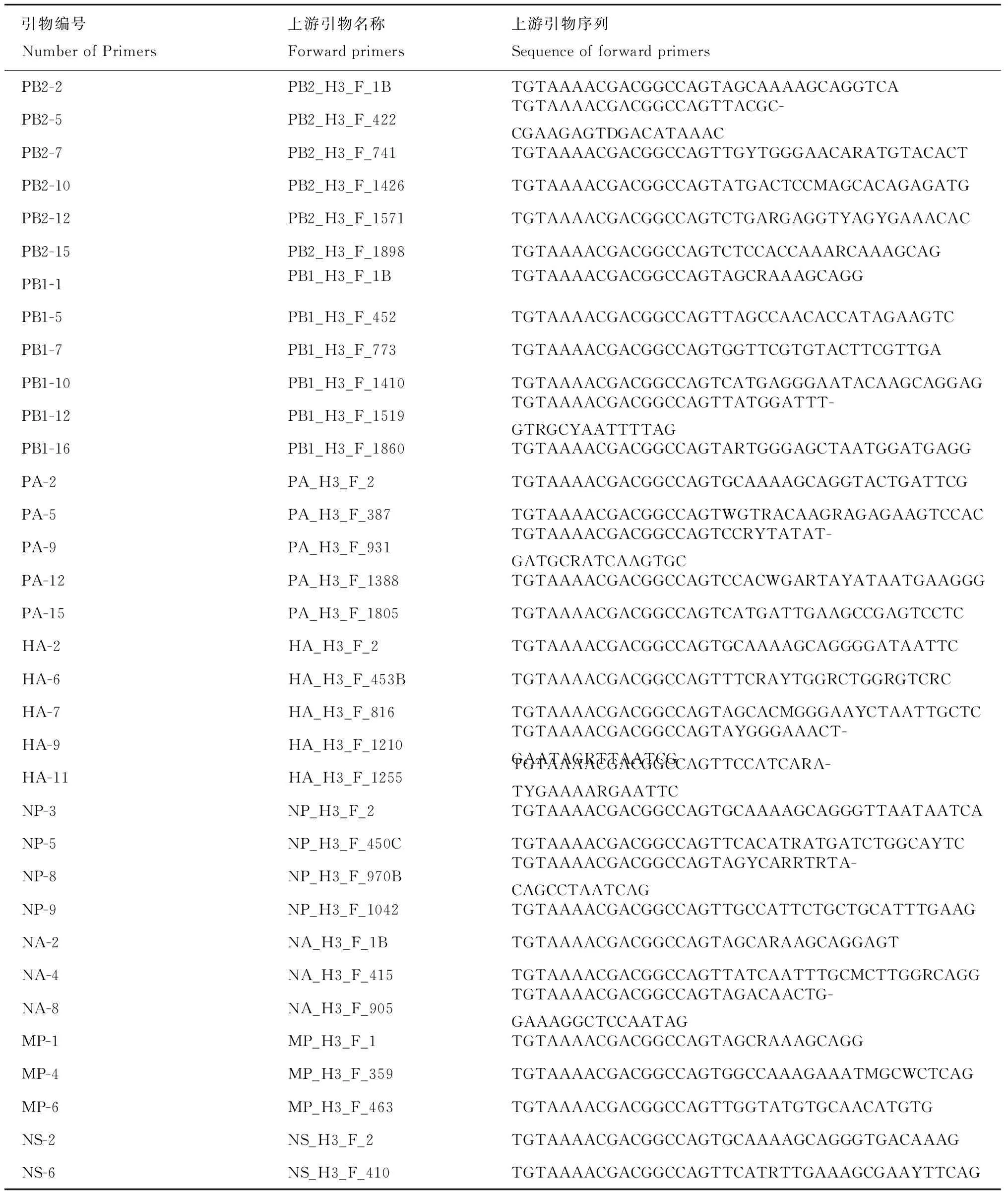
表1 用于季节性H3N2亚型流感病毒测序的PCR引物
续表1
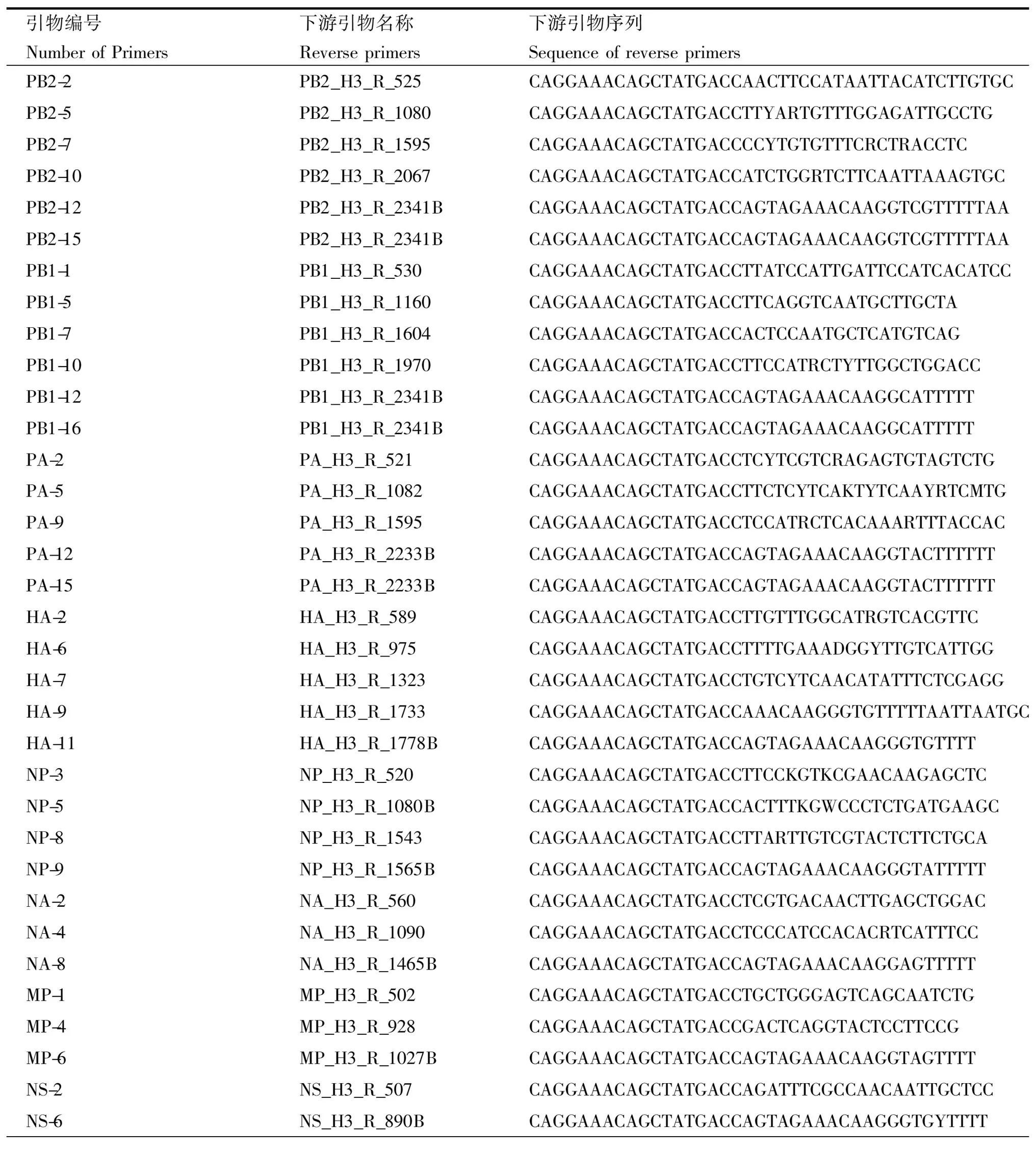
引物编号NumberofPrimers下游引物名称Reverseprimers下游引物序列SequenceofreverseprimersPB2-2PB2_H3_R_525CAGGAAACAGCTATGACCAACTTCCATA-ATTACATCTTGTGCPB2-5PB2_H3_R_1080CAGGAAACAGCTATGACCTTYARTGTTTG-GAGATTGCCTGPB2-7PB2_H3_R_1595CAGGAAACAGCTATGACCCCYTGTGTTT-CRCTRACCTCPB2-10PB2_H3_R_2067CAGGAAACAGCTATGACCATCTGGRTCT-TCAATTAAAGTGCPB2-12PB2_H3_R_2341BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGTCGTTTTTAAPB2-15PB2_H3_R_2341BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGTCGTTTTTAAPB1-1PB1_H3_R_530CAGGAAACAGCTATGACCTTATCCATT-GATTCCATCACATCCPB1-5PB1_H3_R_1160CAGGAAACAGCTATGACCTTCAGGTCAAT-GCTTGCTAPB1-7PB1_H3_R_1604CAGGAAACAGCTATGACCACTCCAATGCT-CATGTCAGPB1-10PB1_H3_R_1970CAGGAAACAGCTATGACCTTCCATRCTYT-TGGCTGGACCPB1-12PB1_H3_R_2341BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGCATTTTTPB1-16PB1_H3_R_2341BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGCATTTTTPA-2PA_H3_R_521CAGGAAACAGCTATGACCTCYTCGT-CRAGAGTGTAGTCTGPA-5PA_H3_R_1082CAGGAAACAGCTATGACCTTCTCYTCAK-TYTCAAYRTCMTGPA-9PA_H3_R_1595CAGGAAACAGCTATGACCTCCATRCTCA-CAAARTTTACCACPA-12PA_H3_R_2233BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGTACTTTTTTPA-15PA_H3_R_2233BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGTACTTTTTTHA-2HA_H3_R_589CAGGAAACAGCTATGACCTTGTTTGGCA-TRGTCACGTTCHA-6HA_H3_R_975CAGGAAACAGCTATGACCTTTT-GAAADGGYTTGTCATTGGHA-7HA_H3_R_1323CAGGAAACAGCTATGACCTGTCYTCAACA-TATTTCTCGAGGHA-9HA_H3_R_1733CAGGAAACAGCTATGACCAAACAAGGGT-GTTTTTAATTAATGCHA-11HA_H3_R_1778BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGGTGTTTTNP-3NP_H3_R_520CAGGAAACAGCTATGACCTTCCKGT-KCGAACAAGAGCTCNP-5NP_H3_R_1080BCAGGAAACAGCTATGACCACTTTKGWC-CCTCTGATGAAGCNP-8NP_H3_R_1543CAGGAAACAGCTATGACCTTARTTGTCG-TACTCTTCTGCANP-9NP_H3_R_1565BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGGTATTTTTNA-2NA_H3_R_560CAGGAAACAGCTATGACCTCGTGACAACT-TGAGCTGGACNA-4NA_H3_R_1090CAGGAAACAGCTATGACCTCCCATCCA-CACRTCATTTCCNA-8NA_H3_R_1465BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGAGTTTTTMP-1MP_H3_R_502CAGGAAACAGCTATGACCTGCTGGGAGT-CAGCAATCTGMP-4MP_H3_R_928CAGGAAACAGCTATGACCGACTCAGG-TACTCCTTCCGMP-6MP_H3_R_1027BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGTAGTTTTNS-2NS_H3_R_507CAGGAAACAGCTATGACCAGATTTCGC-CAACAATTGCTCCNS-6NS_H3_R_890BCAGGAAACAGCTATGACCAGTAGAAA-CAAGGGTGYTTTT
[1] Bedford T.,Riley S.,Barr I.G.,et al. Global circulation patterns of seasonal influenza viruses vary with antigenic drift[J]. Nature,2015,523(7559):217-220. doi: 10.1038/nature14460.
[2] 黄维娟,成艳辉,李希妍,等. 2011-2012年度中国H3N2亚型流感病毒病原学特征分析[J]. 病毒学报,2013,29(3):258-264. doi: 10.13242/j.cnki.bingduxuebao.002384.
[3] Glenn T.C. Field guide to next-generation DNA sequencers[J]. Mol Ecol Resour,2011,11(5):759-769. doi: 10.1111/j.1755-0998.2011.03024.x.
[4] 柳延虎,王璐,于黎. 单分子实时测序技术的原理与应用[J]. 遗传,2015,37(3):259-268. doi: 10.16288/j.yczz.14-323.
[5] 舒跃龙. 中国流感疫苗接种所面临的挑战与应对策略[J]. 中华微生物学和免疫学杂志,2016,36(1):1-2. doi: 10.3760/cma.j.issn.0254-5101.2016.01.001.
Sequencing of seasonal influenza A (H3N2) virus
LiXiyan,ZhaoXiang,TanMinju,LanYu,ChengYanhui,HuangWeijuan,WangDayan,ShuYuelong
NationalInstituteforViralDiseaseControlandPrevention,CollaborationInnovationCenterforDiagnosisandTreatmentofInfectiousDiseases,ChineseCenterforDiseaseControlandPrevention;KeyLaboratoryforMedicalVirology,NationalHealthandFamilyPlanningCommission,Beijing102206,China
WangDayan,Email:dayanwang@cnic.org.cn
ObjectiveTosequencingthewholegenomeofseasonalinfluenzaA(H3N2)viruses.MethodsOptimizedandreducedthePCRprimersofA(H3N2)virus,andimprovedthepurificationmethodofPCRproduction.Usethismethod, 194A(H3N2)virusesweresequenced.ResultsOptimizedsequencingmethodwasusedforfullgenomesequencingofinfluenzaA(H3N2)virus, 8segmentsofallA(H3N2)viruseswereobtained.Thisoptimizedmethodreducedthetimerequiredforsequencing,andsavedexperimentalcost.ConclusionsWeshouldcarryoutthesequencinganalysisofinfluenzaA(H3N2)viruses,totimelyidentifythevariationofviruses,andtoselectmoreappropriatevaccinestrainsforhumanpopulation.
Influenzavirus;H3N2Subtype;Sequencing;Wholegenome
王大燕,Email:dayanwang@cnic.org.cn
10.3760/cma.j.issn.1003-9279.2016.05.013
流感病毒;H3N2亚型;测序;全基因组
国家科技重大专项“H7N9等新型流感病毒跨种传播机制研究” (2014ZX10004002-001-003)
2016-03-16)

