羽衣甘蓝不同组织及柱头发育实时荧光定量PCR内参基因的筛选
李 晗 李治龙 李晓屿 李玉花 蓝兴国
(东北林业大学生命科学学院,哈尔滨 150040)
羽衣甘蓝不同组织及柱头发育实时荧光定量PCR内参基因的筛选
李 晗 李治龙 李晓屿 李玉花 蓝兴国*
(东北林业大学生命科学学院,哈尔滨 150040)
以羽衣甘蓝(Brassicaoleraceavar.acephala)S13-bS13-b自交不亲和系为试材,利用实时荧光定量PCR(qRT-PCR)技术检测Actin、Cpi6、EF-1β、GAPDH、Tub-α3、Tub-α6、Ubc7候选内参基因在不同组织和5个不同发育时期柱头的表达情况,并运用geNorm和BestKeeper软件统计分析候选内参基因的表达稳定性。在不同组织中,geNorm软件统计分析的结果表明Tub-α6和EF-1β的表达较稳定;BestKeeper软件统计分析的结果表明Ubc7和Tub-α6的表达较稳定。在不同发育柱头中,geNorm软件统计分析的结果表明Actin和Ubc7的表达较稳定;BestKeeper软件统计分析的结果表明EF-1β和Tub-α6的表达较稳定。以筛选到的内参基因Tub-α6和Actin,分别分析羽衣甘蓝柱头S-位点糖蛋白基因(SLG)在不同组织和不同发育时期柱头的表达水平,结果显示出SLG主要在柱头中表达,而且SLG在柱头发育成熟时期表达量达到最高。
羽衣甘蓝;实时荧光定量PCR;内参基因;柱头发育;SLG
羽衣甘蓝(Brassicaoleraceavar.acephala)属于十字花科(Brassicaceae)芸薹属(Brassica)植物,由于其叶色彩鲜艳且头部形状酷似牡丹,被称为“叶牡丹”[1]。由于其具有较强的耐寒性,是我国北方重要的绿化观赏植物[2~3]。在育种方面,由于羽衣甘蓝大部分栽培品种都具有自交不亲和性(Self-incompatibility,SI),人们在育种过程中主要采用SI系进行羽衣甘蓝杂交种的生产[4]。
在芸薹属植物SI上的研究发现,SI反应与柱头的生长发育密切相关[5]。而且,SI相关的SLG(S-locus glycoprotein)、SRK(S-locus receptor kinase)、MLPK(M-locus protein kinase)和ARC1(Armadillo Repeat-Containing protein 1)基因表达具有柱头组织表达特异性,并且与柱头发育成熟性正相关[6~13]。因此,定量分析基因在不同组织及柱头发育阶段表达的水平,对于阐述基因在SI及柱头发育方面的功能是十分重要的。
实时荧光定量PCR(Real-time fluorescence quantitative PCR,RT-qPCR)是近些年来发展的一种定量分析基因表达的方法[14~15]。由于该方法重复性好、定量精确及高通量等优点,已广泛应用于基因的表达研究。在本研究中,我们选择了Actin、Cpi6(cysteine proteinase inhibitor 6)、EF-1β(elongation factor 1-beta)、GAPDH(glyceraldehyde-3-phosphate dehydrogenase)、Tub-α3(tubulin alpha-3)、Tub-α6(tubulin alpha-6)、Ubc7(ubiquitin conjugating enzyme7)候选内参基因,通过geNorm[16]和BestKeeper[17]软件统计分析候选内参基因的表达稳定性,以期筛选出表达最稳定的内参基因,为进一步开展基因表达的研究奠定基础。
1 材料与方法
1.1 材料
试验于2014年2~6月在东北林业大学生命科学学院发育生物学实验室完成。羽衣甘蓝(Brassicaoleraceavar.acephala)自交不亲和系(S13-bS13-b)由东北林业大学花卉生物工程研究所提供。选取羽衣甘蓝的根、嫩茎、嫩叶、当天开花未散粉的柱头、花柱、子房、花药、花瓣为不同组织材料,选取不同花蕾长度(0~4、4~6、6~8、8~10、>10 mm)的柱头为不同发育时期材料,液氮速冻后保存于-80℃冰箱内。
Phase Lock GelTM(PLG)Heavy、TRIzol A+购自天根生化科技有限公司;Premix exTaqDNA聚合酶、dNTP购自大连宝生物(TaKaRa)公司;DNaseⅠ,RNase-free购自Thermo公司;High-Capacity cDNA Reverse Transcription Kit试剂盒、标准96孔板、光学膜、Power SYBY® Green PCR Master Mix购自美国应用生物系统(ABI)公司;RNase Inhibitor购于哈尔滨欧比特科技开发有限公司;荧光定量PCR所用机器为ABI 7500。
1.2 PCR引物设计
依据实时定量PCR引物设计原则,分别设计Actin、Cpi6、EF-1β、GAPDH、Tub-α3、Tub-α6、Ubc7候选内参基因和SLG13-b引物,相关信息在表1。

表1 羽衣甘蓝候选内参基因和SLG引物及相关信息
1.3 总RNA的提取和cDNA的合成
将羽衣甘蓝材料于液氮中迅速研磨,将研磨好的材料放于700 μL TRIzol A+中;匀浆样品在室温下震荡15 min;将Phase Lock GelTM(PLG)置于离心机中,12 000×g离心30 s;将匀浆样品转入离心后的PLG中,加入140 μL氯仿,剧烈震荡10 min;4℃,12 000×g离心15 min;将PLG上层含有RNA的水相转移至一个新的RNase Free离心管;向得到的水相加入等体积异丙醇,彻底混合均匀,室温放置30 min;4℃,12 000×g离心10 min;弃掉上清,加入75%乙醇对沉淀进行洗涤;4℃,7 500×g离心5 min;室温放置晾干,加入适量RNase Free H2O充分溶解RNA。利用NanoDrop2000超微分光光度计(Thermo公司)测定OD260/280值,检测RNA的浓度和纯度,并将RNA浓度调整为1 μg·μL-1。使用DNaseⅠ去除基因组DNA:RNA 1 μL(1μg·μL-1)、10×reaction buffer with MgCl21 μL、DNaseⅠ 1 U、RNase-free Water 7 μL,反应条件为37℃,30 min,加入EDTA(50 mmol·L-1)1 μL,65℃,10 min;利用去除基因组DNA的羽衣甘蓝总RNA进行反转录cDNA,反应体系为20 μL,其中10×RT Buffer 2 μL、25×dNTP Mix(100 mmol·L-1)0.8 μL、10×RT Random Primers 2 μL、MultiScribeTMReverse Transcriptase 1 μL、RNase Inhibitor 1 μL、Nuclease-free H2O 2.2 μL、上述去基因组RNA 11 μL。反应条件为25℃,10 min;37℃,120 min;85℃,5 min。
1.4 目的基因片段的PCR扩增
利用设计的引物进行PCR扩增,总体积20 μL,其中Premix exTaq为10 μL、10 μmol·L-1上下游引物各0.5 μL、cDNA 0.5 μL。PCR反应条件为:94℃预变性2 min;94℃变性30 s,60℃退火30 s,72℃延伸30 s,30个循环;72℃延伸7 min。0.8%琼脂糖凝胶电泳检测PCR产物。
1.5 qRT-PCR分析
取10 μL反转录的cDNA稀释于200 μL ddH2O中作为反应模板。反应体系总体积为20 μL,其中SYBR Green Mix(ABI)10 μL、10 μmol·L-1上下游引物各0.8 μL、稀释后cDNA 8.4 μL。95℃预变性10 min,95℃变性15 s,60℃延伸1 min,40个扩增循环,溶解曲线分析:95℃、15 s,60℃、1 min,95℃、15 s,60℃、15 s。每个样品进行3次生物学重复和2次技术重复。
1.6 数据分析
利用geNorm软件和BestKeeper软件对候选内参基因的表达稳定性进行统计分析。将候选基因的Ct值用Microsoft Excel计算出2-ΔCT值并输入geNorm软件,计算得出平均表达稳定指数M值。M值小于1.5的基因被认为表达相对稳定,且M值越小,表达越稳定,反之则不稳定;根据内参基因标准化因子的配对差异分析Vn/n+1判定内参基因合适的数目[16]。BestKeeper软件将Ct值输入程序,获得标准差(SD)和调节系数标准差SD[±x-fold],其值越小则表示越稳定,越大则表示越不稳定(Pfaffl 2001)。计算相对表达量的方法为2-ΔΔCT法,默认扩增效率E为2,ΔCt=Ct样品-Ct内参,ΔΔCt=ΔCt最小-ΔCt样品[18]。
2 结果与分析
2.1 羽衣甘蓝柱头及组织RNA的提取
分别提取不同发育阶段的柱头及根、茎、叶、花柱、子房、花药和花瓣的RNA。OD260/280值均在1.8~2.0。琼脂糖凝胶电泳显示,柱头RNA良好无明显降解(图1),可以用于后续实验。

图1 羽衣甘蓝柱头总RNA的电泳检测Fig.1 Gel electrophoresis of total RNA from stigmas in B.oleracea var. acephala
2.2 候选内参基因的RT-PCR扩增分析
提取大于10 mm花蕾内的柱头RNA,去除基因组DNA后,通过反转录试剂盒获得cDNA。分别对Actin、Cpi6、EF-1β、GAPDH、Tub-α3、Tub-α6、Ubc7候选内参基因进行PCR,利用琼脂糖凝胶电泳进行检测。结果显示出,扩增出的内参基因条带单一,并与预期结果一致(图2,表1)。说明各候选内参基因的扩增条带特异性较好,可以用于qRT-PCR的分析。

图2 7个候选内参基因的PCR扩增产物Fig.2 PCR products of 7 candidate reference genes M.DL1000; 1.Cpi6; 2.GAPDH; 3.Tub-α6; 4.Tub-α3; 5.EF-1β; 6.Actin; 7.Ubc7
2.3 候选内参基因的qRT-PCR表达分析
分别对Actin、Cpi6、EF-1β、GAPDH、Tub-α3、Tub-α6、Ubc7候选内参基因进行qRT-PCR检测。图3显示在不同发育阶段的柱头样品中,7个候选内参基因的熔解曲线有明显的单一峰。而且,图4显示出扩增曲线良好,可以满足qRT-PCR的要求。7个候选内参基因在羽衣甘蓝不同组织中表达的Ct值在17~31间,其中GAPDH表达量较高,Ubc7表达量较低(图5:A);在不同发育时期的柱头中,也发现GAPDH表达量较高,Ubc7表达量较低(图5:B)。

图3 7个候选内参基因的溶解曲线Fig.3 Melting curves of 7 candidate reference genes A.Actin; B.Cpi6; C.EF-1β; D.GAPDH; E.Tub-α3; F.Tub-α6; G.Ubc7
图4 7个候选内参基因的扩增曲线Fig.4 Amplification curves of 7 candidate reference genes A.Actin; B.Cpi6; C.EF-1β; D.GAPDH; E.Tub-α3; F.Tub-α6; G.Ubc7

图5 7个候选基因在不同组织(A)和不同发育时期柱头(B)的Ct值变化 T1~T8分别代表根、茎、叶、柱头、花柱、子房、花药和花瓣;S1~S5分别代表花蕾大小在0~4、4~6、6~8、8~10 mm和大于10 mm的柱头 下同。Fig.5 The Ct value of 7 candidate reference genes in different tissues(A) and developmental stigmas(B) of ornamental kale T1-T8 represented root,stem,leaf,stigma,style,ovule,anther and petal; S1-S5 represented stigma at different bud size,with S1=0-4 mm bud size,S2=4-6 mm bud size,S3=6-8 mm bud size,S4=8-10 mm bud size,S5=over 10 mm bud size The same as below.
2.4不同组织候选内参基因表达稳定性的统计分析
为了筛选羽衣甘蓝不同组织的最佳内参基因,首先采用geNorm软件对7个候选内参基因的表达稳定性进行分析。将7个候选基因的Ct值用Microsoft Excel计算出2-ΔCT值并输入geNorm软件,计算得出平均表达稳定指数M值。M值小于1.5的基因被认为表达相对稳定,且M值越小,表达越稳定。图6A的结果表明表达稳定的候选内参基因顺序为Tub-α6/EF-1β(0.69)>Actin(0.74)>Ubc7(0.79)>Cpi6(0.82)>Tub-α3(0.91)>GAPDH(0.98)。这个结果说明Tub-α6和EF-1β是表达较为稳定的内参基因,而GAPDH表达最不稳定。此外,通过geNorm软件计算出配对变异值Vn/n+1。Vn/n+1为选择阈值,即当Vn/n+1<0.15时,n个基因即可满足内参基因的要求。图6B的结果显示出V4/5为0.144,即需要选择4个内参基因Tub-α6、EF-1β、Actin和Ubc7。
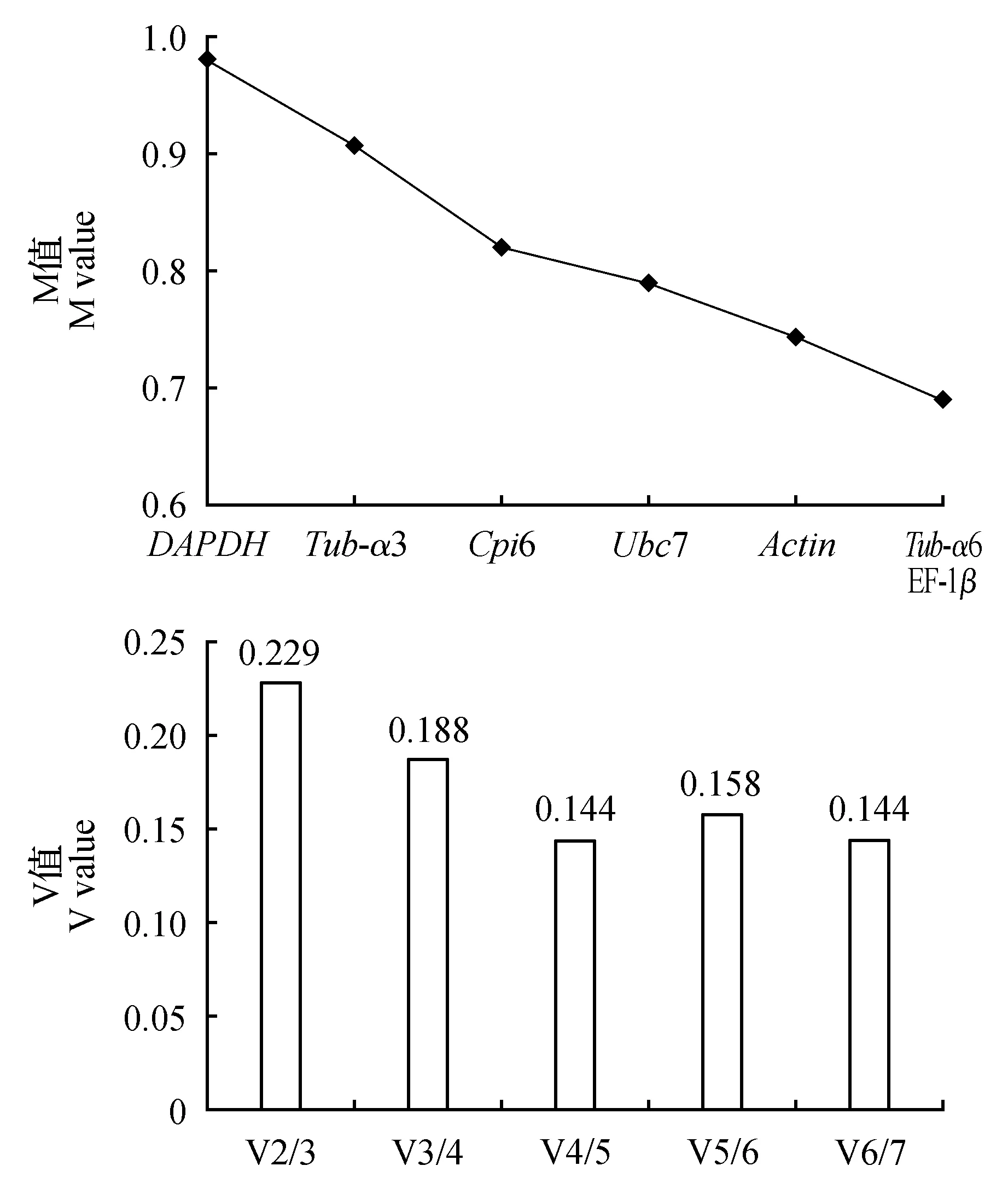
图6 不同组织候选内参基因的M值(A)和V值(B)Fig.6 M values(A) or V values(B) of reference genes indifferent tissues by geNorm analysis
BestKeeper软件是以标准差(SD)和调节系数标准差SD[±x-fold]来评判基因的表达稳定性,其值越小则表示越稳定,越大则表示越不稳定。如表2所示,在羽衣甘蓝不同组织中7个候选基因的表达稳定性为顺序为Ubc7(0.41)>Tub-α6(0.43)>Actin(0.58)>Tub-α3(0.68)>EF-1β(0.72)>Cpi6(0.92)>GAPDH(1.00),这结果表明BestKeeper软件默认Ubc7和Tub-α6是较为稳定的候选内参基因。综合geNorm软件和BestKeeper软件分析的结果,认为Tub-α6是羽衣甘蓝不同组织中表达稳定的最佳候选内参基因。
2.5不同发育时期柱头内参基因表达稳定性的统计分析
为了分析羽衣甘蓝不同发育时期柱头的最佳候选内参基因,采用geNorm软件对7个候选内参基因的表达稳定性进行分析。图7A的结果表明表达稳定的候选内参基因顺序为Actin/Ubc7(0.193)>Cpi6(0.330)>Tub-α6(0.436)>EF-1β(0.486)>Tub-α3(0.530)>GAPDH(0.577)。这个结果表明Actin和Ubc7是表达稳定的内参基因。而且所有的V值均小于0.15,因此认为选择Actin和Ubc7两个内参基因可满足稳定性需求(图7B)。此外,BestKeeper软件分析结果7个候选基因稳定性顺序为EF-1β(0.33)>Tub-α6(0.35)>Cpi6(0.36)>Actin(0.41)>GAPDH(0.41)>Tub-α3(0.45)>Ubc7(0.51),其中表达较为稳定的是EF-1β和Tub-α6(表3),且所有候选基因均符合内参稳定性要求即std dev[±CP]小于1。综合geNorm软件和BestKeeper软件分析的结果,认为Actin是羽衣甘蓝柱头发育时期中表达稳定的最佳候选内参基因。
表2利用BestKeeper软件对羽衣甘蓝不同组织7个候选内参基因的统计分析
Table2SummarystatisticsgeneratedbytheBestKeeperanalysisfor7candidatereferencegenesinthedifferenttissuesofornamentalkale

Ubc7Tub⁃α6ActinTub⁃α3EF⁃1βCpi6GAPDHn8888888geoMean[CP]28.7319.6825.0720.0222.5720.8018.09arMean[CP]28.7419.6825.0820.0422.5820.8318.13min[CP]27.7818.8224.1818.6021.2519.1316.73max[CP]29.9520.9725.9521.2223.7121.9220.18stddev[±CP]0.410.430.580.680.720.921.00CV[%CP]1.422.182.323.383.204.415.53min[x-fold]-1.94-1.81-1.86-2.68-2.50-3.19-2.57max[x-fold]2.322.451.832.312.202.174.26stddev[±x-fold]1.331.351.501.601.651.892.00Stabilityrank1234567
表3利用BestKeeper软件对羽衣甘蓝不同发育时期柱头组织7个候选内参基因的统计分析
Table3SummarystatisticsgeneratedbytheBestKeeperanalysisfor7candidatereferencegenesinthedifferentdevelopmentalstigmasofornamentalkale

EF⁃1βTub⁃α6Cpi6ActinGAPDHTub⁃α3Ubc7n5555555geoMean[CP]23.4920.6518.3625.6124.1721.8030.30arMean[CP]23.4920.6518.3625.6224.1721.8130.31min[CP]23.1920.2517.8624.9223.5621.3129.53max[CP]24.3121.3618.7526.0324.8722.6330.83stddev[±CP]0.330.350.360.410.410.450.51CV[%CP]1.401.671.981.581.702.041.67min[x-fold]-1.23-1.32-1.41-1.61-1.53-1.40-1.71max[x-fold]1.771.641.311.341.631.781.45stddev[±x-fold]1.261.271.291.321.331.361.42Stabilityrank1234567

图7 不同发育时期柱头候选内参基因的M值(A)和V值(B)Fig.7 M values(A) or V values(B) of reference genes in different developmental stigmas by geNorm analysis
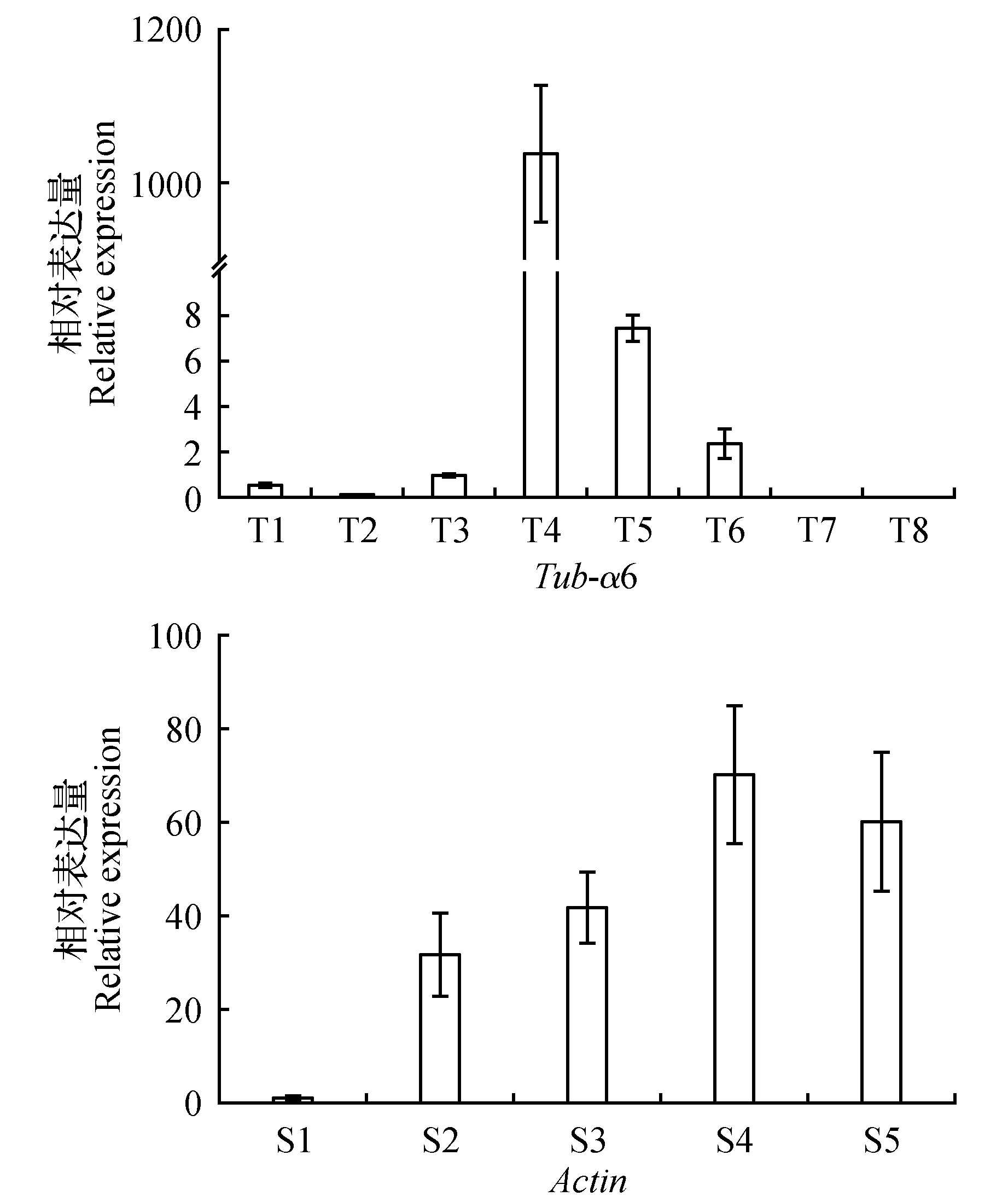
图8 SLG基因在不同组织(A)和不同发育阶段柱头(B)的表达分析Fig.8 Expression analysis of SLG gene in different tissues(A) and developmental stigmas(B) of ornamental kale
2.6SLG在不同组织和不同发育时期柱头的表达分析
SLG基因表达具有柱头组织表达特异性,并且与柱头发育成熟性正相关[6~7]。因此,通过对SLG的表达分析可以验证筛选到的内参基因的可行性。在不同组织中,以候选基因Tub-α6作为内参基因,结果发现SLG主要在柱头组织中表达并且表达量是其它组织表达量的1 000倍左右(图8:A);在不同发育时期的柱头中,以候选基因Actin作为内参基因,发现SLG主要随着柱头的成熟而表达量增高,而且在柱头接近成熟的时候(8~10 mm大小的花蕾)达到表达量高峰(图8:B)。上述结果说明,在羽衣甘蓝不同组织和不同发育时期柱头的荧光定量PCR研究中,可以采用Tub-α6和Actin作为内参基因进行分析。
3 讨论
在qRT-PCR分析中,内参基因的选择是定量分析的前提[19]。目前,GAPDH[20]、Actin[21]、18s rRNA[22]、EF-1α(elongation factor 1-alpha)[23]、ubiquitin[24]等是目前常被选择的内参基因。而且发现,在不同物种或同一物种的不同组织选择的内参基因可能会不同,如在拟南芥不同组织、发育及胁迫反应中选择的最佳内参基因不同[25~27];在白菜(BrassicarapaL. ssp.pekinensis)花芽发育中表达最稳定的基因是Tubulin和GAPDH[28],而在不同干旱条件下表达最稳定的内参基因是Actin和GAPDH[29]。此外,在内参基因筛选统计分析中,目前主要采用geNorm、Bestkeeper和Normfinder[30]等软件。这些软件主要采取不同的统计算法对内参基因稳定性进行分析。在本文的研究中,候选基因Actin、Cpi6、EF-1β、GAPDH、Tub-α3、Tub-α6、Ubc7的表达水平在不同组织中差异较大,但在不同发育时期柱头中表达情况基本稳定(图5)。采用geNorm和Bestkeeper两种软件对候选基因表达数据进行分析。在不同组织表达分析中,geNorm软件将Tub-α6和EF-1β排在最稳定的位置,Bestkeeper软件将Ubc7和Tub-α6排在比较稳定的位置,由于两个软件内参稳定性的算法不同,因此内参基因稳定性排序不同是可能的,最终认为Tub-α6是不同组织中最佳的内参基因;在不同发育阶段的柱头中,geNorm软件将Actin和Ubc7排在最稳定的位置,Bestkeeper软件将EF-1β和Tub-α6排在比较稳定的位置,且所有的候选基因均符合内参稳定性要求即std dev[±CP]小于1,最终认为在不同发育时期柱头中最佳内参基因是Actin。
利用筛选到的内参基因对SLG在不同组织和不同发育阶段的柱头进行表达分析,发现SLG在柱头中的表达量是其它组织表达量1 000倍左右,而且也发现SLG在柱头接近成熟时表达量达到最高,这个研究结果与预期的结果较为一致,同时也说明了筛选的内参基因可以应用到其它目标基因的定量表达分析中。这些研究结果的发现对基因在不同组织及柱头发育阶段的表达研究,提供了更多的线索。
1.Li Y H,Yu X Y.Pollination with laser-irradiated pollens breaks cross-incompatibility between zicaitai(Brassicacampestrisvar.purpurea) and ornamental kale(Brassicaoleraceavar.acephala) to produce hybrids with the aid of ovule culture[J].Scientia Horticulturae,2006,108(4):397-402.
2.解莉楠,丁兵,李玉花.羽衣甘蓝新品种‘红罗裙’和‘白罗裙’[J].园艺学报,2007,34(4):1071.
Xie L N,Ding B,Li Y H.New ornamental kale cultivars of ‘Hongluoqun’ and ‘Bailuoqun’[J].Acta Horticulturae Sinica,2007,34(4):1071.
3.王江,丁兵,李玉花.观赏羽衣甘蓝新品种‘粉玉’[J].园艺学报,2014,41(11):2369-2370.
Wang J,Ding B,Li Y H.A new ornamental kale cultivar ‘Fenyu’[J].Acta Horticulturae Sinica,2014,41(11):2369-2370.
4.Lan X G,Yang J,Cao M M,et al.Isolation and characterization of a J domain protein that interacts with ARC1 from ornamental kale(Brassicaoleraceavar.acephala)[J].Plant Cell Reports,2015,34(5):817-829.
5.Horisaki A,Niikura S.Developmental and environmental factors affecting level of self-incompatibility response inBrassicarapaL.[J].Sexual Plant Reproduction,2008,21(2):123-132.
6.Nasrallah J B,Kao T H,Goldberg M L,et al.A cDNA clone encoding an S-locus-specific glycoprotein fromBrassicaoleracea[J].Nature,1985,318(6043):263-267.
7.Stein J C,Howlett B,Boyes D C,et al.Molecular cloning of a putative receptor protein kinase gene encoded at the self-incompatibility locus ofBrassicaoleracea[J].Proceedings of the National Academy of Sciences of the United States of America,1991,88(19):8816-8820.
8.Goring D R,Rothstein S J.The S-locus receptor kinase gene in a self-incompatibleBrassicanapusline encodes a functional serine/threonine kinase[J].The Plant Cell,1992,4(10):1273-1281.
9.Gu T S,Mazzurco M,Sulaman W,et al.Binding of an arm repeat protein to the kinase domain of the S-locus receptor kinase[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(1):382-387.
10.Takasaki T,Hatakeyama K,Suzuki G,et al.The S receptor kinase determines self-incompatibility inBrassicastigma[J].Nature,2000,403(6772):913-916.
11.Murase K,Shiba H,Iwano M,et al.A membrane-anchored protein kinase involved inBrassicaself-incompatibility signaling[J].Science,2004,303(5663):1516-1519.
12.蓝兴国,杨佳,赵昕,等.羽衣甘蓝ARC1的基因分离、表达及与SRK相互作用的分析[J].园艺学报,2011,38(12):2342-2348.
Lan X G,Yang J,Zhao X,et al.Isolation expression of ARC1 from ornamental kale and interaction analysis between ARC1 and SRK[J].Acta Horticulturae Sinica,2011,38(12):2342-2348.
13.Indriolo E,Safavian D,Goring D R.The ARC1 E3 ligase promotes two different self-pollen avoidance traits inArabidopsis[J].The Plant Cell,2014,26(4):1525-1543.
14.Bustin S A.Quantification of mRNA using real-time reverse transcription PCR(RT-PCR):trends and problems[J].Journal of Molecular Endocrinology,2002,29(1):23-39.
15.Nolan T,Hands R E,Bustin S A.Quantification of mRNA using real-time RT-PCR[J].Nature Protocols,2006,1(3):1559-1582.
16.Vandesompele J,Depreter K,Pattyn F,et al.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J].Genome Biology,2002,3(7):research0034
17.Pfaffl M W.A new mathematical model for relative quantification in real-time RT-PCR[J].Nucleic Acids Research,2001,29(9):e45.
18.Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTMethod[J].Methods,2001,25(4):402-408.
19.Bustin S A,Beaulieu J F,Huggett J,et al.MIQE précis:practical implementation of minimum standard guidelines for fluorescence-based quantitative real-time PCR experiments[J].BMC Molecular Biology,2010,11:74.
20.Magneschi L,Kudahettige R L,Alpi A,et al.Expansin gene expression and anoxic coleoptile elongation in rice cultivars[J].Journal of Plant Physiology,2009,166(14):1576-1580.
21.Bracha-Drori K,Shichrur K,Lubetzky T C,et al.Functional analysis ofArabidopsispostprenylation CaaX processing enzymes and their function in subcellular protein targeting[J].Plant Physiology,2008,148(1):119-131.
22.Brentner L B,Mukherji S T,Merchie K M,et al.Expression of glutathione S-transferases in poplar trees(Populustrichocarpa) exposed to 2,4,6-trinitrotoluene(TNT)[J].Chemosphere,2008,73(5):657-662.
23.Bomal C,Bedon F,Caron S,et al.Involvement of Pinus taeda MYB1 and MYB8 in phenylpropanoid metabolism and secondary cell wall biogenesis:a comparative in planta analysis[J].Journal of Experimental Botany,2008,59(14):3925-3939.
24.Aquea F,Gutierrez F,Medina C,et al.A novel Otubain-like cysteine protease gene is preferentially expressed during somatic embryogenesis inPinusradiata[J].Molecular Biology Reports,2008,35(4):567-573.
25.Guénin S,Mauriat M,Pelloux J,et al.Normalization of qRT-PCR data:the necessity of adopting a systematic,experimental conditions-specific,validation of references[J].Journal of Experimental Botany,2009,60(2):487-493.
26.Kitashiba H,Liu P,Nishio T,et al.Functional test of Brassica self-incompatibility modifiers inArabidopsisthaliana[J].Proceedings of the National Academy of Sciences of the United States of America,2011,108(44):18173-18178.
27.Indriolo E,Tharmapalan P,Wright S I,et al.The ARC1 E3 ligase gene is frequently deleted in self-compatible Brassicaceae species and has a conserved role inArabidopsislyrataself-pollen rejection[J].The Plant Cell,2012,24(11):4607-4620.
28.Xu X Y,Yang Z P,Sun X L,et al.Selection of reference genes for quantitative real-time PCR during flower bud development in CMS7311 of heading Chinese cabbage(BrassicarapaL.ssp.pekinensis)[J].Acta Physiologiae Plantarum,2014,36(3):809-814.
29.Qi J N,Yu S C,Zhang F L,et al.Reference gene selection for real-time quantitative polymerase chain reaction of mRNA transcript levels in Chinese cabbage(BrassicarapaL.ssp.pekinensis)[J].Plant Molecular Biology Reporter,2010,28(4):597-604.
30.Andersen C L,Jensen J L,Ørntoft T F.Normalization of real-time quantitative reverse transcription-PCR data:a model-based variance estimation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets[J].Cancer Research,2004,64(15):5245-5250.
Central Universities Fundamental Research Special Fund Project(DL13CA13);National Natural Science Foundation of China(31070275)
introduction:Li Han(1991—),female,master student,mainly engaged in plant developmental biology.
date:2015-12-17
SelectionofReferenceGenesforReal-timeFluorescenceQuantitativePCRinDifferentTissuesandStigmaDevelopmentfromOrnamentalKale
LI Han LI Zhi-Long LI Xiao-Yu LI Yu-Hua LAN Xing-Guo*
(College of Life Sciences,Northeast Forestry University,Harbin 150040)
We used real-time quantitative polymerase chain reaction to test the expression ofActin, cysteine proteinase inhibitor 6(Cpi6), elongation factor 1-beta(EF-1β), glyceraldehyde-3-phosphate dehydrogenase(GAPDH), tubulin alpha-3(Tub-α3), tubulin alpha-6(Tub-α6) and ubiquitin conjugating enzyme7(Ubc7), and then identified the most suitable reference genes in a given set of tissues and different developmental stigmas of ornamental kale(Brassicaoleraceavar.acephala)S13-bS13-bhomozygotes by using the geNorm and BestKeeper software programs. By a geNorm analysis,Tub-α6 andEF-1βwere the most suitable reference genes among the given set of tissues, andActinandUbc7 were the most stable genes during stigma development. By a BestKeeper analysis,Ubc7 andTub-α6 were the most suitable reference genes among the given set of tissues, andEF-1βandTub-α6 were the most stable genes during stigma development.Tub-α6 andActinwere the most suitable reference genes among the given set of tissues and during stigma development, respectively. Furthermore, the expression ofSLGnormalized withTub-α6 orActinshowed thatSLGwas predominantly expressed in stigma among the given set of tissues and reached its highest level at approaching anthesis during stigma development.
Brassicaoleraceavar.acephala;real-time quantitative PCR;reference genes;stigma development;SLG
中央高校基本科研业务费专项基金项目(DL13CA13);国家自然科学基金项目(31070275)
李晗(1991—),女,硕士研究生,主要从事植物发育生物学研究。
* 通信作者:E-mail:lanxingguo@126.com
2015-12-17
* Corresponding author:E-mail:lanxingguo@126.com
S635
A
10.7525/j.issn.1673-5102.2016.04.012

