玉米籽粒发育相关基因ZmMADS-RIN的克隆及表达分析
夏 雪 马晨雨 白青河 冯园园 王顺友 王 喜 周青利 席章营
河南农业大学农学院 / 小麦玉米作物学国家重点实验室, 河南郑州450002
玉米籽粒发育相关基因ZmMADS-RIN的克隆及表达分析
夏 雪 马晨雨 白青河 冯园园 王顺友 王 喜 周青利 席章营*
河南农业大学农学院 / 小麦玉米作物学国家重点实验室, 河南郑州450002
玉米籽粒发育及产量是玉米重要的经济性状。本研究以玉米骨干自交系郑58为试材, 采用同源克隆法得到一个与玉米籽粒发育相关的基因ZmMADS-RIN。该基因的cDNA全长859 bp, 开放阅读框为768 bp, 编码255个氨基酸, 与玉米B73中所对应的cDNA的编码区序列相比, 共有6个SNP位点的差异, 3个氨基酸残基发生了变化。生物信息学分析表明, ZmMADS-RIN蛋白的相对分子量为29.23 kD, 理论等电点为8.84, 是一种亲水性蛋白, 不含信号肽序列, 也不含跨膜结构; 具有5个磷酸化位点; 二级结构中含有50.20%的α-螺旋(alpha helix)、27.45%的无规则卷曲(random coil)、14.51%的延伸链(extended strand)和7.84%的β-转角(beta turn)。该蛋白位于细胞核内; 其氨基酸序列中含有高度保守的MADS结构域、相对保守的K结构域、低保守的I结构域和最不稳定的C结构域, 是典型的MIKC 型MADS-box蛋白; 系统进化树分析表明, ZmMADS-RIN蛋白属于AGL6分支, 与水稻基因OsMADS6的相似性最高, 为89%。ZmMADS-RIN基因在玉米自交系郑58的籽粒中特异表达, 在根及不同时期的叶片中没有检测到表达信号。荧光定量PCR结果显示, ZmMADS-RIN基因的表达量在玉米籽粒发育的不同阶段呈一定规律的变化, 在授粉后0~20 d, 呈上调表达趋势, 授粉后25 d表达量迅速下降至较低水平, 而在授粉后30~40 d则检测不到其表达。推测该基因可能与玉米籽粒的发育有关。
玉米; 籽粒发育; ZmMADS-RIN; 基因克隆; RT-PCR
MADS-box基因编码一种重要的转录因子, 参与调节真核生物的发育过程和信号转导[1], 该种基因的名称是由酿酒酵母细胞调节因子 MCM1 (Minichromosome maintenance 1)、拟南芥花器官特征基因 AG (AGAMOUS)、金鱼草花器官特征基因DEF (DEFICIENS)和人类血清应答因子SRF (Serum Response Factor)的蛋白首字母的缩写组成的[2-3]。研究表明, 这种转录因子中都含有一个由 56~58个氨基酸组成的高度保守的MADS-box结构域, 故将具有这一结构域的基因统称为MADS-box基因[2-3]。
目前, 已经从不同的植物中分离到了大量的MADS-box基因, 例如, 拟南芥中有132个MADS-box基因, 水稻中有97个MADS-box基因, 番茄中有 36个 MADS-box基因[4-5], 大豆中有 106个MADS-box基因[6]等。大量研究证明, 植物中的MADS-box基因表达的蛋白能与真核基因启动子区域中的顺式作用元件特异结合, 从而使靶基因以特定的强度在特定的时间和空间表达[7], 并在生物体中通过调节其他基因的转录来发挥其调控作用。植物MADS-box基因的功能主要集中在开花时间的调控、分生组织和花器官功能决定等方面, 包括营养器官如叶和根的发育甚至根瘤的形成[8-9]、顶端分生组织的分化[10-11]、花器官的决定[2]、激素信号的转导[12]、开花时间的控制[13]、光合作用和营养代谢的调控[14]、以及非生物胁迫应答[4]等。近年来有关MADS-box基因对果实的发育和成熟[15-17]以及胚的发育[18-19]的调节作用的研究越来越多, 在水稻[20]、大豆[15]、番茄[16,21-22]、拟南芥[23-24]、草莓[25-26]等物种上均有报道, 说明这个基因家族具有不同的调节功能[27-28]。
众所周知, 植物的果实是人类和动物食物中的重要组成部分, 因此, 研究果实的发育和成熟对于确保人类的食物安全具有重要意义。番茄通常被作为一种研究果实发育和成熟的模式作物, 研究番茄果实成熟机制的文献比较多, 也发现了许多番茄果实成熟缺陷的突变体, 如成熟抑制(ripening inhibitor, rin)、从不成熟(never ripe, Nr)、不成熟(nonripening, nor)、颜色不成熟(color nonripening, cnr)等[17,29]。其中rin突变体具有较大的萼片, 并且抑制果实成熟。这种突变型是由于 2个 MADS-box转录因子(SlMADS-MC和 SlMADS-RIN)的功能缺失造成的。其中SlMADS-MC参与萼片的发育, 而SlMADS-RIN则调节果实成熟, 是番茄果实成熟过程中的一个重要的正向调控因子, 其与乙烯的生物合成、感知乙烯和乙烯响应有关[17]。该基因的发现, 对于研究果实发育与成熟机制具有重要的意义。
在玉米中, 有142个MADS-box基因被注释在植物转录因子数据库(plant transcription factor database PlnTFDB)中, 其中的36个已经被克隆[4,30]。它们中的大多数都参与花发育的调控, 如卵细胞、小花、小穗的发育以及花粉管生长、花药开裂和花粉成熟[4]等。Malcomber等[31]发现玉米的LEAFY HULL STERILE1 (LHS1)基因在小穗、小花中不同的表达模式导致了花序形态结构的变化。ZMM8和 ZMM14这两个基因只在发育小穗的上位花中表达, 对上位小花分生组织的分化起决定性作用[7]。ZAG3 (即bde)[32]能够影响玉米分生组织的发育和决定性, 在bde突变体中, 上部花分生组织促进形成额外的镶嵌或者融合花器官, 而下部分生组织则产生一些额外的花分生组织。Becker等[7]发现玉米的 ZAG2基因在胚珠中特异表达, 该基因在胚珠和心皮的内表面上表达, 其氨基酸序列与水稻胚珠特异基因OsMADS13[33]的同源性为 65.31%, 属于 MADS-box基因家族中的AGAMOUS(AG)-like亚家族。相对而言, MADS-box基因对于玉米籽粒发育调控的研究非常少。
为了研究玉米中MADS-box基因对籽粒发育的调控, 本研究利用番茄中调节果实成熟发育的基因LeMADS-RIN (AF448522)[17,34]的氨基酸序列对玉米B73的all maize protein sequences数据库(http://www.maizegdb.org/)进行同源搜索, 发现一个未知其功能的 MADS-box蛋白(GenBank登录号为 ACF80378,其对应的基因 GenBank登录号为 BT035373.1), 其氨基酸序列与番茄LeMADS-RIN的氨基酸序列的同源性为 53.57%, 经序列比对分析发现, 该基因的氨基酸序列与辣椒中调节果实成熟发育的基因 CaRIN (DQ999998)[3 5]、草莓中果实特异表达的基因FaMADS9 (AF484683)[26]、葡萄中籽粒发育相关基因VvMADS39 (XM_002263003)[36]和香蕉中果实发育与成熟相关的基因MuMADS1 (DQ060444)[37]的氨基酸序列的同源性分别为52%、51%、51%和52%, 说明它与以上调控果实发育的基因有着相对较高的同源性, 并且该基因中含有 MADS-box基因家族高度保守的MADS结构域, 以及相对较保守的K结构域,故将其命名为ZmMADS-RIN。本研究以玉米骨干自交系郑58为试验材料, 从其籽粒中克隆了 ZmMADSRIN基因, 并进行表达分析, 旨在为进一步研究该基因调控玉米籽粒发育和成熟机制奠定基础。
1 材料与方法
1.1 材料
1.1.1 试验材料 玉米骨干自交系郑58, 由河南农业大学农学院提供。将其种子播种于装有蛭石的花盆中, 长至三叶期时, 用无菌水清洗根和叶片,再用干净的吸水纸吸干, 液氮冷冻后保存于-80℃冰箱中。
将玉米骨干自交系郑58的种子于2015年6月种植于河南农业大学郑州毛庄科教园区(34°51′54.66″ N, 113°35′12.11″ E), 在大田中随机选取生长良好且长势一致的植株, 分别于五叶期、十叶期采集其叶片, 3次重复; 自交授粉当天标记授粉日期, 随后取授粉后0、5、10、15、20、25、30、35、40 d的籽粒, 液氮冷冻后保存于-80℃冰箱中备用。
1.1.2 主要试剂和菌株 柱式植物 RNA提取试剂盒CAT#: 71203购自北京天恩泽基因科技有限公司; FastQuant RT Kit (with gDNase)第1链合成试剂盒购自天根生化科技有限公司; Premix Taq (LA Taq Version 2.0 plus dye)、pMD18-T Vector Cloning Kit均购自 TaKaRa (大连)公司; 大肠杆菌 DH5α Competent Cell、琼脂糖凝胶纯化回收试剂盒、细菌生长培养基配制所需试剂(Tryptone, Yeart Extract, 琼脂糖)均购自康为世纪公司; 实时荧光定量试剂盒GoTaq qPCR Master Mix购自Promega (北京)生物技术有限公司。
1.2 方法
1.2.1 RNA提取和cDNA合成 取适量试验材料,置于经高温处理并用液氮预冷过的研钵中, 加液氮研磨, 按照天恩泽 CAT#: 71203试剂盒的操作说明提取玉米骨干自交系郑 58的幼苗长至三叶期的根和叶片, 长至五叶期、十叶期的叶片, 以及授粉后0、5、10、15、20、25、30、35、40 d的籽粒的总RNA,并用分光光度计检测 RNA的纯度, 用 1.5%琼脂糖凝胶电泳检测 RNA的完整性, 于-80℃冰箱保存。用FastQuant RT Kit (with gDNase)第1链合成试剂盒反转录合成 cDNA第 1链, 并以此为模板, 以玉米18S基因[38]作为内参对照设计引物18S-F、18S-R (表1), PCR扩增玉米18S基因, 扩增片段长度为174 bp, PCR反应结束后取10 μL反应物, 用1.5%琼脂糖凝胶电泳检测第1链合成的质量。将反转录产物储存于-20℃冰箱中用于下一步实验。
1.2.2 特异性引物设计及克隆 根据目前已公布的玉米 B73 all maize protein sequences数据库(http://www.maizegdb.org/)中 MADS-box蛋白(GenBank登录号为 ACF80378, 其对应的基因 GenBank登录号为BT035373.1)的cDNA序列, 利用Primer 5.0在开放阅读框两端设计引物ZmMADS-RIN-F、ZmMADS-RIN-R(表1), 以玉米骨干自交系郑 58授粉后 5 d的籽粒的cDNA为模板, 扩增ZmMADS-RIN基因的全长cDNA序列, PCR产物经1.5%琼脂糖凝胶电泳检测, 并使用琼脂糖凝胶纯化回收试剂盒纯化回收目的条带, 连接pMD-18T载体, 转化DH5α感受态细胞并涂板, 将菌液PCR检测的阳性克隆送华大基因科技公司测序。
1.2.3 ZmMADS-RIN基因的生物信息学分析、序列比对以及进化树分析 利用在线工具 ProtParam tool分析ZmMADS-RIN基因编码的蛋白的理化性质;利用 Signalp 4.1 server软件预测信号肽; 利用TMHMM Server v.2.0在线软件分析跨膜结构; 利用在线工具 SOPMA软件分析蛋白的二级结构; 运用Netphos 2.0 server软件预测磷酸化位点; 用PSORT II Prediction预测亚细胞定位; 运用ProtScale在线分析软件预测疏水性/亲水性; 利用 SMART网站(http://smart.embl-heidelberg.de/)分析蛋白的结构域;使用DNAMAN Version 6.0软件将ZmMADS-RIN与B73的all maize protein sequences数据库中GenBank登录号为 BT035373.1的基因的编码区序列和对应的氨基酸序列进行差异分析, 同时与番茄 LeMADSRIN (AF448522)[17,34]、辣椒CaRIN (DQ999998)[35]、草莓 FaMADS9 (AF484683)[26]、葡萄 VvMADS39 (XM_002263003)[36]和香蕉MuMADS1 (DQ060444)[37]基因的氨基酸序列比对分析; 用MEGA 6软件结合NCBI网站(http://www.ncbi.nlm.nih.gov/)上的蛋白数据库将 ZmMADS-RIN蛋白序列与玉米中已克隆的36个MADS-box蛋白序列[4,30], 以及番茄中的13个、水稻中的7个和拟南芥中的15个, 共72个MADS-box基因的蛋白序列进行分子进化树分析。
1.2.4 ZmMADS-RIN基因相对表达量分析 以玉米骨干自交系郑 58不同部位(三叶期的根和叶片,五叶期和十叶期的叶片, 以及授粉后5 d的籽粒)的cDNA 为模板, 通过 RT-PCR 反应, 进行ZmMADS-RIN基因器官特异性表达分析。反应体系含Premix Taq (LA Taq Version 2.0 plus dye) 12.5 µL,引物 ZmMADS-RIN-F、ZmMADS-RIN-R (表1)(10 μmol L–1)各0.8 μL, cDNA模板2 μL, 加水至终体积25 μL。反应条件为95℃ 180 s; 94℃ 30 s, 55℃ 50 s, 72℃ 60 s, 35个循环; 72℃ 600 s; 4℃保存。同时以玉米18S基因[38]作为内参对照, 将扩增产物在1.5%的琼脂糖凝胶上电泳分离。
以玉米骨干自交系郑58不同生长时期的籽粒的cDNA为模板, 以玉米18S基因[38]作为内参基因, 将目的基因的 cDNA序列与基因组 DNA序列比对,在基因的编码序列中跨内含子设计荧光定量PCR引物Q-PCR-F、Q-PCR-R (表1), PCR产物长度为199 bp。按照 Promega的实时荧光定量试剂盒 GoTaq qPCR Master Mix的说明书操作, 反应体系含GoTaq qPCR Master Mix 2 ×12.5 µL, 引物 Q-PCR-F、Q-PCR-R (表1)(10 μmol L–1)各0.8 μL, cDNA模板2 μL, 加水至终体积25 μL。使用Biorad chorom 4实时荧光定量PCR仪扩增, 反应条件为94℃ 180 s; 94 ℃ 10 s, 58℃ 20 s, 72℃ 20 s, 40个循环; 72℃读取荧光值; 循环结束后 60℃保温 60 s; 进行熔解曲线分析, 由熔解曲线判定PCR反应的特异性。进行3 次PCR重复, 参照基因的2–ΔΔCt法[39]计算结果。
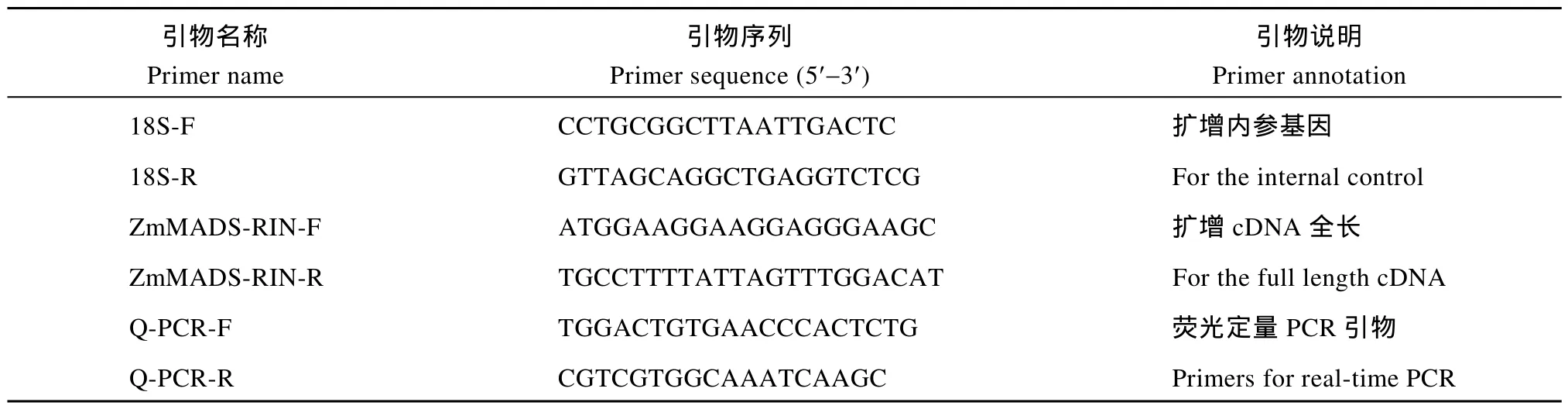
表1 基因克隆及其荧光定量PCR分析所用引物Table1 Primers used for gene cloning and analysis of real-time PCR
2 结果与分析
2.1 玉米自交系郑58总RNA的提取与cDNA第1链的合成、检测
用1.5%琼脂糖凝胶电泳检测, 提取的玉米各部分的RNA条带清晰, 说明RNA完整性良好, 无明显降解。对经FastQuant RT Kit (with gDNase)试剂盒反转录合成的cDNA第1链进行18S基因[38]内参对照扩增检测, 琼脂糖凝胶电泳结果显示, 目的条带清晰, 说明反转录的cDNA质量良好。
2.2 ZmMADS-RIN基因的克隆
以反转录的授粉后5 d的籽粒的cDNA第1链为模板, 利用设计的引物 ZmMADS-RIN-F、ZmMADS-RIN-R进行聚合酶链式反应扩增, 扩增产物用1.5%琼脂糖凝胶电泳检测得到1条约1000 bp的条带, 和预测的目的片段大小一致(图1)。目的条带经回收、连接、转化后, 挑取单克隆进行菌液检测。将阳性菌液测序经BioXM 2.6处理发现, 该基因的cDNA全长为859 bp, 开放阅读框为768 bp, 将其与玉米B73的all maize protein sequences数据库中BT035373.1对应的cDNA的编码区序列比对(图2)发现, 共有 6个核苷酸(SNP)位点的差异, 对应有3个氨基酸残基发生了变化。
2.3 ZmMADS-RIN蛋白生物信息学分析
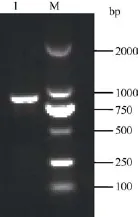
图1 ZmMADS-RIN基因的cDNA扩增结果Fig.1 Amplification results of ZmMADS-RIN cDNA
ZmMADS-RIN基因编码的蛋白生物信息学分析发现, 该蛋白由 255个氨基酸组成, 分子式为C1275H2028N380O381S14, 相对分子量为29.23 kD, 理论等电点(pI)为 8.84, 其不稳定参数为 49.18, 属于不稳定蛋白。该基因编码的多肽含亮氨酸(Leu, L)最多, 达 11.4%; 其次是谷氨酸(Glu, E), 为 9.80%。其中,带负电荷的氨基酸(Asp +Glu)残基总数为29个, 带正电荷的氨基酸(Arg +Lys)残基总数为34个, 不含吡咯赖氨酸(Pyl, O)和硒半胱氨酸(Sec, U), 其总的疏水性平均系数(GRAVY)为-0.704, 属于亲水性蛋白。该蛋白不含信号肽序列, 属于非分泌蛋白; 也不含跨膜结构, 为非跨膜蛋白。该蛋白的二级结构类型为α-螺旋(alpha helix)50.20%; 无规则卷曲(random coil)和延伸链(extended strand)分别为27.45%和14.51%; β-转角(beta turn)的比例最小, 为7.84%。该蛋白序列中含有16个丝氨酸(S), 其中5个可能是磷酸化位点。该蛋白位于细胞核内(细胞核: 60.9%, 细胞质: 17.4%, 线粒体: 17.4%, 细胞骨架: 4.3%)。

图2 郑58 ZmMADS-RIN与B73 BT035373.1的编码区的差异分析Fig.2 Variable analysis between ZmMADS-RIN of Zheng 58 and BT035373.1 of B73 in coding region方框内表示具有差异的编码序列。The differences in coding region are marked with box.
ZmMADS-RIN蛋白的亲水性和疏水性预测分析(图3)显示, 在33~50位氨基酸表现极强的疏水性,其他序列则均表现亲水性, 总体来说表现亲水性,与之前预测的总的疏水性平均系数(GRAVY)值的预测结果相一致。
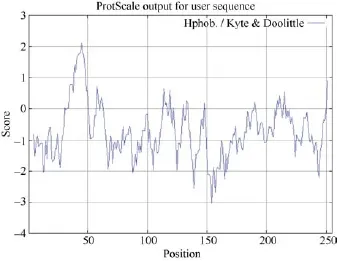
图3 ZmMADS-RIN蛋白的亲水性/疏水性预测Fig.3 Prediction of hydrophilicity/hydrophobicity for ZmMADS-RIN protein
该蛋白含有 MADS结构域(1~60), 具有与特异DNA结合和蛋白质二聚化等功能; 以及 K结构域(118~175), 形成 3个 α螺旋组成的卷曲—卷曲(coiled-coil)结构, 参与蛋白质间的相互作用。将ZmMADS-RIN 蛋 白 与 番 茄 LeMADS-RIN (AAM15775.1)[17,34]、辣椒CaRIN (ABJ98752.1)[35]、草莓FaMADS9 (AAO49380.1)[26]、葡萄VvMADS39 (XP_002263039.1)[36]和香蕉MuMADS1 (AAY53908.1)[37]的氨基酸序列比对(图4), 发现这6个物种的氨基酸序列中的MADS结构域和K结构域在物种间具有高度的相似性。
进化树(图5)分析表明, ZmMADS-RIN基因的蛋白序列属于AGL6分支, 其他的玉米MADS-box基因的蛋白序列则属于不同的亚家族。ZmMADS-RIN基因的蛋白序列与AGL6分支中的一个调控胚珠发育的水稻基因OsMADS6[40]的相似度最高, 为89%。

图4 玉米ZmMADS-RIN蛋白与其他植物物种蛋白的氨基酸序列比对Fig.4 Homology analysis of the deduced amino acid sequences from ZmMADS-RIN and those of other plant species
2.4 ZmMADS-RIN基因表达分析
2.4.1 组织特异性表达分析 半定量 RT-PCR分析结果(图6)表明, 该基因主要在玉米籽粒中表达,而在三叶期的根和叶片、五叶期以及十叶期的叶片中均没有检测到表达信号。
2.4.2 在籽粒发育过程中的表达分析 图7表明,该基因的表达量在玉米自交系郑58籽粒发育的不同阶段呈现一定规律的变化, 在授粉后0~20 d, 籽粒中ZmMADS-RIN基因的表达量呈上调趋势, 且在授粉后20 d达最高峰, 之后迅速下降, 在授粉后25 d已降到较低水平, 而在授粉后30~40 d则检测不到其表达。
3 讨论
序列分析结果显示, ZmMADS-RIN基因所推导的氨基酸序列有高度保守的MADS结构域, 相对保守的K结构域, 低保守的I结构域和最不稳定的C结构域(图4), 说明该蛋白是典型的 MIKC型MADS-box蛋白。系统进化分析结果显示, ZmMADSRIN基因的蛋白序列属于 AGL6分枝, 其蛋白序列与 AGL6分支中的一个调控胚珠发育的水稻基因OsMADS6[40]的蛋白序列的相似度最高, 为89%。组织特异性表达分析显示, ZmMADS-RIN基因在玉米籽粒中高度表达。以上结果表明, ZmMADS-RIN基因可能参与籽粒的发育。一般来说, MADS-box基因的表达部位与其功能密切相关。本研究中 ZmMADSRIN基因的这种表达模式与其他植物中调控果实发育的MADS-box基因的表达模式相似, 即主要在生殖器官中表达。如水稻中调控胚珠发育的基因OsMADS6[40]在花和发育中的种子中高度表达, 但是在根、叶和悬浮细胞中没有检测到, OsMADS13[33]主要在胚珠中大量表达, 而 OsMADS21[41]只在发育的种子中表达; 大豆种子发育调控的基因GmAGL15[15]在幼胚中表达量最高; 番茄中与果实发育有关的基因TAG1、TAGL1、TAGL2和TAGL11[42]只在花和果实中表达, SlMADS1[29]在萼片和果实中高度表达, LeMADS-RIN[17,34]主要在果实中表达; 拟南芥中调控籽粒发育的基因SHP1和SHP2[28]在籽粒中高度表达, 调控果实开裂。
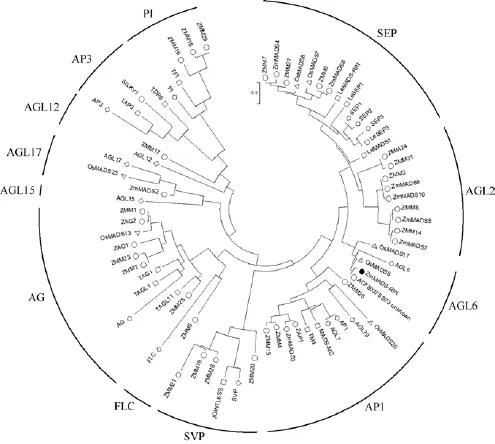
图5 玉米、番茄、水稻和拟南芥中MADS-box蛋白的进化树分析Fig.5 Phylogenetic analysis of MADS-box proteins from Zea mays, Solanum lycopersicum, Oryza sativa, and Arabidopsis thaliana
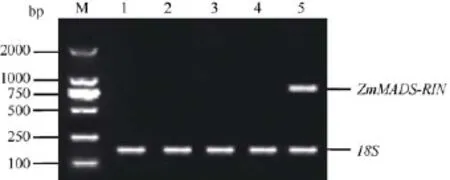
图6 ZmMADS-RIN基因在不同器官中的表达Fig.6 RT-PCR analysis of ZmMADS-RIN gene in different organs
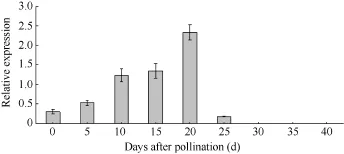
图7 ZmMADS-RIN基因在不同授粉天数的籽粒中的表达Fig.7 Expression of ZmMADS-RIN gene in developing kernel
近年来的研究表明, 大量MADS-box基因与果实发育和成熟相关, 因此研究其在果实中的表达趋势, 对其功能的发掘有重要的意义。研究发现, 水稻的 OsMADS6基因[40]在胚珠中减数分裂时期大量表达, 授粉后10 d的胚乳中表达量最高, 授粉后20 d在胚乳中的表达量略有降低; 而OsMADS13基因[33]在胚珠中高度表达, 并随着胚珠的发育表达量不断提高。大豆的 GmAGL15基因[15]在大豆发育的种子中都有表达, 它在花后15 d的幼胚中表达量比较高,出现第一个峰值, 而在花后30 d的幼胚中表达量最高, 然后迅速下降, 这种表达模式明显与种子的发育过程有关。番茄的 TAG1基因受精前在胚珠和心皮中高度表达, 受精后大大降低, 然后随着果实的发育逐渐减少直至没有; TAGL1基因和TAGL11基因在花和种子中的表达模式相似, 优先表达于发育的种子中, 受精后仍然高度表达, 且没有明显变化; TAGL2基因在种子中高度表达, 但是随着种子的成熟而不断减少[42]; SlMADS1基因[29]的转录物主要积累在果实中, 并随着果实的发育和成熟而不断减少; 而LeMADS-RIN基因[17,34]的表达量在果实中随着果实的成熟而不断提高。由此看来, 不同的MADS-box家族基因在植物的果实发育过程中的表达趋势不尽相同。本研究表明, ZmMADS-RIN基因的表达量在玉米自交系郑58籽粒发育的不同阶段呈现一定规律的变化, 在授粉后0~20 d, 籽粒中ZmMADS-RIN基因的表达量呈上调趋势, 且在授粉后 20 d达最高峰,之后迅速下降, 在授粉后 25 d已降到较低水平, 而在授粉后30~40 d则检测不到其表达。这些结果表明, ZmMADS-RIN基因可能与玉米籽粒的发育有关。同时, 参考张冬梅等[43]的研究结果, 玉米骨干自交系郑58的籽粒灌浆速率整体表现为快–快–慢的节奏,即籽粒的平均灌浆速率表现出在授粉后前20 d迅速增快, 授粉后20~30 d缓慢加快, 而在授粉后30~40 d明显减慢的趋势。很明显, 玉米骨干自交系郑 58的籽粒的平均灌浆速率的变化趋势与本研究中ZmMADS-RIN基因的表达趋势基本一致, 说明ZmMADS-RIN基因很可能与玉米籽粒的平均灌浆速率有关。至于该基因在调控玉米籽粒发育过程的机制中的确切作用, 将在后续研究中探讨。
4 结论
玉米骨干自交系郑58的ZmMADS-RIN基因与玉米B73中所对应的cDNA的编码区序列相比, 共有6 个SNP位点的差异, 对应的氨基酸序列有3个氨基酸残基发生了变化。ZmMADS-RIN蛋白是一种亲水性蛋白, 不含信号肽序列, 位于细胞核内, 具有高度保守的MADS结构域, 相对保守的K结构域, 低保守的I结构域和最不稳定的 C结构域, 是典型的 MIKC型MADS-box蛋白, 与水稻基因OsMADS6的蛋白序列的相似性最高。ZmMADS- RIN基因在玉米籽粒中高度表达, 其表达量在玉米籽粒发育的不同阶段呈现一定的变化规律, 授粉后0~20 d表达上调, 出现一个明显的表达高峰, 授粉后 25 d表达量迅速下降至较低水平,而在授粉后30~40 d则检测不到其表达。ZmMADS-RIN基因很可能与玉米籽粒的平均灌浆速率有关。
[1] Ng M, Yanofsky M F.Function and evolution of the plant MADS-box gene family.Nat Rev Genet, 2001, 2: 186–195
[2] Alvarez-Buylla E R, Liljegren S J, Pelaz S, Gold S E, Burgeff C, Ditta G S, Vergara-Silva F, Yanofsky M F.MADS-box gene evolution beyond flowers: expression in pollen, endosperm, guard cells, roots and trichomes.Plant J, 2000, 24: 457–466
[3] de Bodt S, Raes J, Florquin K, Rombauts S, Rouzé P, Theißen G, Van de Peer Y.Genome wide structural annotation and evolutionary analysis of the type I MADS-box genes in plants.J Mol Evol, 2003, 56: 573–586
[4] Zhang Z B, Li H Y, Zhang D F, Liu Y H, Fu J, Shi Y S, Song Y C, Wang T Y, Li Y.Characterization and expression analysis of six MADS-box genes in maize (Zea mays L.).J Plant Physiol, 2012, 169: 797–806
[5] Leseberg C H, Eissler C L, Wang X, Johns M A, Duvall M R, Mao L.Interaction study of MADS-domain proteins in tomato.J Exp Bot, 2008, 59: 2253–2265
[6] Shu Y J, Yu D S, Wang D, Guo D L, Guo C H.Genome-wide survey and expression analysis of the MADS-box gene family in soybean.Mol Biol Rep, 2013, 40: 3901–3911
[7] Becker A, Theißen G.The major clades of MADS-box genes and their role in the development and evolution of flowering plants.Mol Phylogenet Evol, 2003, 29: 464–489
[8] Ku A T, Huang Y S, Wang Y S, Ma D F, Yeh K W.IbMADS1 (Ipomoea batatas MADS-box 1 gene) is involved in tuberous root initiation in sweet potato (Ipomoea batatas).Ann Bot, 2008, 102: 57–67
[9] Benlloch R, Roque E, Ferrándiz C, Cosson V, Caballero T, Penmetsa R V, Beltrán J P, Cañas L A, Ratet P, Madueño F.Analysis of B function in legumes: PISTILLATA proteins do not require the PI motif for floral organ development in Medicago truncatula.Plant J, 2009, 60: 102–111
[10] Ferrario S, Shchennikova A V, Franken J, Immink R G H, Angenent G C.Control of floral meristem determinacy in petunia by MADS-box transcription factors.Plant Physiol, 2006, 140: 890–898
[11] 韩利涛, 姜伟, 杨守萍, 喻德跃, 盖钧镒.大豆细胞质雄性不育系 MADS-box基因的分离分析.作物学报, 2010, 36: 905–910
Han L T, Jiang W, Yang S P, Yu D Y, Gai J Y.Isolation and analysis of MADS-box gene from soybean (Glycine max L.Merr.) cytoplasmic male sterile line.Acta Agron Sin, 2010, 36: 905–910 (in Chinese with English abstract)
[12] Kaufmann K, Muiño J M, Jauregui R, Airoldi C A, Smaczniak C, Krajewski P, Angenent G C.Target genes of the MADS transcription factor SEPALLATA3: Integration of developmental and hormonal pathways in the Arabidopsis flower.PLoS Biol, 2009, 7: 854–875
[13] Michaels S D, Ditta G, Gustafson-Brown C, Pelaz S, Yanofsky M, Amasino R M.AGL24 acts as a promoter of flowering in Arabidopsis and is positively regulated by vernalization.Plant J, 2003, 33: 867–874
[14] Mara C D, Irish V F.Two GATA transcription factors are downstream effectors of floral homeotic gene action in Arabidopsis.Plant Physiol, 2008, 147: 707–718
[15] 汪潇琳, 陈艳萍, 喻德跃.MADS-box基因GmAGL15在大豆种子发育过程中的表达.作物学报, 2008, 34: 330–332
Wang X L, Chen Y P, Yu D Y.Expression of the MADS-box geneGmAGL15 in seed development of soybean.Acta Agron Sin, 2008, 34: 330–332 (in Chinese with English abstract)
[16] Ito Y, Kitagawa M, Ihashi N, Yabe K, Kimbara J, Yasuda J, Ito H, Inakuma T, Hiroi S, Kasumi T.DNA-binding specificity, transcriptional activation potential, and the rin mutation effect for the tomato fruit-ripening regulator RIN.Plant J, 2008, 55: 212–223
[17] Vrebalov J, Ruezinsky D, Padmanabhan V, White R, Medrano D, Drake R, Schuch W, Giovannoni J.A MADS-box gene necessary for fruit ripening at the tomato Ripening-Inhibitor (Rin) locus.Science, 2002, 296: 343–346
[18] Battaglia R, Brambilla V, Colombo L, Stuitje A R, Kater M M.Functional analysis of MADS-box genes controlling ovule development in Arabidopsis using the ethanol-inducible alc gene-expression system.Mechanisms Development, 2006, 123: 267–276
[19] Moyle R, Fairbairn D J, Ripi J, Crowe M, Botella J R.Developing pineapple fruit has a small transcriptome dominated by metallothionein.J Exp Bot, 2005, 56: 101–112
[20] Dreni L, Jacchia S, Fornara F, Fornari M, Ouwerkerk P B F, An G, Colombo L, Kater M M.The D-lineage MADS-box gene Os-MADS13 controls ovule identity in rice.Plant J, 2007, 52: 690–699
[21] Mazzucato A, Olimpieri I, Siligato F, Picarella M E, Soressi G P.Characterization of genes controlling stamen identity and development in a parthenocarpic tomato mutant indicates a role for the DEFICIENS ortholog in the control of fruit set.Physiol Plant, 2008, 132: 526–537
[22] 刘菊华, 徐碧玉, 张静, 金志强.MADS-box转录因子的相互作用及对果实发育和成熟的调控.遗传, 2010, 32: 893–902
Liu J H, Xu B Y, Zhang J, Jin Z Q.The interaction of MADS-box transcription factors and manipulating fruit development and ripening.Hereditas (Beijing), 2010, 32: 893–902 (in Chinese with English abstract)
[23] Colombo M, Masiero S, Vanzulli S, Lardelli P, Kater M M, Colombo L.AGL23, a type I MADS-box gene that controls female gametophyte and embryo development in Arabidopsis.Plant J, 2008, 54: 1037–1048
[24] Dinneny J R, Weigel D, Yanofsky M F.A genetic framework for fruit patterning in Arabidopsis thaliana.Development, 2005, 132: 4687–4696
[25] Seymour G B, Ryder C D, Cevik V, Hammond J P, Popovich A, King G J, Vrebalov J, Giovannoni J J, Manning K.A SEPALLATA gene is involved in the development and ripening of strawberry (Fragaria×ananassa Duch.) fruit, a nonclimacteric tissue.J Exp Bot, 2011, 62: 1179–1188
[26] 周厚成, 李刚, 赵霞, 康兆茹, 郭蔼光.森林草莓全基因组MADS-box转录因子基因的鉴定及凤梨草莓果实FaMADS1基因克隆.植物生理学报, 2014, 50: 1630–1638
Zhou H C, Li G, Zhao X, Kang Z R, Guo A G.Genome-wide sequence identification of MADS-box transcription factor gene family from Fragaria vesca and cloning of FaMADS1 gene from F.ananassa fruit.Plant Physiol J, 2014, 50: 1630–1638 (in Chinese with English abstract)
[27] Ferrándiz C, Liljegren S J, Yanofsky M F.Negative regulation of the SHATTERPROOF genes by FRUITFULL during Arabidopsis fruit development.Science, 2000, 289: 436–438
[28] Liljegren S J, Ditta G S, Eshed Y, Savidge B, Bowman J L, Yanofsky M F.SHATTERPROOF MADS-box genes control seed dispersal in Arabidopsis.Nature, 2000, 404: 766–770
[29] Dong T T, Hu Z L, Deng L, Wang Y, Zhu M K, Zhang J L, Chen G P.A tomato MADS-box transcription factor, SlMADS1, acts as a negative regulator of fruit ripening.Plant Physiol, 2013, 163: 1026–1036
[30] Münster T, Deleu W, Wingen L U, Ouzunova M, Cacharrón J, Faigl W, Werth S, Kim J T T, Saedler H, Theißen G.Maize MADS-box genes galore.Maydica, 2002, 47: 287–301
[31] Malcomber S T, Kellogg E A.Heterogeneous expression patterns and separate roles of the SEPALLATA gene LEAFY HULL STERILE1 in grasses.Plant Cell, 2004, 16: 1692–1706
[32] Thompson B E, Bartling L, Whipple C, Hall D H, Sakai H, Schmidt R, Hake S.bearded-ear encodes a MADS box transcription factor critical for maize floral development.Plant Cell, 2009, 21: 2578–2590
[33] Lopez-Dee Z P, Wittich P, Pè M E, Rigola D, Buono I D, Gorla M S, Kater M M, Colombo L.OsMADS13, a novel rice MADS-box gene expressed during ovule development.Dev Genet, 1999, 25: 237–244
[34] Moore S, Vrebalov J, Payton P, Giovannoni J.Use of genomics tools to isolate key ripening genes analyse fruit maturation in tomato.J Exp Bot, 2002, 53: 2023–2030
[35] Dong T T, Chen G P, Tian S B, Xie Q L, Yin W C, Zhang Y J, Hu Z L.A non-climacteric fruit gene CaMADS-RIN regulates fruit ripening and ethylene biosynthesis in climacteric fruit.PLoS One, 2014, 9: e95559
[36] Wang L, Yin X J, Cheng C X, Wang H, Guo R R, Xu X Z, Zhao J, Zheng Y, Wang X P.Evolutionary and expression analysis of a MADS-box gene superfamily involved in ovule development of seeded and seedless grapevines.Mol Genet Genomics, 2015, 290: 825–846
[37] Liu J H, Xu B Y, Hu L F, Li M Y, Su W, Wu J, Yang J H, Jin Z Q.Involvement of a banana MADS-box transcription factor gene in ethyleneinduced fruit ripening.Plant Cell Rep, 2009, 28: 103–111
[38] Wang X T, Wu L J, Zhang S F, Wu L C, Ku L X, Wei X M, Xie L L, Chen Y H.Robust expression and association of ZmCCA1 with circadian.Plant Cell Rep, 2011, 30: 1261–1272
[39] Rajeevan M S, Ranamukhaarachi D G, Vernon S D, Unger E R.Use of real-time quantitative PCR to validate the results of cDNA array and differential display PCR technologies.Methods, 2001, 25: 443–451
[40] Zhang J, Nallamilli B R, Mujahid H, Peng Z H.OsMADS6 plays an essential role in endosperm nutrient accumulation and is subject to epigenetic regulation in rice (Oryza sativa).Plant J, 2010, 64: 604–617
[41] Lee S, Kim J, Son J S, Nam J, Jeong D H, Lee K, Jang S, Yoo J, Lee J, Lee D Y, Kang H G, An G.Systematic reverse genetic screening of T-DNA tagged genes in rice for functional genomic analyses: MADS-box genes as a test case.Plant Cell Physiol, 2003, 44: 1403–1411
[42] Busi V M, Bustamante C, Angelo C D, Hidalgo-Cuevas M, Boggio S B, Valle E M, Zabaleta E.MADS-box genes expressed during tomato seed and fruit development.Plant Mol Biol, 2003,52: 801–815
[43] 张冬梅, 刘洋, 赵永锋, 祝丽英, 黄亚群, 郭晋杰, 陈景堂.不同杂种优势群玉米籽粒灌浆速率分析.中国农业科学, 2014, 47: 3323–3335
Zhang D M, Liu Y, Zhao Y F, Zhu L Y, Huang Y Q, Guo J J, Chen J T.Analysis of maize grain filling rate in different heterotic groups.Sci Agric Sin, 2014, 47: 3323–3335 (in Chinese with English abstract)
Cloning and Expression Analysis of ZmMADS-RIN Gene for Regulating the Kernel Development of Maize
XIA Xue, MA Chen-Yu, BAI Qing-He, FENG Yuan-Yuan, WANG Shun-You, WANG Xi, ZHOU Qing-Li, and XI Zhang-Ying*
College of Agronomy, Henan Agricultural University / State Key Laboratory of Wheat and Maize Crop Science, Zhengzhou 450002, China
Kernel development is the most important character of maize.In this study, a maize gene named ZmMADS-RIN encoding MADS-box protein relating to kernel development was isolated.The full length cDNA of ZmMADS-RIN is 859 bp, and it contains an open reading frame (ORF) with 768 nucleotides and encodes 255 amino acid residues.The variable analysis of ZmMADS-RIN in coding region showed that six nucleotides and three amino acid residues were different between Zheng 58 and B73.Bioinformatics analyses showed that ZmMADS-RIN was a hydrophilic protein with a molecular weight of 29.23 kD, a theoretical isoelectric point of 8.84, and five phosphorylation sites, but signal peptide sequence and transmembrane domain were not found.The secondary structure of ZmMADS-RIN protein contained 50.20% of alpha helix, 27.45% of random coil, 14.51% of extended strand and 7.84% of beta turn.ZmMADS-RIN protein was tested to be a typical MIKC-type protein localized in nucleus with highly conserved MADS domain, relatively conserved K domain, less weakly conserved I domain and most variable C domain.Phylogenetic analysis indicated that ZmMADS-RIN protein belongs to the same branch of AGL6, and shows high similarity of 89% to OsMADS6 protein from Oryza sativa.RT-PCR results showed that ZmMADS-RIN expressed in kernel specially, and not in root or leaf during different phases.In addition, qRT-PCR results indicated that the expression of ZmMADS-RIN increased continually from zero day to 20 days after pollination, and reached the highest level at the 20th day, then decreased significantly.And it could not be detected during 30th-40th days.These results demonstrated that ZmMADS-RIN might be associated with theregulation of developing kernel in maize.
Maize; Kernel development; ZmMADS-RIN; Gene cloning; RT-PCR
10.3724/SP.J.1006.2016.01656
本研究由国家自然科学基金项目(31371629)资助。
This study was supported by the National Natural Science Foundation of China (31371629).
*通讯作者(Corresponding author): 席章营, E-mail: xizhangying@163.com
联系方式: E-mail: xiaxue282522@163.com
稿日期): 2016-02-04; Accepted(接受日期): 2016-06-20; Published online(
日期): 2016-07-04.URL: http://www.cnki.net/kcms/detail/11.1809.S.20160704.0826.020.html

