华北落叶松种子园控制授粉子代遗传多样性分析
孙文婷,于大德,董明亮,赵 健,王小平,张鸿景,张金凤*
(1 林木育种国家工程实验室/林木、花卉遗传育种教育部重点实验室/国家林业局树木花卉育种与生物工程重点开放实验室/北京林业大学 生物科学与技术学院,北京100083;2 北京园林绿化局,北京100013;3河北省林业科学研究院,石家庄 050061)
华北落叶松种子园控制授粉子代遗传多样性分析
孙文婷1,于大德1,董明亮1,赵 健1,王小平2,张鸿景3,张金凤1*
(1 林木育种国家工程实验室/林木、花卉遗传育种教育部重点实验室/国家林业局树木花卉育种与生物工程重点开放实验室/北京林业大学 生物科学与技术学院,北京100083;2 北京园林绿化局,北京100013;3河北省林业科学研究院,石家庄 050061)
以华北落叶松控制授粉群体的全部子代为材料,母本相同的个体视为同一家系,利用18对SSR分子标记对7个家系257个个体进行扩增,分析其遗传多样性及其遗传分化水平。结果表明:(1)18个SSR位点共检测到72个等位基因,平均等位基因数4个,有效等位基因数(Ne) 为1.247~3.411。(2) 7个家系的平均有效等位基因数为2.135个,观测杂合度(Ho)为0.518,期望杂合度(He)为0.502,Shannon信息指数0.846,其中55号家系遗传多样性水平最高,56号的遗传多样性水平最低。(3)遗传分化系数(GST)为0.113,各家系群体处于中等遗传分化水平,AMOVA分析结果显示82%的遗传变异存在于家系内,18%的遗传变异存在于家系间。(4)聚类分析结果表明,55号与59号家系的遗传距离最近,具有较近的亲缘关系,49号家系与其他家系的遗传距离最远。(5)结合遗传多样性及遗传分化水平结果,估算获得选择核心家系数及核心个体数,选择5个家系均可获得96%以上的遗传多样性,对于个体数较少的家系,选择15~20个个体;对于个体数较多的家系,选择35个个体,即可获得96%以上的遗传多样性。该研究结果对华北落叶松种子园育种群体的选择及其遗传多样性的保护具有重要意义。
华北落叶松;家系;SSR分子标记;遗传多样性
华北落叶松(Larixgmeliniivar.principis-rupprechtiiMayr.)属于松科(Pinaceae)落叶松属(Larix),天然群体主要分布在中国河北、山西两省,北京和内蒙古最南部也有少量分布。华北落叶松生长速度快、木材材质好、用途广、抗腐朽耐力强,是营造经济生态林的良好树种,是中国华北地区山地的主要造林树种。
种子园在实现林木良种化的过程中发挥着重要作用[1],不仅是提供大量优质种子的繁殖基地,本身又是由低级向高级发展的育种系统中的一个重要环节[2]。种子园的建立是针叶树育种最有效的方法之一[3]。华北落叶松的育种主要采用种子园形式,中国现有国家级落叶松良种基地18个,大多建有种子园。通常种子园中通过不断加大选择强度对上下世代进行选择,以此获得具有目标性状的家系或无性系。虽然可以将生长较差的个体淘汰,但群体的遗传基础可能变窄,导致遗传多样性降低,影响种子园良种的繁殖力度以及对生态系统的适应性[4]。为了更好地利用并维持种子园群体较宽的遗传基础,了解种子园各群体的遗传多样性水平尤为重要。
SSR分子标记,即简单重复序列(simple sequence repeat),属于共显性分子标记,具有数量丰富、多态性高、通用性好等特点[5-6],每个微卫星DNA的核心序列结构相同,重复单位数目10~60个,其高度多态性主要来源于串联数目的不同[7]。已证明很多植物中存在微卫星高信息量,同时是位点特异的分子标记[8],因此,在落叶松属遗传多样性的研究方面有广泛的应用。范英明等[9]对河北省8个华北落叶松天然群体312个个体的遗传多样性进行了SSR分子标记分析;贯春雨等[10]运用了RAPD、SSR对日本落叶松与兴安落叶松的杂种进行了遗传多样性分析。杨秀艳等[11]开发了日本落叶松的EST-SSR标记并对二代优树进行了遗传多样性分析;姚宇等[12]利用SSR标记分析了去劣疏伐对长白落叶松初级无性系种子园的遗传多样性影响。然而,该技术在华北落叶松种子园遗传多样性研究方面的应用较少,仅于大德等[13]对国家落叶松良种基地华北落叶松种子园的一至三代群体的遗传多样性进行了分析,研究表明一至三代群体遗传多样性水平没有明显差异,遗传变异主要存在于群体内,且子代与亲代遗传分化较小。
杂交育种是创造新的遗传型的育种方式,在林木育种中应用较为普遍。人工控制杂交集中了父母双方的有利基因,利用杂种优势,达到育种目标[1],但由于这种方法集中利用了父母本少数的优良性状,导致子代家系间亲缘关系相近,遗传基础相对较窄,所以还应考虑群体的遗传多样性水平。有关华北落叶松种子园控制授粉子代群体的遗传多样性方面的研究未见报道。本研究利用18对SSR分子标记对华北落叶松种子园控制授粉群体的遗传多样性进行分析,从分子水平上探讨了全同胞子代的遗传多样性水平和遗传分化程度,并提出家系及家系内个体的选择策略,旨在为进一步家系、品种选择和资源的有效利用提供理论指导。
1 材料和方法
1.1 材 料
试验材料取自于河北省承德市围场县龙头山国家华北落叶松良种基地种子园。该种子园始建于1976年,该场地处E117°45′、N41°56′,平均海拔1 386 m。在一代园子代测定林138个半同胞家系中,选择7个优良单株作为母本,5个优良单株作为父本进行人工控制授粉得到二代全同胞种子园。
2012年5月在良种基地采集二代全同胞种子园中的全部个体,按照相同的母本将子代分为7个家系,共257个个体。剪取顶端带有幼嫩叶子的枝条,每个单株3~5枝,装入带有硅胶的自封袋中,标记编号。带回实验室后,将嫩叶收集到自封袋,放入
-80 ℃冰箱储存待用。材料详细信息见表1。
1.2 方 法
1.2.1 SSR引物及PCR扩增 取各样本针叶进行液氮研磨,采用改良CTAB法[14]提取基因组DNA。利用0.8%琼脂糖凝胶电泳和Nanodrop2 000分光光度计对DNA浓度和纯度进行检测,最终稀释至50 ng/μL,-20 ℃保存待用。
研究所用18对SSR引物由本实验室自行设计开发,其中10对引物来自于董明亮[15]利用华北落叶松愈伤组织转录组测序序列设计开发的多态性引物,另外8对来自范英明[9]利用NCBI中松科物种EST序列开发的EST-SSR标记(表2)。引物由北京擎科生物科技公司合成。
PCR体系通过优化后,最终确定总体积为25 μL,其中dNTP (2.5 mmol/L) 2 μL、10×buffer(Mg2+plus) 2.5 μL、引物F、R(10 μmol/L)各1 μL、模板(50 ng/μL)2 μL、Taq酶(5U/μL) 0.2 μL、ddH2O 16.3 μL。PCR扩增程序如下:94 ℃预变性5 min,94 ℃变性30 s,在引物最适退火温度下退火30 s,72 ℃延伸45 s,共29个循环,72 ℃延伸5 min。扩增产物利用8%非变性聚丙烯酰胺凝胶进行电泳,通过银染及显色后,对目的条带进行人工判读,统计等位基因数据。
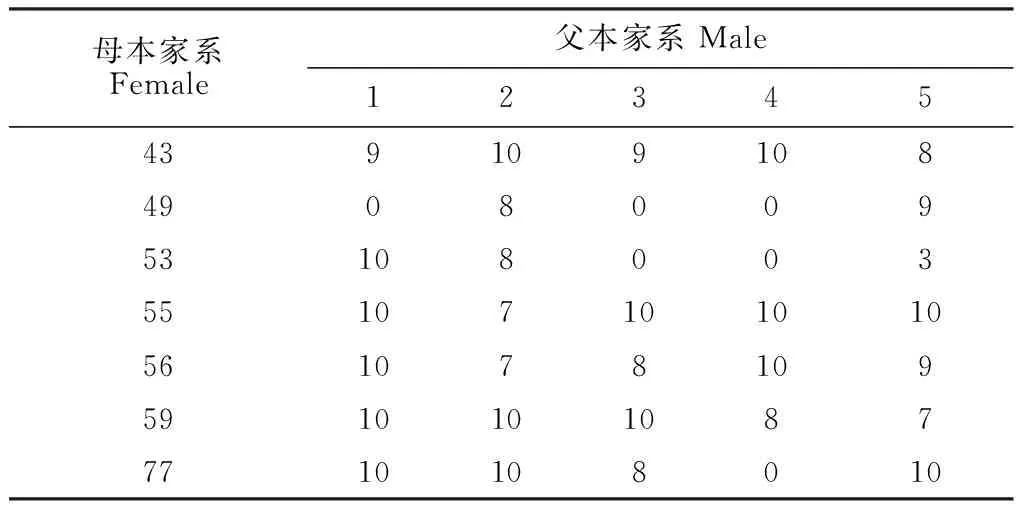
表1 华北落叶松种子园控制授粉子代统计表
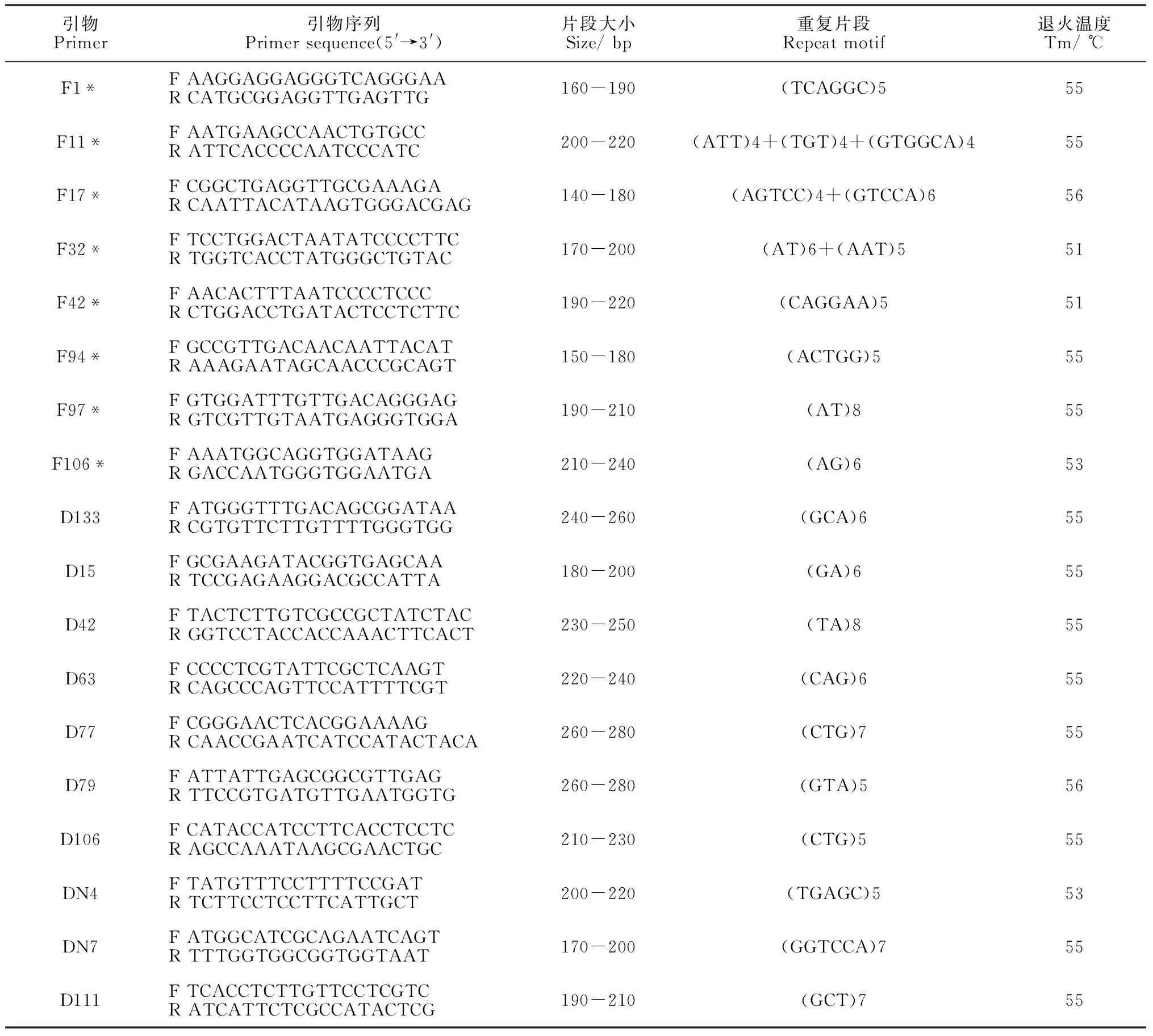
表2 18对SSR引物信息
注:“*”标注的引物来自范英明等[9],其他引物来自董明亮等[15]
Note: * were from Fan Yingmingetal[9],and the others were from Dong Mingliang et al[15]
1.2.2 数据处理及分析 利用POPGENE Version 1.32[16]软件分析各家系的多样性,计算各位点的等位基因数(Na)、有效等位基因数(Ne)、Shannon信息指数(I)、观测杂合度(Ho)、期望杂合度(He)等参数,以及分析各个群体的遗传多样性。利用PIC_CAL 0.6计算各个位点的多态信息含量(PIC)。通过GenALEx6.5和Excel2007计算各家系的Nei’s遗传一致度(GI)和遗传距离(GD),并利用分子方差分析(AMOVA)[17]对家系间与家系内的遗传变异分配情况进行估算。根据Nei’s遗传距离,通过NTSYSpc2.1软件采用非加权组平均法[18]进行聚类分析。
各个群体的遗传分化程度主要由总基因多样度(HT)、群体内基因多样度(HS)、群体间基因多样度(DST)、遗传分化系数(GST)等[19]参数进行衡量。涉及遗传参数计算公式如下:
DST=HS-HT;GST=DST/HT
式中S为总的群体数,wk、qk(i)为第k个群体的权重及其等位基因Ai的频率观察值,HS为把各个群体视为理想群体的期望杂合体频率平均值,HT为把整个群体视为理想群体的期望杂合体频率。
核心家系数量的估计是将家系按照1、2、3、4、5、6个家系样本共6个级别从7个华北落叶松家系中进行随机抽样,并以此代表整个华北落叶松群体,每个级别重复7次,然后运用POPGENE 软件计算相关群体的等位基因数,最后进行群体捕获曲线的拟合,找到等位基因数稳定时对应的家系数量,即核心家系数量。家系内核心个体数的估计是将7个家系内的个体,按照5、10、15、20、25、30、35、40、44株9个等级进行随机抽样(家系中个体数不足者以其个体数最高值代替),利用Excel产生的随机数抽取相应的样本,每个级别重复10次,统计每个级别的平均等位基因数和Shannon信息指数,并进行曲线的拟合,找出所需的等位基因数、Shannon信息指数对应的各家系群体内个体数量。
2 结果与分析
2.1 SSR位点的多态性
利用18对SSR引物对257个个体进行扩增,共检测到72个等位基因(表3),等位基因变异范围为2~6个,平均每个位点为4个,其中,引物D77和F106检测出6个等位基因,F11和DN4检测到2个等位基因。有效等位基因数为1.247~3.411,平均为2.312个,其中引物D63有效等位基因数最小(1.247),D133有效等位基因数最多(3.411)。有效等位基因数的差异说明各位点对群体遗传多样性的检测能力是不同的。18个位点的观测杂合度和期望杂合度平均为0.505和0.536,其中,D15、D63、 D77、D133、F32、F17、D79、F106、DN4的期望杂合度高于观测杂合度,说明这些位点表现为杂合子缺失。由表3可以看出,Shannon信息指数与多态信息含量在不同SSR位点存在一定差异。Shannon信息指数范围为:0.416~1.345,平均为0.957。多态信息量平均值为0.475,仅有2对引物的PIC值小于0.25,说明这2对引物的等位基因过于集中;有8对SSR引物的PIC值大于0.5,属于高度多态信息位点。综合上述数据信息,18对SSR引物能够检测出相对较多的等位位点,能够较好地反映群体的遗传多样性。
2.2 华北落叶松不同家系的遗传多样性
利用18对SSR引物对7个家系的257个个体进行遗传多样性分析,平均有效等位基因为2.135个,最高为55号家系(2.331),最低为49号家系(1.980)。7个家系的平均期望杂合度为0.502,除43、56、77号家系外,其他家系的期望杂合度均在0.5以上,遗传多样性水平较高。55号家系的遗传多样性水平最高,He为0.550,56号家系的遗传多样性水平最低,He为0.463。从表4中可以看出,43、56、59号家系的期望杂合度高于观测杂合度,说明这3个家系内存在杂合子过剩的现象。Shannon信息指数为0.778~0.931,平均值为0.846,这些群体具有较大的遗传变异,55号家系的变异最大,I为0.931。等位基因数、有效等位基因数、期望杂合度以及Shannon信息指数在7个家系中都存在一定的差异,说明这些群体存在丰富的遗传多样性。
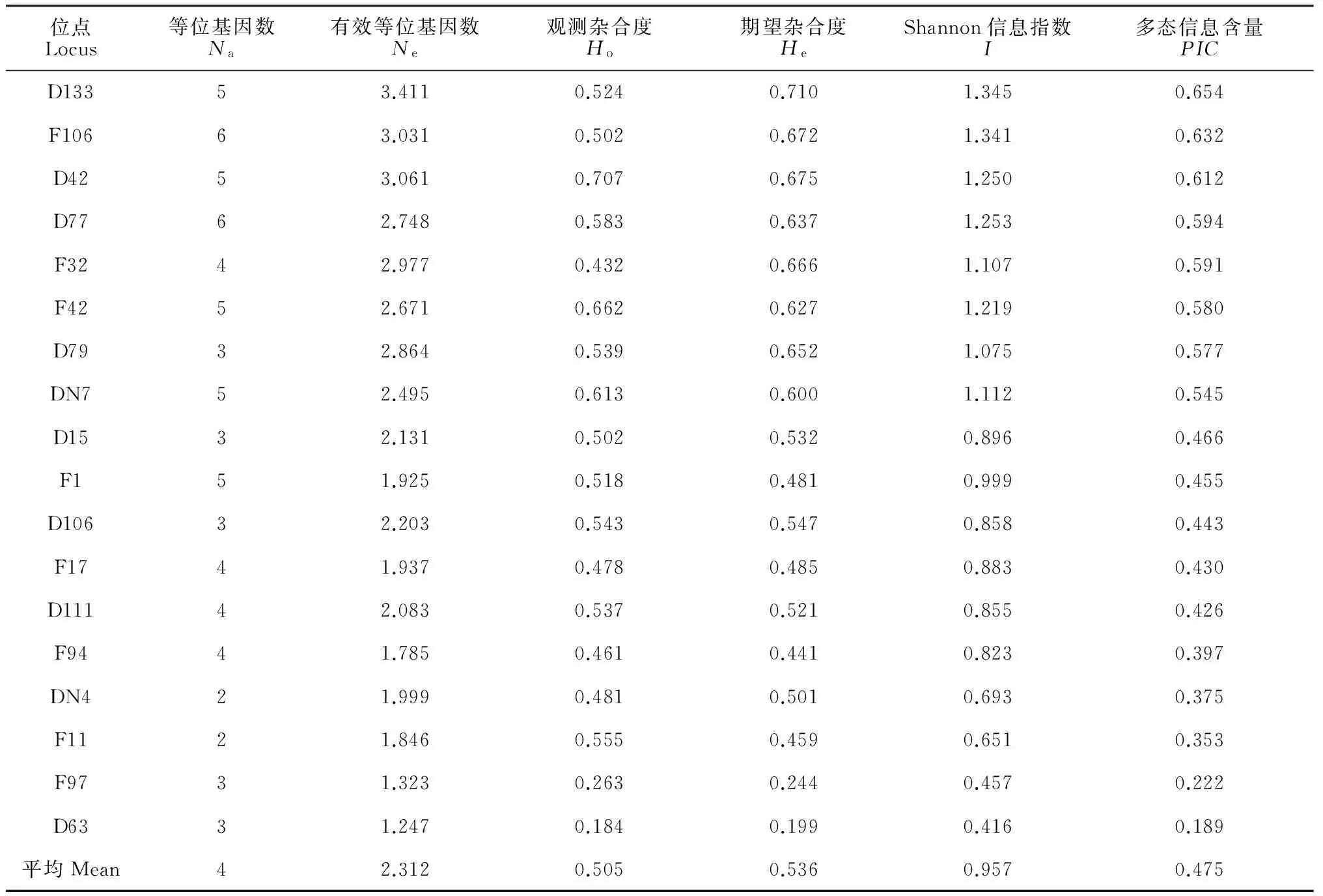
表3 18个SSR位点的遗传多样性信息
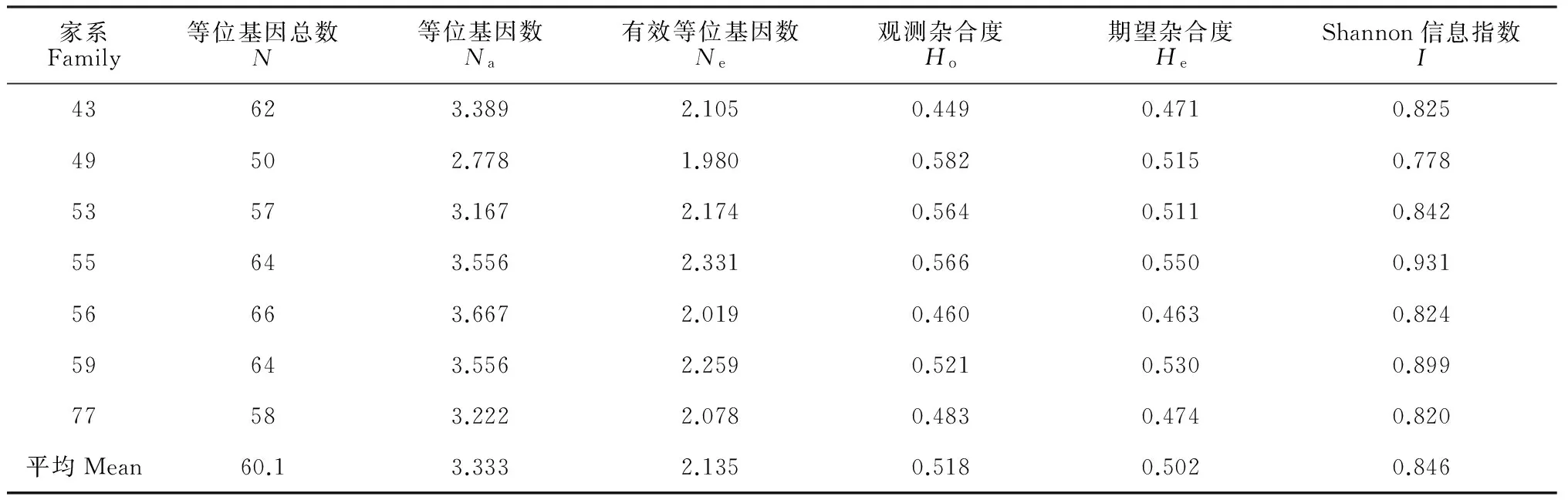
表4 7个华北落叶松家系的遗传多样性
2.3 华北落叶松不同家系的遗传分化
遗传分化系数是衡量种群间和种群内遗传分化程度的重要参数,当HT与HS相等时,GST为0,表明所有群体中等位基因频率相等。当HS等于0时,群体内部没有变异,群体中均为纯合体,这时GST为1[19]。由表5可知,平均总基因多样度(HT)为0.561,其中,引物D79的HT值最高(0.839);D63的HT值最低(0.203),说明各引物间的多样性水平具有一定的差异。总基因多样度(HT)与群体内基因多样度(HS)比较接近,从而使群体间基因多样度(DST)较小,平均值仅为0.071,遗传分化系数(GST)平均为0.113,说明华北落叶松各家系处于中等遗传分化程度,含有丰富的等位基因频率。
通过AMOVA分析表明(表6),华北落叶松的遗传变异主要存在于家系内部,占82%,18%的遗传变异存在于家系间,说明华北落叶松7个家系的遗传变异主要存在于家系内部。
2.4 华北落叶松不同家系的遗传一致度和遗传距离
表7显示,遗传距离的变化范围为0.046~0.217,遗传一致度的变化范围为0.805~0.955,两者变化范围较小。其中49号家系与53号家系、56号家系的遗传距离最远(0.217和0.203),遗传一致度最低(0.805和0.816);59号家系与55号家系的遗传距离最近(0.046),遗传一致度最高(0.955)。
基于Nei’s遗传距离对7个家系的UPGMA聚类结果图1显示。
在遗传距离0.1时,7个家系分为3组,第一组由49号家系独立组成,第二组由56号家系独立组成,第三组由43号、55号、59号、77号和53号5个家系组成,其中55号和59号家系的遗传关系最近,这与Nei’s遗传一致度和遗传距离的分析结果是一致的。
2.5 华北落叶松核心种质资源策略
2.5.1 核心家系估算 华北落叶松7个家系按照1、2、3、4、5、6个家系共6个级别进行随机抽样,以抽样群体代表整个华北落叶松群体,由图2可知,等位基因数随着抽样群体数量的增加而增加,6个不同取样群体的平均等位基因数(Na)为3.34、3.60、3.76、3.80、3.85、3.88,占全部群体平均等位基因数的83.5%、90.0%、94.0%、95.1%、96.2%、97.1%。因此,选择5个群体时就可以获得96%以上的等位基因。
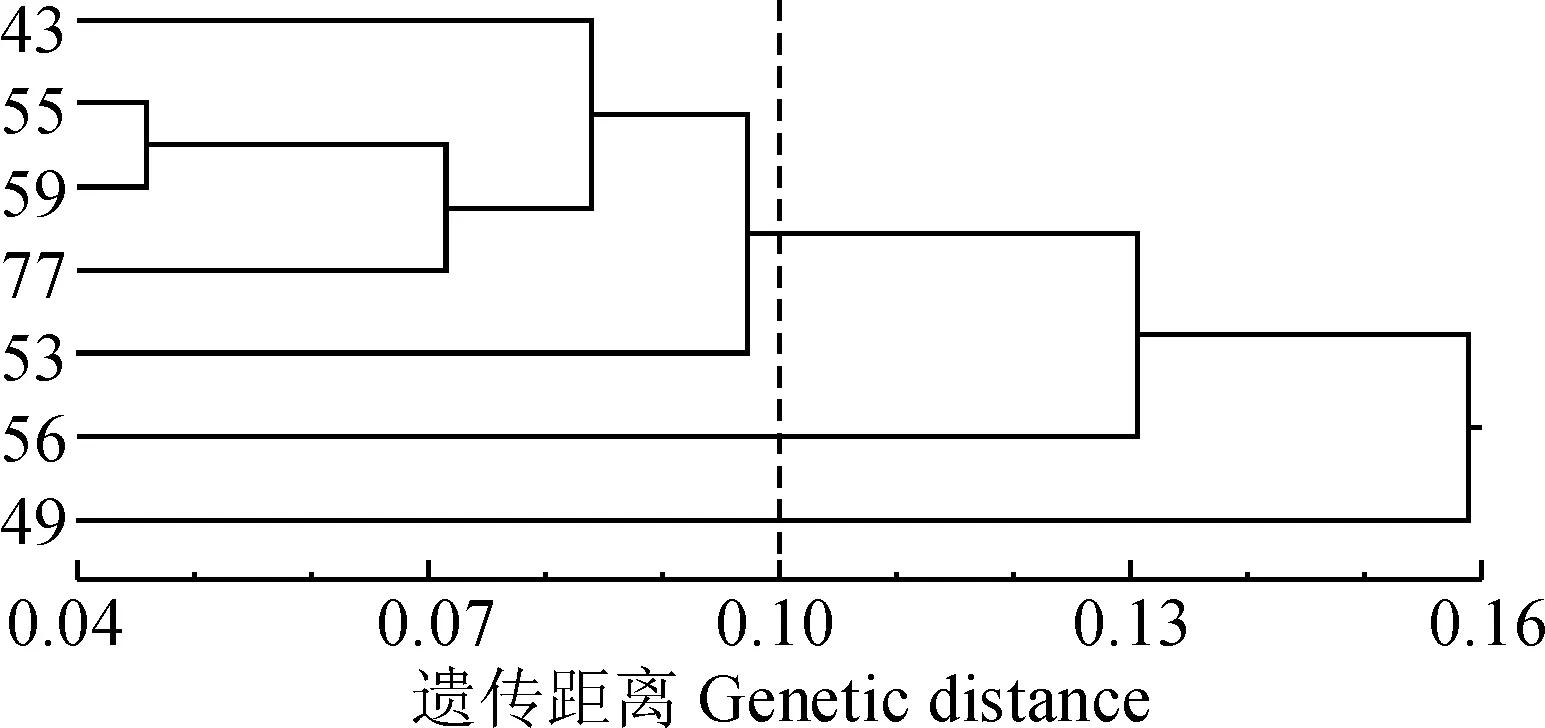
家系编号同表1图1 7个华北落叶松家系的聚类分析Number of families is the same as Table 1Fig. 1 A dendrogram of seven families of L. gmelinii var. principis-rupprechtii
2.5.2 核心个体数估算 华北落叶松7个家系内的个体按照5、10、15、20、25、30、35、40、44株9个等级进行随机抽样(家系中个体数不足者以其个体数最高值代替),获得的等位基因数与Shannon信息指数见图3、图4。从图中可以看出,随着选择个体数的增加,等位基因数与Shannon信息指数也不断增加。所有家系分别选择15株个体均可获得80%以上的等位基因数。49号和53号家系总的样本数少于25,在选择15株和20株时可以获得98%以上的等位基因,其他家系个体数均大于35,当取35株时,43号、55号、56号、59号、77号家系分别可以获得97.3%、96.6%、97.1%、98.3%、100%的等位基因。Shannon信息指数的变化趋势与观察等位基因数基本一致,其中选择35个个体时,56号家系的Shannon信息指数较高,可以获得全部的遗传多样性信息。综合上述结果,个体数较少的49号和53号家系选择15~20个个体,其他家系均选择35个个体即可实现稳定遗传多样性的目的。
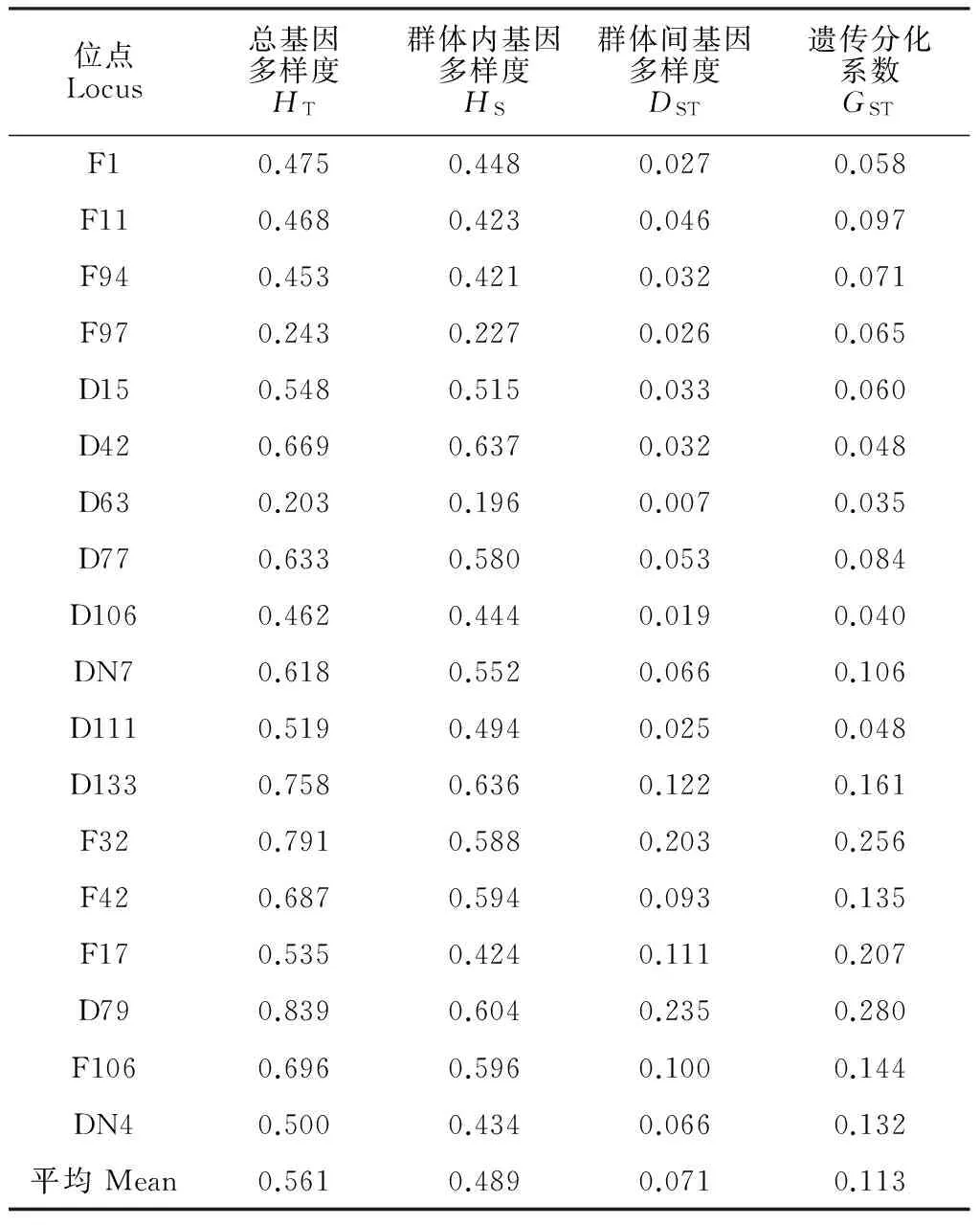
表5 7个华北落叶松家系的遗传分化
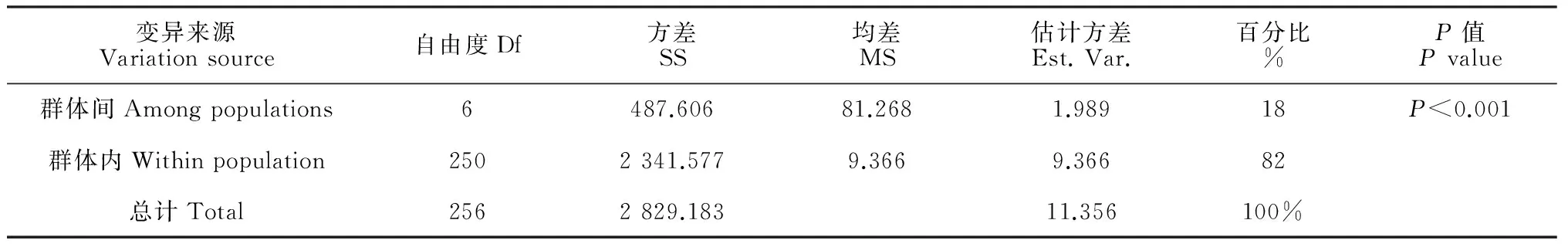
表6 7个华北落叶松家系的AMOVA分析
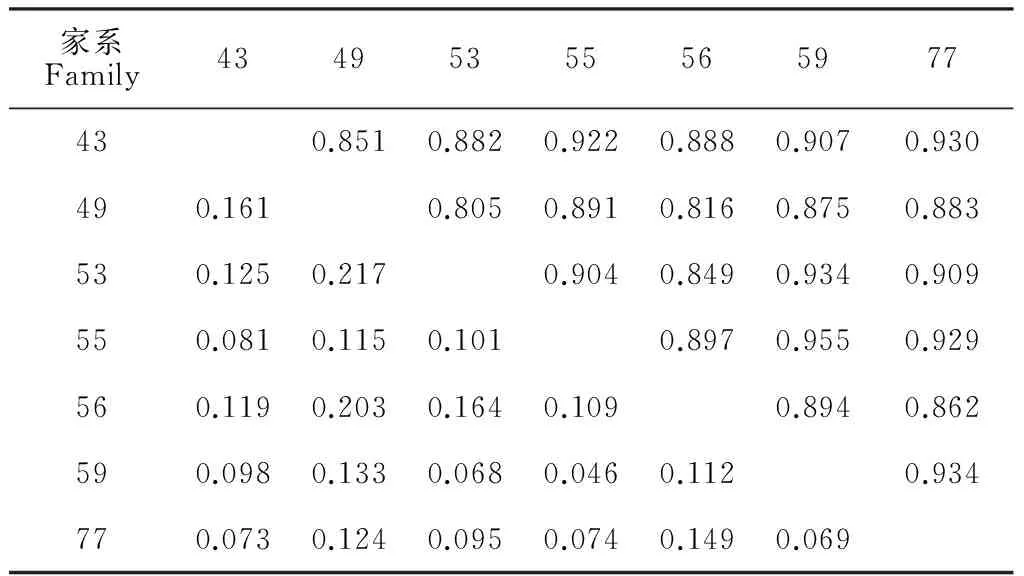
表7 华北落叶松7个家系Nei’s遗传一致度(GI) (对角线上方)和遗传距离(GD)(对角线下方)
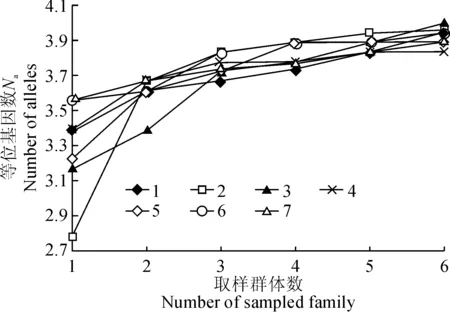
图例数字表示重复次数图2 等位基因数(Na)与抽样群体数的关系Legend represents the number of repeatsFig. 2 The relationship between the number of alleles and the number of sampled populations
3 讨 论
3.1 华北落叶松控制授粉群体的遗传多样性
遗传增益和遗传基础在种子园生产中极其重要,但两者又相互制约,保证较高的遗传增益是种子园建立的重要基础,同时保持较高的遗传多样性水平和较宽的遗传基础,提高种质资源对环境的适应性也是至关重要的。
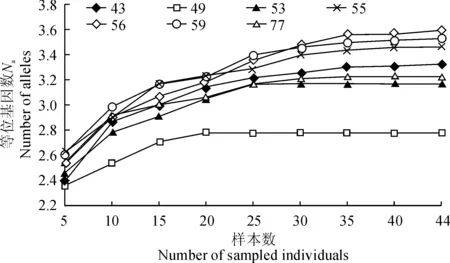
图例数字表示家系编号图3 每个家系抽取不同数量个体所含等位基因数的变化Legend represents the number of familiesFig. 3 The relationship between the number of alleles and the number of sampled individuals
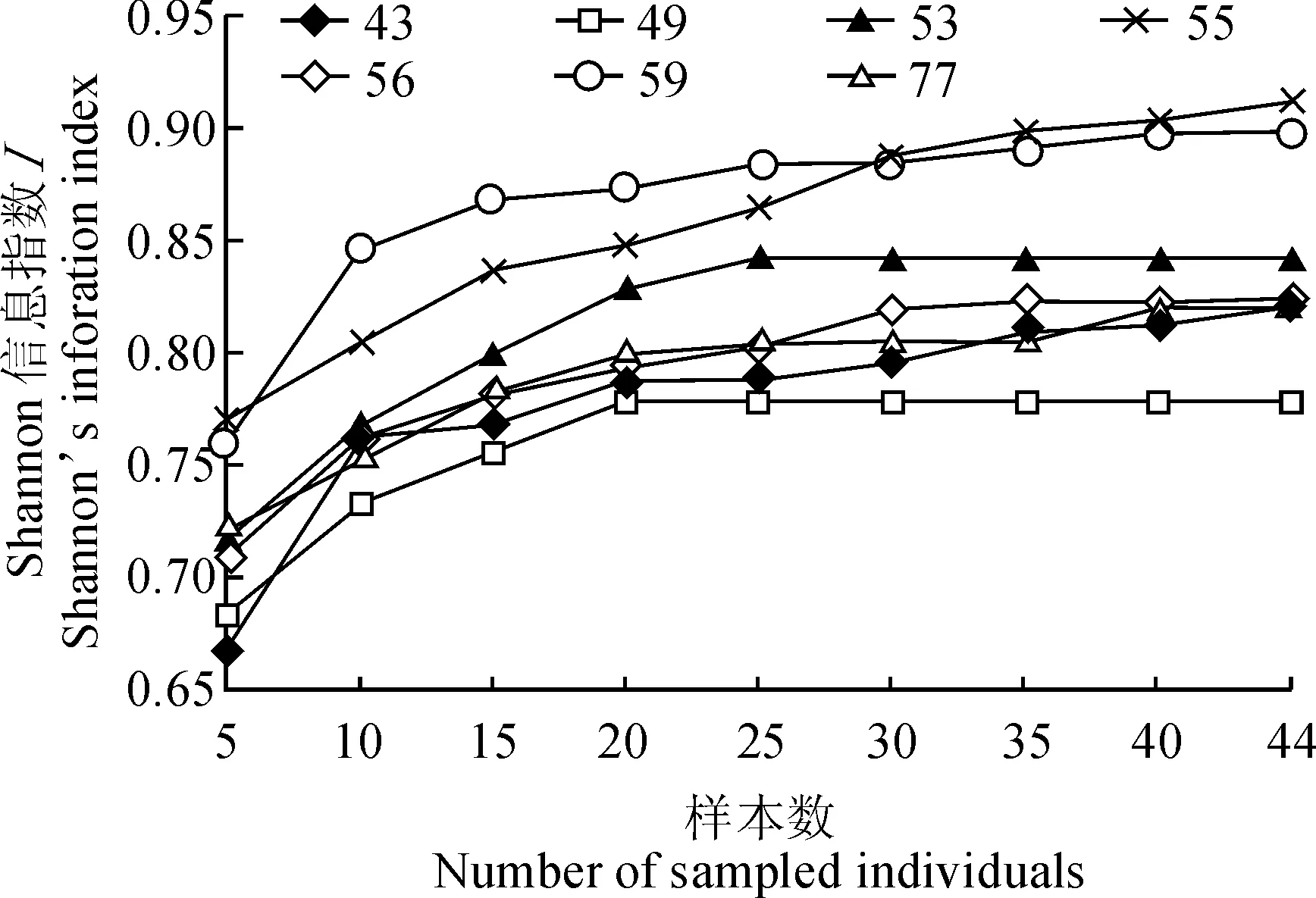
图例数字表示家系编号图4 每个家系抽取不同数量个体的Shannon信息指数(I)的变化情况Legend represents the number of familiesFig. 4 The relationship between Shannon’s information index and the number of sampled individuals
本研究利用18对SSR引物257个个体扩增,平均每个位点的等位基因数为4个,有效等位基因数为2.312个,7个家系的平均观测杂合度为0.518,平均期望杂合度为0.502,Shannon信息指数为0.846,说明华北落叶松控制授粉群体存在丰富的遗传变异。在华北落叶松遗传多样性的研究中,Di等[20]对6个山西省华北落叶松天然群体的遗传多样性进行了研究,其中6个群体平均期望杂合度为0.181,Shannon信息指数为0.268,总体遗传多样性低于华北落叶松控制授粉群体。现较多研究者认为,随着改良选择力度的增大,种子园的遗传基础和遗传多样性并不会降低,反而比天然林群体要高一些[21],本研究与天然林对比的结果与此观点是一致的。董明亮等[15]对北京市华北落叶松人工林的5个优树群体进行了遗传多样性分析,观测杂合度为0.429,期望杂合度为0.440,Shannon信息指数为0.756,人工林群体的遗传多样性同样低于华北落叶松控制授粉群体,同时也进一步印证了上述观点。杨秀艳等[11]对4个日本落叶松种子园的二代优树群体进行遗传多样性水平研究,群体的观测杂合度与期望杂合度分别为0.590 2和0.570 2,Shannon信息指数为1.096 6,与控制授粉群体相比,二代优树群体遗传多样性较高,说明人工选择强度的逐代增加,使每一代生长优势明显的杂合个体更多地被选择,这在一定程度上可以增加群体的遗传多样性水平,扩宽群体的遗传基础。
3.2 华北落叶松控制授粉群体的遗传分化水平
遗传分化系数(GST)能很好地度量群体相对基因分化程度[17],当GST为0~0.05时,群体属于较低遗传分化水平,分化程度可以忽略不计;当GST为0.05~0.15时,群体属于中等遗传分化水平,当GST为0.15~0.25时,群体具有较高的遗传分化水平,当GST大于0.25时,群体具有相当高的遗传分化水平[22]。本研究中7个群体的平均总基因多样度为0.561,群体间基因多样度为0.071,群体内基因多样度为0.489,遗传分化系数为0.113,说明这些群体的遗传分化处于中等程度。研究涉及的18个位点中,仅有4个位点的GST小于0.05,其他位点均在0.05以上,说明这些位点可以较大程度地反映华北落叶松控制授粉群体的遗传分化水平。与自由授粉群体[13](FST=0.0176)相比,控制授粉群体的遗传分化程度高一些,处于较高的遗传分化水平。可能的原因是,种子园中自由授粉的群体主要由几个优势父本与母本进行随机杂交,父本数量较少,导致遗传分化程度较低,而控制杂交授粉的父母本是一定的,可以产生各种等位基因的子代,另外,AMOVA分析表明,82%的遗传变异存在于家系内,说明控制授粉可以产生较多的遗传变异,因此遗传分化程度相对较高。
Nei’s遗传距离和遗传一致度是群体或个体间亲缘关系的指标,群体间遗传距离越小,说明亲缘关系越近,反之亦然[19]。7个家系聚类分析表明49号家系与其他家系的遗传距离最远,55号与59号遗传距离最近,亲缘关系较近,同时遗传一致度也有相同的结论。营建生态林或造林绿化应考虑家系间的亲缘关系,选择适当的家系可以提高整个群体对病害、虫害的抗性,同时保证遗传多样性不会丧失。
3.3 华北落叶松控制授粉群体核心种质
核心种质是保存种质资源的一个核心群体,以最少数量的种质资源使群体的遗传多样性得到最大程度的保存[24]。一般来说,遗传多样性水平会随着群体数的增加而增大,但群体数达到一定水平后,遗传多样性将趋于稳定,因此,在不降低遗传多样性的情况下获得最小数量的群体可以大大降低研究成本。白端霞[25]对4个枣的栽培品种的核心种质进行了构建,选择初始种质的30%构成核心种质资源,其观测等位基因与Shannon信息指数分别可以保留94.97%和94.32%。张龙进[26]从73份山茱萸种子资源中选择18个山茱萸作为核心种质,通过鉴定得出18个山茱萸与原始种质具有相同的遗传结构。刘娟等[27]利用ISSR标记构建了新疆野杏的核心种质,当选择原种质的22.96%的样品时,观测等位基因数、有效等位基因数和Shannon信息指数均可达到99%以上,可以代表原种质的遗传多样性水平。另外,遗传变异主要存在于群体间的群体与主要分布于群体内的群体具有完全不同的取样方法,本研究群体遗传变异主要存在于家系内(82%),因此,选择群体时尽量包含分布区的所有群体,并且每个群体需要相对较多的个体数目,从而达到有效的选择目的[24]。本研究结果表明华北落叶松控制授粉群体选择5个家系就可以保证96%以上的遗传多样性,对于每个家系中选择的个体数,当个体数较少(小于25)时,选择15~20个个体,当个体数较多(大于35)时,可以选择35个个体,就可以保持遗传多样性的相对稳定。在遗传多样性水平不降低的情况下,选择适当的群体和个体数可以大量节省资金成本,减少时间资源的浪费,在提高育种效率的同时达到良种改良和生态保持的共赢。华北落叶松的控制授粉群体家系内存在丰富的遗传变异,杂交子代间较宽的遗传基础有助于新品种的培育。本研究利用SSR分子标记揭示了华北落叶松控制授粉子代具有相对较高的遗传多样性以及遗传分化水平,同时对育种选择的核心家系数量和核心个体数量进行了估算,为今后华北落叶松的良种选育和种质资源的有效利用提供了理论基础。
[1] 陈晓阳,沈熙环. 林木育种学[M]. 北京:高等教育出版社, 2005,110-132.
[2] 沈熙环. 油松、华北落叶松良种选育实践与理论[M]. 北京: 科学出版社, 2014,2-7.
[3] 李义良, 赵奋成, 张应中, 等.分子标记在松树遗传与进化研究中的应用[J].分子植物育种,2009, 7(5): 1 004 - 1 009.
LI Y L, ZHAO F C, ZHANG Y Z,etal. Applications of Molecular marker on the research of pine genetics and evolution [J].MolecularPlantBreeding, 2009, 7(5): 1 004 - 1 009.
[4] 于大德. 华北落叶松种子园遗传多样性及亲本分析[D]. 北京:北京林业大学, 2014.
[5] XIN W, WANG L H ,etal. Development of Simple Sequence Repeat (SSR) markers of sesame (Sesamumindicum) from a genome Survey[J].Molecules, 2014, 19: 5 150-5 162.
[6] HEMANT K Y, ALOK R, MEHAR H,etal. EST-derived SSR markers inJatrophacurcasL.: development, characterization, polymorphism, and transferability across the species/genera [J].TreeGenetics&Genomes, 2011, 7:207-219.
[7] 李汝玉.简单序列重复(SSR)及其在农作物研究中的应用[J].山东农业科学,2004,29(6):11-15.
LI R Y. Simple sequence repeats (SSR) and its application in crop research [J].ShandongAgriculruralSciences, 2004, 29(6):11-15.
[8] C. R, H. S. Abundance and DNA sequence of two-base repeat regions in tropical tree genomes [J].Genome, 1991, 34(1): 66-71.
[9] 范英明, 张登荣, 于大德, 等.河北省华北落叶松天然群体遗传多样性分析[J]. 植物遗传资源学报, 2014, 15(3): 465-471.
FAN Y M, ZHANG D R, YU D D,etal. Genetic diversity and population structure ofLarixprincipisrupprechtiiMayr in Hebei Province [J].JournalofPlantGeneticResources, 2014, 15(3): 465-471.
[10] 贯春雨, 张含国, 等.落叶松杂种F1代群体遗传多样性的RAPD、SSR分析[J].经济林研究, 2010, 28(4): 8-14.
GUAN C Y, ZHANG H G,etal. RAPD and SSR analysis of genetic diversity of hybrid F1 progeny population inLarixspp [J].NonwoodForestResearch, 2010, 28(4):8-14.
[11] 杨秀艳, 孙晓梅, 张守攻, 等.日本落叶松EST-SSR标记开发及二代优树遗传多样性分析[J].林业科学, 2011, 47(11): 52-58.
YANG X Y, SUN X M, ZHANG S G,etal. Development of EST-SSR markers and genetic diversity analysis of the second cycle elite population inLarixkaempferi[J].ScientiaSilvaeSinicae, 2011, 47(11): 52-58.
[12] 姚 宇, 张含国, 张 振, 等.去劣疏伐对长白落叶松初级无性系种子园SSR 遗传多样性的影响[J].中南林业科技大学学报, 2013, 33(3): 40-46.
YAO Y, ZHANG H G, ZHANG Z,etal. Effects of rogue thinning on genetic diversity by SSR in aLarixolgensisclone seed orchard [J].JournalofCentralSouthUniversityofForestry&Technology, 2013, 33(3): 40-46.
[13] 于大德, 袁定昌, 等.华北落叶松种子园不同世代间遗传多样性变化[J].植物遗传资源学报, 2014, 15(5): 940-947.
YU D D, YUAN D C,etal. Genetic diversity ofLarixprincipis-rupprechtiiMayr. seed orchard among generations [J].JournalofPlantGeneticResources, 2014, 15(5): 940-947.
[14] DOYLE J J. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J].Phytochem.Bull., 1987, 19: 11-15.
[15] 董明亮, 高嘉玥, 孙文婷, 等.北京市华北落叶松优树群体遗传多样性分析[DB/OL]. (2016-4-6) [2016-5-9]. http://www.cnki.net/.
[16] YEH F C, YANG R, BOYLE T J,etal. Pop Gene32, Microsoft Windows-based freeware for population genetic analysis, version1. 32 [M]. Edmonton: Molecular Biology and Biotechnology Centre, University of Alberta, 2000.
[17] PEAKALL R O D, SMOUSE P E. Genalex 6: genetic analysis in Excel. Population genetic software for teaching and research [J].MolecularEcologyNotes, 2006, 6(1): 288-295.
[18] NEI M. Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genetics, 1978, 89(3): 583-590.
[19] 徐刚标.植物群体遗传学[M]. 北京:科学出版社, 2009. 163-200.
[20] DI X Y, LI X N, WANG Q X,etal. Genetic diversity of natural populations ofLarixprincipis-rupprechtiiin Shanxi Province, China [J].BiochemicalSystematicsandEcology, 2014, 54: 71-77.
[21] 艾 畅. 马尾松无性系种子园遗传多样性及其自由授粉子代父本组成研究[D]. 南京: 南京林业大学, 2004.
[22] WRIGHT S. The interpretation of population structure by F-statistics with special regard to systems of mating [J].Evolution, 1965:395-420.
[23] NEI M, TAJIMA F, TATANO Y. Accuracy of estimated phylogenetic trees from molecular data[J].JournalofMolecularEvolution, 1983 19:153.
[24] 卫尊征.小叶杨遗传资源评价及重要性状的SSRs关联分析[D].北京: 北京林业大学,2010:6.
[25] 白端霞. 枣种质资源遗传多样性和分子评价及其核心种质的构建[D]. 保定: 河北农业大学, 2008.
[26] 张龙进.山茱萸种质资源遗传多样性分析及核心种质构建方法研究[D]. 西安: 陕西师范大学, 2012.
[27] 刘 娟,廖 康,等.利用ISSR分子标记构建新疆野杏核心种质资源[J].中国农业科学, 2015, 48(10): 2 017-2 028.
LIU J, LIAO K,etal. The core collection construction of Xinjiang wild apricot based on ISSR molecular markers [J].ScientiaAgriculturalSinica, 2015, 48(10): 2 017-2 028.
(编辑:宋亚珍)
Genetic Diversity of Control-pollinated Progenies in Seed Orchard ofLarixgmeliniivar.principis-rupprechtiiMayr.
SUN Wenting1, YU Dade1, DONG Mingliang1, ZHAO Jian1,WANG Xiaoping2, ZHANG Hongjing3, ZHANG Jinfeng1*
(1 National Engineering Laboratory for Tree Breeding/Key Laboratory of Genetics and Biotechnology Laboratory of State Forestry Administration/College of Biological Science and Biotechnology, Beijing Forestry University, Beijing 100083,China; 2 Beijing Municipal Bureau of Landscape and Forestry, Beijing 100013,China; 3 Hebei Forestry Research Institute, Shijiazhuang 050061,China)
In order to further understand the genetic diversity and genetic divergence of control-pollinated progenies ofLarixgmeliniivar.principis-rupprechtii, we utilized 18 pairs of SSR primers in 257 samples from 7 families. (1) A total of 72 alleles were detected with a mean of 4 alleles per primer and effective number of alleles of each SSR marker ranged from 1.247-3.411. (2) The number of effective alleles was equally 2.135 in each family. The observed heterozygosity (Ho), expected heterozygosity (He) and Shannon information index (I) were 0.518, 0.502 and 0.846, respectively. Among the 7 families, the family of 55 had the richest genetic diversity, while the family of 56 had the lowest one. (3) Gene differentiation coefficient was 0.113, illustrating the intermediate-level of genetic differentiation. The result of AMOVA showed that 82% of genetic variation was within the populations (families), while the remaining 18% was among populations (families). (4) 49 family was far from others in the clustering analysis; 55 family was the nearest to 59 family so that they had the closest relationship. (5) According to the results of the genetic diversity and genetic divergence, the core of families and individuals were estimated. The 5 families selected was accounting for 96% of genetic diversity. In addition, 15-20 individuals can be selected in a family with less number of individuals, 35 individuals can be selected when a family had more individuals. The result of this study has important theoretical and practical significance in elected populations for breeding and protected genetic diversity in seed orchard ofLarixgmeliniivar.principis-rupprechtii.
Larixgmeliniivar.principis-rupprechtiiMayr.; families; SSR; genetic diversity
1000-4025(2016)08-1662-09
10.7606/j.issn.1000-4025.2016.08.1662
2016-05-09;修改稿收到日期:2016-07-12
北京林业大学青年教师科学研究中长期项目(2015ZCQ-SW-02);北京市园林绿化局计划项目(CEG-2015-01-3);国家自然科学基金(31370658);教育部“长江学者和创新团队发展计划”(IRT13047);河北省科技计划项目(16226309D);河北省重要乡土树种种质资源创制与利用技术研究
孙文婷(1990-),女,在读硕士研究生,主要从事植物遗传育种方向研究。E-mail:bjsunwenting@163.com
*通信作者:张金凤,教授,博士生导师,主要从事针叶树良种繁育研究。E-mail:zjf@bjfu.edu.cn
Q346+.5; Q789
A

