基于细菌同源蛋白预测细菌最适生长温度的研究
丛华剑武栓虎田健初晓宇伍宁丰
(1.烟台大学,烟台 264005;2.中国农业科学院生物技术研究所,北京 100081)
基于细菌同源蛋白预测细菌最适生长温度的研究
丛华剑1,2武栓虎1田健2初晓宇2伍宁丰2
(1.烟台大学,烟台 264005;2.中国农业科学院生物技术研究所,北京 100081)
不同细菌有不同的最适生长温度,而基因序列与其最适生长温度密切相关。为探究其相关性,选取92个具有不同最适生长温度的细菌的全基因组序列为研究材料,通过寻找92个细菌共有的同源蛋白,并计算共有同源蛋白中氨基酸的频率,发现共有同源蛋白的氨基酸频率特征与其最适生长温度存在着显著的相关关系,其中蛋白质序列中的螺旋结构与其最适生长温度关系最大。该研究为揭示细菌对温度的适应机制,以及对蛋白质稳定性相关的分子设计具有重要的意义。
细菌;最适生长温度;同源蛋白;氨基酸频率
细菌生长在不同的自然环境里,在某个特定的温度下,它的生长速度最快,这个温度被称为其最适生长温度。根据最适生长温度的不同,细菌可以被分为嗜热菌、中温菌及嗜冷菌,其中嗜热菌的最适生长温度一般在40℃以上,嗜冷菌的最适生长温度在20℃以下,而大多数中温菌最适生长温度在30℃左右。细菌适应不同最适生长温度的机制一直是人们针对细菌研究的热点。2003年,Makarova等[1]通过系统发生学分析方法寻找到一些可能对嗜热起关键作用的基因,并找到了一些与嗜热表型特征相关的同源基因簇。2007年,Li等[2]发现了部分原核生物基因序列与其最适生长温度的关系。2011年,Kim 等[3]发现了某一藻类中的耐热基因,该基因的表达使其在高温下有更好的生存能力。2012年,Jensen等[4]基于全基因组序列,利用贝叶斯的统计方法对细菌的最适生长温度范围进行了预测,得到了准确的结果。2014年,Hu[5]发现了对细菌在低温下正常生长起关键作用的酶。通过大量研究表明,细菌在最适生长温度方面的差异,与其基因序列的差异密切相关,然而对于影响其最适生长温度的序列特征,目前仍缺乏有效的研究和分析,需要从基因组学的层面对其进行系统挖掘。
本研究为了探究基因序列与其最适生长温度的关系,将运用生物信息学的方法确定不同细菌间共有的同源蛋白,并对这些共有同源蛋白进行序列分析,从中分析与其最适生长温度相关的分子特征,为揭示细菌对温度的适应机制以及利用蛋白质的分子设计来提高其稳定性等方面奠定基础。
1 材料与方法
1.1 材料
数据集采用Jensen等[4]在实验中采用的92个细菌(表1),其全基因组序列和最适生长温度均来自NCBI数据库(http://www.ncbi.nlm.nih.gov/)。
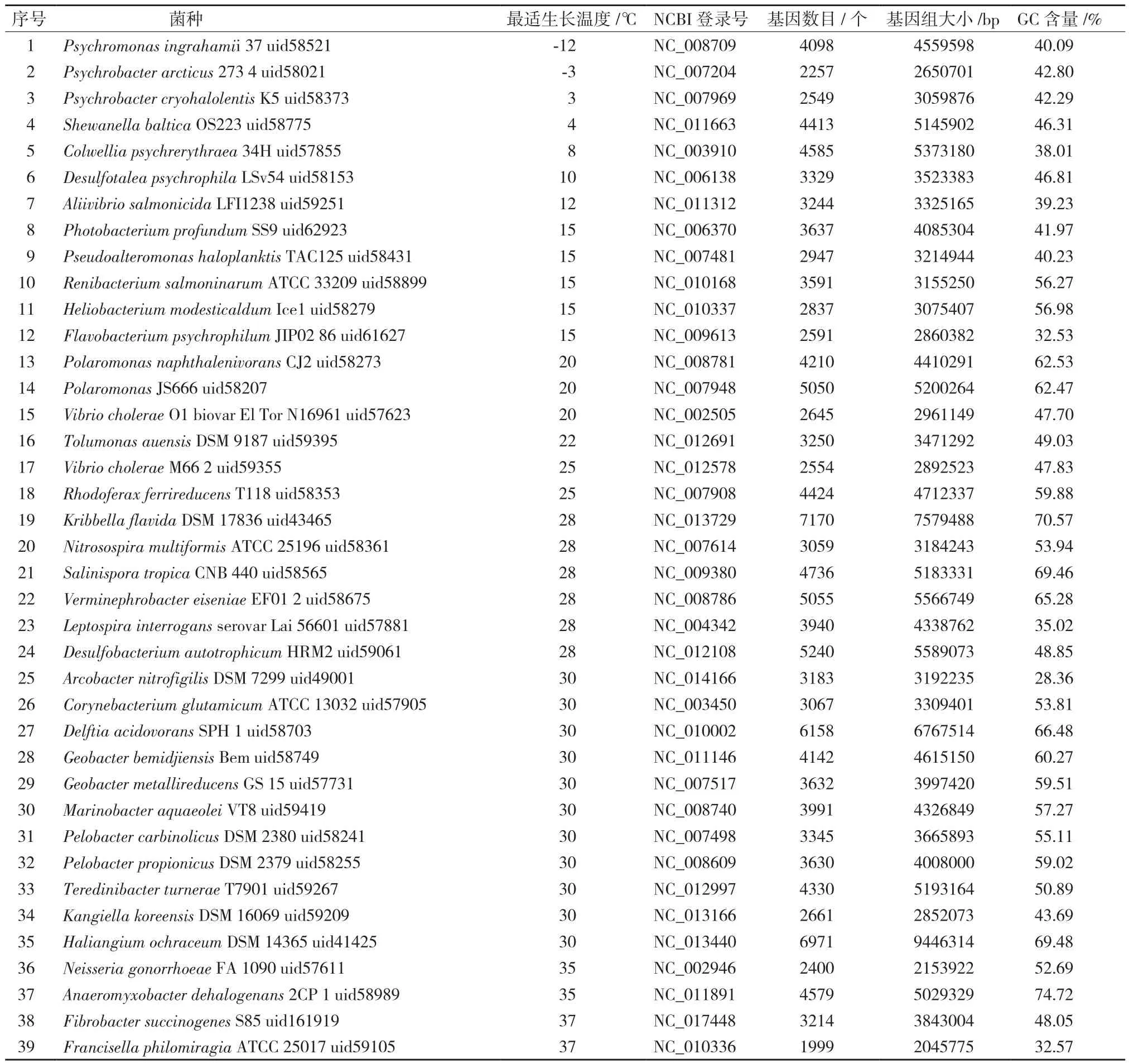
表1 92个细菌的详细信息

续表
1.2 方法
1.2.1 提取共有同源蛋白 在NCBI数据库中,获取全部92个细菌的全部基因组信息,选取基因数目最多的一个细菌,通过RSD[6]软件(Reciprocal Smallest Distance,https://github.com/todddeluca/ reciprocal_smallest_distance/)找到该菌与其他所有细菌之间共有的同源蛋白,参数divergence和E-value thresholds分别取值0.2和1e-20,divergence是散度阈值,用来判断基因之间的“距离”,E-value是期望值阈值,用来判断结果的显著性。通过整理之后可以得到所有细菌的共有的同源蛋白,然后对其进行序列比对[7]确定蛋白质的功能。
1.2.2 计算氨基酸频率 提取共有同源蛋白中每一种氨基酸的频率,并把其频率值作为特征,采用随机森林的方法来模拟其与最适生长温度的相关性。相关系数采用20-倍的交叉验证的方法进行,首先把数据分为20份,每一次交叉验证选取其中一份作为测试集,其余19份数据作为训练集,通过训练集建立随机森林模型,然后用测试集进行验证,通过20次交叉验证后,可以得到最终的相关系数,相关系数计算公式如下:
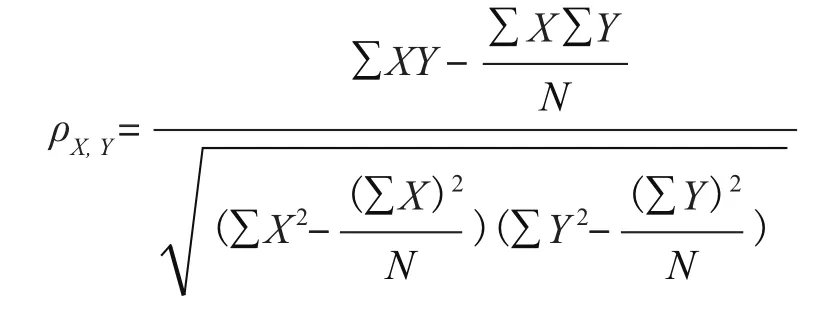
式中,ρ表示相关系数值,X与Y分别表示同源蛋白的氨基酸频率与温度值,N为样本数量。通过计算可以得到每一个同源蛋白的氨基酸频率与最适生长温度之间的相关系数。
1.2.3 进化树的构建 利用与最适生长温度相关性最高的同源蛋白的序列、全部共有同源蛋白序列以及菌株的16S rDNA来构建进化树,采用的工具为MEGA[8](Molecular Evolutionary Genetics Analysis,http://www.megasoftware.net/),先对所有的同源蛋白进行序列比对,然后选择构建Maximum Likelihood Tree,因为根据最大似然估计的方法,概率总和最大的那棵树最有可能是反映真实情况的系统发生树。Bootstrap replications值选择1 000,1 000次重复可以充分保证结果的可靠性。其他参数为默认值,以此可以构建出反应进化关系的进化树。
1.2.4 确定对与最适生长温度关系最大的序列位置
选取与最适生长温度相关性最高的10个共有同源蛋白,采用ClusterW[9,10](http://www.clustal.org/)对其序列进行比对后,逐列分析确定与其最适生长温度相关的关键位置。具体方法是根据比对结果,一次删除一列,计算相关系数,与原相关系数进行比较,得到差值,差值的绝对值越大,说明这个位置与最适生长温度的关系越大。
从与最适生长温度相关性最高的前10个同源蛋白中,提取每个同源蛋白中对最适生长温度影响较大的前5%的位置,每个同源蛋白合并可得到92个“子序列”,将10个同源蛋白的“子序列”合并成92个“特征序列”,“特征序列”中的每一个位置都是影响细菌最适生长温度的关键序列。我们将对得到的“特征序列”进行后续的氨基酸指数和蛋白质二级结构方面的分析。
1.2.5 计算氨基酸指数 氨基酸指数[11]是代表每种氨基酸物理或化学性质的一套定量值标,现每种氨基酸已有544个氨基酸指数,分别代表某种氨基酸的544种性质。在获取全部544种氨基酸指数后,分别计算每个“特征序列”在每种氨基酸指数下的加权平均值,这样可以获取一个92行544列的矩阵,然后计算某种氨基酸指数与最适生长温度的相关性,共可获取544个氨基酸指数与最适生长温度的相关指数。
2 结果
2.1 共有同源蛋白的提取
不同的微生物通常含有不同的基因数量,为了使不同微生物间具有可比性,本研究首先提取了不同物种间共有的直系同源基因,这些基因一般是由共同的祖先进化而来。不同物种间的直系同源基因一般具有类似的功能,通常编码生命必需的酶、辅酶或关键调控蛋白的基因,往往具有功能保守、进化缓慢的特征[12,13],这样的特点便于利用其来分析基因序列与最适生长温度的关系等相关研究。表2为提取的53个共有的同源基因信息,这些基因是生物中的看家基因,如methionyl-tRNA formyltransferase、glutamine-fructose-6-phosphate aminotransferase、Polyribonucleotide nucleotidyltransferase等都是细菌中非常重要的转移酶,30S ribosomal protein S13、50S ribosomal protein L13等都是参与细胞翻译过程的核糖体亚单位的重要组成蛋白。它们在维持生命的过程中起到关键的作用。

表2 53个共有同源基因信息

图1 53个同源蛋白中氨基酸频率与温度的相关系数
2.2 氨基酸频率与最适生长温度的相关性
53个共有同源蛋白中氨基酸的频率与最适生长温度的相关系数,并按相关系数大小进行排序,结果(图1)显示,与温度相关性最高的同源蛋白,相关系数为0.86,对应的蛋白质名称为:Polyribonucleotide nucleotidyltransferase,相关性最低的同源蛋白,相关系数为0.67,对应蛋白质名称为:Ribulose-phosphate 3-epimerase,全部53个共有同源蛋白的相关系数都在0.65以上,说明共有同源蛋白的氨基酸频率与其最适生长温度具有很高的相关性。我们把53个共有同源蛋白相连,计算氨基酸频率与其最适生长温度的相关系数为0.90,再次验证了共有同源蛋白中氨基酸频率与最适生长温度之间具有较高的相关性。
2.3 氨基酸指数分析
图2为相关系数最高的前30个氨基酸指数(图2-A)和最低的后30个氨基酸指数(图2-B)的对比图,图中颜色越深的点表示相关系数值越大。图2-A显示,除了排在第9位的氨基酸指数呈现负相关的关系,具体表现为随着“特征序列”最适生长温度的增加,氨基酸指数值逐渐变小,其他的氨基酸指数均呈现正相关的关系,表现为随着“特征序列”最适生长温度的增加,氨基酸指数值逐渐变大。由此可见,蛋白质中与其最适生长温度相关的特征序列中的氨基酸选择性有明显的倾向性,在相关系数最高的前30个氨基酸指数中,其相关系数绝对值均在0.70以上。10个与最适生长温度相关性最高的氨基酸指数(表3)显示,最适生长温度较高的微生物中蛋白质中的特征序列倾向选择分子量大,且疏水性强的氨基酸。
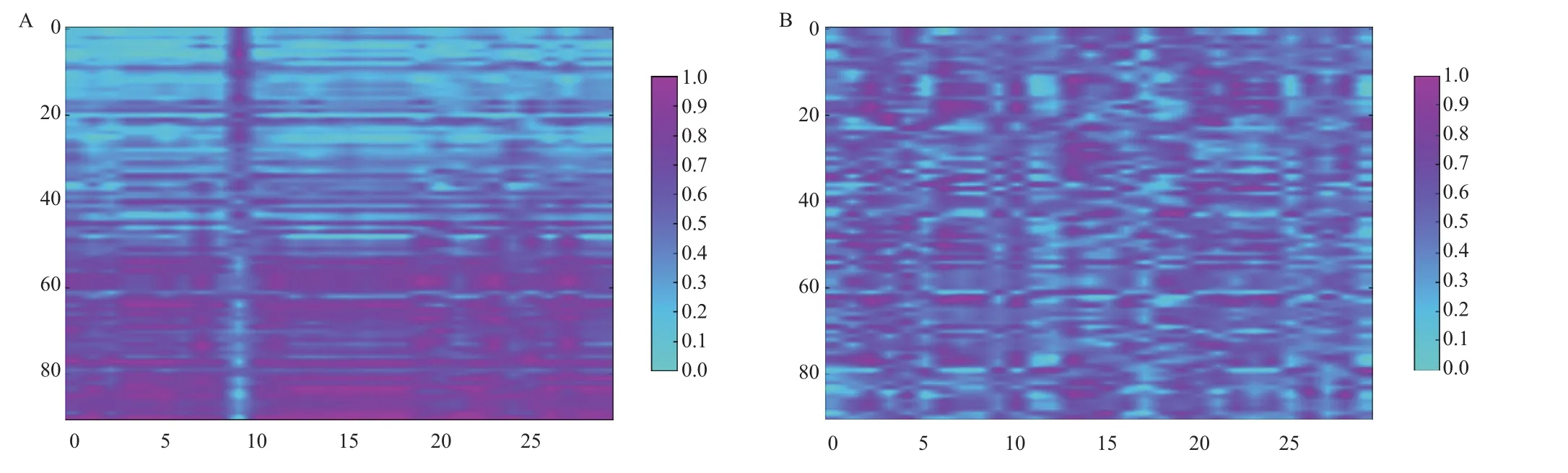
图2 相关系数最高的前30个氨基酸指数(A)和最低的30个氨基酸指数(B)
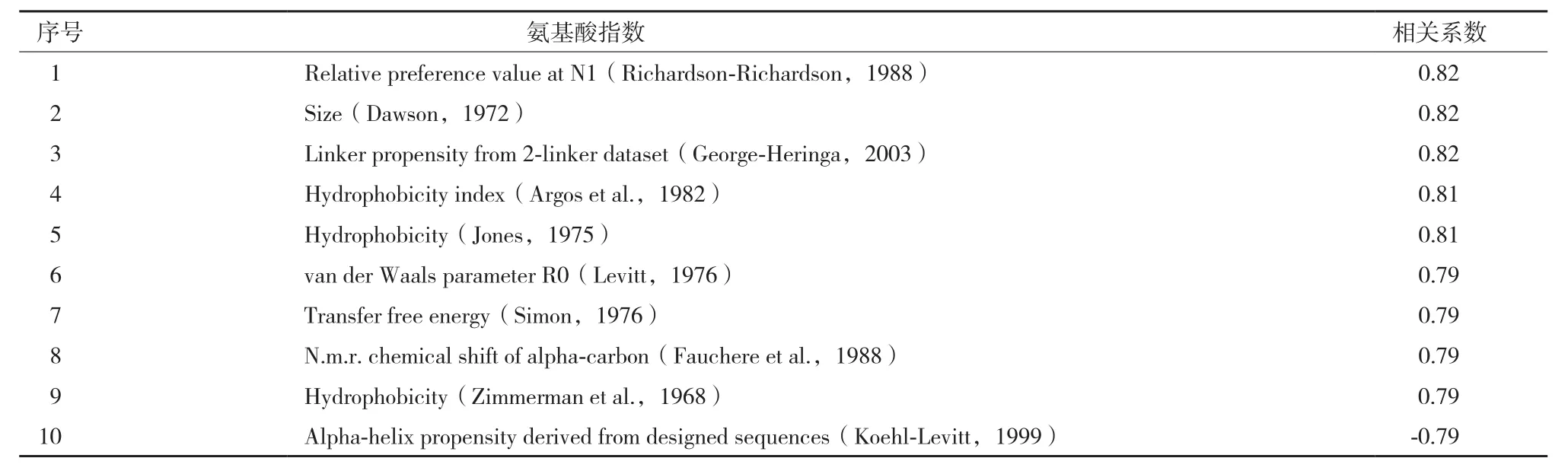
表3 相关性最大的前10个氨基酸指数
2.4 蛋白质二级结构信息
对于蛋白质二级结构上的分析,本研究主要探究“特征序列”的每个氨基酸位于蛋白质的区域,观察其是否有明显的位置特征。对蛋白质的二级结构的预测,采用的工具是PSIPRED[14](http://bioinf. cs.ucl.ac.uk/psipred/)。

图3 利用Polyribonucleotide nucleotidyltransferase所建的进化树
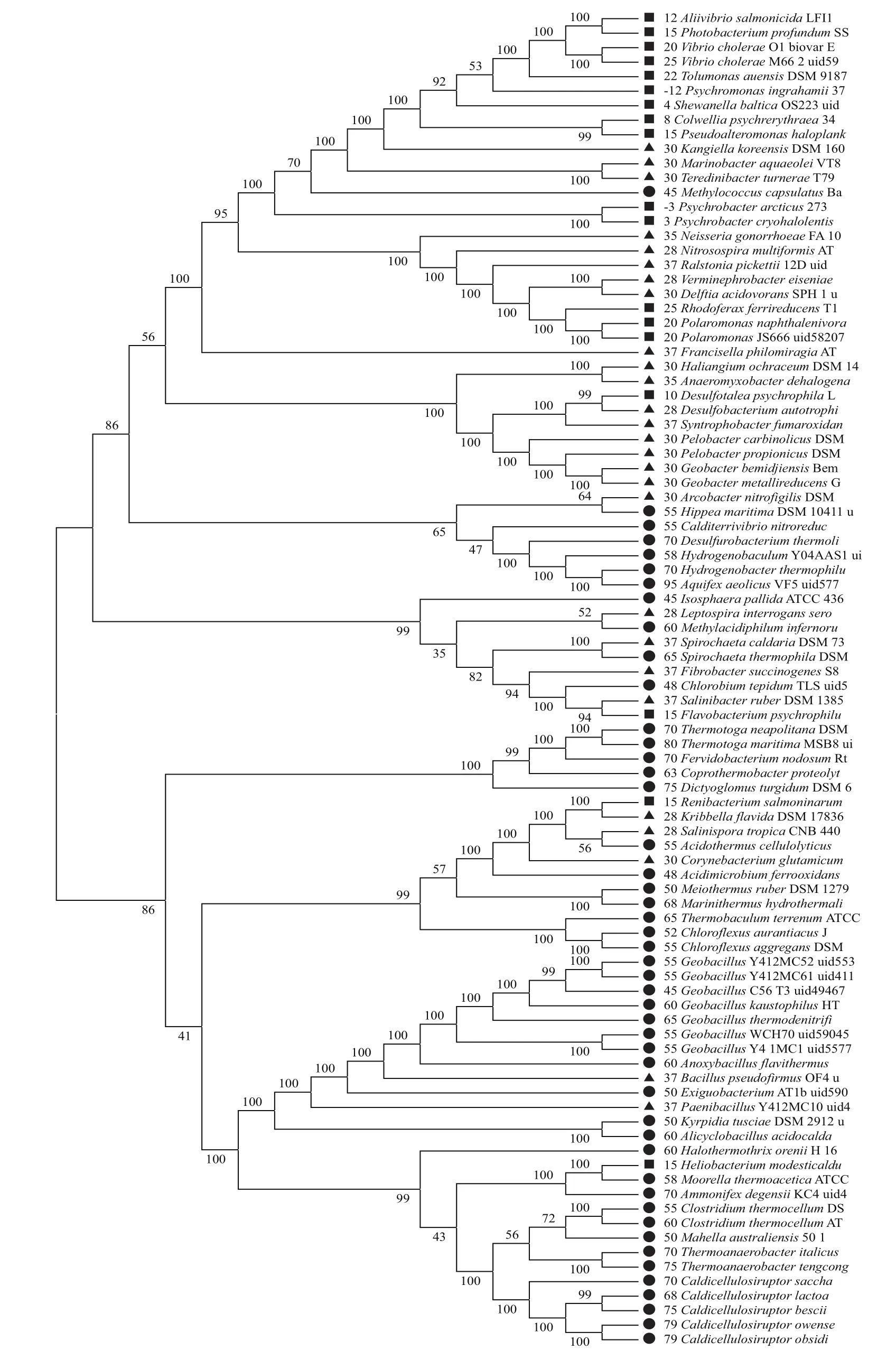
图4 全部同源蛋白构建的进化树

图5 基于16 S基因构建的进化树
对最适生长温度的影响比较大的氨基酸主要分布于α螺旋及loop区,比例分布分别为46.10%和37.13%,β折叠部分只占了16.77%。而“特征序列”所在的完整的蛋白序列中,α螺旋、loop区及β折叠所占的比例分别为:35.30%、46.37%及18.33%。研究发现,“特征序列”中α螺旋所占比例增加,loop区所占比例减少,β折叠部分基本保持不变,可见对最适生长温度有较大影响的位置对α螺旋部分有一定选择偏好性。
2.5 进化分析
我们选取与最适生长温度相关性最高的同源蛋白以及全部蛋白来构建进化树(图3,图4)。通过观察与最适生长温度相关性最高的同源蛋白所建的进化树(图3),发现较为明显的异常值有8个,分别占总数的8.7%。全部共有同源蛋白相连所建的以及进化树(图4),可以发现较为明显的异常值有5个,占总数的5.4%。利用92个细菌的16S rDNA构建的进化树(图5),其明显的异常值有7个,占总数的7.6%,其值均不足10%。因此通过进化树很好地将高温、中温以及低温3类细菌进行了分类,且3个进化树分类效果相当。
3 讨论
本研究通过氨基酸频率特征来探究共有同源蛋白中一些对温度有影响的关键位置,并对其进行分析发现,细菌中的一些特殊基因序列对其最适生长温度有较大影响,这对蛋白质热稳定性相关实验有比较重要的指导意义。然而,实际上仍有很多因素对蛋白质热稳定性有较大影响,国内外学者都对其进行了大量的研究。其中Zeldovich等[15]的研究发现、Ile、Val、Tyr、Trp、Arg、Glu及Leu(IVYWREL)在蛋白序列中的比例与蛋白质的最适生长温度有较高的相关性,说明氨基酸的组成比例与蛋白质的最适生长温度具有相关性。除了基因序列的影响之外,蛋白质的空间结构作用力的影响也是一个重要的因素[16],通过分析蛋白质的三级结构,并对其进行分子动力学模拟,可以更直观地分析在高温情况下蛋白质的热稳定性情况。Mitra等[17]的研究就是通过这样的方法对蛋白质进行设计,提高了纤维素酶的热稳定性,Akcapinar等[18]则是对突变后的耐高温蛋白质进行分析,从蛋白质活性中心空间作用力改变的角度解释了突变对其热稳定性的提高的机制。在国内,对细菌最适生长温度以及蛋白质热稳定性的研究也正不断开展[19-21],在基因组的热稳定性机制,蛋白质稳定性的机理,理性设计蛋白质热稳定性等方面都取得了不错的研究成果。所以,在后续工作中将对这些因素进行更深入的研究,同时也将选取一个更大的数据库来获取影响最适生长温度的特征,通过生物信息学的方式来对蛋白质的热稳定性进行系统的预测分析。利用生物信息学的方法,可以更高效地获得并处理大量的基因组数据,这将对理解蛋白质的热稳定性以及蛋白质的分子设计具有重要的意义。
4 结论
本研究通过利用生物信息学的方法,从基因组的层面上对细菌的最适生长温度进行了分析,利用氨基酸频率作为特征,发现了同源蛋白的氨基酸频率与细菌最适生长温度有较高的相关性;通过构建进化树分析发现,共有同源蛋白可以更好地对不同最适生长温度的细菌进行分类。通过分析氨基酸指数发现,不同最适生长温度的细菌中,同源蛋白对氨基酸的选择有偏好性。通过分析蛋白质二级结构发现,对细菌最适生长温度影响较大的位置的氨基酸普遍位于α螺旋以及loop区,并且偏好选择于α螺旋部位。
[1]Makarova KS, Wolf YI, Koonin EV. Potential genomic determinants of hyper thermophily[J]. Trends Genet, 2003, 19:172-176.
[2]Li W, Zou H, Tao M. Sequences downstream of the start codon and their relations to G02+02C content and optimal growth temperature in prokaryotic genomes[J]. Antonie Van Leeuwenhoek, 2007, 92(4):417-427.
[3]Kim E, Park H S, Jung Y, et al. Identification of the high-temperature response genes from Porphyra seriata(rhodophyta)expression sequence tags and enhancement of heat tolerance of Chlamydomonas(chlorophyta)by expression of the Porphyra htr2 gene 1[J]. Journal of Phycology, 2011, 47(4):821-828.
[4] Jensen DB, Vesth TC, Hallin TC, et al. Bayesian prediction of bacterial growth temperature range based on genome sequences[J]. BMC Genomics, 2012, 13(suppl7):53.
[5]Hu J. Polynucleotide phosphorylase is required for Escherichia coliO157:H7 growth above refrigerated temperature[J]. Foodborne Pathogens & Disease, 2014, 11(3):177-185.
[6] Wall DP, Deluca T. Ortholog detection using the reciprocal smallest distance algorithm[J]. Methods In Molecular Biology, 2007, 396:95-110.
[7]Altschul SF. Basic local alignment search tool[J]. J Mol Biol,1990, 215:403-410.
[8] Tamura K, Stecher G, Peterson D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30:2725-2729.
[9]Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Res, 1994, 22:4673-4680.
[10]Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2. 0[J]. Bioinformatics, 2007, 23:2947-2948.
[11] Kawashima S, Kanehisa M. AAindex:amino acid index database[J]. Nucleic Acids Res, 2000, 28(1):374.
[12] Sonnhammer EL, Koonin EV. Orthology, paralogy and proposed classification for paralog subtypes[J]. Trends in Genetics, 2002,18(12):619-620.
[13]Gabaldn T, Dessimoz C, Huxley-Jones J, et al. Joining forces in the quest for orthologs[J]. Genome Biology, 2009, 10(9):403.
[14]Jones DT. Protein secondary structure prediction based on positionspecific scoring matrices[J]. J Mol Biol, 1999, 292:195-202.
[15]Zeldovich KB, Berezovsky IN, Shakhnovich EI. Protein and DNA sequence determinants of thermophilic adaptation[J]. Plos Computational Biology, 2007, 3(1):62-72.
[16]Ratakonda S, Anand A, Dikshit K, et al. Crystallographic structure determination of B10 mutants of Vitreoscilla hemoglobin:role of Tyr29(B10)in the structure of the ligand-binding site[J]. Acta Crystallographica Section F-Structural Biology and Crystallization Communications, 2013, 69:215-222.
[17]Mitra S, Mukhopadhyay BC, Mandal AR, et al. Cloning,overexpression, and characterization of a novel alkali-thermostable xylanase from Geobacillus sp. WBI[J]. Journal of Basic Microbiology, 2015, 55(4):527-537.
[18]Akcapinar GB, Venturini A, Martelli PL, et al. Modulating the thermostability of Endoglucanase I from Trichoderma reesei using computational approaches[J]. Protein Engineering Design & Selection, 2015, 28(5):127-135.
[19]田健, 王平, 伍宁丰, 范云六. 理性设计提高蛋白质热稳定性的研究进展[J]. 生物技术进展, 2012, 4:233-239.
[20]张健, 张琳, 王维. 通过蛋白质序列比对探讨细菌的热适应机制[J]. 安徽农业科学, 2011, 21:12646-12648.
[21] 盛多红. 超嗜热古菌基因组的热稳定性[J]. 生命科学,2014, 1:64-71.
(责任编辑 李楠)
Prediction of Optimal Growth Temperature of Bacterium Based on the Homologous Proteins
CONG Hua-jian1,2WU Shuan-hu1TIAN Jian2CHU Xiao-yu2WU Ning-feng2
(1. Yantai University,Yantai 264005;2. Biotechnology Research Institute,Chinese Academy of Agricultural Sciences,Beijing 100081)
The optimal temperature for each bacterium differs,which is related to its gene sequence. In order to explore the correlation between them,the known genome sequences of 92 bacteria with own different optimal temperatures were selected as the study material,then the common homologous protein from 92 bacteria were searched,and frequencies of the amino acids in homologous protein were calculated. A significant correlation between the frequency of the amino acid in homologous protein and the optimal growth temperature was realized. The analysis of the sites in homologous genes showed that the helix regions in the protein sequence were the most correlated with its optimal growth temperature. This study presents important significance on understanding the mechanism of the bacterial adaption to the temperature as well as designing the mutation to improve the protein stability.
bacterium;optimal growth temperature;homologous protein;amino acid frequency
10.13560/j.cnki.biotech.bull.1985.2016.03.025
2015-05-19
国家自然科学基金项目(31371748)
丛华剑,男,硕士,研究方向:生物信息学;E-mail:conghuajian1991@163.com
武栓虎,教授,硕士生导师, 研究方向: 数字图像处理,数字信号处理,基因信息学,模式识别, 视频图像压缩,小波分析与应用,E-mail:wushuanhu@163.com;田健,博士,副研究员,硕士生导师,研究方向:蛋白质分子设计与改良、微生物重要基因资源挖掘等,E-mail:tianjian@caas.cn

