海洋石油降解菌的筛选及复合菌系的构建
吴秉奇 刘淑杰 陈福明 周楚莹
(深圳清华大学研究院 深圳环境微生物资源开发与应用工程实验室,深圳 518057)
海洋石油降解菌的筛选及复合菌系的构建
吴秉奇 刘淑杰 陈福明 周楚莹
(深圳清华大学研究院 深圳环境微生物资源开发与应用工程实验室,深圳 518057)
为采用生物法治理海洋石油污染,以原油为唯一碳源,从深圳海域5个采样点取样,通过富集、涂布平板分离高效石油降解菌,并以复配、正交等方式构建石油降解复合菌系;通过生理生化实验和16S rRNA 基因序列分析对菌株进行鉴定;采用单因素实验对复合菌系降解石油的条件进行优化,并使用气相色谱-质谱法(GC-MS)研究其对石油的降解特性。结果显示,共分离得到22株高效石油降解菌,对石油的降解率为34.5%-52.2%;由S1-30、S1-38和S2-13构建了复合菌系SQ1,对石油降解率可达68.3%,3株菌分别鉴定为棒杆菌(Corynebacterium sp.)、迪茨氏菌(Dietzia sp.)和拉布伦茨氏菌(Labrenzia sp.);经优化,SQ1在最适条件下(30℃、pH7.6、石油浓度20 g/L),11 d内对石油的去除率高达73.5%;GC-MS结果表明,复合菌系SQ1对石油总烷烃的去除率为91.7%,对较难降解的C21-C35烷烃组分的降解率接近100%。研究表明,复合菌系SQ1在海洋石油污染的生物修复中具有较强的应用潜力。
石油降解菌;复合菌系;降解率;鉴定;条件优化
石油是一种重要的战略性资源,近年来,随着海洋石油开采业的发展和远洋运输业的繁荣,海上溢油事故时有发生,严重污染了海洋环境,给海洋生态平衡带来了严重的威胁[1]。2010年,美国墨西哥湾钻井平台“深水地平线”发生爆炸,使墨西哥湾沿岸生态环境遭遇了“灭顶之灾”;同年,我国大连新港“7.16”爆炸事故导致上万吨原油泄漏进入渤海湾,这是我国历史上发生的最为严重的海上溢油事故[2,3]。据统计联合国环境规划署报告,每年流入海洋的石油约为2×106-2×107t,海洋石油污染已成为一个世界性的环境问题[4]。
治理海洋石油污染主要有物理方法、化学方法及生物方法,其中,生物修复法是利用微生物对污染物的降解作用达到去除油污的目的,具有成本低廉、无二次污染、可原位修复等显著优点[5,6],筛选优良的微生物资源用于石油污染的修复,已受到人们的广泛关注。Hassanshahiand等[7]分离到一株柴油食烷菌(Alcanivorax dieselolei)PG-12;郭平等[8]从表层海水分离到一株厦门栖东海菌(Donghicola xiamenensis)HD-1;姜肸等[9]从南海筛选到6株石油降解菌,分属于芽孢杆菌(Bacillus)、假单胞菌(Pseudomonas)、交替单胞菌(Alteromonas)等菌属,均表现出良好的石油降解效果。但由于石油是由各类烷烃、芳烃及其他成分构成的复杂混合物,不同的菌株对不同碳链的烷烃、芳烃的降解情况差别较大[10],利用菌株间的共生、协同等作用构建复合菌,则可扩大对石油类底物的利用范围和效率,进而实现对污染物的高效降解[11,12]。张海玲等[13]采用4株细菌构建了石油降解菌系;Van Hamme 等[14]将假单胞菌与红球菌混合培养构建复合菌系;Li等[15]富集得到了菌群KO5-2,相比于单一菌株,均显著提高了原油的生物降解率。目前,采用复合菌系修复海洋石油污染的过程中,尚缺乏对环境因素作用的系统分析;单一菌株与复合菌系降解特性比较方面的研究也相对较少。
近岸海域与人类的关系较为密切,该区域内的石油降解菌较远洋中可能存在较大的差别,目前尚未有专门针对该区域的石油降解复合菌系的研究。本研究即是以原油为唯一碳源,从深圳近岸海域筛选高效石油降解菌,采用复配、正交试验等方法构建石油降解复合菌系,并对其降解特性展开充分的研究,旨在为工业化菌剂的开发及应用提供参考及借鉴。
1 材料与方法
1.1材料
1.1.1海水及石油样品 海水样品取自深圳市蛇口港和盐田港,在113.9°E-114.2°E,22.4°N-22.5°N范围内5个不同位点取样;实验用油为惠州大亚湾石化工业区提供的原油,25℃下密度约为0.9 g/mL。
1.1.2培养基及试剂 MMC培养基[16](g/L):NaCl 24,MgSO4·7H2O 7,NH4NO31,KCl 0.7,KH2PO42,Na2HPO43,pH7.4,1.0×105Pa灭菌30 min。补加微量元素混合液(g/L):CaCl20.02,FeCl3·6H2O 0.5,CuSO40.005,MnCl2·4H2O 0.005,ZnSO4·7H2O 0.1。固体培养基另加琼脂15-20 g。平板表面均匀涂抹一层原油,制备成油平板。
2216E培养基(g/L)(用于菌种的培养):蛋白胨5,酵母膏1,柠檬酸铁0.10,NaCl 19.45,MgCl25.98,CaCl23.24,KCl 0.55,Na2CO30.16,KBr 0.080,SrCl2·6H2O 0.034,H3BO30.022,Na2SiO3·9H2O 0.004,NaF 0.002 4,NaNO30.001 6,Na2HPO40.008,pH 7.4-7.8,固体培养基另加琼脂15-20 g。
石油培养基(用于降解菌的富集、降解率的测定试验):每100 mL MMC培养基中加入1 g原油作为唯一碳源。
兔血平板培养基(用于表面活性剂的定性检测):固体2216E培养基融化后冷却至50℃,加入2-3滴无菌兔血摇匀后倒平板。
本研究所用的酶、dNTP等分子生物学试剂购自北京全式金生物技术有限公司;GC-MS实验所用流动相、标准品等购自国药集团;其他试剂均为国产分析纯。
1.2方法
1.2.1石油降解菌的筛选及产表面活性剂测定 取5 mL海水样品加入100 mL石油培养基中,30℃、180 r/min培养7 d,取5 mL培养液转接至新鲜的石油培养基,重复以上步骤,连续转接富集培养4次,培养基中原油量依次提升至1.0、1.5和2.0 g。将乳化现象明显的样品梯度稀释,取0.1 mL涂布于石油平板,30℃培养3-5 d,挑取周围有透明圈的菌落于2216E平板纯化、待测。
将各菌株用无菌牙签依次挑至兔血平板培养基上,30℃培养2-3 d,根据菌落周围透明圈的大小判断表面活性剂的浓度[17]。将活化后的菌株以5%的比例接种到石油培养基中,以不接菌的培养基作为空白对照,30℃、180 r/min培养5 d。采用铂金环式表面张力测定仪测定培养液的表面张力。
1.2.2石油降解率的测定 石油浓度的测定采用紫外法,参考蒋瑞萍[18]的方法制作吸光度-石油浓度的标准曲线,略有改动。在一系列50 mL的容量瓶中加入不同量的原油母液(1 g/L),用二氯甲烷定容,260 nm处测定吸光度。为保证结果准确,加标回收率控制在95%-105%之间。
各菌株接至2216E培养基中培养至对数期,离心收集菌体,用无菌MMC洗涤2次,平板涂布法测定细胞数目并稀释浓度≈108CFU/mL,以5%的比例(V/V)接种至石油培养基中,30℃、180 r/min振荡培养7 d,以未接菌的培养基作为对照。向各样品中加入50 mL二氯甲烷,200 r/min振荡20 min,再于40℃下超声15 min萃取其中的残余油污,离心去除菌体碎片。从二氯甲烷相吸取0.2 mL 溶液定容于50 mL容量瓶,260 nm处测定吸光度,根据标准曲线计算样品中残余的石油浓度:
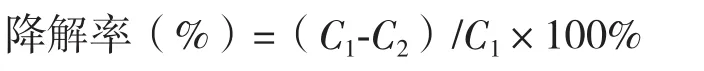
其中,C1为对照中石油浓度,C2为样品中石油浓度。每个样品3次重复,下同。
1.2.3石油降解复合菌系的构建 根据降解率和产表面活性剂情况,优选5株细菌分别接种至2216E中培养至对数末期,种子液离心、洗涤后用平板涂布法调节为一致的浓度(108CFU/mL)。各菌以等比例随机配伍的方式接至石油培养基中,总接种量为5%(体积比),30℃、180 r/min培养7 d后测定石油降解率。选择降解率最高的1个组合,通过3水平(0.5%、1.0%和2.0%)的正交实验确定各菌株的接种量。
1.2.4菌株的鉴定 生理生化实验及16S rRNA基因序列分析参照吴秉奇等[19]方法,PCR产物由上海生工生物工程有限公司测序,结果提交GenBank获取登录号,采用MEGA软件构建系统发育树。
1.2.5石油降解条件的优化 在石油培养基中接种复合菌系,摇床培养7 d,设置不同的培养条件。(1)培养温度:22、26、30、34和38℃;(2)培养基初始pH值:6.4、7.0、7.6、8.2和8.8;(3)摇床转速:160、180、200、220和240 r/min;(4)初始油浓度:1.0、5.0、10.0、20.0和40.0 g/L。石油降解率的测定方法同1.2.2。
将以上因素设置为最佳条件,定时取样测定复合菌系对石油的降解情况,同时采用平板计数法对培养液中细菌进行菌落计数。
1.2.6复合菌系对石油的降解特性 采用GC-MS法测定石油生物降解后各烷烃组分的残留情况。将复合菌系以最佳接种量接种至石油培养基,30℃、180 r/min条件下培养7 d。培养液前处理同1.2.2,取待测液用干燥无水Na2SO4脱水,再经0.22 μm有机系滤膜过滤,氮气吹干后用二氯甲烷重新溶解。GC-MS运行条件:全扫描模式;色谱柱为RYX-5 (30 m×0.25 mm×0.25 μm);柱温45℃,以 5℃/min的速度升至300℃,停留20 min;进样口、离子源、四级杆温度分别为250、230和150℃;载气为氦气;进样量1 μL。数据分析用安捷伦5975-7890A工作站。
1.2.7数据统计及分析 使用SPSS 18.0和ORIGIN 8.6软件对数据进行整理;采用新复极差法,在0.05水平上分析差异显著性(P<0.05)。菌株16S rRNA基因序列通过DNAMAN 7.02拼接后进行BLAST分析,利用MEGA 6程序的Neighbor-joining算法构建系统发育树。
2 结果
2.1石油降解菌的筛选及产表面活性剂情况
对5个取样点的样品进行驯化,选择乳化现象较为明显的样品分离细菌,共获得68株石油降解菌,分别测定各菌株对石油的降解率及培养液的表面张力。如图1所示,共有22株细菌对石油的去除情况较好,接种7 d后,菌株S1-3A、S1-12、S1-38等3株细菌对石油的降解率超过50%,其中S1-38可达52.2%。同时发现,菌株S1-5、S1-12、S1-22、S1-30、S1-37、S1-41B和S2-13等7株细菌在兔血平板上可产生较为明显的透明圈,其培养液的表面张力相比于对照也有大幅下降,最高达44.6%,初步推断以上菌株能够产生表面活性剂,促进石油的乳化、分散;其余接菌处理培养液的表面张力也有不同程度的降低。
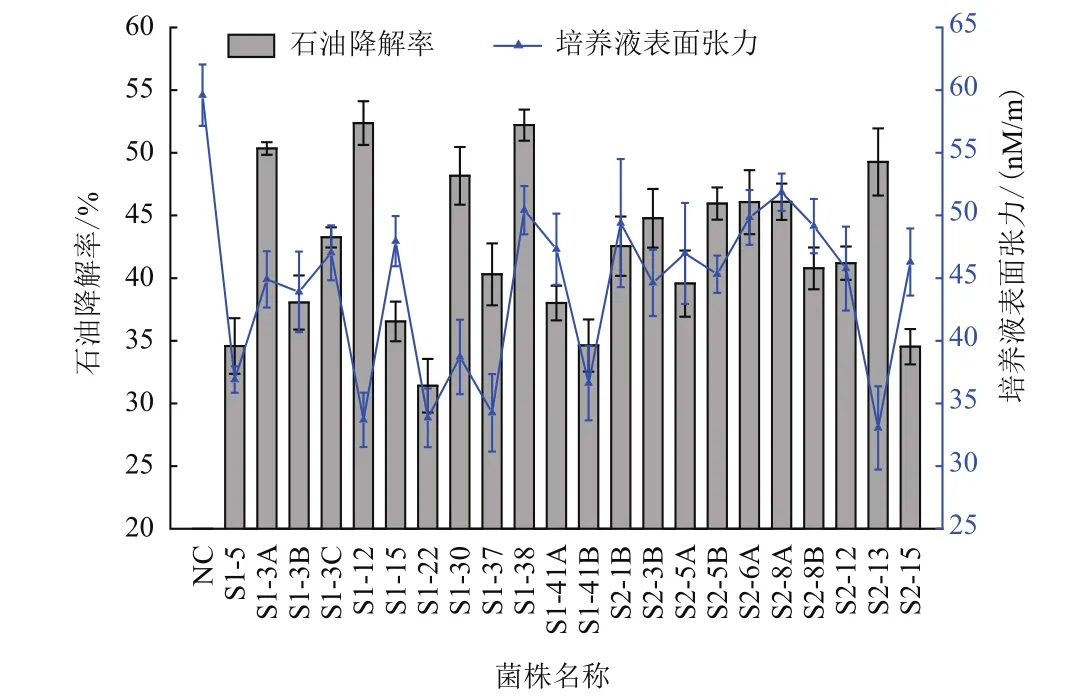
图1 菌株对石油的降解率及培养液表面张力测定
2.2石油降解复合菌系的构建
根据降解石油及产表面活性剂情况,排除菌株间的拮抗作用,选择S1-3A、S1-12、S1-30、S1-38、S2-13等5株细菌,通过等比例接种的方式分别构建二阶至五阶复合菌系,共计25种组合,测定各组合对石油的降解率,结果见表1。
结果(表1)显示,复合菌对石油的降解率在53.4%-63.7%之间,均高于单一菌株;凡同时接种S1-38和S2-13的组合均具有较高石油降解率,由S1-38、S1-30、S2-13等3株细菌构成的复合菌系对石油的去除情况最好,7 d内对石油降解率可达63.7%。设计3因素3水平的正交实验对该组合中各菌株的接种量加以优化,结果见表2。
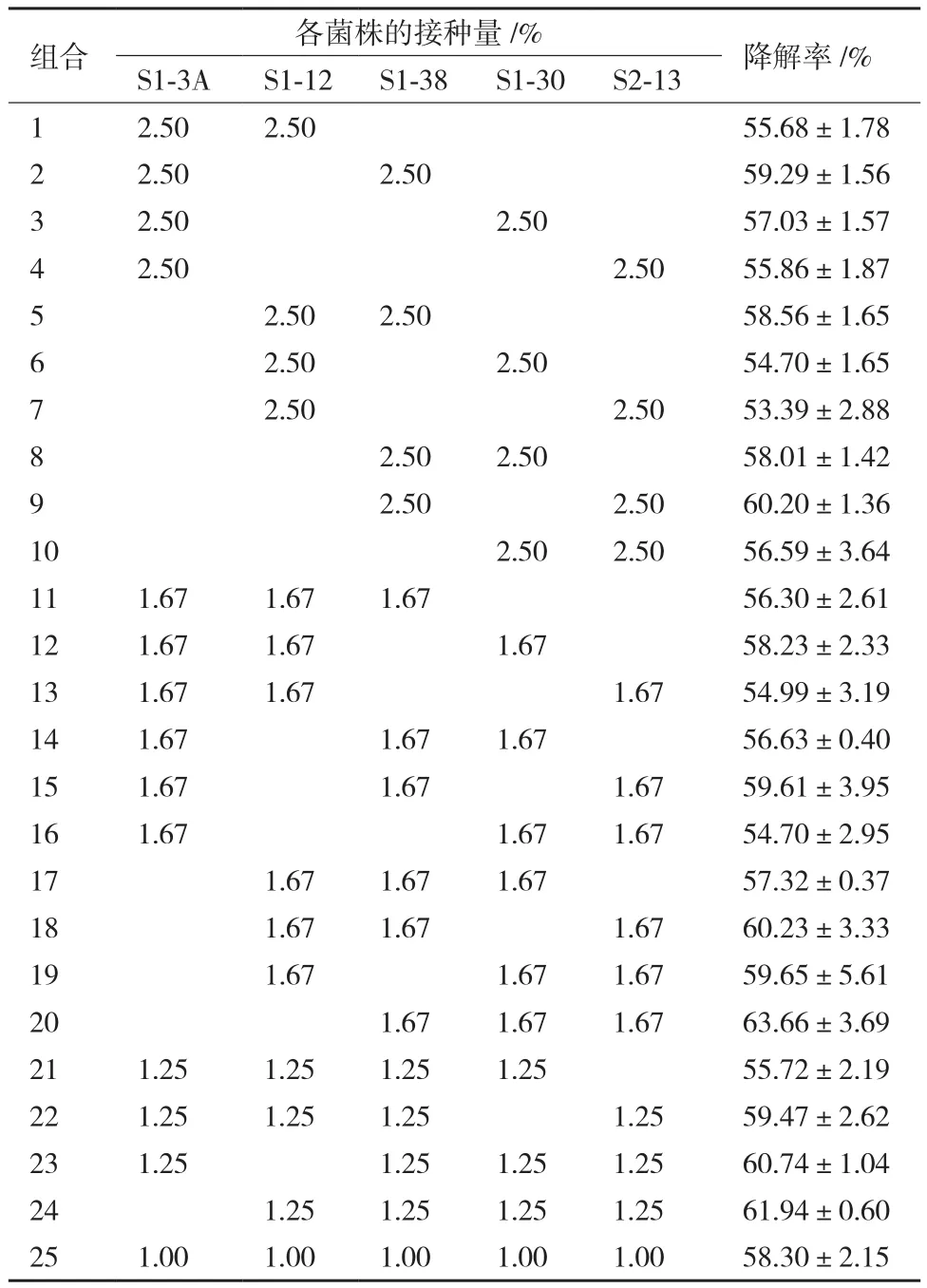
表1 各组复合菌系对原油的降解效果
表2中R值显示,接种量对S1-38的影响最大,其次为 S1-30,对S2-13的影响较小;当S1-30取2.0%水平,S1-38取1.0%水平,S2-13取1.0%水平时,各自具有最高的K值,理论上采用该接种量时,复合菌应该具有最高的石油降解率。按此接种量制备复合菌系,培养7 d测得其对石油的降解率为68.3%,高于正交实验中的任一组合,与预期相符,将该菌系命名为SQ1。
2.3石油降解菌的鉴定
3株细菌在2216E 平板上培养48-72 h后,S1-30呈不透明的淡黄色圆形菌落,直径0.5-1.5mm,表面光滑、凸起、湿润;镜检为(1 000×)G+,菌体呈短杆状,(0.3-0.6)×(1.0-4.0)μm,单生或簇生。S1-38呈淡红色圆形菌落,直径0.2-1.0 mm,表面光滑、凸起,有黏液,边缘整齐;镜检为G+,菌体呈球状,(0.5-1.0)×(0.5-1.0)μm。S2-13呈灰白色不规则圆形菌落,直径2.0-3.0 mm,凸起;镜检为G-,菌体呈杆状,(0.5-1.0)×(1.0-5.0)μm,单生。生理生化实验结果见表3。
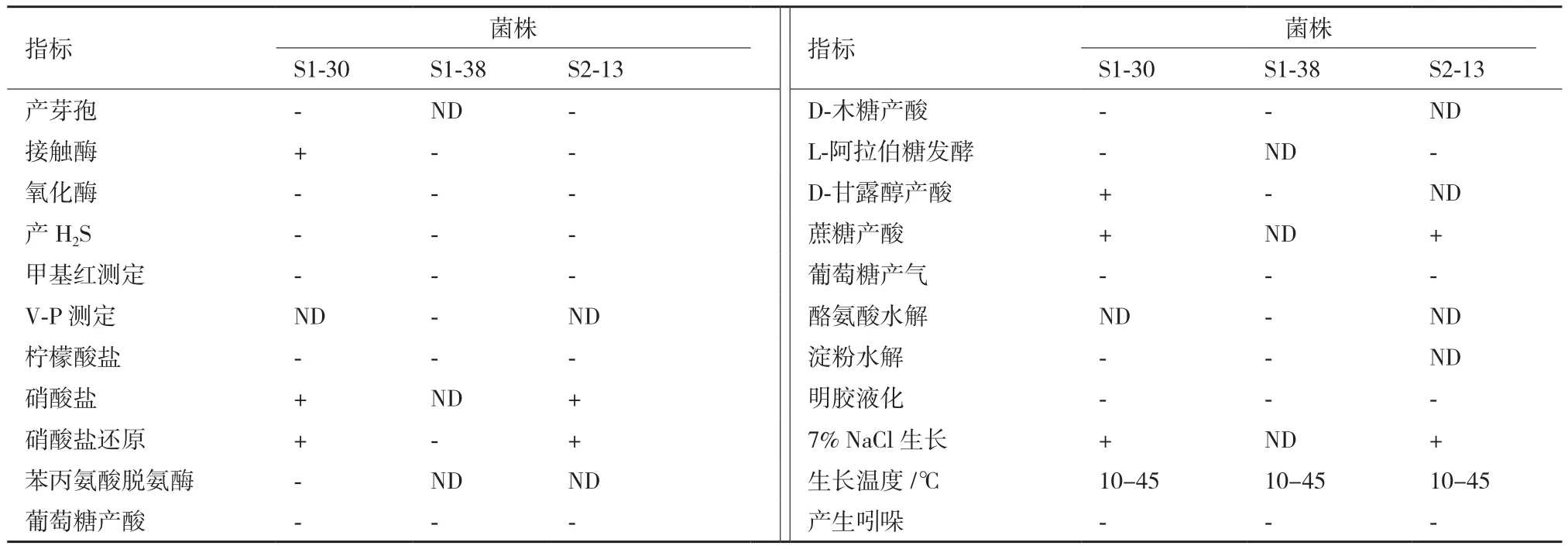
表3 3株细菌的生理生化特征
采用通用引物扩增3株细菌的16S rRNA基因序列,得到大小约为1 400-1 500 bp的片段,测序后将序列与GenBank中近缘模式种进行BLAST分析,选择相似度较高的菌株序列采用邻接法构建系统发育树,以Methanococcus vannielii作为外群,结果见图2。
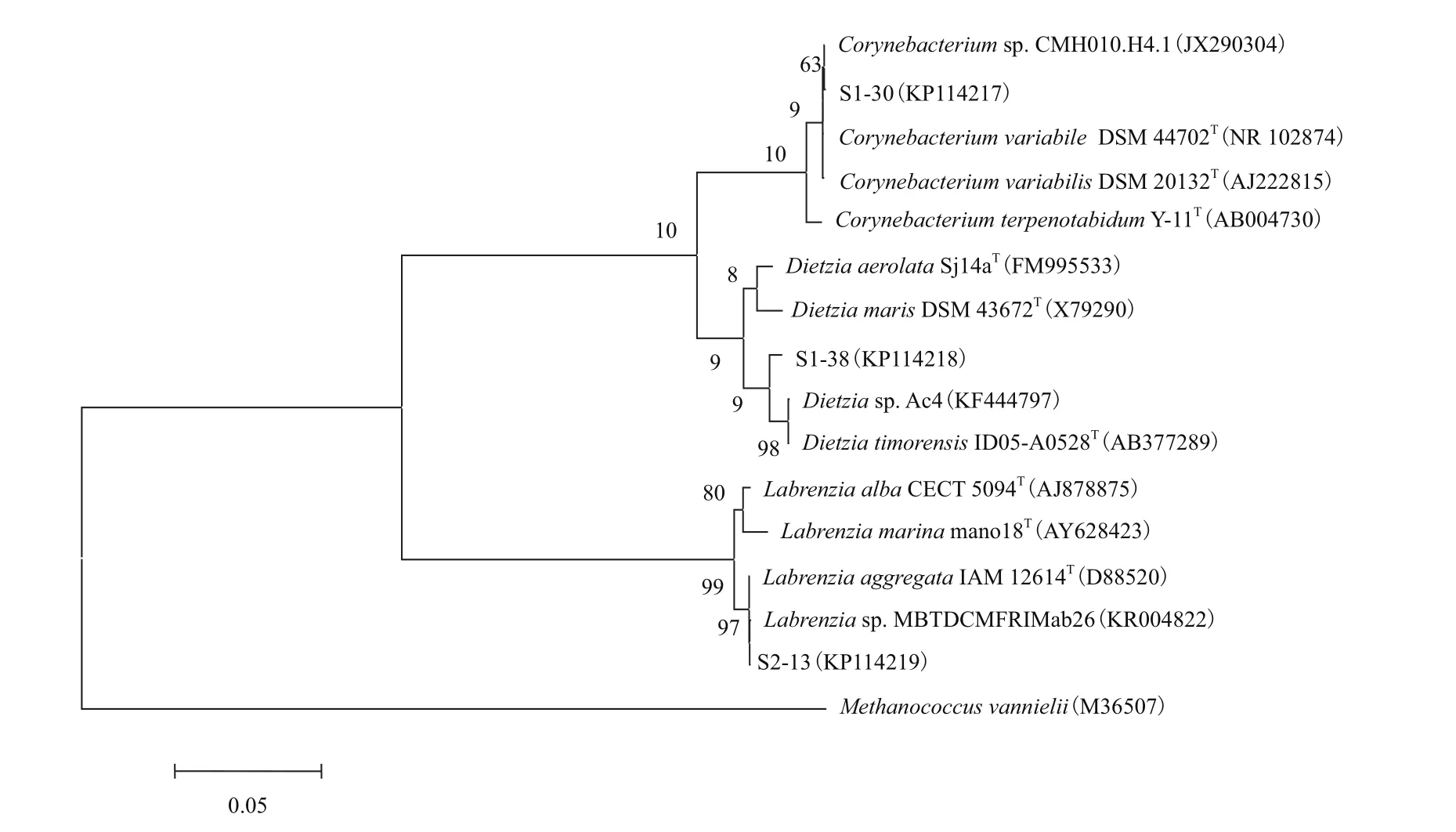
图2 基于16S rRNA基因的菌株系统发育树
由图2可见,菌株S1-30与棒杆菌属(Corynebacterium)下的几种细菌模式种聚为一个类群,与菌株Corynebacterium variabile DSM 44702T、Corynebacterium sp. CMH010.H4.1和Corynebacterium variabilis DSM 20132T的相似度均为99%;S1-38与迪茨氏菌属(Dietzia)下几种细菌模式种聚为同一类群,与Dietzia timorensis ID05-A0528T、Dietzia sp. Ac4等菌株的相似度均为99%;S2-13与拉布伦茨氏菌属(Labrenzia)细菌聚为同一类群,与菌株Labrenzia sp. MBTDCMFRIMab26的相似度达100%。结合生理生化实验结果,将菌株S1-30鉴定为棒杆菌,将S1-38鉴定为迪茨氏菌,将S2-13鉴定为拉布伦茨氏菌;GenBank登录号分别为KP114217、KP114218、KP114219。
2.4SQ1降解石油条件的优化
通过单因素实验,测定培养温度、摇床转速(通气量)、初始pH值、初始石油浓度等4种因素对复合菌系SQ1降解石油的影响,结果见图3。
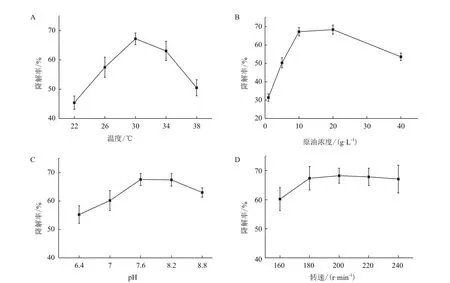
图3 环境因素对SQ1降解石油的影响
如图3-A所示,30℃是SQ1降解石油的最适温度,降解率为67.2%;过低或过高的温度对SQ1降解石油均具有负面影响,22℃和38℃时的去除率仅为45.4%和50.5%。石油浓度(图3-B)对SQ1降解石油的影响同样显著,当石油浓度为1 g/L时,其生物降解率仅为31.4%;随着浓度的提升,SQ1对石油的去除率也逐渐升高,当浓度为20 g/L时,其降解率可达67.3%;但当石油的浓度进一步升至40 g/L时,SQ1对石油的降解率出现明显的下降。图3-C所示,初始pH方面,SQ1在pH为7.0-8.8范围内对石油均有较高的降解率(>60%),当pH为7.6时,SQ1对石油的降解率最高,可达67.6%。通气量(图3-D)对石油去除率的影响较小,当摇床转速达到180 r/min以上时,SQ1对石油的降解情况无显著差别。
2.5培养过程中石油降解及菌系SQ1的变化
根据2.4的结果,将温度、pH、转速和石油浓度分别调节至 30℃、7.6、200 r/min和20 g/L,定时取样测定SQ1对石油的降解和反应体系中各细菌的生长情况。
结果(图4)显示,复合菌系SQ1对石油表现出持续的降解能力。接种后1-7 d内,石油的降解情况呈指数增长,此后石油的生物降解速率有所下降,培养11 d时,石油的去除率达到最高(73.5%)。在石油的降解的过程中,复合菌系SQ1中3种细菌的生长状况差异较大,S1-38在培养1-5 d内大量生长,7 d后活菌数呈不断下降的趋势;S2-13在培养3-9 d内活菌激增,此后有小幅下降;S1-30的生长速度较其他2株菌慢,但其活菌数始终处于增长状态,表现出持续的石油降解能力。
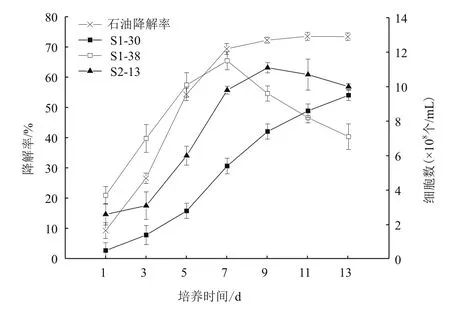
图4 培养时间对石油降解及细菌生长的影响
2.6残油组分的GC-MS分析
采用GC-MS法对石油生物降解后的残留组分定量分析,结果见图5。
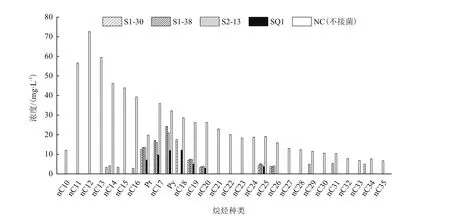
图5 降解后石油中烷烃含量
图5显示,菌株S1-30、S1-38、S2-13和复合菌系SQ1对石油总烷烃的去除率分别为87.55%、73.11%、87.55%和91.69%,均能够降解C10-C35之间的大部分正构烷烃,其中对C10-C13、C21-C24、C27、C30、C32、C33等烷烃的降解率为100%。相比于单一细菌,复合菌系SQ1降解烷烃的能力更强,对C10-C16和C21-C35之间(除C25)烷烃的降解率也达到了100%,对本研究中较难以降解的C16-C20之间烷烃的去除效果也要好于单一细菌。
3 讨论
已有报道表明,海洋环境中存着数百种微生物能够参与石油烃类的降解,其中大部分为细菌,如:不动杆菌(Acinetobacter)、假单胞菌、红球菌(Rhodococcus)、嗜盐单胞菌(Halomonas)、食烷菌(Alcanivorax)、海杆菌(Marinobacter)和 微杆菌(Microbacterium)等[7,20,21]。本研究从深圳近海分离到的22株石油降解菌,对石油的降解率最高为52.4%,与其他已报道的菌株相比,其降解能力处于中间水平[8,9,22],但本研究分离到菌株广泛分布于副球菌(Paracoccus)、拉布伦茨氏菌、鞘氨醇盒菌(Sphingopyxis)、假单胞菌以及迪茨氏菌等菌属,其中,拉布伦茨菌在海洋石油降解方面的作用为首次报道。
研究表明,分泌表面活性剂是一些菌株能够高效利用石油烃的重要因素,例如:菌株Pseudomonas sp. BP10即是通过产表面活性剂降低了培养液表面的张力,提高了对石油的摄取作用[23];Bacillus methylotrophicus USTBa不仅能够分泌表面活性剂,其产物对某些细菌还具有一定的拮抗作用[24]。依据在血平板上产生透明圈的大小和培养液的表面张力降低幅度,本研究将S1-12等7株细菌初步判定为表面活性剂高产菌,但7株细菌中S1-12、S1-30和S2-13等3株菌的石油降解能力明显强于另外4株,这说明除了产表面活性剂外,菌株的生长状态、分泌的降解酶系等因素与石油降解能力也密切相关。有报道表明,细菌产表面活性剂的多少与降解石油的能力呈正相关[25,26],但本研究的结果却有所不同,如:S1-3A和S1-38虽然表面活性剂的产生特征不明显,但对石油的降解能力却较强,这可能与其细胞表面特性有关,也可能是分泌的其他代谢产物的缘故。由于培养条件、底物浓度等外界因素与细菌产表面活性剂息息相关[24],本研究菌株所产表面活性剂的类型及产量还需通过红外法、薄层层析法等手段做进一步分析。
依据对石油的降解能力和产表面活性剂的情况,本研究选择5株石油降解菌进行复配,并通过正交实验优化接种量,构建了由棒杆菌S1-30、迪茨氏菌S1-38和拉布伦茨氏菌S2-13组成的复合菌系SQ1。结果显示,SQ1对石油的降解率可达68.3%,相比于单一细菌至少高出16.1%,GC-MS结果也表明SQ1对石油的去除效果要好于单一菌株。与其他报道的复合菌系相比,SQ1的降解能力略低于Bao等[27]采用苍白杆菌(Ochrobactrum sp.)和类短短芽孢杆菌(Brevibacillus parabrevis)构建的四元复合菌系(79%);在2%的原油浓度下,与Sathishkumar[28]构建的四元菌系降解能力相近(72%-76%);高于张海玲等[12]构建的复合菌系(61.3%)。但SQ1对石油总烷烃的去除率可达91.7%,对石油组分降解情况较为特殊。一般来说,石油中的短链和中链组分更易于生物降解[29,30],但SQ1对石油中C21-C35等长链烷烃的降解率几乎为100%,对C10-C16等中链烷烃的降解却不是很彻底,这可能是由于SQ1中的3株细菌含有长链烷烃降解需要的特殊酶系基因[31]。此外,3株细菌表现出明显的协同作用,一方面是由于菌株S1-30和S2-13通过产生表面活性剂,提高了S2-13对各种碳源的利用,也可能是3株细菌在底物利用和代谢途径上具有互补性[32]。
本研究发现,反应体系的温度、石油浓度及pH 对SQ1降解石油的影响较为显著。温度不仅可以影响微生物本身的代谢活性,对石油的物理状态、化学组成也具有一定的影响,直接关系到石油的生物降解率;而作为唯一碳源,石油浓度过低则养分不足,过高又会对微生物产生毒害;pH是通过对微生物的代谢及酶活性的调节产生影响[28,33]。总体来看,SQ1适于在中等温度、较高的石油浓度以及偏弱碱性的环境下发挥降解作用,这与我国南方的海洋环境较为接近。此外,微生物生物量的增长与石油污染的降解密切相关[2,34],从体系中3株细菌在生长情况来看,迪茨氏菌S1-38在培养初期即大量生长,这可能是因为其他2株细菌通过产表面活性剂乳化了石油,刺激了S1-38的快速摄取利用,推测其在石油降解初期发挥主要作用;此后拉布伦茨氏菌S2-13和棒杆菌S1-30进入对数期;9 d后,由于细菌进入稳定期和衰亡期,石油降解产物也可能对细菌产生反馈抑制,反应体系中石油的降解较为缓慢,但由于S1-30生物量始终处于增长状态,对复合菌系SQ1持续降解石油起到一定的作用。
本研究分离到的石油降解菌全部来自近岸海域,与人类活动的关系更为密切,其环境适应能力较强。构成菌系SQ1的3株细菌中,棒杆菌和迪茨氏菌在海洋石油降解方面的作用已有所报道[22,35],但有关拉布伦茨氏菌的报道主要集中于降解利用氨基酸、产酶等方面[36,37],本研究是首次发现其在海洋石油降解方面的作用,有关3株细菌在复合菌系SQ1中的具体作用及代谢规律等,还需要做进一步研究。另外,由于海洋环境的复杂性,SQ1对石油污染的修复效果也有待现场试验加以证实。
4 结论
从深圳近岸海域筛选得到22株高效石油降解菌,分布于副球菌、拉布伦茨氏菌、鞘氨醇盒菌、假单胞菌以及迪茨氏菌等菌属,实验室条件下,7 d内对石油的降解率最高为52.2%。
采用棒杆菌S1-30、迪茨氏菌S1-38和拉布伦茨氏菌S2-13由构建了高效石油降解复合菌系SQ1,对石油的降解率可达68.3%,比接种单菌高出16.1%。
SQ1的最适环境因素为30℃、石油浓度20 g/L、pH7.6。SQ1可降解石油大部分的烷烃组分,对长链烷烃的去除率几乎达到100%,在海洋石油污染修复中具有较强的应用潜力。
[1]Head IM, Jones DM, Röling WF. Marine microorganisms make a meal of oil[J]. Nature Reviews Microbiology, 2006, 4(4):173-82.
[2] 王鑫, 王学江, 刘免, 等. 高效石油降解菌群构建及降解性能[J]. 海洋环境科学, 2014, 33(4):576-579.
[3]陈澎, 李颖, 余小凤, 等. “7. 16”大连新港石油管道爆炸事故中的热红外溢油监测[J]. 环境工程学报, 2013, 7(2):796-800.
[4]崔志松, 李倩, 高伟, 等. 复合菌液在模拟溢油岸滩修复中的应用[J]. 应用与环境生物学报, 2013, 19(2):324-329.
[5]Mukherjee AK, Bordoloi NK. Bioremediation and reclamation of soil contaminated with petroleum oil hydrocarbons by exogenously seeded bacterial consortium:a pilot-scale study[J]. Environmental Science and Pollution Research International, 2011, 18(3):471-478.
[6]Al-Saleh E, Drobiova H, Obuekwe C. Predominant culturable crude oil-degrading bacteria in the coast of Kuwait[J]. International Biodeterioration & Biodegradation, 2009, 63(4):400-406.
[7] Hassanshahian M, Emtiazi G, Cappello S. Isolation and characterization of crude-oil-degrading bacteria from the Persian Gulf and the Caspian Sea[J]. Marine Pollution Bulletin, 2012, 64(1):7-12.
[8]郭平, 曹滨霞, 张君, 等. 海洋石油降解菌的筛选与降解性能研究[J]. 科学技术与工程, 2015, 15(11):152-154.
[9]姜肸, 高伟, 李倩, 等. 南海高效石油降解菌的筛选及降解特性研究[J]. 环境科学学报, 2012, 32(7):1572-1578.
[10]左丽敏, 马晓阳, 李智民, 等. 土壤中石油烃与降解菌群的相互作用研究[J]. 安全与环境工程, 2015, 22(4):63-68.
[11]何丽媛, 党志, 唐霞, 等. 混合菌对原油的降解及其降解性能的研究[J]. 环境科学学报, 2010, 30(6):1220-1227.
[12]Owsianiak M, Chrzanowski Ł, Szulc A, et al. Biodegradation of diesel/biodiesel blends by a consortium of hydrocarbon degraders:effect of the type of blend and the addition of biosurfactants[J]. Bioresour Technol, 2009, 100(3):1497-1500.
[13]张海玲, 杨琴, 赵敏. 石油降解菌群构建的研究[J]. 油气田环境保护, 2013, 23(3):19-21.
[14]Van Hamme JD, Ward OP. Physical and metabolic interactions of Pseudomonas sp. strain JA5-B45 and Rhodococcus sp. strain F9-D79 during growth on crude oil and effect of a chemical surfactant on them[J]. Applied & Environmental Microbiology, 2001, 67 (10):4874-4879.
[15]Li Z, Gu G, Zhao C, et al. Degradation characteristics and community structure of a hydrocarbon degrading bacterial consortium[J]. China Petroleum Processing & Petrochemical Technology, 2015, 17(3):15-24.
[16]刘陈立, 邵宗泽. 海洋石油降解微生物的分离鉴定[J]. 海洋学报, 2005, 27(4):114-120.
[17]Najafi AR, Rahimpour MR, Jahanmiri AH, et al. Enhancing biosurfactant production from an indigenous strain of Bacillus mycoides by optimizing the growth conditions using a response surface methodology[J]. Chemical Engineering Journal, 2010,163(3):188-194.
[18]蒋瑞萍, 孙丽丽, 解开治, 等. 采用紫外分光光度法评价石油烃降解菌的降解能力[J]. 石化技术与应用, 2015, 33(1):70-74.
[19]吴秉奇, 刘淑杰, 张淼, 等. 接种耐镉细菌对青葙吸收积累土壤中镉的影响[J]. 生态学杂志, 2014, 12(12):3409-3415.
[20]Petrikov K, Delegan Y, Surin A, et al. Glycolipids of Pseudomonas and Rhodococcus oil-degrading bacteria used in bioremediation preparations:Formation and structure[J]. Process Biochemistry, 2013, 48(5-6):931-935.
[21]Ibrahim ML, Ijah UJJ, Manga SB, et al. Production and partial characterization of biosurfactant produced by crude oil degrading bacteria[J]. International Biodeterioration & Biodegradation,2013, 81(1):28-34.
[22]Zhang X, Xu D, Zhu C, et al. Isolation and identification of biosurfactant producing and crude oil degrading Pseudomonas aeruginosa strains[J]. Chemical Engineering Journal, 2012, 209 (41):138-146.
[23] Kumari B, Singh SN, Singh DP. Characterization of two biosurfactant producing strains in crude oil degradation[J]. Process Biochemistry, 2012, 47(12):2463-2471.
[24]Chandankere R, Yao J, Cai M, et al. Properties and characterization of biosurfactant in crude oil biodegradation by bacterium Bacillus methylotrophicus USTBa[J]. Fuel, 2014, 122(8):140-148.
[25]Haassanshahian M, Zeynalipour MS, Musa FH. Isolation and characterization of crude oil degrading bacteria from the Persian Gulf(Khorramshahr provenance)[J]. Marine Pollution Bulletin,2014, 82(1-2):39-44.
[26]Souza EC, Vessoni-Penna TC, de Souza Oliveira RP. Biosurfactantenhanced hydrocarbon bioremediation:An overview[J]. International Biodeterioration and Biodegradation, 2014, 89(2):88-94.
[27]Bao MT, Wang LN, Sun PY, et al. Biodegradation of crude oil using an efficient microbial consortium in a simulated marine environment[J]. Marine Pollution Bulletin, 2012, 64(6):1177-1185.
[28]Sathishkumar M, Binupriya AR, Baik SH, et al. Biodegradation of crude oil by individual bacterial strains and a mixed bacterial consortium isolated from hydrocarbon contaminated areas[J]. Clean-Soil Air Water, 2008, 36(1):92-96.
[29]Mukred AM, Hamid AA, Hamzah A, et al. Development of three bacteria consortium for the bioremediation of crude petroleum-oil in contaminated water[J]. Online Journal of Biological Sciences,2008, 8(4):73-79.
[30]Nikolopoulou M, Pasadakis N, Kalogerakis N. Enhanced bioremediation of crude oil utilizing lipophilic fertilizers[J]. Desalination, 2007, 211(11):286-295.
[31]刘晔, 周卫民, 牟伯中, 等. 长链烷烃降解菌的降解特性[J].微生物学杂志, 2005, 25(6):14-18.
[32]Bouchez M, Blanchet D, Vandecasteele JP. Degradation of polycyclic aromatic hydrocarbons by pure strains and by defined strain associations:inhibition phenomena and cometabolism[J]. Applied Microbiology and Biotechnology, 1995, 43(1):156-164.
[33]Rahman KSM, Thahira-Rahman J, Lakshmanaperumalsamy P, et al. Towards efficient crude oil degradation by a mixed bacterial consortium. [J]. Bioresource Technology, 2002, 85(3):257-261.
[34]Papazi A, Kotzabasis K. Bioenergetic strategy of microalgae for the biodegradation of phenolic compounds—Exogenously supplied energy and carbon sources adjust the level of biodegradation[J]. Journal of Biotechnology, 2007, 129(4):706-716.
[35]Gao XX, Gao W, Cui ZS, et al. Biodiversity and degradation potential of oil-degrading bacteria isolated from deep-sea sediments of South Mid-Atlantic Ridge[J]. Marine Pollution Bulletin,2015, 97(1-2):373-380.
[36]郭彦岑, 尹群健, 卓书辉, 等. 琼胶酶高产细菌的分离及鉴定[J]. 西北师范大学学报:自然科学版, 2012, 48(6):76-81.
[37]Ghani NA, Norizan SN, Chan XY, et al. Labrenzia sp. BM1:a quorum quenching bacterium that degrades N-acyl homoserine lactones via lactonase activity[J]. Sensors, 2014, 14(7):11760-11769.
(责任编辑 马鑫)
Screening of Marine Crude Oil-degrading Bacteria and Construction of Microbial Consortium
WU Bing-qi LIU Shu-jie CHEN Fu-ming ZHOU Chu-ying
(Research Institute of Tsinghua University in Shenzhen,Shenzhen Environmental Microbiology Resources Development and Application Engineering Laboratory,Shenzhen 518057)
For the purpose of controlling marine oil contamination by biological treatment technology,using crude oil acting as sole carbon source and enrichment and spread plate method,high-performance oil-grading bacteria were isolated from five sampling points in the sea near Shenzhen,and bacterial consortium was constructed by mixing and orthogonal experiments. Physiological and biochemical experiments and 16S rRNA gene sequence analysis were used to identify the strains. Single-factor experiment was employed to optimize the conditions of oil biodegradation by the consortium,and gas chromatography and mass spectrum(GC-MS)were utilized to analyze its biodegradation characteristics. The results showed that 22 strains of high-performance oil-degrading bacteria were isolated,and the degrading rates varied from 34.5% to 52.2%. The degrading rate by microbial consortium SQ1 composed of S1-30,S1-38,and S2-13 strains reached 68.3%. These three strains were identified as Corynebacterium sp.,Dietzia sp. and Labrenzia sp. SQ1 was able to degrade the oil by 73.5% in 11 days under optimized conditions,referring to 30℃,pH7.6,oil concentration 20 g/L. The GC-MS results showed that consortium SQ1 was able to degrade the total alkane by 91.7%,and the more refractory C21-C35 by nearly 100%. The study shows that consortium SQ1 has great application potential of bioremediation for marine oil contamination.
oil-degrading bacteria;microbial consortium;degradation rate;identification;condition optimization
10.13560/j.cnki.biotech.bull.1985.2016.08.027
2015-10-22
深圳市科技计划项目(JCYJ20120817102941279)
吴秉奇,男,硕士,研究方向:环境微生物;E-mail:wubingqi@163.com
刘淑杰,女,硕士,研究方向:环境保护技术;E-mail:10734452@qq.com

