代谢工程改造谷氨酸棒杆菌生成丙酮酸
于莹 许湄雪 刘金雷 范荣 冯惠勇 李天明
(河北科技大学,石家庄 050018)
代谢工程改造谷氨酸棒杆菌生成丙酮酸
于莹 许湄雪 刘金雷 范荣 冯惠勇 李天明
(河北科技大学,石家庄 050018)
旨在增加谷氨酸棒杆菌代谢过程中丙酮酸的积累、减少副产物的生成,利用同源重组的方法,敲除了丙酮酸代谢过程中支流代谢途径的关键基因:丙酮酸醌氧化还原酶pqo基因、丙酮酸脱氢酶pdh基因和乳酸脱氢酶lldh基因,获得了基因缺失工程菌株。利用4.5%的葡萄糖复合培养基,工程菌经摇瓶发酵48 h,丙酮酸的浓度达到14.6 g/L,而野生菌株仅为0.45 g/L;工程菌的糖酸转化率为33.18%,比野生菌提高了32.5倍。
丙酮酸;谷氨酸棒状杆菌;丙酮酸醌氧化还原酶;丙酮酸脱氢酶;乳酸脱氢酶
丙酮酸不仅是生物体内糖代谢途径中重要的有机酸之一,而且也是合成多种化合物的前体,广泛应用于食品、农药、生化等工业中[1]。其中,丙酮酸作为酸味添加剂在食品工业中具有很大的发展潜力。丙酮酸是合成丙酮酸钙的主要原料,丙酮酸钙有减肥的特效也容易被人体吸收,因此,在食品中添加丙酮酸钙受到广大消费者的青睐。随着对丙酮酸的需求量不断扩大,对丙酮酸的研究具有十分重要的意义。
生产丙酮酸的方法主要有化学合成法,酶转化法和微生物发酵法[2]。由于发酵法具有产品纯度高、成本低、转化率高、对环境污染小等优点,已成为国内外最受青睐的生产方法。自然界中有很多的微生物可以合成丙酮酸,如:球拟酵母、假丝酵母、酿酒酵母、肠杆菌属等。代谢工程可以有效地改造微生物的代谢途径,从而获得优良高产菌株。目前,已有文献[3]报导利用代谢工程技术对大肠杆菌和酵母进行定向改造获得丙酮酸高产菌株。
谷氨酸棒杆菌(Corynebacterium glutamicum)作为一种重要的工业生产菌,被广泛应用于氨基酸、有机酸、糖醇的发酵生产,是典型的安全模式菌[4-13]。在谷氨酸棒杆菌代谢过程中,葡萄糖经糖酵解途径生成磷酸烯醇式丙酮酸,磷酸烯醇式丙酮酸在丙酮酸激酶的作用下生成丙酮酸,丙酮酸在碳源和能量代谢途径中具有关键节点的作用,是合成多种氨基酸和有机酸的前体(图1)。Blombach等[14]发现缺失丙酮酸脱氢酶pdh基因的谷氨酸棒杆菌相比于野生型,其胞内的丙酮酸含量具有明显升高;在进一步的研究中,Blombach等[15]发现通过抑制丙酮酸醌氧化还原酶pqo基因能够影响L-缬氨酸的产量,进而提高了丙酮酸的积累,这些研究为实现细胞内大量积累丙酮酸提供了方向。
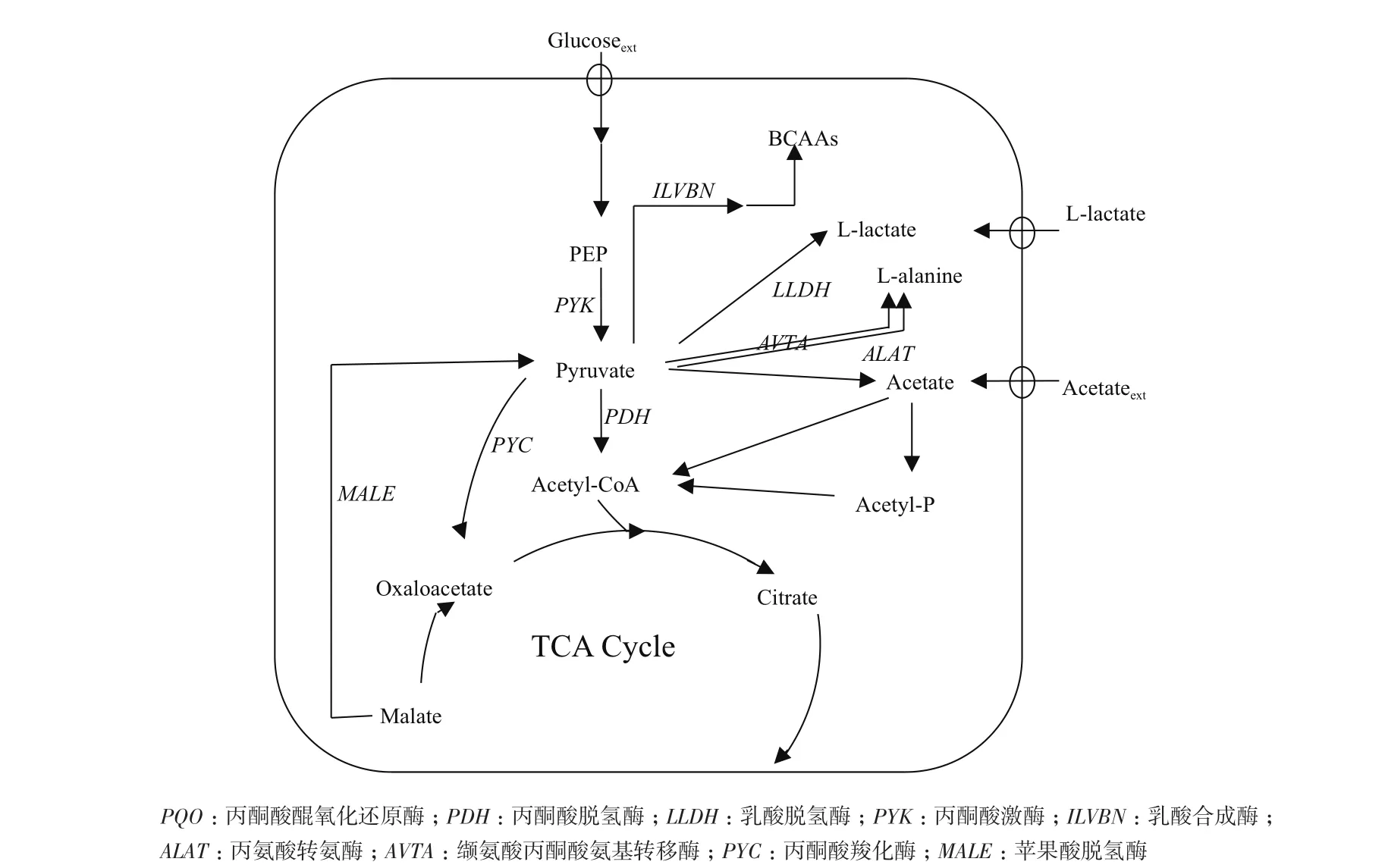
图1 Corynebacterium glutamicum ATCC 13032代谢途径
本研究利用两次同源重组方法和蔗糖致死基因sacB反向筛选技术,敲除控制丙酮酸代谢过程中支流代谢途径的关键基因:丙酮酸醌氧化还原酶pqo基因、丙酮酸脱氢酶pdh基因和乳酸脱氢酶lldh基因[16-19],以期遏制由丙酮酸生成乙酸、乙酰辅酶A 和L-乳酸的支路代谢途径,使丙酮酸产量得到积累,减少副产物的生成,从而为谷氨酸棒杆菌工业化生产丙酮酸奠定基础。
1 材料与方法
1.1材料
1.1.1菌株与质粒 本实验所用的菌株、质粒见表1。
1.1.2主要试剂 High-Fidelity DNA 聚合酶购自北京全式金生物技术有限公司;限制性内切酶、T4连接酶购自NEB公司;质粒提取试剂盒、DNA回收试剂盒购自天根生化科技(北京)有限公司;PCR引物由上海英潍捷基生物技术有限公司合成;其他试剂均为国产分析纯,购自生工生物(上海)股份有限公司。
1.1.3仪器与设备 PCR 扩增仪,Eppendorf Mastercycler gradient;全自动凝胶成像系统,Bio-Rad Molecular Imager Gel DOC XR;台式高速离心机,Eppe-ndorf Mini Spin;高速冷冻离心机,Thermo Sorvall Evolution RC;酶标仪,Bioteke Powerwave;高效液相色谱仪,Agilent 1260 Infinity。

表1 本实验所用的菌株和质粒及寡核苷酸
1.1.4培养基 LB培养基:1%胰蛋白胨,0.5%酵母提取物,1%氯化钠,调pH至7.0,固体培养基需加2.2%的琼脂。
发酵培养基:4.5%葡萄糖,0.5%乙酸钠,0.1%NHNO3,0.3%(NH4)2SO4,0.1% KH2PO4,0.05% MgSO4,0.02% CaCl2,0.001% FeSO4,0.001% MnSO4,0.0002% CuSO4,0.0001% ZnSO4,0.0002%生物素。
1.2方法
1.2.1基因敲除打靶质粒的构建 pqo基因敲除自杀质粒构建方法为:以谷氨酸棒杆菌基因组为模板,利用PCR扩增出含有pqo基因的上、下同源臂片段,通过Overlapping PCR使 pqo基因的上、下同源臂基因融合,然后将所获得的基因片段纯化后与pK18mobsacB空质粒经HindⅢ和NdeⅠ双酶切后,将同源臂基因片段连接到质粒上,获得自杀质粒pK18mobsacB△pqo。本研究用于pdh基因敲除的自杀质粒pK18mobsacB△pdh和用于lldh基因敲除的自杀质粒pK18mobsacB△lldh利用类似方法获得。
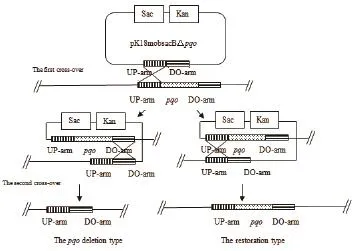
图2 同源交换原理示意图
1.2.2敲除系统用于谷氨酸棒杆菌基因的敲除 谷氨酸棒杆菌的基因敲除是利用负筛选标记通过两次同源重组的方法来实现的,原理示意图如图2所示。以获得突变菌株C. glutamicum△pqo为例,首先将pK18mobsacB△pqo经电转化转入野生型谷氨酸棒杆菌感受态细胞中,用含有卡那霉素的LB固体培养基30℃恒温培养48-60 h,经菌落PCR验证筛选出第一次同源重组的工程菌,即pK18mobsacB△pqo经同源重组整合到染色体上。再将其接到LB液体培养基中,摇床振荡自由培养12 h后,涂布于含有10%蔗糖的LB固体培养基上,利用蔗糖致死基因sacB负筛选出第二次同源重组的转化子,将其一一对应分别扩培到含10%蔗糖和30.0 μg/mL Kan抗性的LB固体培养基中,培养1 d左右。最后挑选在蔗糖板上生长而Kan板上不长的菌落,利用菌落PCR验证筛选出阳性工程菌C. glutamicum△pqo。利用此方法将pK18mobsacB△pdh转入C. glutamicum△pqo中,获得C. glutamicum△pqo△pdh。将pK18mobsacB△lldh转入C. glutamicum△pqo△pdh中,获得C. glutamicum△pqo△pdh△lldh。
1.2.3基因缺失突变菌株生长情况验证 将验证正确的工程菌C. glutamicum△pqo、C. glutamicum△pqo △pdh、C. glutamicum△pqo△pdh△lldh和野生型菌株接种到摇瓶中作种子过夜培养,后收集菌体,各株菌分别接种到含1.5%葡萄糖的LB培养基、含1.5%葡萄糖和0.5%乙酸钠的LB培养基中,同时摇瓶振荡培养,而后通过测定OD值,定期记录菌株生长情况。
1.2.4丙酮酸的发酵方法 从LB固体培养基上挑取工程菌C. glutamicum△pqo、C. glutamicum△pqo △pdh、C. glutamicum△pqo△pdh△lldh与野生型菌株,分别接种到LB培养基中作种子,30℃、180 r/min培养过夜,后将培养物离心收集菌体,转移到含有发酵培养基的三角瓶中,以4.5%的葡萄糖作为碳源,相同条件继续摇床振荡培养,发酵48 h,整个发酵过程中定时调节pH,使之维持在7.0。
1.2.5高效液相色谱检测丙酮酸的含量 将待测发酵液经转速8 000 r/min离心10 min,取上清液经0.22 μm醋酸纤维素滤膜过滤后,利用高效液相色谱法分析丙酮酸的含量。检测条件为:美国伯乐Aminex HPX-87H Column,流动相0.005 mol/L H2SO4,检测波长210 nm,流速0.6 mL/min,柱温55℃,进样量20 μL。
2 结果
2.1基因敲除打靶质粒的构建与验证
利用Overlapping PCR将目的基因的上、下同源臂基因片段融合搭接,后与pK18mobsacB空质粒同时双酶切,获得用于基因打靶的重组质粒,而后通过双酶切鉴定上下同源臂是否连接到载体上。重组质粒pK18mobsacB△pqo的双酶切验证结果(图3-A)显示,经限制性内切酶酶切后,分别得到大小为5 400 bp的质粒和大小为2 000 bp的同源臂片段,与理论值一致,表明pqo基因的上、下同源臂基因连接至pK18mobsacB空质粒。重组质粒pK18mobsacB△pdh的双酶切验证结果(图3-B)显示,经限制性内切酶酶切后,分别得到大小为5 400 bp的质粒和大小为2 200 bp的同源臂片段,与理论值一致,表明pdh基因的上、下同源臂基因连接至pK18mobsacB空质粒。重组质粒pK18mobsacB△lldh的双酶切验证结果(图3-C)显示,经限制性内切酶酶切后,分别得到大小为5 400 bp的质粒和大小为1 800 bp的同源臂片段,与理论值一致,表明lldh基因的上、下同源臂基因连接至pK18mobsacB空质粒。
2.2谷氨酸棒杆菌基因敲除菌株的验证
由图2同源重组示意图可知,谷氨酸棒杆菌经过两次同源重组,能够出现一种情况是基因缺失突变菌株,另一种情况是基因恢复菌株,因而需要通过PCR对发生第2次同源重组的突变菌株进行验证。将打靶质粒pK18mobsacB△pqo经电转化转入野生型谷氨酸棒杆菌感受态细胞中,用含有卡那霉素的固体LB培养基筛选得到第一次同源重组的工程菌,再利用蔗糖致死基因sacB基因负筛选出第二次同源重组的转化子,最后得到C. glutamicum△pqo。通过NCBI查找谷氨酸棒杆菌ATCC13032基因组序列中pqo基因序列,设计敲除验证引物,分别以基因敲除菌株C. glutamicum△pqo与野生菌株的基因组DNA为模板,进行PCR验证,结果(图4-A)显示,野生菌扩增出大小为850 bp的pqo基因片段,而C. glutamicum△pqo菌株没有扩增片段,说明C. glutamicum△pqo工程菌株基因组中的pqo基因已被敲除。同样,将pK18mobsacB△pdh转入C. glutamicum△pqo中,获得C. glutamicum△pqo△pdh,分别以C. glutamicum△pqo和C. glutamicum△pqo△pdh为模板,PCR验证pdh基因片段,结果如图4-B所示。将pK18mobsacB△lldh转入C. glutamicum△pqo△pdh中,获得C. glutamicum△pqo△pdh△lldh,并分别以C. glutamicum△pqo△pdh和C. glutamicum△pqo△pdh △lldh为模板,PCR验证lldh基因片段,结果如图4-C所示。
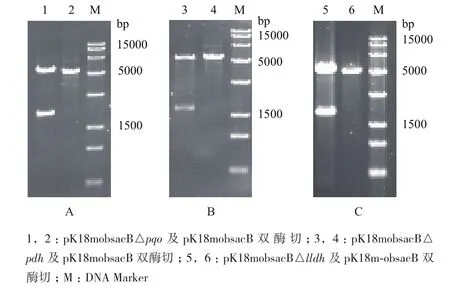
图3 重组质粒pK18mobsacB△pqo(A)、pK18mobsacB △pdh(B)、pK18mobsacB△lldh(C)的酶切验证
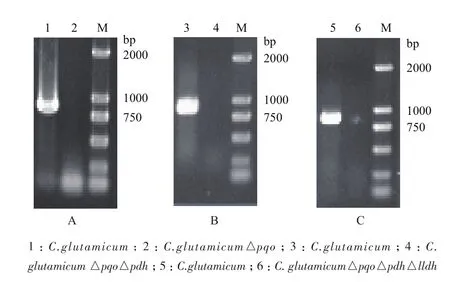
图4 工程菌株C. glutamicum△pqo(A)、C. glutamicum △pqo△pdh(B)、C. glutamicum△pqo△pdh△lldh (C)PCR验证
2.3工程菌生长特性研究
C. glutamicum△pqo、C. glutamicum△pqo△pdh、C. glutamicum△pqo△pdh△lldh与野生菌以等OD值分别接种到含1.5%葡萄糖的LB培养基、含1.5%葡萄糖和0.5%乙酸钠的LB培养基中。生长状况如图5所示,生长曲线(图5-A)显示在不含乙酸的培养基中,野生型C. glutamicum和C. glutamicum △pqo生长,而敲除了pdh基因的C. glutamicum△pqo△pdh和C. glutamicum△pqo△pdh△lldh无法正常生长;生长曲线(图5-B)显示在含乙酸的培养基中,由于乙酸的补给,使pdh基因缺失菌株C. glutamicum△pqo △pdh和C. glutamicum△pqo△pdh△lldh与野生型C. glutamicum和C. glutamicum △pqo菌株生长的趋势一致。这就说明由于敲除了pdh基因,使丙酮酸脱氢酶失活,遏制了丙酮酸生成乙酰辅酶A,影响了代谢途径的TCA循环,只有在培养基中添加0.5%乙酸钠才能正常生长,进而验证了乙酸钠是工程菌能够正常生长所必需的碳源。
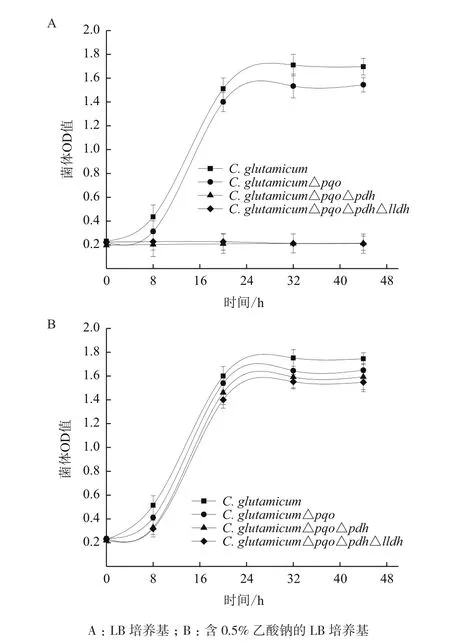
图5 野生菌与工程菌的生长情况
2.4工程菌丙酮酸的发酵
为了进一步验证重组菌种中丙酮酸产量,分别将C. glutamicum△pqo、C. glutamicum△pqo△pdh、C. glutamicum△pqo△pdh△lldh与野生菌C. glutamicum同时以4.5%的葡萄糖作为碳源进行摇瓶发酵,48 h后收集发酵液,利用高效液相色谱法测定发酵液中丙酮酸的含量。结果(图6)显示,由于工程菌C. glutamicum△pqo△pdh△lldh敲除了丙酮酸代谢过程中的3个关键基因,遏制了副产物的形成,因而积累的丙酮酸含量要明显高于野生型菌株、仅敲除一个基因的C. glutamicum△pqo和敲除两个基因的C. glutamicum△pqo△pdh。经计算可知野生型菌株发酵后丙酮酸含量仅为0.45 g/L,而工程菌C. glutamicum△pqo△pdh△lldh发酵后丙酮酸含量为14.6 g/L,工程菌比野生菌提高了31.4倍;工程菌株的糖酸转化率为33.18%,比野生菌提高了32.5倍。
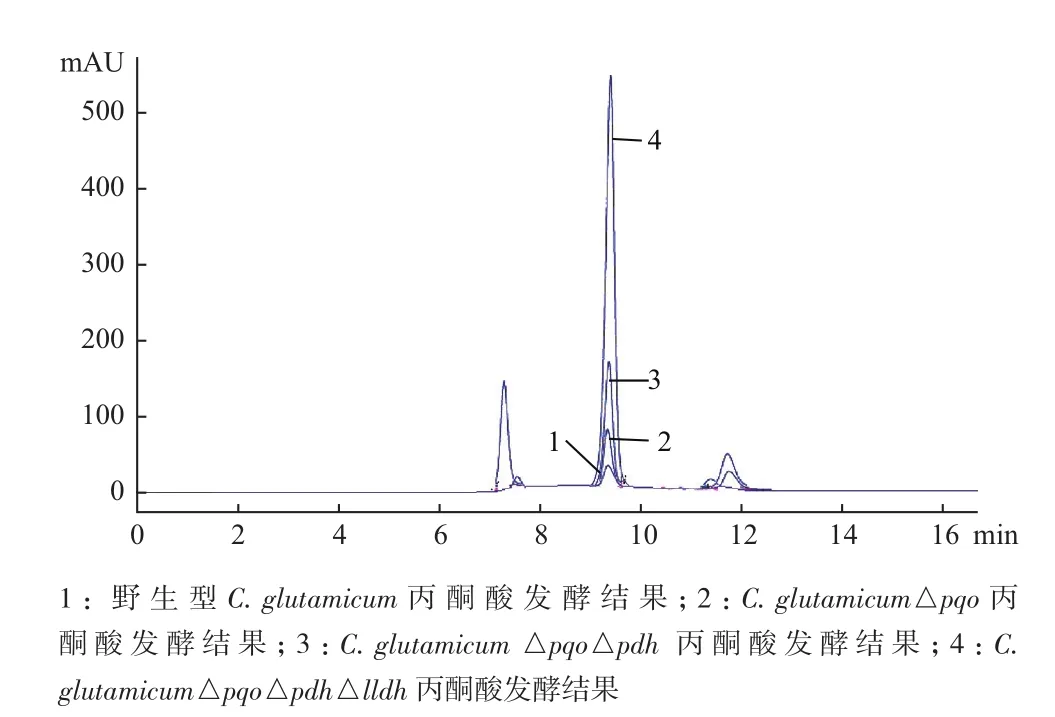
图6 野生菌与工程菌丙酮酸发酵液相对比图
3 讨论
传统的发酵法生产丙酮酸,产量和糖酸转化率较低,远远不能满足日益增加的市场需求。近年来应用缺陷性酵母菌和大肠杆菌来生产丙酮酸成为人们研究的热点。Li等[20]利用酵母发酵生产丙酮酸,通过复杂的发酵设置维持培养基中维生素的浓度来提高丙酮酸的产量;Zelic等[21]利用大肠杆菌工程菌进行摇瓶发酵,能够达到每摩尔葡萄糖生产1.9 mol的丙酮酸;Zhu等[22]改造的用于丙酮酸生产的大肠杆菌,在最优发酵条件下丙酮酸产量能达到90 g/L。而谷氨酸棒杆菌是食品级微生物、鲁棒性好,更适合工业生产条件,利用的代谢工程改造谷氨酸棒杆菌生产丙酮酸,目前还未见报道。本研究根据谷氨酸棒杆菌的代谢途径设计策略,敲除pqo基因使其以葡萄糖为碳源不能生成乙酸;以pqo基因缺失的工程菌C. glutamicum△pqo为基础菌株敲除编码丙酮酸脱氢酶pdh基因,有效的阻止了乙酰辅酶A的生成,影响了TCA循环,只有在含乙酸钠的培养基中才能够稳定生长;敲除编码乳酸脱氢酶的lldh基因,减少了副产物乳酸的生成,从而提高了丙酮酸的积累量。获得的基因缺失工程菌C. glutamicum △pqo△pdh△lldh,以4.5%的葡萄糖为碳源,进行摇瓶发酵,48 h后,工程菌中丙酮酸的浓度为14.6 g/L,比野生菌株提高了14.15 g/L;工程菌的糖酸转化率为33.18%,比野生菌提高了32.5倍,生产效率为0.3 g/(L·h)。具Zhu[22]和Li[20]的报道,大肠杆菌工程菌发酵罐发酵的生产率为1.23 g/(L·h),酵母菌发酵罐发酵的生产率为1.875 g/(L·h)。与大肠杆菌工程菌和酵母菌工程菌相比,本研究还有一定差距,一是因为摇瓶发酵条件没有发酵罐的优越,二是因为丙酮酸形成后通过合成缬氨酸、丙氨酸等支流代谢丙酮酸分解消耗,如编码生成乙酰乳酸的乳酸合成酶ilvbn基因、编码生成L-丙氨酸的丙氨酸转氨酶alat基因和缬氨酸丙酮酸氨基转移酶avta基因、编码生成草酰乙酸的丙酮酸羧化酶pyc基因等,影响了丙酮酸的积累。另外,强化丙酮酸合成代谢的基因,如编码苹果酸脱氢酶male基因将苹果酸代谢生成丙酮酸等,将有利于丙酮酸的积累,提高丙酮酸的产量。
4 结论
本研究利用两次同源重组方法和蔗糖致死基因sacB反向筛选技术,敲除了控制丙酮酸代谢过程中支流代谢途径的关键基因,获得基因缺失工程菌C. glutamicum △pqo△pdh△lldh,进而遏制了由丙酮酸生成乙酸、乙酰辅酶A和L-乳酸的支路代谢途径,使丙酮酸积累量大大增加,以4.5%的葡萄糖为碳源,进行摇瓶发酵,48 h后,工程菌中丙酮酸的浓度为14.6 g/L,比野生菌株提高了14.15 g/L;工程菌的糖酸转化率为33.18%,比野生菌提高了32.5倍,生产效率为0.3 g/(L·h)。
[1]郭英, 付涛. 发酵法生产丙酮酸的工艺条件优化研究[J]. 石家庄职业技术学院学报, 2010, 22(2):26-28.
[2]刘立明, 李寅, 堵国成, 等. 生物技术法生产丙酮酸的研究进展[J]. 生物工程学报, 2002, 18(6):651-655.
[3]Wieschalka S, Blombach B, EikmannsI BJ. Engineering Corynebacterium glutamicum for the production of pyruvate[J]. Applied Microbiology and Biotechnology, 2012, 94:449-459.
[4]吴新阳, 裴广胜, 等. 不同抑制剂对谷氨酸棒状杆菌磷酸烯醇式丙酮酸羧化酶酶活的影响[J]. 生物技术通报, 2013(7):172-178.
[5]张君胜, 王扬, 杨晓志, 张力. 谷氨酸棒杆菌关键酶活性与苏氨酸高产的关系[J]. 湖南农业科学, 2011(9):23-25.
[6]Wieschalka S, Blombach B, Bott M, et al. Bio-based production of organic acids with Corynebacterium glutamicum[J]. Microbial Biotechnology, 2012, 6:87-102.
[7]Lin LH, Hu XQ, Xu DQ, et al. Co-expression of feedback-resistant threonine dehydratase and acetohydroxy acid synthase increase L-isoleucine production in Corynebacterium glutamicum[J]. Metabolic Engineering, 2012, 14:542-550.
[8]Radoš D, Turner DL, Fonseca LL, et al. Carbon flux analysis by13C nuclear magnetic resonance to determine the effect of CO2on anaerobic succinate production by Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2014,80(10):3015-3024.
[9]Bommareddy RR, Chen Z, Rappert S, et al. A de novo NADPH generation pathway for improving lysine production of Corynebacterium glutamicum by rational design of the coenzyme specificity of glyceraldehyde 3-phosphate dehydrogenase[J]. Metabolic Engineering, 2014, 25:30-37.
[10]Vogt M, Haas S, Klaffl S, et al. Pushing product formation to its limit:metabolic engineering of Corynebacterium glutamicum for L-leucine overproduction[J]. Metabolic Engineering, 2014, 22:40-52.
[11]Baumgart M, Unthan S, Rückert C, et al. Construction of a prophage-free variant of Corynebacterium glutamicum ATCC 13032 for use as a platform strain for basic research and industrial biotechnology[J]. Applied and Environmental Microbiology,2013, 79(19):6006-6015.
[12]Zhang Y, Shang XL, Lai SHJ, et al. Development and application of an arabinose-inducible expression system by facilitating inducer uptake in Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2012, 78(16):5831-5838.
[13]Yamamoto S, Gunji W, Suzuki H, et al. Overexpression of genes encoding glycolytic enzymes in Corynebacterium glutamicum enhances glucose metabolism and alanine production under oxygen deprivation conditions[J]. Applied and Environmental Microbiology, 2012, 78(12):4447-4457.
[14]Blombach B, Schreiner ME, Holátko J, et al. L-valine production with pyruvate dehyrogenase complex-deficient Corynebacterium glutamicum[J]. Appl Environ Microbiol, 2007, 73(7):2079-2084.
[15]Blombach B, Schreiner ME, Bartek T, et al. Corynebacterium glutamicum tailored for high-yield L-valine production[J]. Appl Microbiol Biotechnol, 2008, 79:471-479.
[16] Litsanov B, Brocker M, Bott M. Toward homosuccinate fermentation:metabolic engineering of Corynebacterium glutamicum for anaerobic production of succinate from glucose and formate[J]. Applied and Environmental Microbiology, 2012, 78(9):3325-3337.
[17] Yamamoto S, Gunji W, Suzuki H, et al. Overexpression of genes encoding glycolytic enzymes in Corynebacterium glutamicum enhances glucose metabolism and alanine production under oxygen deprivation conditions[J]. Applied and Environmental Microbiology, 2012, 78(12):4447-4457.
[18]Zahoor A, Lindner SN, Wendisch VF. Metabolic engineering of Corynebacterium glutamicum aimed at alternative carbon sources and new products[J]. Computational and Structural Biotechnology Journal, 2012, 3:1-11.
[19]Litsanov B, Kabus A, Brocker M, et al. Efficient aerobic succinate production from glucose in minimal medium with Corynebacterium glutamicum[J]. Microbial Biotechnology, 2012, 5(1):116-128.
[20]Li Y, Chen J, Lun SY. Biotechnological production of pyruvic acid[J]. Applied Microbiology and Biotechnology, 2001, 57:451-459.
[21]Zelić B, Gerharz T, Bott M, et al. Fed-batch process for pyruvate production by recombinant Escherichia coli YYC202 strain[J]. Eng in life Sci, 2003, 3:299-305.
[22]Zhu Y, Eiteman MA, Altman R, Altman E. High glycolytic flux improves pyruvate production by a metabolically engineered Escherichia coli strain[J]. Appl Environ Microbiol, 2008, 74(21):6649-6655.
(责任编辑 马鑫)
Metabolic Engineering for Modifying Corynebacterium glutamicum to Produce More Pyruvate
YU Ying XU Mei-xue LIU Jin-lei FAN Rong FENG Hui-yong LI Tian-ming
(Hebei University of Science and Technology,Shijiazhuang 050018)
In order to increase the accumulation of pyruvate while decrease the production of by-products during the metabolic process of Corynebacterium glutamicum,the homologous recombination was employed to knock out the key genes of pyruvate:quinone oxidoreductase (pqo),pyruvate dehydrogenase(pdh)and L-lactate dehydrogenase(lldh)in the tributary metabolic pathways,thus the gene deletion mutation strains were constructed. Using compound medium with 4.5% glucose as carbon sources,the concentration of pyruvate with mutation strain at shake flask fermentation for 48 hours reached 14.6 g/L,while that with the wild type only was 0.45 g/L. The conversion rate of sugar to acid with the mutation strain was 33.18% and 32.5 times higher than the wild type.
pyruvate;Corynebacterium glutamicum;pyruvate:quinone oxidoreductase;pyruvate dehydrogenase;L-lactate dehydrogenase
10.13560/j.cnki.biotech.bull.1985.2016.08.033
2015-12-08
国家科技支撑计划(2015BAD15B05)
于莹,女,硕士研究生,研究方向:代谢工程与合成生物学;E-mail:503572523@qq.com
李天明,男,助理研究员,研究方向:代谢工程与合成生物学;E-mail:iamltm2000@hotmail.com

