芸薹属栽培种ALS1-3的克隆及序列分析
孙妍妍,曲高平,胡胜武
(西北农林科技大学 农学院,陕西 杨凌 712100)
芸薹属栽培种ALS1-3的克隆及序列分析
孙妍妍,曲高平,胡胜武
(西北农林科技大学 农学院,陕西 杨凌 712100)
[摘要]【目的】 揭示6种芸薹属植物乙酰乳酸合成酶(ALS)基因的结构特征,为ALS酶的遗传操作和抗除草剂种质创制奠定基础。【方法】 利用特异PCR方法,对甘蓝型油菜(Brassica napus L.)、甘蓝(B.oleracea)、芥菜型油菜(B.juncea)、白菜型油菜(B.rapa)、埃塞俄比亚芥(B.carinata)、黑芥(B.nigra)等6个芸薹属16份材料的ALS基因进行扩增、克隆测序及生物信息学分析。【结果】 除1个B.nigra材料未扩增出任何产物外,其余参试材料中均有PCR扩增产物,共成功克隆了30个ALS基因(GenBank登录号为KM816807~KM816836),且克隆的基因ALS1、ALS2、ALS3均不含内含子,只有一个开放阅读框。ALS1基因开放阅读框长为1 968 bp,编码655个氨基酸;ALS2基因开放阅读框长为1 914 bp,编码637个氨基酸;ALS3基因开放阅读框长为1 959 bp,编码652个氨基酸。参试材料中,在ALS1基因中检测到7个SNP,其中4个导致氨基酸突变;在ALS2基因中检测到3个SNP,其中2个导致氨基酸突变;在ALS3基因中检测到25个SNP,其中2个导致氨基酸突变。在3类ALS蛋白中,ALS1和ALS3遗传距离较近,与ALS2的遗传距离相对较远。ALS1基因定位于C01染色体上,ALS2和ALS3基因定位于A01染色体上。【结论】 参试的6个芸薹属不同材料间ALS1、ALS2、ALS3基因序列及其编码的蛋白序列均存在差异。
[关键词]芸薹属植物;乙酰乳酸合成酶基因;基因克隆;生物信息学;基因序列分析
油菜是我国乃至全世界的主要油料作物,随着我国国民经济的快速发展,农村青壮年劳动力向城市大量转移,轻简化、机械化已成为油菜产业发展的主要方向。杂草的综合有效治理是实现油菜机械化、轻简化的重要技术环节[1],选育和推广抗除草剂油菜品种是克服油菜田间草害最为经济有效的措施。目前,抗除草剂油菜主要分为两大类:转基因抗除草剂油菜(主要抗性基因有抗草胺膦的bar和抗草甘膦的aroA)和非转基因抗除草剂油菜。Swanson等[2]和Hattori等[3]采用小孢子化学诱变的方法获得2个抗咪唑啉酮类除草剂的油菜突变体PM1和PM2。PM1和PM2均由ALS基因点突变所致,其中PM1是BnALS1基因上的第653位丝氨酸(Ser)突变为天冬酰胺(Asn),PM2是基因BnALS3的第574位色氨酸(Trp)突变为亮氨酸(Leu)(对应拟南芥序列)。2004年江苏省农业科学院在油菜和大豆轮作喷施咪唑啉酮类除草剂的试验田中,发现了自然突变的除草剂抗性油菜新种质M9,该突变体抗性稳定、抗性效应明显[4-5]。胡茂龙等[6]研究表明,M9抗性由1个显性核基因控制,其BnALS1的抗性突变位点是Ser-653-Asn(对应拟南芥序列)。目前,国外生产上大面积使用转基因抗除草剂油菜品种,而非转基因油菜主要是由PM1和PM2选育得到的抗咪唑啉酮类油菜。
乙酰乳酸合成酶(Acetolactate synthase,ALS)抑制剂类除草剂是发展最快、品种最多、市场最大的一类选择性除草剂,该类除草剂由于生物活性高、杀草谱广、对人和动物十分安全而应用于多种作物[7-9]。乙酰乳酸合成酶(ALS)是控制植物体内合成支链氨基酸(包括亮氨酸、异亮氨酸、缬氨酸)公共途径的关键酶,该酶由催化亚基和调控亚基组成[10-16],它同时催化2个平行反应,催化2分子丙酮酸缩合形成2-乙酰乳酸并释放出CO2,最终生成缬氨酸和亮氨酸;催化1分子丙酮酸和1分子2-氧丁酸缩合形成乙酰羟基丁酸,最终生成异亮氨酸[17]。细菌、真菌、藻类和植物中均含有ALS,植物ALS由核基因编码,定位于叶绿体[18]。ALS是磺酰脲类、咪唑啉酮类等5类高选择性、低毒化学除草剂的作用靶标[19-21],因此ALS基因是抗除草剂遗传操作的一个重要候选基因,具有重要的研究价值。
甘蓝型油菜(BrassicanapusL.)基因组内存在5个ALS基因拷贝(ALS1-ALS5),芸薹属植物中ALS基因以多基因家族的形式存在,其多样性超过烟草和拟南芥中的ALS基因[22]。ALS1和ALS3享有广泛的同源性,它们可能是编码植物生长和发育所必需的ALS酶。ALS2与ALS1和ALS3在DNA序列上差异较大,在成熟蛋白编码区、N端叶绿体转运肽的编码区及基因上游非编码区具有独特的特征,并且只在成熟的胚珠和不成熟的种子胚胎中表达,表明ALS2酶可能在种子的发育中起特殊作用。ALS4和ALS5的编码区被打断,可能是缺失功能ALS基因。分析B.napus的二倍体祖先B.rapa和B.oleracea表明,ALS2、ALS3和ALS4起源于B.rapa,而ALS1和ALS5起源于B.oleracea[22]。
本研究拟对6个不同芸薹属植物的ALS基因进行克隆测序及生物信息学分析,以明确这6个主要芸薹属植物ALS基因的特征特性,为ALS酶的遗传操作和抗除草剂种质创制奠定基础。
1材料与方法
1.1材料
供试材料包括4个Brassicanapus材料:中双9号、SH11、秦油3号、新型甘蓝型油菜;2个B.oleracea材料:特选中甘11、晚丰;3个B.juncea材料:渭源大黄芥、关中芥菜、黔西牛尾油菜;4个B.rapa材料:金秋66、彬县义门油菜、Opava、Rapido;2个B.carinata材料:Dodalla、09H2901003;1个B.nigra材料:Sizaja。上述材料均由西北农林科技大学油菜研究中心提供,2013年9月播种于陕西杨凌西北农林科技大学农学院标本区,10月28日取嫩叶,用于基因组DNA的提取。
ExTap酶、dNTP Mixture、10×ExTapBuffer及pMD 19-T Vector由大连宝生物TaKaRa公司生产;6×DNA Loading Buffer和琼脂糖凝胶DNA回收试剂盒及DH5α大肠杆菌感受态细胞均由天根生化科技(北京)有限公司生产;引物合成和测序均由上海立菲生物技术有限公司完成。
1.2ALS基因的克隆
叶片全基因组DNA采用CTAB法提取。利用胡茂龙等[6]设计的3对引物:BnALS1-F1/BnALS1-R1、BnALS2-F1/BnALS2-R1、BnALS3-F1/BnALS3-R1分别扩增ALS1、ALS2和ALS3基因序列。PCR反应体系:模板DNA 2 μL,ExTaq酶(5 U/μL)0.15 μL,10×ExTaqBuffer 2 μL,上下游引物(10 μmol/L)各0.75 μL,ddH2O补足20 μL。PCR循环参数:94 ℃预变性5 min;94 ℃变性40 s,55 ℃退火40 s,72 ℃延伸2.5 min,34个循环;72 ℃延伸10 min。PCR扩增产物经1.0%琼脂糖凝胶电泳分离,然后切下目的条带,用离心柱型琼脂糖凝胶DNA回收试剂盒回收纯化(具体步骤参照试剂盒说明书)。回收产物经pMD 19-T Vector连接、DH5α大肠杆菌感受态转化、蓝白斑筛选、菌落PCR鉴定后,每个材料每个基因分别随机选取 3~5 个阳性克隆送至上海立菲生物技术有限公司测序。将克隆成功的30条核酸序列提交至GenBank数据库中,并获得登录号。
1.3生物信息学分析
利用NCBI网站(http://www.ncbi.nlm.nih.gov/)进行核酸和蛋白质的Blast分析,利用Clustal X 2.0.12软件进行核酸和蛋白质的序列比对,利用MEGA 5.1软件,采用极大似然法(Maximum-likelihood,ML)构建ALS蛋白系统发育进化树。利用在线软件http://brassicadb.org/brad/对ALS基因进行定位。
2结果与分析
2.1ALS基因的克隆
试验结果显示,除在1个B.nigra材料中未扩增出任何产物外,其他参试材料中均有PCR扩增产物(图1~3)。
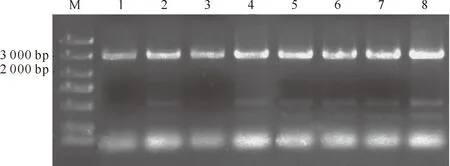
图 1 ALS1基因扩增结果 M. Marker Ⅲ;1.中双9号;2.SH11;3.秦油3号;4.新型甘蓝型油菜;5.特选中甘11;6.晚丰;7.Dodalla;8.09H2901003Fig.1 PCR amplification of ALS1 M. Marker Ⅲ;1.Zhongshuang No.9;2.SH11;3.Qinyou No.3;4.New type Brassica napus; 5.Texuanzhonggan No.11;6.Wanfeng;7.Dodalla;8.09H2901003
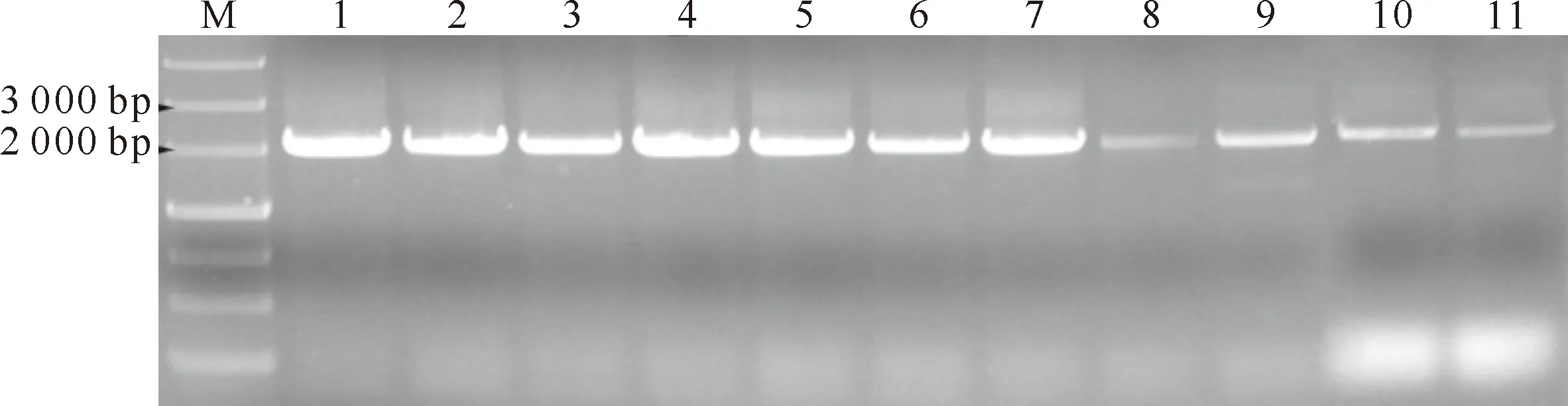
图 2 ALS2基因扩增结果 M.Marker Ⅲ;1.中双9号;2.SH11;3.秦油3号;4.新型甘蓝型油菜;5.渭源大黄芥; 6.关中芥菜;7.黔西牛尾油菜;8.金秋66;9.彬县义门油菜;10.Opava;11.RapidoFig.2 PCR amplification of ALS2 M.Marker Ⅲ;1.Zhongshuang No.9;2.SH11;3.Qinyou No.3;4.New type Brassica napus;5.Weiyuandahuangjie; 6.Guanzhongjiecai;7.Qianxiniuweiyoucai;8.Jinqiu 66;9.Binxianyimenyoucai;10.Opava;11.Rapido
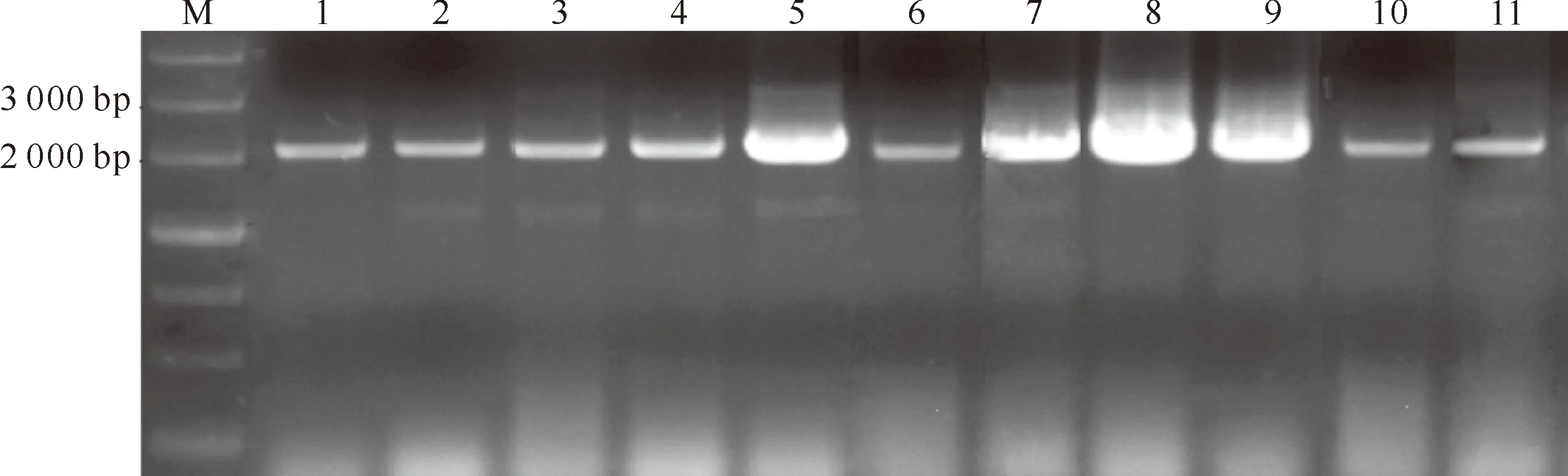
图 3 ALS3基因扩增结果 M.Marker Ⅲ;1.中双9号;2.SH11;3.秦油3号;4.新型甘蓝型油菜;5.渭源大黄芥;6.关中芥菜; 7.黔西牛尾油菜;8.金秋66;9.彬县义门油菜;10.Opava;11.RapidoFig.3 PCR amplification of ALS3 M.Marker Ⅲ;1.Zhongshuang No.9;2.SH11;3.Qinyou No.3;4.New type Brassica napus;5.Weiyuandahuangjie; 6.Guanzhongjiecai;7.Qianxiniuweiyoucai;8.Jinqiu 66;9.Binxianyimenyoucai;10.Opava;11.Rapido
在参试的B.napus、B.oleracea、B.carinata中扩增得到ALS1基因(图1),在B.napus、B.rapa、B.juncea中扩增得到ALS2(图2)和ALS3基因(图3)。对扩增目的片段进行回收、克隆、测序,共成功克隆了30个ALS基因,并在GenBank数据库中登录,获得登录号KM816807-KM816836(表1)。经过NCBI的Blast分析,结果显示所克隆基因分别与NCBI上已公开的B.napusALS1(Z11524)、ALS2(Z11525)、ALS3(Z11526)基因的一致率达到99%,证明克隆所得基因与预期结果相符。
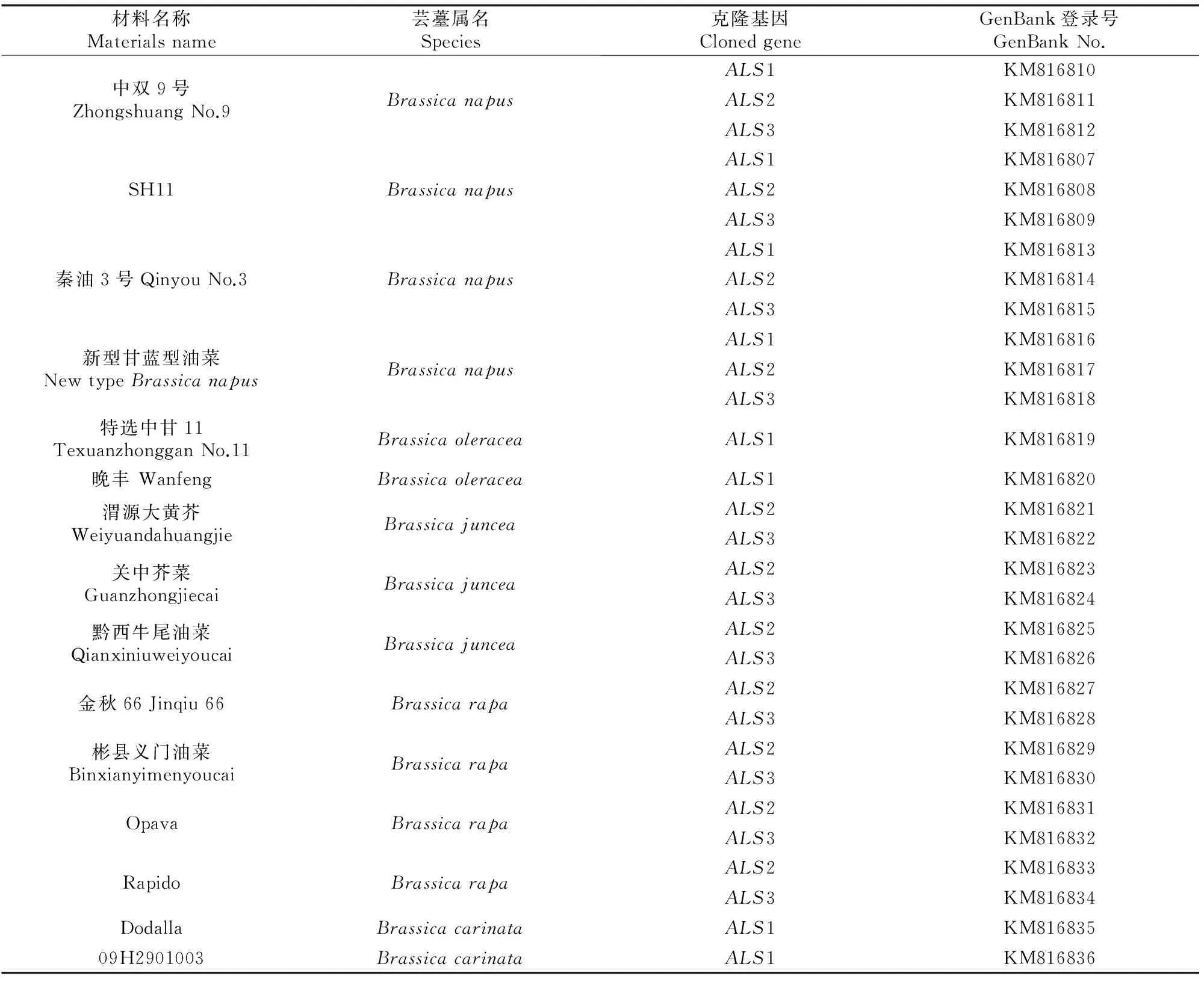
表 1 参试材料及成功克隆的ALS基因Table 1 Plant materials and cloned ALS genes
2.2ALS基因的序列分析
利用NCBI网站的ORF Finder软件对2.1节克隆得到的基因序列进行分析,结果表明:5种芸薹属植物中所克隆的ALS基因均不含内含子,只有1个开放阅读框。ALS1基因开放阅读框长为1 968 bp,编码655个氨基酸;ALS2基因开放阅读框长为1 914 bp,编码637个氨基酸;ALS3基因开放阅读框长为1 959 bp,编码652个氨基酸。利用Clustal X 2.0.12软件分析表明,5个芸薹属不同种材料的ALS基因核酸序列存在差异(表2~4),导致由其编码的氨基酸也存在差异。对8个材料ALS1基因的比对分析发现了7个SNP,其中4个SNP可导致氨基酸突变,B.napus材料SH11的ALS1基因与其他B.napus材料间存在3个SNP,但只有1个SNP导致氨基酸突变。对11个材料ALS2基因的比对分析发现了3个SNP,其中2个SNP导致氨基酸突变。对11个材料ALS3基因的比对分析发现了25个SNP,但仅有2个SNP导致氨基酸突变。从SNP数量来看,在自然条件下ALS3发生同义突变的概率最大,而发生错义突变的概率最小;ALS2序列相对保守,发生同义突变的概率最小,但最易发生错义突变。
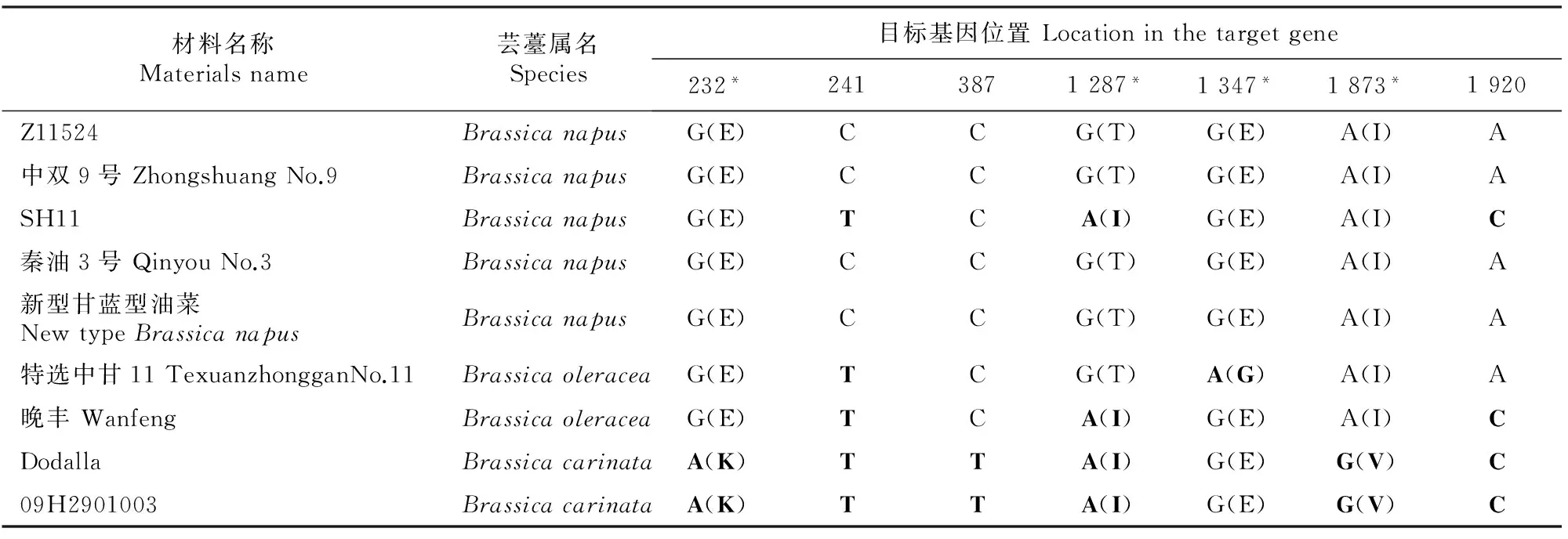
表 2 ALS1核酸和蛋白质序列变异Table 2 Mutations of ALS1 DNA and protein sequences
注:Z11524是NCBI上已公开的ALS1序列;黑体表示突变位点;目标基因位置中括弧外为编码核苷酸,括弧内为引起突变的氨基酸;*表示此位点发生错义突变。下表同。
Note:Z11524 is the sequence ofALS1 published in NCBI;Mutations are showed in bold;outside brackets are the coding nucleotides,in brackets are the mutation amino acid.*means missense mutation at this site.The same below tables.
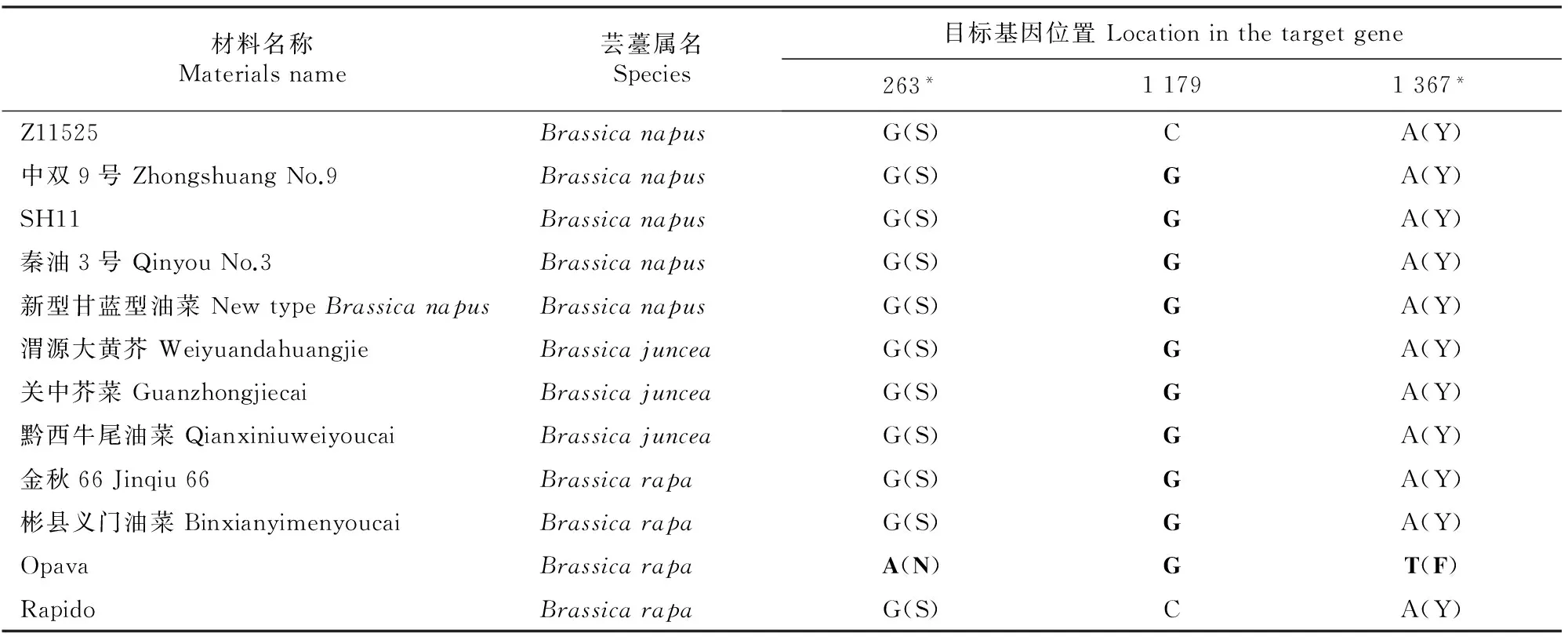
表 3 ALS2核酸和蛋白质序列变异Table 3 Mutations of ALS2 DNA and protein sequences
注:Z11525是NCBI上已公开的ALS2序列。
Note:Z11525 is the sequence ofALS2 published in NCBI.
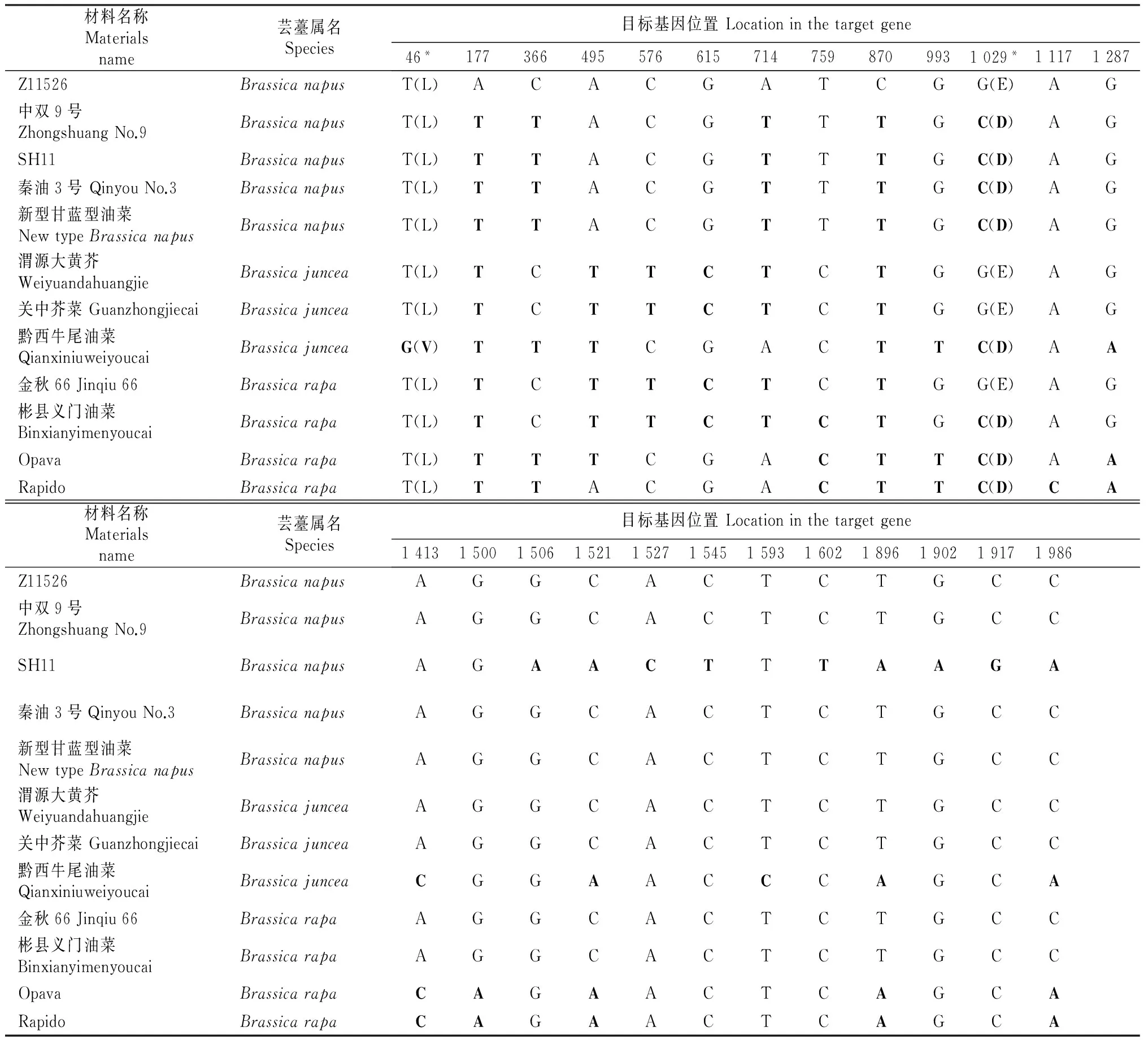
表 4 ALS3核酸和蛋白质序列变异Table 4 Mutations of ALS3 DNA and protein sequences
注:Z11526是NCBI上已公开的ALS3序列。
Note:Z11526 is the sequence ofALS3 published in NCBI.
2.3ALS蛋白系统发育进化树的构建
利用MEGA 5.1软件,采用极大似然法(Maximum-likelihood,ML)对2.1节成功克隆的30个ALS基因编码的蛋白质序列构建系统发育进化树,结果见图4。图4表明,ALS1、ALS2、ALS3独自聚为一类,然后ALS1和ALS3聚在一起,最后与ALS2聚在一起,说明ALS1和ALS3遗传距离较近,与ALS2的遗传距离相对较远。8个材料共编码了4种不同ALS1蛋白(SH11、晚丰、Dodalla和09H2901003、其他4个材料)。11个材料编码了2种ALS3蛋白,其中渭源大黄芥、关中芥菜、金秋66编码的ALS3与基因库中Z11526编码的相同;其他8个材料编码的ALS3相同。11个材料共编码了2种ALS2蛋白,Opava编码了1种ALS2,其他10个材料编码的ALS2相同。
2.4ALS基因的定位预测
利用网站http://brassicadb.org/brad/对B.oleracea和B.rapa材料的ALS基因进行染色体定位,结果表明(1)B.oleracea材料ALS1基因编码区序列(第1-1 968碱基)定位于C01染色体上(第17 124 680-17 122 713碱基);(2)B.rapa材料ALS2基因编码区(第199-1 217碱基)定位于A01染色体上(第14 732 437-14 731 422碱基);(3)B.rapa材料ALS3基因编码区(第1-1 959碱基)也定位于A01染色体上(第14 183 751-14 181 793 碱基)。ALS基因编码区序列与定位在该染色体区间内的碱基序列一致率高达99%,说明定位结果真实可靠。
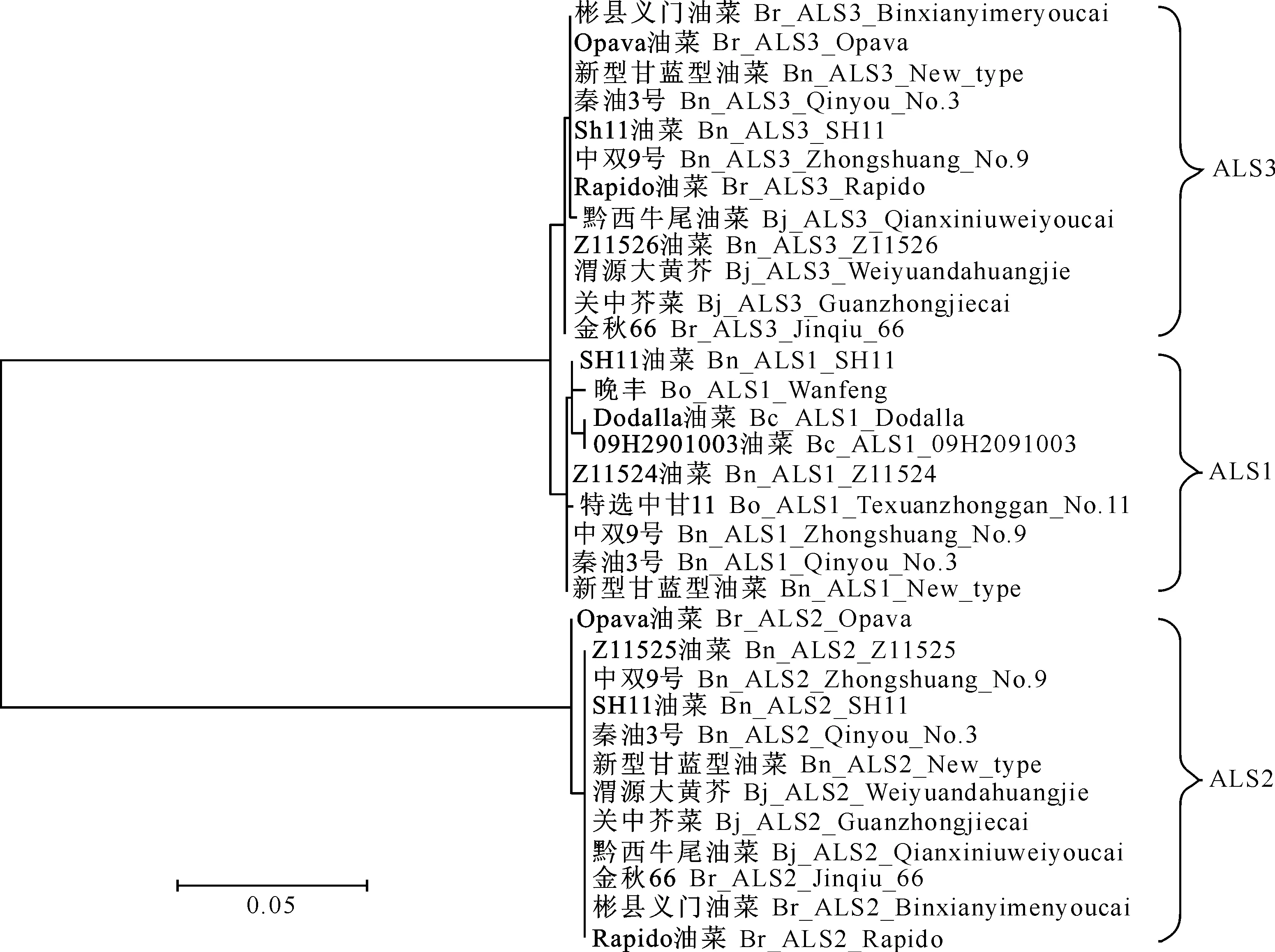
图 4 5种芸薹属植物ALS1、ALS2、ALS3蛋白的系统发育进化树 每个分支上的数值为自展检验值(1 000次重复)Fig.4 Phylogenic analysis of ALS1,ALS2,and ALS3 proteins from five Brassica species The number for each interior branch is the bootstraps value (1 000 replicates)
3结论与讨论
在以轻简化、机械化为油菜产业发展主要方向的大环境下,抗除草剂油菜品种的选育和推广工作势在必行,而乙酰乳酸合成酶(ALS)基因作为抗除草剂遗传操作的一个重要候选基因得到了人们的日益重视。本研究对6个不同芸薹属16个植物材料ALS基因进行PCR扩增,其中15个材料的ALS基因克隆成功,仅1个B.nigra材料未扩增成功,其原因可能是ALS1基因定位于C基因组中,ALS2和ALS3基因定位于A基因组中,而B.nigra材料只包含B基因组。本研究所用的ALS1、ALS2、ALS3基因特异引物在B.nigra中均无PCR扩增产物,因此B.nigra的ALS基因有待进一步研究。
对15个不同材料ALS1、ALS2、ALS3基因的分析发现,ALS1和ALS3同源性较高(达到95%以上),这2个基因中的突变位点较多,其中一部分形成错义突变;而ALS2与ALS1和ALS3同源性较低,在DNA序列上差异较大,相对保守,极少出现核苷酸的突变。Rutledge等[22]认为,ALS1和ALS3可能是编码植物生长和发育所必需的ALS酶,而ALS2在成熟蛋白编码区、N端叶绿体转运肽的编码区及基因上游非编码区具有独特的特征,可能具有不同于ALS1和ALS3的功能。本研究结果与Rutledge等[22]的上述观点相符合。经过对15种材料ALS蛋白的比对分析,未发现以往报道的对抗除草剂有贡献的SNP位点。以往报道的对除草剂抗性有贡献的位点主要为Ala122、Pro197、Ala205、Asp376、Trp574和Ser653[23-24](以拟南芥序列为参照)。本研究所发现的ALS突变位点是否对除草剂有贡献,尚有待于进一步研究。
[参考文献]
[1]张宏军,张佳,刘学,等.我国油菜田农药的登记及应用概况 [J].湖北农业科学,2008,47(7):846-851.
Zhang H J,Zhang J,Liu X,et al.The review of the registration and application of pesticide of canola in China [J].Hubei Agricultural Sciences,2008,47(7):846-851.
[2]Swanson E B,Herrgesell M J,Arnoldo M,et al.Microspore mu-tagenesis and selection:canola plants with field tolerance to the imidazolinones [J].Theoretical and Applied Genetics,1989,78(4):525-530.
[3]Hattori J,Brown D,Mourad G,et al.An acetohydroxy acid sy-nthase mutant reveals a single site involved in multiple herbicide resistance [J].Mol Gen Genet,1995,246(4):419-425.
[4]高建芹,浦惠明,龙卫华,等.抗咪唑啉酮油菜种质的发现与鉴定 [J].植物遗传资源学报,2010,11(3):369-373.
Gao J Q,Pu H M,Long W H,et al.Identification of imidazolidone resistant oilseed rape mutant [J].Journal of Plant Genetic Resoures,2010,11(3):369-373.
[5]浦惠明,高建芹,龙卫华,等.油菜抗咪唑啉酮性状的遗传及其应用 [J].中国油料作物学报,2011,33(1):15-19.
Pu H M,Gao J Q,Long W H,et al.Studies on inheritance of imidazolinones resistance inBrassicanapusand its utilization [J].Chinese Journal of Oil Crop Sciences,2011,33(1):15-19.
[6]胡茂龙,浦惠明,高建芹,等.油菜抗乙酰乳酸合成酶抑制剂类除草剂突变体M9抗性基因的分离与鉴定 [J].中国农业科学,2012,45(20):4326-4334.
Hu M L,Pu H M,Gao J Q,et al.Inheritance and gene cloning of an ALS inhabiting herbicide resistant mutant line M9 inBrassicanapus[J].Scientia Agricultura Sinica,2012,45(20):4326-4334.
[7]张一宾.抑制乙酰乳酸合成酶(ALS)除草剂世界市场及品种发展概述 [J].现代农药,2005,6(4):28-31.
Zhang Y B.Summary of global market and development of ALS inhibitors [J].Modern Agrochemicals,2005,6(4):28-31.
[8]苏少泉.抗除草剂作物的发展 [J].农药研究与应用,2008,12(5):1-6.
Su S Q.The development of crops [J].Agrochemicals Research & Application,2008,12(5):1-6.
[9]薛思佳,邹金山.新型乙酰乳酸合成酶(ALS)抑制剂的研究进展 [J].化学世界,2000,41(8):399-403.
Xue S J,Zou J S.Progress on new acetolactate synthase (ALS) inhibitor [J].Chemical World,2000,41(8):399-403.
[10]Umbarger H E.Amino acid biosynthesis and its regulation [J].Annu Rev Biochem,1978,47:533-606.
[11]McCourt J A,Duggleby R G.Acetohydroxyacid synthase and its role in the biosynthetic pathway for branched-chain amino acids [J].Amino Acids,2006,31:173-210.
[12]Ray T B.Site of action of chlorsulfuron:inhibition of valine and isoleucine biosynthesis in plants [J].Plant Physiol,1984,75:827-831.
[13]Schloss J V.Acetolactate synthase,mechanism of action and its herbicide binding site [J].Pestic Sci,1990,29(3):283-292.
[14]Shaner D L,Anderson P C,Stidham M A.Imidazolinones:potent inhibitors of acetohydroxyacid synthase [J].Plant Physiol,1984,76:545-546.
[15]Stidham M A,Shaner D L.Imidazolinone inhibition of acetohydroxyacid synthase in vitroandinvivo[J].Pestic Sci,1990,29:335-340.
[16]Duggleby R G,McCourt J A,Guddat L W.Structure and mechanism of inhibition of plant acetohydroxyacid synthase [J].Plant Physiol Biochem,2008,46:309-324.
[17]Chen H,Saksa K,Zhao F Y,et al.Genetic analysis of pathway regulation for enhancing branched-chain amino acid biosynthesis in plants [J].The Plant Journal,2010,63:573-583.
[18]赵跃芳.乙酰羟酸合成酶亚基间相互作用的研究 [D].天津:南开大学,2013.
Zhao Y F.Study on subunits interaction of acetohydroxyacid synthase [D].Tianjin:Nankai University,2013.
[19]Binder S,Knill T,Schuster J.Branched-chain amino acid metabolism in higher plants [J].Physiologia Plantarum,2007,129(1):68-78.
[20]Zhou Q Y,Liu W P,Zhang Y S,et al.Action mechanisms of acetolactate synthase-inhibiting herbicides [J].Pest Biochem Physiol,2007,89:89-96.
[21]McCourt J A,Pang S S,King-Scott J,et al.Herbicide-binding sites revealed in the structure of plant acetohydroxyacid synthase [J].Proc Natl Acad Sci USA,2006,103(3):569-573.
[22]Rutledge R G,Quellet T,Hattori J,et al.Molecular characterization and genetic origin of theBrassicanapusacetohydroxyacid synthase multigene family [J].Mol Gen Genet,1991,229:31-40.
[23]Tan S,Evants R R,Dahmer M L,et al.Imidazolinone-tolerant crops:history,current status and future [J].Pest Management Science,2005,61:246-257.
[24]Heap I.The international survey of herbicide resistant weeds [J/OL] Integrated Pest Management,2014,3:281-301,[2014-10-18].http://www.weedscience.org.
DOI:网络出版时间:2016-06-0816:2110.13207/j.cnki.jnwafu.2016.07.015
[收稿日期]2014-12-05
[基金项目]现代农业产业技术体系建设专项(CARS-13);“十一五”国家科技支撑计划项目(2010BAD01B02)
[作者简介]孙妍妍(1988-),女,吉林长春人,在读硕士,主要从事油菜遗传育种研究。E-mail:15929800833@163.com [通信作者]胡胜武(1966-),男,陕西柞水人,教授,博士生导师,主要从事油菜遗传育种研究。 E-mail:swhu83251@nwsuaf.edu.cn
[中图分类号]S503.53
[文献标志码]A
[文章编号]1671-9387(2016)07-0101-08
Cloning and sequence analysis ofALS1-3 genes from cultivatedBrassicaspecies
SUN Yanyan,QU Gaoping,HU Shengwu
(CollegeofAgronomy,NorthwestA&FUniversity,Yangling,Shaanxi712100,China)
Abstract:【Objective】 This study revealed the acetolactate synthetase (ALS) genes from six Brassica species to lay foundation for genetic manipulation of ALS and creation of herbicide resistant materials.【Method】 Specific PCR method was used to isolate ALS genes from 16 accessions of six Brassica species,including Brassica napus L.,B.oleracea,B.juncea,B.rapa,B.carinata,and B.nigra.Bioinformatics and the isolated ALS genes and their encoding products were also analyzed. 【Result】 Except for one B.nigra accession,ALS genes from other 15 accessions were successfully amplified.In total,30 ALS genes were cloned and their GenBank numbers (from KM816807 to KM816836) were obtained.ALS1,ALS2 and ALS3 had no introns and only one open reading frame (ORF).ORF of ALS1 was 1 968 bp in length,encoding a peptide of 655 amino acids.ORF of ALS2 was 1 914 bp in length,encoding a peptide of 637 amino acids.ORF of ALS3 was 1 959 bp in length,encoding a peptide of 652 amino acids.Seven single nucleotide polymorphisms (SNP) were detected among ALS1,and four of them resulted in amino acid change.Three SNPs were detected among ALS2,and two of them resulted in amino acid change.Twenty-five SNPs were detected among ALS3,and two of them resulted in amino acid change.Genetic distance between ALS1 and ALS3 was relatively closer than that with ALS2.ALS1 was located in C01 chromosome,while ALS2 and ALS3 were located in A01 chromosome.【Conclusion】 Nucleic acid differences in ALS1,ALS2,and ALS3 were detected among six Brassica species,resulting in changes of several amino acids of the encoded proteins.
Key words:Brassica species;acetolactate synthetase (ALS) gene;gene cloning;bioinformatics;sequence analysis
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20160608.1621.030.html

