几类主要病原需氧放线菌菌属的SecA1基因分析研究
牟丽丽,涂云华,明春艳,夏茂宁,孟俞辰,康颖倩
几类主要病原需氧放线菌菌属的SecA1基因分析研究
牟丽丽1,涂云华2,明春艳1,夏茂宁1,孟俞辰1,康颖倩1
1.贵州医科大学微生物学教研室,贵阳550004;2.贵州医科大学附属医院皮肤性病专科,贵州550004
摘要:目的了解几类主要病原需氧放线菌不同菌属间SecA1基因碱基突变特点,为病原需氧放线菌的实验室诊断,分类及致病性提供一定的实验室理论数据。方法以162株需氧放线菌为实验研究对象,通过分子生物学方法对需氧放线菌SecA1基因扩增测序,及收集NCBI数据库中已发表的需氧放线菌SecA1基因,运用多序列对比及DNAStar软件对需氧放线菌SecA1序列进行特异性基序分析及SecA1蛋白的二级结构和表位特性预测。结果需氧放线菌不同菌属间SecA1序列在300~350 bp区域有着特异性差异,经预测该区域编码的SecA1蛋白二级结构属间大致相同,而蛋白质柔性区域、亲水性及表面可及性等性质在属间存在差异,其中以Mycobacteria菌属的差异较为明显。结论需氧放线菌SecA1基因不同菌属间具有特异性的碱基突变,这种突变导致属间SecA1蛋白部分性质有差异,与各菌属致病性可能存在着一定的相关性。
关键词:需氧放线菌;SecA1基因;碱基突变;SecA1 蛋白
Supported by the National Natural Science Fund of China (Nos. 31060006 & 31260029), the Social Development Research Project of Guizhou (No.[2011] 3017), and the Social Development & Livelihood Plan of Guiyang (No.[2011103]16)
需氧放线菌(aerobic actinomycetes)是一群专性需氧,革兰氏染色阳性,呈丝状分支,生长速度相对较缓慢的一类微生物。放线菌广泛存在于世界各地富含腐生物质的土壤中和其它自然环境中。然而,大量增加的临床病例证实有部分放线菌是导致人类疾病的诱因,尤其导致一些机体严重的免疫系统障碍[1-3]。病原需氧放线菌如Nocardia属的菌株可引起免疫缺陷或非免疫缺陷的宿主疾病[4-6],其中对人或动物致病的主要有星形诺卡氏菌(Nocardiaasteroides)和巴西诺卡氏菌(Nocardiatransvalensis),星形诺卡氏菌是需氧放线菌中最常见的动物病原体,最常见的动物疾病为牛乳腺炎[7-8],也曾被报道引起鱼类感染[9-10];其他可引起人兽共患病的病原需氧放线菌如Mycobacteria菌属的牛型分支杆菌(Mycobacteriumbovis)为牛的致病菌,可引起牛的结核感染,而人由于食入未经消毒或已污染此菌的牛乳也可被感染[11]。
在需氧放线菌等原核生物的系统发育分析及分类地位的确定中,16S rRNA 基因作为一种理想的研究材料而被广泛应用。然而随着研究的深入,16S rRNA 基因也表现出了不容忽视的缺点:高保守性使其不能很好的区分属内不同的物种;在基因组内的多拷贝性使确定其序列的准确性降低[12]。为弥补16S rRNA基因的缺点,人们开始尝试使用其他不同的看家基因(gyrB、recA基因等) 进行放线菌的分子生物学分析。 Fischer[13]扩增了分枝杆菌属29个种的47株ATCC标准菌和59株临床分离菌的SecA1基因序列,首次发现分泌相关基因可以用于区分分枝杆菌属内的不同菌种。Conville等[14]分析比较了诺卡氏菌属的29个种的16S rRNA以及SecA1基因序列,发现SecA1基因能更好更准确地区分和鉴别诺卡氏菌属内的菌种。本课题组在前期研究中[15]在对戈登氏属的23个模式种分别进行了SecA1基因和16S rRNA系统进化关系的比较发现,SecA1作为看家基因用来进行种水平的鉴定以系统进化的研究,是非常理想的靶标。本研究在分析比较几类主要病原需氧放线菌菌属SecA1基因过程中,发现SecA1基因300~350 bp区域在不同菌属间的基因突变各具其特点。
1材料与方法
1.1菌株来源用于实验分析的菌株共162株,其中实验测序的菌株30株(日本千叶大学真菌医学研究所赠予),包括Nocardia属12株、Gordonia属18株;在NCBI(The National Center for Biotechnology Information)数据库中收集含SecA1基因的不同种属的需氧放线菌132株,分别包括Gordonia菌34株、Mycobacteria菌48株、Nocardia菌35株、Tsukamurella菌7株、Rhodococcus菌7株、Streptomyces菌1株。1株Streptomyces菌为白黄链霉菌分离自澳大利亚红树植物组织[8],为环境中分离的需氧放线菌,作为对照株,其余菌株为病原需氧放线菌。
1.2培养基和试剂高氏一号固体培养基(Difco Laboratories),CTAB(四川金山制药公司),PCR试剂盒(Fermentas公司),Taq-DNA多聚酶(上海英俊)1.3需氧放线菌DNA提取及SecA1基因扩增根据文献[16]的方法提取需氧放线菌DNA,细菌SecA1基因扩增通用引物SecA1-f: 5′-GTAAAACGACGGCCAGGACAGYGAGTGGATGGG YCGSGT GCACCG-3′,SecA1-r: 5′-CAGGAAACAGCTATGACGCGGACGATGTAGTCC TTGTC-3′分别为前后引物(上海英俊生物技术有限公司合成)。以SecA1-f、SecA1-r为引物,用PCR试剂盒在26 μL 反应体系中扩增,上、下游引物及DNA各1 μL,反应条件为预变性94 ℃ 4 min,然后变性94 ℃ 30 s、 退火 58 ℃ 50 s、 延伸72 ℃ 1 min 循环35次,最后72 ℃ 延伸10 min,将产物于1.0% 琼脂糖中电泳后在紫外灯下观察,并将扩增产物进行测序。
1.4SecA1基因序列分析收集NCBI数据库中已发表的不同种属氧放线菌的SecA1基因,分别包括Gordonia菌属、Mycobacteria菌属、Nocardia菌属、Tsukamurella菌属、Rhodococcus菌属、Streptomyces菌属的SecA1基因。
将本研究中扩增的SecA1基因测序序列及NCBI数据库中收集的SecA1基因序列在DNAMAN中进行多序列对比,分析各菌属间特异的碱基基序。
1.5不同菌属间特异性碱基区域对应的氨基酸序列分析将SecA1基因对应的氨基酸序列在DNAMAN中进行多序列对比,分析不同菌属间特异性碱基区域对应的氨基酸序列的变化。
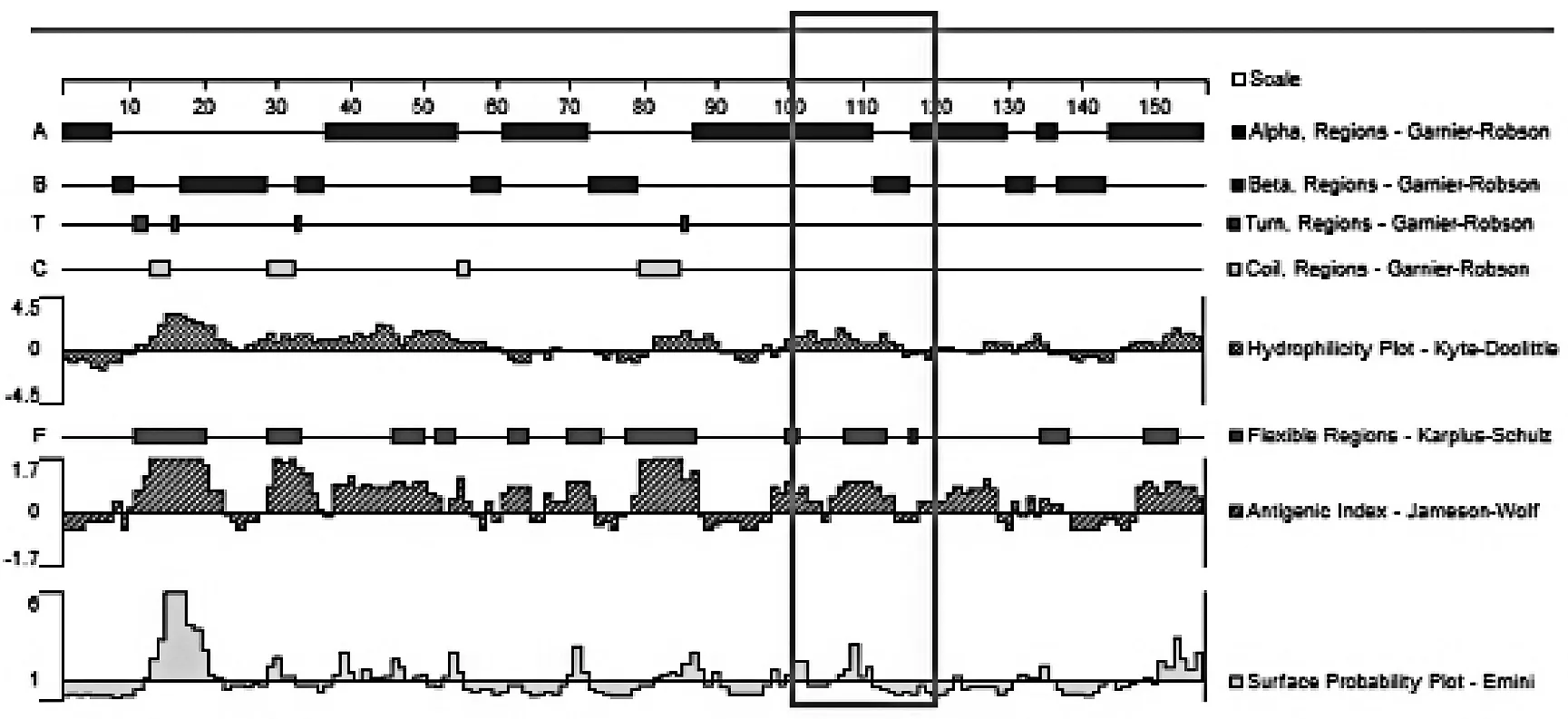
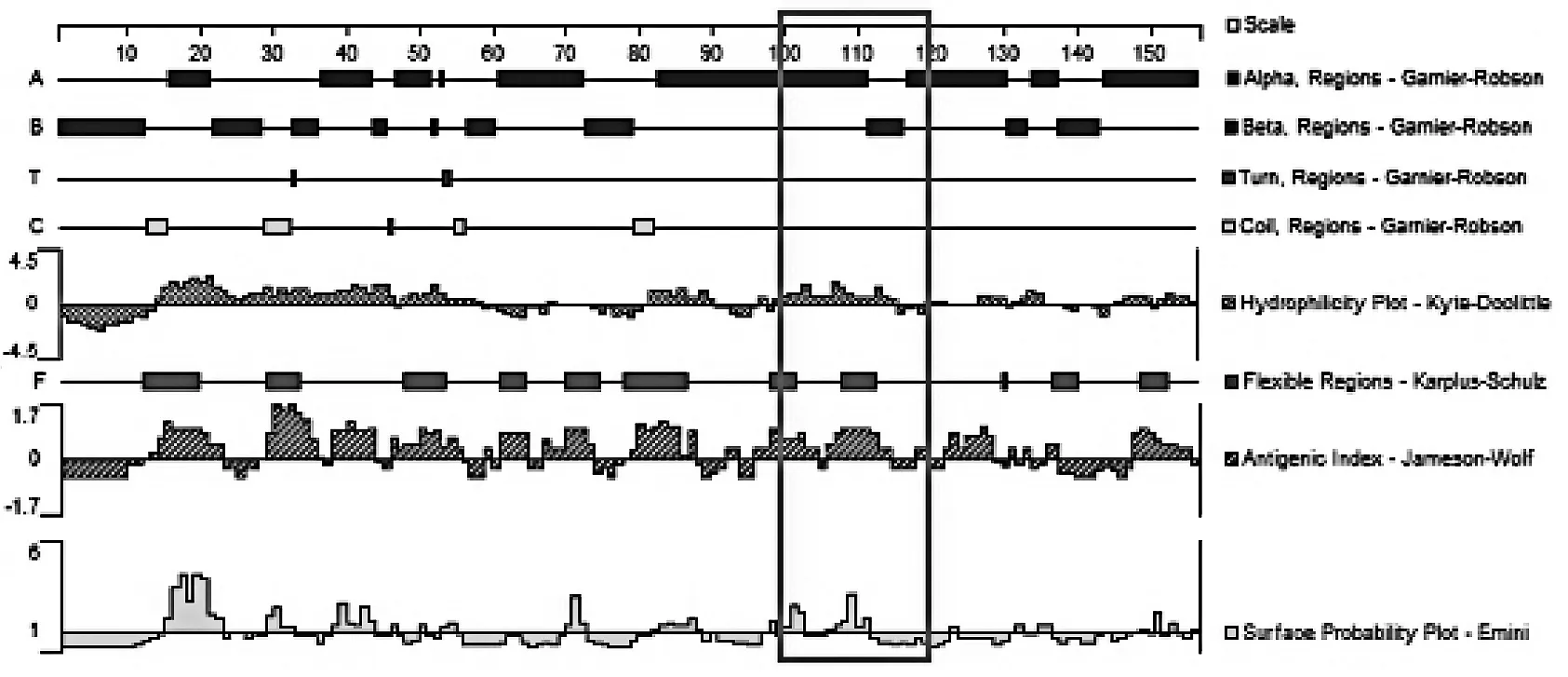
采用DNAStar软件的Protean模块对不同菌属间特定区域的SecA1蛋白进行预测。分别以Garnier-Robson法预测其二级结构,以Karplus-Schulz法预测其柔性区域。以Kyte-Doolittle方法进行亲水性区域分析,以Emini方案进行氨基酸位于分子表面可能性分析,以Jameson-Wolf法进行抗原指数分析。
2结果
2.1不同需氧放线菌菌属间SecA1序列分析经过DNAMAN多序列对比后发现,碱基序列在300~350 bp区域存在碱基的突变,且不同需氧放线菌菌属间有着特异性差异。本研究选择各菌属的标准菌株的SecA1碱基序列作为其菌属代表序列,且各菌属的代表序列与同属的序列有着80%相似率。
几类病原放线菌菌属间在SecA1基因300~350 bp区域,属间特异性碱基区域比对结果见表1,在300~350 bp基因区域,共有24处碱基突变,其中颠换频率为54.2%,转换频率为46.8%。(见表2)
表1不同需氧放线菌菌属SecA1基因特异性碱基位点
Tab.1SecA1 gene specific motifs among different genera of aerobic actinomycetes
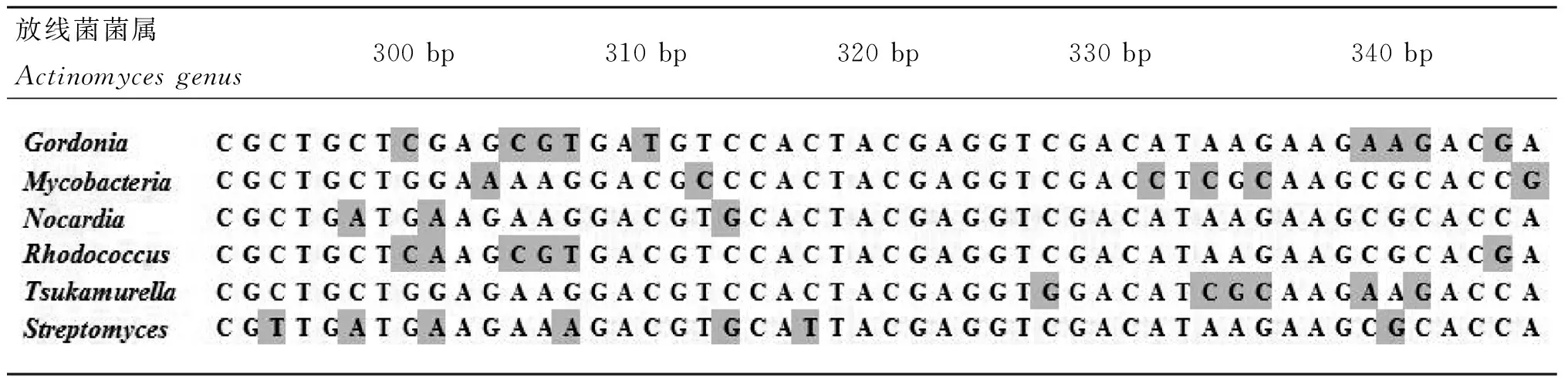
放线菌菌属Actinomycesgenus300bp310bp320bp330bp340bp
表2需氧放线菌菌属的SecA1基因突变情况
Tab.2SecA1 gene specific motifs mutation features among different genera of aerobic actinomycetes
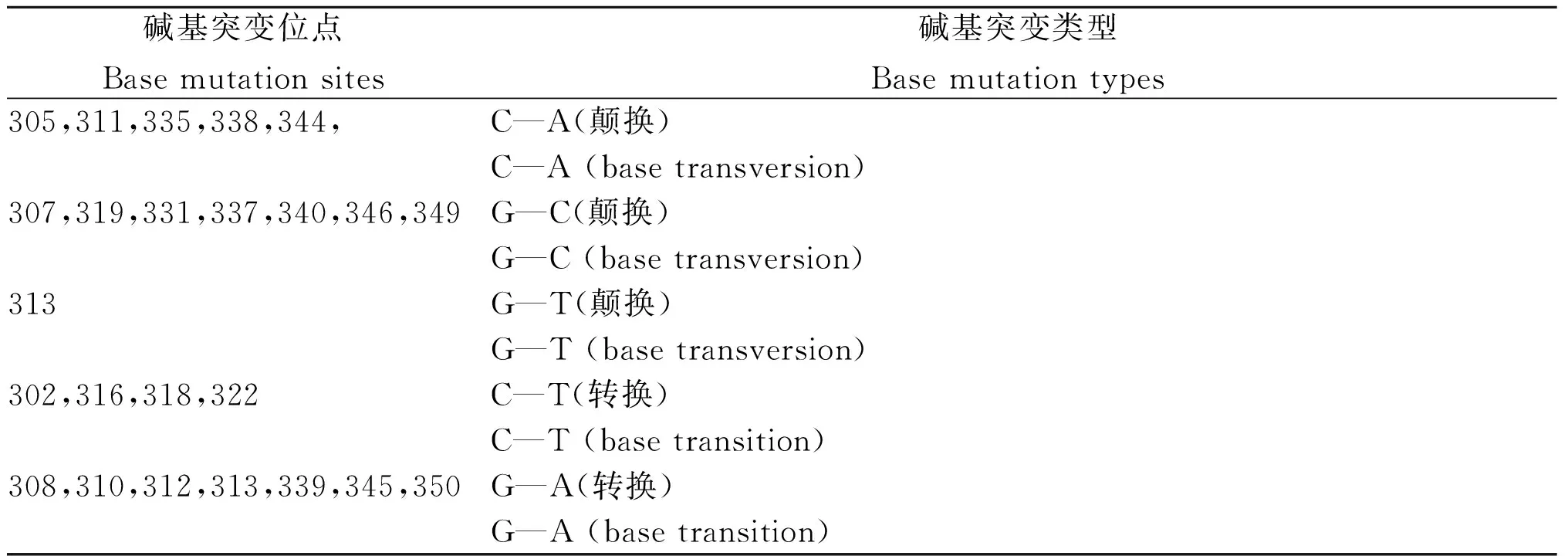
碱基突变位点Basemutationsites碱基突变类型Basemutationtypes305,311,335,338,344,C—A(颠换)C—A(basetransversion)307,319,331,337,340,346,349G—C(颠换)G—C(basetransversion)313G—T(颠换)G—T(basetransversion)302,316,318,322C—T(转换)C—T(basetransition)308,310,312,313,339,345,350G—A(转换)G—A(basetransition)
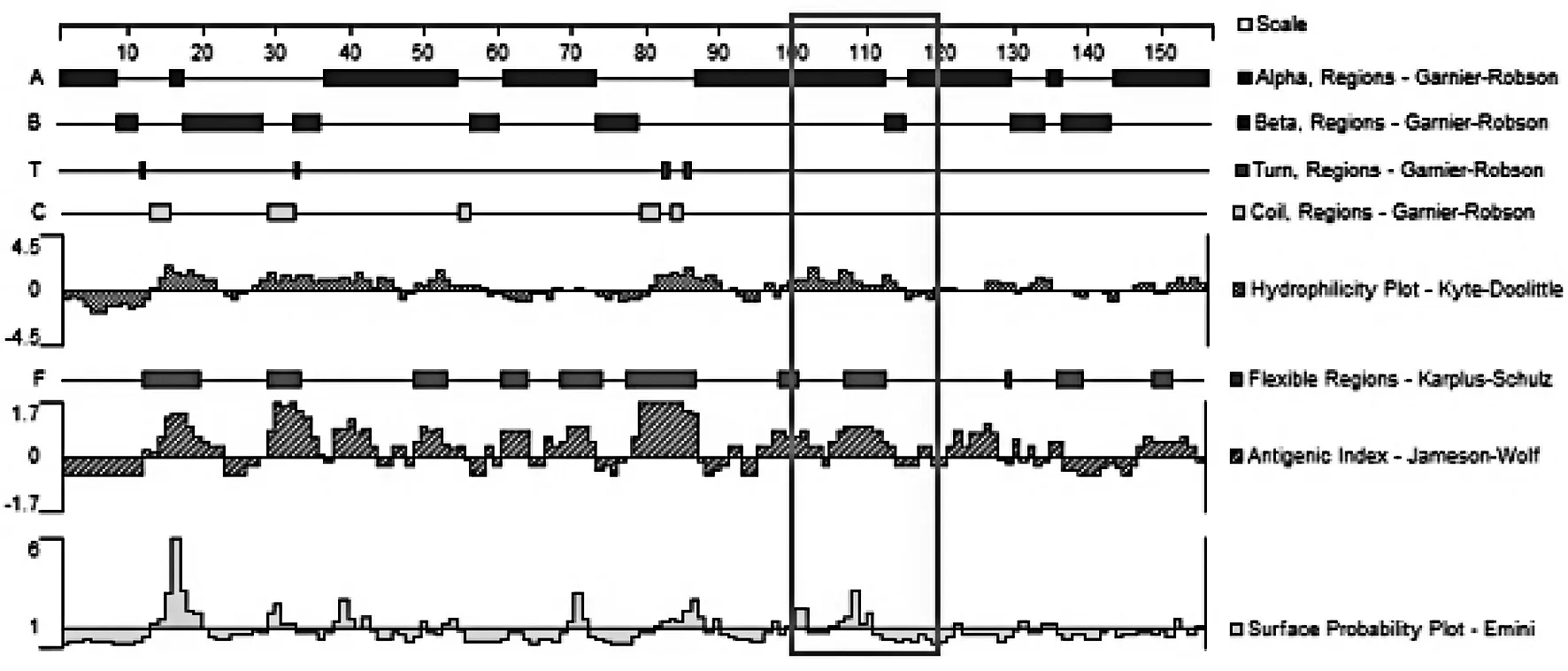
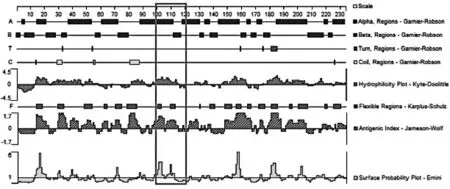
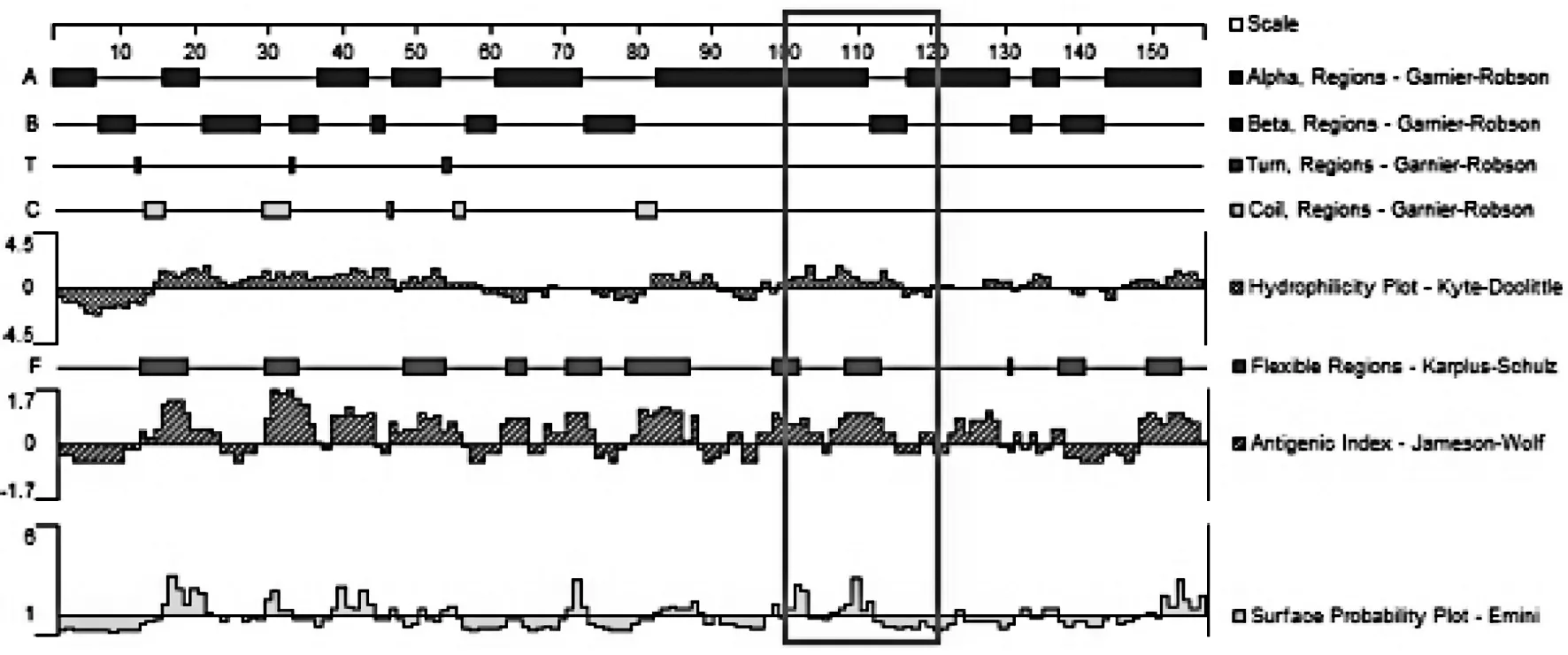
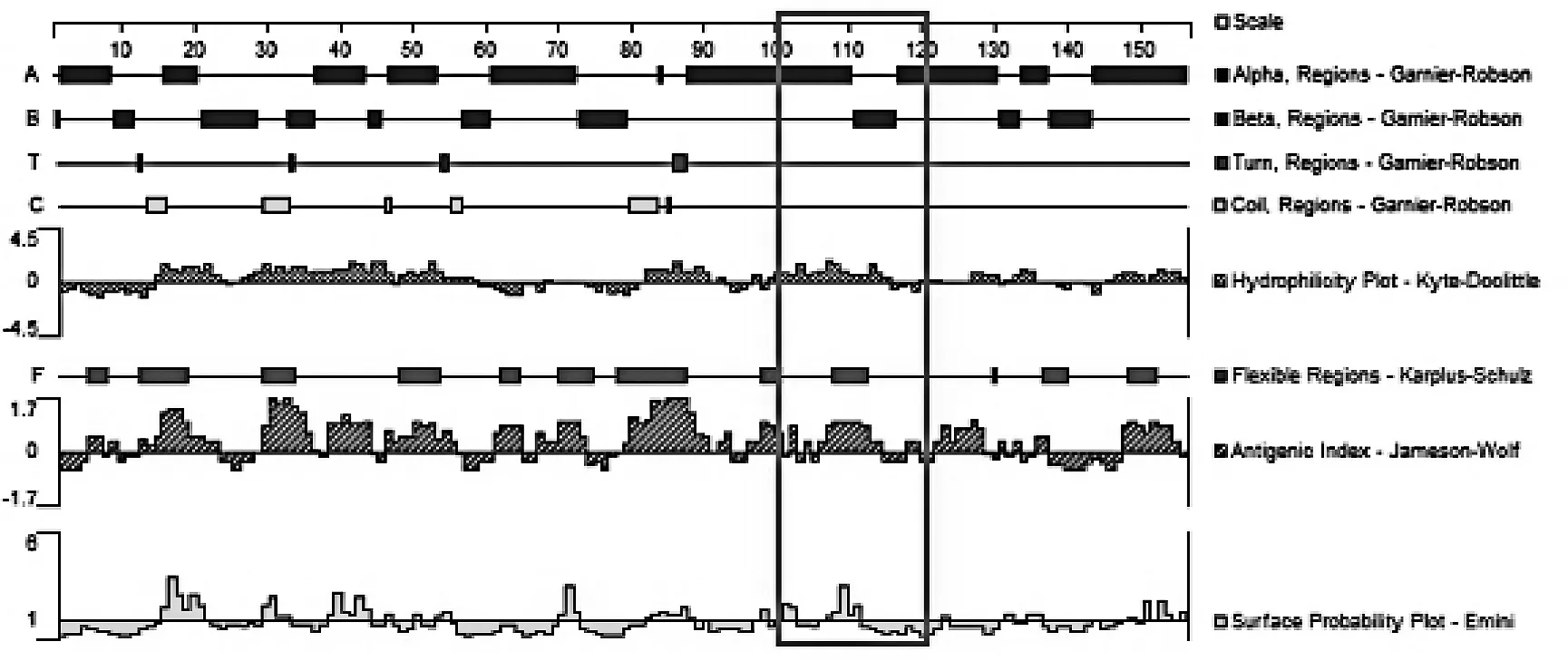
2.2特异性碱基区域对应的氨基酸序列比对病原放线菌菌属间特异性碱基区域对应的氨基酸序列也发生了变化,不同菌属氨基酸序列在100~120 bp共有11处变异,见表3。在100位处,Mycobacteria菌属及Tsukamurella菌属的氨基酸为M(甲硫氨酸),其余菌属为L(亮氨酸);101位:Gordonia菌属、Mycobacteria菌属及Tsukamurella菌属为E(谷氨酸),其余为K(赖氨酸);102位:Gordonia菌属为R(精氨酸)、Rhodococcus菌属为P(脯氨酸)、其余为K(赖氨酸);103位:Rhodococcus菌属为E(谷氨酸),其余为D(天冬氨酸);104位:Mycobacteria菌属为T(苏氨酸)、Nocardia菌属为L(亮氨酸),其余为V(缬氨酸);108位:Rhodococcus菌属为I(异亮氨酸),其余为V(缬氨酸);110位:Mycobacteria菌属为L(亮氨酸),其余为I(异亮氨酸);111位:Gordonia菌属、Nocardia菌属及Streptomyces菌属为K(赖氨酸),其余为R(精氨酸);113位:Gordonia菌属及Tsukamurella菌属为K(赖氨酸),其余为R(精氨酸);115位:Mycobacteria菌属及Rhodococcus菌属为V(缬氨酸),其余为I(异亮氨酸);118位:Tsukamurella菌属为N(天冬酰胺),其余为H(组氨酸)。
表3不同需氧放线菌菌属特异性氨基酸基序
Tab.3Amino acids specific motifs mutation features among different genera of aerobic actinomycetes
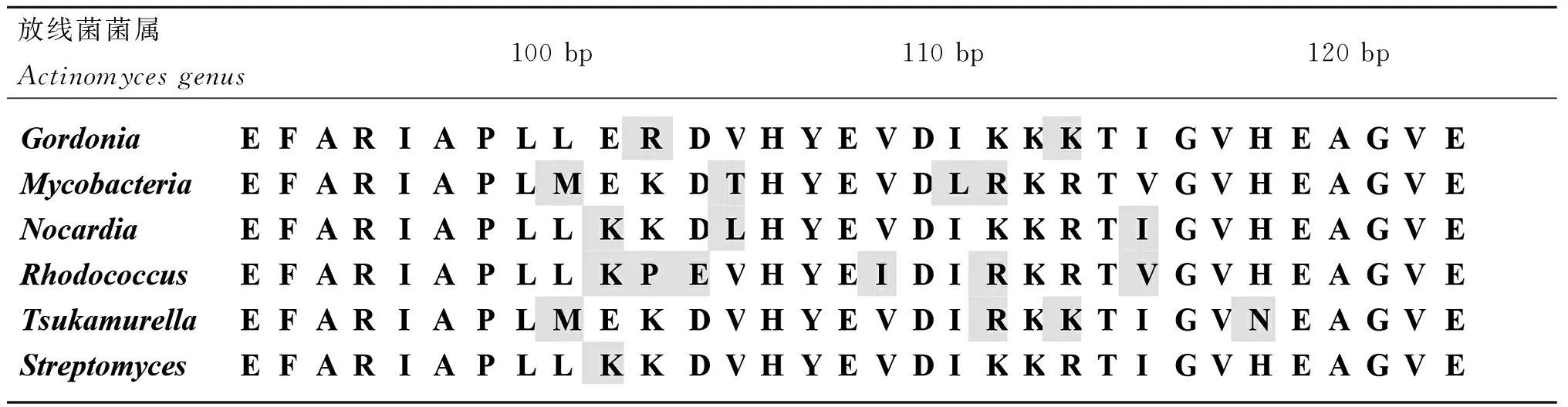
放线菌菌属Actinomycesgenus100bp110bp120bp
(1)Gordonia菌属
(2)Mycobacteria菌属
(3)Nocardia菌属
(4)Rhodococcus菌属
(5)Tsukamurella菌属
(6)Streptomyces菌属图1 氨基酸序列DNAStar分析结果Fig.1 Analysis of the amino acids by DNAStar
3讨论
放线菌广泛分布于自然界中,是微生物中与人的关系非常密切而大有应用前途的一类菌。在我国已有不少科学家对于土壤和极端环境中的放线菌研究比较深入[17-19]。需氧放线菌(aerobic actinomycetes)是一群专性需氧,革兰氏染色阳性,呈丝状分支,生长速度相对较缓慢的一类微生物。在过去它们曾被误认为属于真菌类。广泛存在于世界各地富含腐生物质的土壤中和其它自然环境中。1888年,法国科学家Edmond Nocard首次分离出这类潜在的致病微生物,需氧放线菌能生产新型抗菌制剂,同时也是一类能感染人及动物的潜在病原菌[20],往往通过呼吸了污染的粉尘微粒或表面伤口的侵蚀而引起一些肺部或皮肤等典型感染。大量增加的临床病例证实病原需氧放线菌是导致人类疾病的诱因,尤其导致一些机体严重的免疫系统障碍。如移植患者或艾滋病毒感染患者,且这类微生物感染的抗菌疗法可并发耐药性。与医学相关的重要需氧放线菌如诺卡氏菌属Nocardia是与人类疾病相关的常见放线菌之一。肺部诺卡氏菌感染是在发展中国家中人类诺卡氏菌感染中最为常见的一类疾病,引起肺部急性,亚急性的化脓坏死性感染。其中对人致病的主要是外源性感染星形诺卡氏菌,其次是巴西诺卡氏菌。足菌肿(Mycetoma)和孢子丝菌病(sporotrichoid infection)在热带或亚热带地区(如南非,中非,南美洲,美国南部区域等)比较普遍,其病原菌主要是Nocardiabrasiliensis,也可由其它诺卡氏菌种引起。播散性诺卡氏菌病可由肺部或皮肤向中枢神经系统,骨骼关节,眼部,心肝脾肾胰等部位扩散。红球菌属Rhodococcus是可以引起皮肤感染,肺部感染,菌血症,眼内炎,腹膜炎,前列腺脓肿及静脉置管后败血症的罕见菌属。此外,分支杆菌属Mycobacteria中的结核分支杆菌Mycobacteriumtuberculosis和麻风分支杆菌Mycobacteriumleprae引起的严重病症也众所周知。
在临床上除非是已经怀疑有相应感染可能性,需氧放线菌通常会被漏诊或误诊[20]。对临床病原需氧放线菌分离的了解有助于内科大夫诊断疾病和设计合理正确的用药治疗方案。SecA1等类似的看家基因,在放线菌系统发育分析和分子进化领域里, 发挥了其高分辨率的优势。Sec系统是相当保守的分泌机制,在古菌、细菌及真核生物中都存在。Sec分泌途径在细菌中,尤其是大肠杆菌已得到了广泛的研究[21]。由SecA1基因编码的SecA1蛋白具 ATPase活性,作为前体蛋白转位酶(Protein translocase)的重要成分,为蛋白的跨质膜输送提供驱动力。前体蛋白移位酶被认为是一个高度动态的纳米机器[22],通过它的多聚合体结构可向前移动,位于中心的SecAATPase位移提供能量。
本研究将SecA1基因多序列对比后发现,碱基序列在300~350 bp区域存在碱基的突变,且不同需氧放线菌菌属间有着特异性差异。各菌属间碱基有24处不同,氨基酸有11处不同,这种变异导致其编码的蛋白质在100~120位点间的部分性质在各菌属间并不相同,差异以Mycobacteria菌属较为突出。Streptomyces菌作为环境分离的菌株(对照)与Nocardia属、Gordonia属及Rhodococcus属的蛋白性质大致相同,而Tsukamurella菌属的蛋白质柔性区域增加一个区域,提示该区域容易发生构象变化[23];Mycobacteria菌属的蛋白质亲水性、抗原指数及表面可及性较其余菌属增强,提示该菌属暴露在蛋白质表面的亲水基更容易和其他蛋白质分子结合[24]。Mycobacteria菌属特异的性质可能与该菌属致病性有关。本研究采用DNAStar软件的Protean模块对不同菌属间特定区域的SecA1蛋白进行预测,具体机制还需进一步试验证实。
对于其它一些常见的代表病原需氧放线菌属,如Actinomadurae菌属、Thermoactinomyces菌属、Saccharomonospora相关各属等,尚未对其SecA1基因进行扩增测序分析,GenBank中也未发现储存的相应基因序列信息。因此为了扩大本研究中需氧放线菌属间SecA1基因分析研究,我们后期试验将对这些放线菌属的菌株进行SecA1基因补充扩增测序。本研究对不同菌属需氧放线菌进行SecA1基因多序列分析,分析结果为病原需氧放线菌的临床分类、诊断及致病性提供一定的实验室理论数据。
参考文献:
[1] Walther K, Bruder E, Goldenberger D, et al.Actinomycesneuiiisolated from a 20-month-old girl with cervical lymphadenitis[J]. J Pediatric Infect Dis Soc, 2015, 4(3): e32-37. DOI: 10.1093/jpids/piu096
[2]Henkle E, Winthrop KL. Nontuberculousmycobacteriainfections in immunosuppressed hosts[J]. Clin Chest Med, 2015, 36(1): 91-99. DOI: 10.1016/j.ccm.2014.11.002
[3]Phelippeau M, Delord M, Drancourt M, et al. Respiratory tract isolation ofMycobacteriumeuropaeumfollowing influenza infection in an immunocompromised patient: a case report[J]. J Med Case Rep, 2014, 8: 463. DOI: 10.1186/1752-1947-8-463
[4]Li S, Song XY, Zhao YY, et al. Clinical analysis of pulmonaryNocardiosisin patients with autoimmune disease[J]. Medicine (Baltimore), 2015, 94(39): e1561.DOI: 10.1097/MD.0000000000001561
[5]Tang H, Mao T, Gong Y, et al.Nocardialbrain abscess in an immunocompromised old patient: a case report and review of literature[J]. Int J Clin Exp Med, 2014, 7(5): 1480-1482.
[6]Adjamian N, Kikam A, Wessell KR, et al.Nocardiabrain abscess and CD4+lymphocytopenia in a previously healthy individual[J]. Case Reports Immunol, 2015, 2015: 374956. DOI: 10.1155/2015/374956
[7]Condas LA, Ribeiro MG, Yazawa K, et al. Molecular identification and antimicrobial susceptibility ofNocardiaspp. isolated from bovine mastitis in Brazil[J]. Vet Microbiol, 2013, 167(3/4): 708-712. DOI: 10.1016/j.vetmic
[8]Kogure T, Shimada R, Ishikawa J, et al. Homozygous triplicate mutations in three 16S rRNA genes responsible for high-level aminoglycoside resistance inNocardiafarcinicaclinical isolates from a Canada-wide bovine mastitis epizootic[J]. Antimicrob Agents Chemother, 2010, 54(6): 2385-2390. DOI: 10.1128/AAC.00021-10
[9]Xia L, Zhang H, Lu Y, et al. Development of a loop-mediated isothermal amplification assay for rapid detection ofNocardiasalmonicida, the causative agent of nocardiosis in fish[J]. J Microbiol Biotechnol, 2015, 25(3): 321-327.
[10]Xia L, Cai J, Wang B, et al. Draft genome sequence ofNocardiaseriolaeZJ0503, a fish pathogen isolated fromTrachinotusovatusin China[J]. Genome Announc, 2015, 3(1): e01223-14. DOI: 10.1128/genomeA.01223-14
[11]Wanzala SI, Nakavuma J, Travis DA, et al. Draft genome sequences ofMycobacteriumbovisBZ 31150 andMycobacteriumbovisB2 7505, pthogenic bacteria isolated from archived captive animal bronchial washes and human sputum samples in Uganda[J]. Genome Announc, 2015, 3(5): e01102-15. DOI: 10.1128/genomeA.01102-15
[12]Yang LL, Zhi XY, Li WJ. Phylogenetic trees derived from 16S rRNA,gyrB,sodandrpoBgene sequences ofNocardiopsis[J]. Acta Microbiologica Sinica, 2007, 06: 951-955. (in Chinese)
杨玲玲,职晓阳,李文均. 拟诺卡氏菌16S rRNA,gyrB,sod和rpoB基因的系统发育分析[J]. 微生物学报,2007,06:951-955.
[13] Zelazny AM, Calhoun LB, Li L, et al. Identification ofMycobacteriumspecies bysecA1 sequences[J]. J Clin Microbiol, 2005, 43(3): 1051-1058.
[14] Conville PS, Zelazny AM, Witebsky FG. Analysis ofsecA1 gene sequences for identification ofNocardiaspecies[J]. J Clin Microbiol, 2006, 44(8): 2760-2766.
[15] Kang Y, Takeda K, Yazawa K, et al. Phylogenetic studies ofGordoniaspecies based ongyrBandsecA1 gene analyses[J]. Mycopathologia, 2009, 167 (2): 95-105. DOI: 10.1007/s11046-008-9151-y
[16]Xu LH, Li WJ, Liu ZH, et al. Actinomycete systematics -principles, methods and practices[M]. Beijing: Science Press, 2007: 96-298. (in Chinese)
徐丽华,李文均,刘志恒,等.放线菌系统学—原理、方法及实践[M].北京:科学出版社,2007:96-298.
[17]Kachuei R, Emami M, Mirnejad R, et al. Diversity and frequency ofNocardiaspp. in the soil of Isfahan province, Iran[J]. Asian Pac J Trop Biomed, 2012, 2(6): 474-478. DOI: 10.1016/S2221-1691(12)60079-3
[18]Aghamirian MR, Ghiasian SA. Isolation and characterization of medically important aerobic actinomycetes in soil of iran (2006-2007)[J]. Open Microbiol J, 2009, 3: 53-57. DOI: 10.2174/1874285800903010053
[19]Mohammadipanah F, Hamedi J, Sproer C, et al.Streptomyceszagrosensissp. nov., isolated from soil[J]. Int J Syst Evol Microbiol, 2014, 64(10): 3434-3440. DOI: 10.1099/ijs.0.064527-0
[20]McNeil MM, Brown JM. The medically important aerobic actinomycetes: epidemiology and microbiology[J]. Clin Microbiol Rev, 1994, 7(3): 357-417.
[21]Papanikolau Y, Papadovasilaki M, Ravelli RB, et al. Structure of dimericSecA, theEscherichiacolipreprotein translocase motor[J]. J Mol Biol, 2007, 366(5): 1545-1557.
[22]Vrontou E, Economou A. Structure and function ofSecA, the preprotein translocase nanomotor[J]. Biochim Biophys Acta, 2004, 1694(1/3): 67-80.
[23]Radivojac P, Obradovic Z, Smith DK, et al. Protein flexibility and intrinsic disorder[J]. Protein Sci, 2004, 13(1): 71-80.
[24]Baker EN, Arcus VL, Lott JS. Protein structure prediction and analysis as a tool for functional genomics[J]. Appl Bioinformatics, 2003, 2(3 Suppl): S3-10.
DOI:10.3969/j.issn.1002-2694.2016.04.007
通讯作者:康颖倩,Email: joycekangtokyo@qq.com
中图分类号:R379.1
文献标识码:A
文章编号:1002-2694(2016)04-0349-07
Corresponding author:Kang Ying-qian, Email: joycekangtokyo@qq.com
收稿日期:2015-10-26修回日期:2015-12-25
SecA1 gene sequences for aerobic actinomycetes
MU Li-li1, TU Yun-hua2, MING Chun-yan1,XIA Mao-ning1,MENG Yu-chen1,KANG Ying-qian1
(1.DepartmentofMicrobiology,GuizhouMedicalUniversity,Guiyang550004,China;2.DepartmentofDermatologyandVenerealDisease,theAffiliatedHospitalofGuizhouMedicalUniversity,Guizhou550004,China)
Abstract:We analyzed the SecA1 gene mutation features among different genera of aerobic pathogenic actinomycetes and provided some basic theoretical datum for the clinical diagnosis, classification and pathogenicity of aerobic actinomycetes. One hundred and sixty-two aerobic actinomycetes strains were used in this study. The SecA1 gene sequences of aerobic actinomycetes were amplified by the laboratory or collected in the NCBI database. The gene specific motifs and the SecA1 protein secondary structure were analyzed by multiple sequence alignment and DNAStar. Results showed that each genus had a specific area in SecA1 gene (300~350 bp). The hydrophilicity plot and surface probability of the SecA1 protein properties were various among different genera, and those above characteristics of Mycobacteria were obviously different from other studied genera. In conclusion, the specific SecA1 gene nucleotide mutations exist in each pathogenic actinobacterial genus, which can cause the differences of SecA1 protein properties, which may correlated with the pathogenic characteristics of relevant genera.
Keywords:aerobic actinomycetes; SecA1 gene; nucleotide mutation; SecA1 protein
国家自然基金地方项目(No.31060006,No.31260029),贵州省社会发展科技攻关项目([2011]3017号)和贵阳市科技局社会发展与民生计划([2011103]16号)联合资助

