巴西橡胶树葡萄糖/核糖醇脱氢酶基因的克隆和表达分析
朱晋恒 戚继艳 肖小虎 唐朝荣

摘 要 割胶显著刺激新开割橡胶树胶乳产量,前期的比较蛋白质组学的研究中,已通过双向电泳的方法分离到一个随割胶下调的葡萄糖/核糖醇脱氢酶(GRDH,glucose/ribitol dehydrogenase)。通过搜索本实验室的橡胶树EST数据库和PCR扩增得到该蛋白点对应的全长cDNA序列并命名为HbGRDH1。序列分析显示,cDNA的编码序列(CDS)為882 bp,推测编码294个aa,分子量大小为32.2 ku,等电点为5.18,基因组序列包含4个外显子和3个内含子。通过Realtime PCR和Solexa高通量转录组测序分析发现,HbGRDH1主要在胶乳和雌花中表达,在叶片发育稳定期呈现明显的下调表达趋势;在新开割树胶乳中,前4刀该基因表达变化不明显,而第5刀开始呈现上调表达趋势;在胶乳中,乙烯利处理对该基因表达前12 h无明显影响,在24 h时呈明显的下调表达。本研究结果有助于进一步阐明橡胶树中HbGRDH1基因的功能,特别是其参与橡胶树胶乳再生调控的分子机理。
关键词 巴西橡胶树;葡萄糖和核糖醇脱氢酶;表达分析;割胶;乙烯利;叶片发育
中图分类号 S794.1 文献标识码 A
Abstract Tapping has a significant impact on latex production of rubber trees. In a previous study,two-dimensional proteomics analysis allowed us to isolate a tapping-depressed protein spot in the c-serum of latex,which was identified by tandem mass-spectrometry as a glucose/ribitol dehydrogenase(GRDH). By searching the H. brasiliensis EST database in our laboratory and PCR amplification,we obtained the full-length cDNA sequence corresponding to the GRDH protein spot,and named as HbGRDH1. Sequence analysis revealed that the length of coding domain sequence(CDS)was 882 bp that encoded a protein of 294 amino acids with a molecular weight of 32.2 ku and an isoelectric point of 5.18. The genomic sequence of HbGRDH1 contains four exons and three introns. By fluorescence quantitative RT-PCR and Solexa-sequencing transcriptomes of H. brasiliensis,expressional analyses were conducted. HbGRDH1 was mainly expressed in latex and female flowers,and showed an expression decrease in the fourth process of leaf development. The expression of HbGRDH1 in latex was up-regulated by tapping of virgin rubber trees although not significant for the first four tappings. Ethephon treatment affected little on HbGRDH1 expression in the first 12 hours but decreased obviously later. The results of this study will be beneficial to further studies on the functions of HbGRDH1 in the regulation of latex regeneration.
Key words Hevea brasiliensis; Glucose/ribitol dehydrogenase; Expression; Tapping; Ethephon; Leaf development
doi 10.3969/j.issn.1000-2561.2016.06.011
天然橡胶(Hevea brasiliensis)是一种重要的工业原料,而巴西橡胶树是天然橡胶的主要商业来源[1]。长期以来中国天然橡胶的生产远远不能满足消费的需求,随着对天然橡胶需求的日益增长,如何进一步提高天然橡胶产量已成为中国天然橡胶行业亟待解决的重大课题。
双向电泳技术已被广泛运用于植物蛋白质组学研究[2-3]。本实验室利用双向蛋白质凝胶电泳系统,对未开割橡胶树开割前5刀胶乳c-乳清蛋白质组进行比较分析时,分离得到一个在割胶过程中表达水平发生明显变化的蛋白点。通过二级质谱分析,得到该蛋白的部分氨基酸序列,并注释为葡萄糖/核糖醇脱氢酶(GRDH,glucose/ribitol dehydrogenase)。通过搜索本实验室的胶乳EST数据库,获得了目标基因的拼接序列,并对该基因进行了全长cDNA克隆并命名为HbGRDH1。目前,很少有文献报道GRDH基因,仅王启磊等[4]报道了玉米的葡萄糖和核糖醇脱氢酶基因ZmGRDH的克隆和表达分析,功能预测显示其编码的蛋白具有一个标准的激活位点及一个甘氨酸富集区NAD结合位点,该蛋白家族主要参与氧化还原反应,碳水化合物代谢等过程。割胶是天然橡胶生产的主要方式,对橡胶树而言也是一种机械伤害,随着割胶大量细胞质流出体外,需要消耗大量的原料来进行胶乳的再生[5]。在未开割橡胶树开割过程中,割胶可以在一定程度上提高胶乳产量,有报道割胶能促进内源乙烯的释放[6-7]。乙烯利作为人工合成的能够释放乙烯的植物生长调节剂已被广泛应用于橡胶树割胶生产,可显著提高胶乳产量[8]。乙烯利刺激能够诱导乳管中的一些生化反应,例如蔗糖装载、水分吸收和一些保护性蛋白的合成,这涉及到很多乙烯应答基因。无论是割胶产生的内源乙烯还是乙烯利处理后释放的外源乙烯,都能诱导乳管产生相应的生化反应[9]。为初步确定HbGRDH1基因的功能,本研究利用Solexa测序和Realtime PCR分析了割胶和乙烯利刺激对其表达的影响及其在不同组织和叶片不同发育时期中的表达模式,首次克隆了橡胶树葡萄糖/核糖醇脱氢酶基因,研究了该基因的表达模式,以期进一步揭示该类基因在橡胶树胶乳再生调控中的生理功能。
1 材料与方法
1.1 材料
1.1.1 植物材料 不同组织中的胶乳、树皮、树叶、种子、雌花和雄花材料来自中国热带农业科学院试验场三队种植10 a、割胶3 a的热研7-33-97橡胶树;由于橡胶树一般通过芽接的方式进行繁殖,为保证不同组织材料的遗传一致性,根取自于热研7-33-97橡胶树组培苗。不同发育时期的叶片采自中国热带农业科学院橡胶所种质资源圃,为1年生的热研7-33-97芽接苗。乙烯利处理选用上述不同组织采集所用的橡胶树,割胶处理采用中国热带农业科学院试验场二队种植8 a、达开割标准的热研7-33-97橡胶树。
1.1.2 菌株与试剂 Escherichia coli JM109菌株由本实验室保存。植物总RNA提取试剂盒购自Biotech公司;逆转录试剂盒购自Fermentas公司;克隆载体pMDR18-T vector和Realtime PCR荧光染料试剂SYBRⅡ premix购自TaKaRa公司;HiFi DNA polymerase购自TransGen公司;琼脂糖凝胶回收试剂盒购自Omega公司;引物合成和测序由华大基因生物技术有限公司完成;其他生化试剂和常规试剂均为进口或国产分析纯试剂。
1.2 方法
1.2.1 材料处理 乙烯利处理未开割树3、12、24 h,以0 h为对照。乙烯利处理和割胶具体方法参照文献[7]进行。
1.2.2 核酸的提取与cDNA合成 橡胶树胶乳总RNA的提取参照Tang等[10]方法进行,其他不同组织RNA的提取方法参照Kiefer[11];cDNA第一条链合成采用Fermentas公司逆转录试剂盒,操作步骤按照说明书进行。
1.2.3 巴西橡胶树GRDH基因的克隆 利用蛋白点二级质谱分析所得到肽段对本实验室EST数据库进行搜索,得到一个与目标肽段完全吻合的EST拼接序列并设计全长引物,对含完整读码框(Open Reading Frame, ORF)的cDNA进行PCR扩增。反应体系如下:稀释10×的cDNA第一链模板1 μL、10×PCR Buffer 5 μL、HiFi DNA Polymerase (5 U/L)1 μL、dNTPs(2.5 mmol/L)4 μL、上下游引物各1 μL、ddH2O 37 μL,共50 μL体系。反应程序:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸2 min,共35个循环;72 ℃延伸10 min。PCR扩增程序结束后,配制1.0%琼脂糖凝胶跑电泳检测并切取目的片段,使用琼脂糖凝胶回收试剂盒回收目的片段后连接pMD18-T载体,转化大肠杆菌感受态细胞并挑取阳性克隆送往公司测序。
1.2.4 生物信息学分析 利用生物软件Genetyx(version 5.0.4)和DNAMAN对全长cDNA序列进行比对分析以及阅读框ORF的预测。利用MEGA 4.0软件对来自巴西橡胶树(HbGRDH1),杨树(gi|224123214),水稻(gi|108885236),拟南芥(gi|75309952),苜蓿(gi|357441633),大豆(gi|356505868),蓖麻(gi|255552291),葡萄(gi|147801315),玉米(gi|414590803)在内的10种植物GRDH氨基酸序列进行聚类分析,用Neighbor-Joining分析方法进行分子系统学分析,并进行1 000次bootstrap统计学检验。
1.2.5 基于Solexa测序的表达分析 利用本实验室现有Solexa转录组数据,对HbGRDH1在不同组织、叶片不同发育时期和乙烯利刺激胶乳中的表达模式进行分析:以所克隆HbGRDH1的编码序列(Coding sequence,CDS)作为参考序列,利用程序BWA[12]将各样品fastq格式的EST数据mapping到参考序列上,得到sam文件,然后从服务器将sam文件下载到个人电脑,利用软件Tablet [http://bioinf.scri.ac.uk/tablet/]对sam文件的结果进行统计分析。
1.2.6 实时Realtime PCR分析 利用实时Realtime PCR技术对HbGRDH1基因在割胶处理下的表达模式进行分析并对Solexa测序的结果进行了验证:提取不同刀次胶乳、不同组织、不同发育时期叶片和乙烯利处理不同时间胶乳的RNA进行逆转录合成cDNA,以YLS8作为内参进行Realtime PCR分析,HbGRDH1和内参的引物序列分别是:
全长cDNA扩增引物:HbGRDH1 F:5′-TAG
AATTCAGAATTTGAGTCTC-3′;HbGRDH1 R:5′-AATTATTATGCTATAAGAGGAC-3′。
實时Realtime PCR引物:HbGRDH1 F:5′-GTGAATAATGCCGCCGAACA-3′;HbGRDH1 R:5′-GTCCAGCAACCCTGAATAGCC-3′;YLS8 F:5′-CCTCGTCGTCATCCGATTC-3′;YLS8 R:5′-CAG
GCACCTCAGTGATGTC-3′。
标准曲线的制作以及对该基因在样品的表达分析主要参照Li等[13]方法进行。具体反应体系为:cDNA模板1 μL、上下游引物混合物0.6 μL、2×SYBRII Premix 10 μL、ddH2O 7.8 μL,总体系20 μL;反应程序为94 ℃预变性5 min,94 ℃变性5 s,68 ℃退火及延伸10 s,共40个循环。
2 结果与分析
2.1 HbGRDH1基因的克隆与序列分析
通过对未开割树胶乳c乳清蛋白质组进行比较分析,分离得到一个随着割胶刀次蛋白表达水平明显变化的蛋白点(图1)。再通过二级质谱分析得到对应蛋白点的肽段氨基酸序列,然后利用该肽段序列对本实验室橡胶树EST数据库进行检索,得到与之匹配的EST序列,通过对所得到的EST进行拼接,得到一个注释为GRDH的拼接序列(contig)。通过基因克隆得到了包含完整ORF的该基因全长cDNA序列,命名为HbGRDH1,并提交GenBank获得序列登录号KU323797。序列分析显示,该基因的编码序列(CDS)为882 bp,推测编码294个氨基酸(图2),分子量大小为32.2 ku,等电点为5.18。用HbGRDH1的cDNA序列blast NCBI橡胶树基因组数据库,搜索并下载到HbGRDH1的基因组序列(gi|447939060|)。利用在线软件GSDS2.0分析HbGRDH1基因组序列结构,结果显示该基因包含4个外显子和3个内含子(图3)。
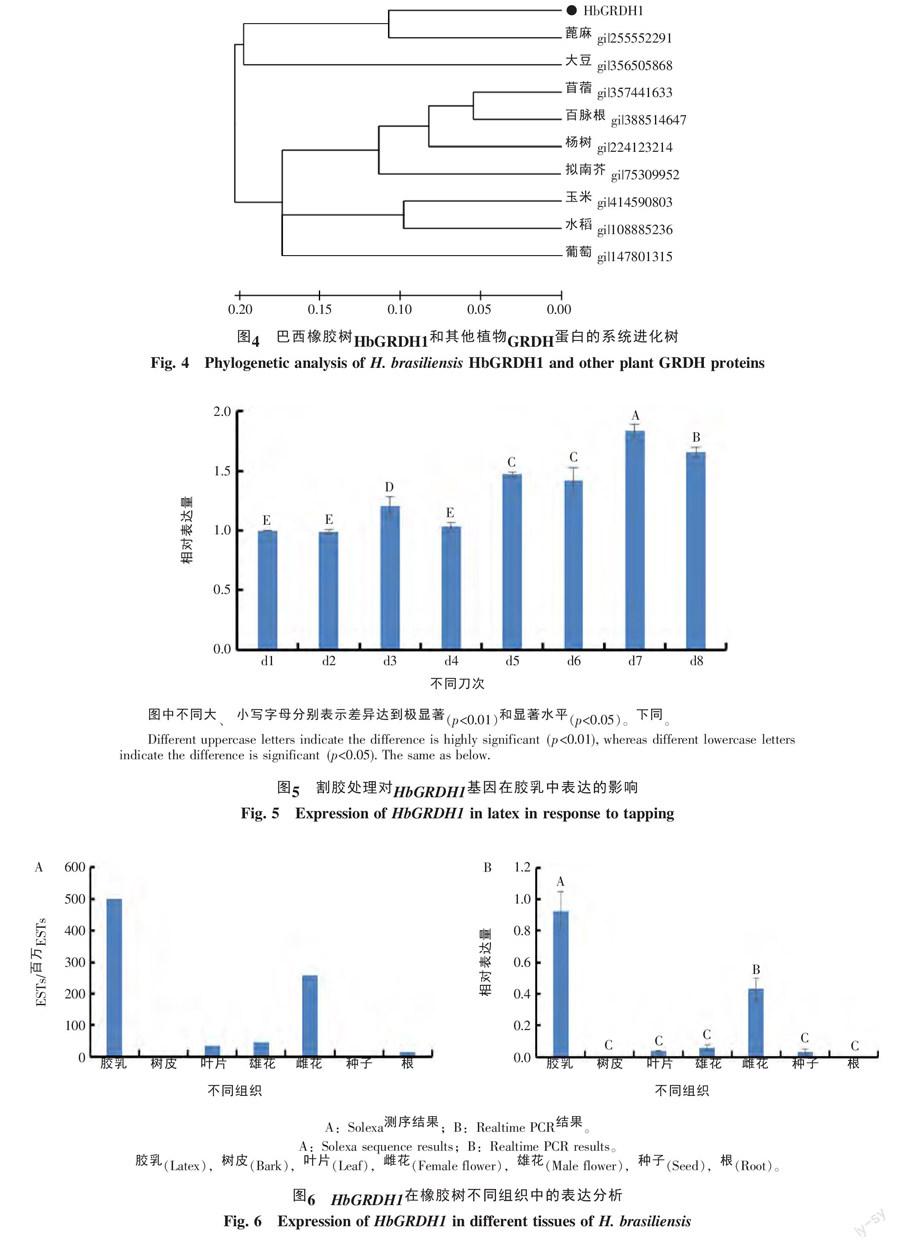
2.2 HbGRDH1蛋白的系统进化分析
利用MEGA 4.0软件对HbGRDH1与其他9种植物的GRDH蛋白进行多序列比对,并构建系统进化树(图4)。所选10种植物的GRDH蛋白可以明显分为2个大的类群,其中橡胶树、蓖麻和大豆的GRDH聚在一起,其它植物的聚在一起,另外橡胶树和蓖麻的GRDH蛋白共同聚在一个小的分支上,这和2种植物同为大戟科的分类一致。
2.3 HbGRDH1在割胶处理下的表达模式分析
在未开割树开割前5刀中HbGRDH1蛋白的表达水平有明显的下降趋势(图5),为了探明HbGRDH1在mRNA水平的表达是否也具有同样的趋势,本研究进一步利用Realtime PCR技术分析了割胶对HbGRDH1基因表达的影响。结果表明,在未开割树开割的前8刀中HbGRDH1的表达呈现上升趋势,表明HbGRDH1在mRNA和蛋白的表达模式不同,推测HbGRDH1基因的表达存在转录后调控模式。
2.4 HbGRDH1的组织特异性表达模式分析
利用Solexa转录组数据对HbGRDH1基因在不同组织中的表达特性进行了分析,并用Realtime PCR技术进行了验证,两者结果一致(p<0.01)。分析结果显示(图6),HbGRDH1在所检测的7种组织中均有表达,其中在胶乳中的表达丰度最高,雌花次之,而在树皮和根中基本不表达。
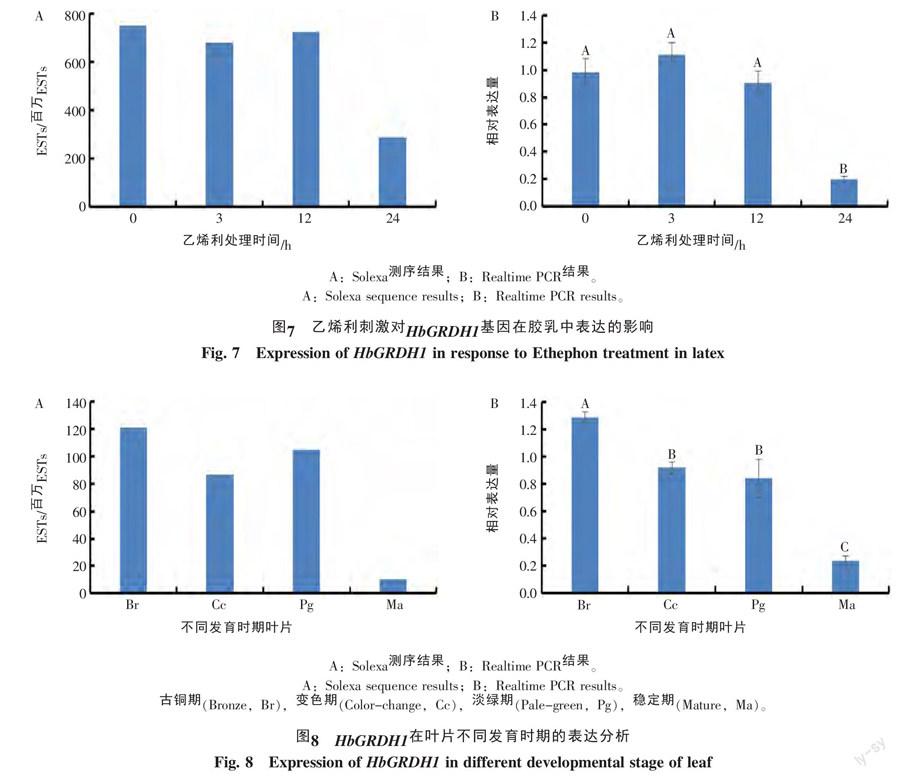
2.5 HbGRDH1在乙烯利处理下的表达模式分析
割胶可以促进胶乳的再生提高胶乳的产量,乙烯利处理同样可以明显提高橡胶树胶乳的产量,为此进一步利用Solexa转录组数据分析了乙烯利刺激对HbGRDH1表达的影响,并用Realtime PCR技术进行了验证,两者结果一致(p<0.05)。结果显示,乙烯利处理对HbGRDH1基因的表达影响较小(图7),仅在处理24 h时HbGRDH1表达量有明显的下降趋势。
2.6 HbGRDH1在叶片发育过程中的表达模式分析
橡胶树叶片是橡胶生物合成原料蔗糖合成的场所,为了研究HbGRDH1基因表达是否参与橡胶树叶片发育,本研究利用Solexa转录组数据对HbGRDH1在叶片4个发育阶段(古铜、变色、淡绿和稳定)中的表达模式进行了分析(图7),并用Realtime PCR技术进行了验证,两者结果一致(p<0.05)。结果表明,在叶片发育的稳定期,HbGRDH1呈明显下调表达。
3 讨论
GRDH是一种以NAD+或NADP+为受体作用于供体CH-OH基团的氧化还原酶,主要参与葡萄糖和核糖醇的代谢[14]。本研究通过对未开割橡胶树开割前5刀胶乳c-乳清的蛋白质组进行比较分析,发现一个随着割胶表达水平明显降低、二级质谱鉴定为GRDH的蛋白点。通过本地EST数据库搜索以及PCR扩增得到该蛋白点对应的全长cDNA,命名为HbGRDH1。通过对来自不同植物的GRDH蛋白序列进行多序列比对及进化分析发现,所选10种植物的GRDH氨基酸序列主要聚为两类,其中橡胶树、蓖麻和大豆聚在一起,且橡胶树和蓖麻的距离最近,这和2种植物同属大戟科植物是一致的。Realtime PCR技术分析显示,在未开割树中HbGRDH1基因的表达随割次增加呈现上调表达的模式,与蛋白质组分析的结果不一致。造成这种结果的原因可能有2个,其一可能是该基因的表达存在明显的转录后调控机制,其二是所克隆的基因不是蛋白质组研究中那个GRDH蛋白点所对应的基因,而是具较高同源性的另一个家族成员。虽然HbGRDH1基因在所检测的7种橡胶组织中都有表达,但表达丰度存在显著差异,在胶乳中表达量最高,雌花次之,树皮和根中基本不表达。在橡胶树叶片发育过程中,随叶片发育进程HbGRDH1呈下调表达。在里氏木霉菌[15]中克隆grd1(glucose-ribitol dehydrogenase 1),并发现该基因产物是纤维素酶基因表达的增强子,能够调控纤维素酶的表达,而纤维素是细胞壁构成和再生的重要组分。橡胶树叶片发育从古铜期到淡绿期,叶片表面积在不断增大,细胞体积增大,细胞壁的一直处于延伸状态,虽然HbGRDH1的表达量在变色期有所下降,但仍然维持较高的表达丰度,而在淡绿期以后,叶片表面积基本停止增長,细胞壁停止延伸,可能不再需要过多的GRDH蛋白调控细胞壁的延伸,所以在稳定期HbGRDH1表达量有显著的下降,推测在橡胶叶片发育过程中HbGRDH1可能参与叶片发育调控。乙烯利虽然能够增加胶乳产量,但是绝大多数与橡胶生物合成相关基因的表达却被其抑制或者无明显变化[16-18],可能与其能延长损伤乳管的闭合时间而增加胶乳排出量,进而促进橡胶的合成代谢有关[19-20]。王旭初等[21]的最新研究指出乙烯利刺激调控胶乳产量不仅表现在基因水平也表现在蛋白水平上,其中转录后修饰在控制橡胶生物合成相关酶的功能上可能发挥重要作用。在乙烯利刺激下HbGRDH1的表达在12 h内没有明显变化,在24 h时却有明显的下调,在胶乳中的表达受到了抑制,但考虑到该基因的表达调控可能存在明显的转录后调控机制,以及该类基因参与纤维素合成调控的事实,因而目前尚不能完全排除该基因参与乙烯利刺激的胶乳再生调控。
参考文献
[1] D′auzac J, Jacob J, Prévot J, et al. The regulation of cis-polyisoprene production(natural rubber)from Hevea brasiliensis[J]. Recent Research Developments in Plant Physiology, 1997, 1: 273-332.
[2] Wade L J, Ghareyazie B, Bennett J. Proteomic analysis of rice leaves during drought stress and recovery[J]. Proteomics, 2002, 2: 1 131-1 145.
[3] Hajheidari M, Eivazi A, Buchanan B B, et al. Proteomics uncovers a role for redox in drought tolerance in wheat[J]. Journal of proteome research, 2007, 6(4): 1 451-1 460.
[4] 王啟磊, 董永彬, 薛晓静. 玉米葡萄糖和核糖醇基因ZmGRDH的克隆及表达分析[C]. 2012年全国玉米遗传育种学术研讨会暨新品种展示观摩会论文及摘要集, 2012.
[5] Pirrello J, Leclercq J, Dessailly F, et al. Transcriptional and post-transcriptional regulation of the jasmonate signalling pathway in response to abiotic and harvesting stress in Hevea brasiliensis[J]. BMC Plant Biology, 2014, 14(1): 341.
[6] Pakianathan S, Tata S, Chon L F, et al. Certain aspects of physiology and biochemistry of latex production[J]. Developments in Crop Science, 1992, 23: 298-323.
[7] Tang C R, Huang D B, Yang J H, et al. The sucrose transporter HbSUT3 plays an active role in sucrose loading to laticifer and rubber productivity in exploited trees of Hevea brasiliensis (para rubber tree)[J]. Plant Cell And Environment, 2010, 33(10): 1 708-1 720.
[8] Jetro N N, Simon G M. Effects of 2-chloroethylphosphonic acid formulations as yield stimulants on Hevea brasiliensis[J]. African Journal of Biotechnology, 2007, 6(5): 523-528.
[9] Putranto R A, Duan C, Kuswanhadi T C, et al. Ethylene response factors are controlled by multiple harvesting stresses in Hevea brasiliensis[J]. PloS one, 2015, 10(4): e0123 618.
[10] Tang C R, Qi J Y, Li H P, et al. A convenient and efficient protocol for isolating high-quality RNA from latex of Hevea brasiliensis(para rubber tree)[J]. Journal of Biochemical and Biophysical Methods, 2007, 70(5): 749-754.
[11] Kiefer E, Heller W, Ernst D. A simple and efficient protocol for isolation of functional RNA from plant tissues rich in secondary metabolites[J]. Plant Molecular Biology Reporter, 2000, 18(1): 33-39.
[12] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1 754-1 760.
[13] Li H, Qin Y, Xiao X, et al. Screening of valid reference genes for real-time RT-PCR data normalization in Hevea brasiliensis and expression validation of a sucrose transporter gene HbSUT3[J]. Plant Sci, 2011, 181(2): 132-139.
[14] Jany K D, Ulmer W, Froschle M, et al. Complete amino acid sequence of glucose dehydrogenase from Bacillus megaterium[J]. FEBS Letters, 1984, 165(1): 6-10.
[15] Schuster A, Kubicek C P, Schmoll M. Dehydrogenase GRD1 represents a novel component of the cellulase regulon in Trichoderma reesei(Hypocrea jecorina)[J]. Applied and Environmental Microbiology, 2011, 77(13): 4 553-4 563.
[16] Yeang H, Arif S a M, Yusof F, et al. Allergenic proteins of natural rubber latex[J]. Methods, 2002, 27(1): 32-45.
[17] Zhu J, Zhang Z. Ethylene stimulation of latex production in Hevea brasiliensis[J]. Plant Signaling & Behavior, 2009, 4(11): 1 072-1 074.
[18] Adiwilaga K, Kush A. Cloning and characterization of cDNA encoding farnesyl diphosphate synthase from rubber tree(Hevea brasiliensis)[J]. Plant Molecular Biology, 1996, 30(5): 935-946.
[19] Yip E, Chin H. Latex flow studies. X. Distribution of metallic ions between phases of Hevea latex and the effects of yield stimulation on this distribution[J]. Journal of the Rubber Research Institute of Malaysia, 1977, 25(1): 31-49.
[20] Tungngoen K, Kongsawadworakul P, Viboonjun U, et al. Involvement of HbPIP2; 1 and HbTIP1; 1 aquaporins in ethylene stimulation of latex yield through regulation of water exchanges between inner liber and latex cells in Hevea brasiliensis[J]. Plant Physiology, 2009, 151(2): 843-856.
[21] Wang X, Wang D, Sun Y, et al. Comprehensive proteomics analysis of laticifer latex reveals new insights into ethylene stimulation of natural rubber production[J]. Scientific Reports, 2015, 5: 13 778.

