海带转录组SSR序列特征及其相关基因功能分析
李秋莹, 张 杰, 姚建亭 王秀良 段德麟
(1. 中国科学院海洋研究所, 山东 青岛 266071; 2. 青岛海洋科学与技术国家实验室实验海洋生物学与生物技术实验室, 山东 青岛 266071; 3. 中国科学院大学, 北京 100049)
海带转录组SSR序列特征及其相关基因功能分析
李秋莹1,2,3, 张 杰1,2,3, 姚建亭1,2, 王秀良1,2, 段德麟1,2
(1. 中国科学院海洋研究所, 山东 青岛 266071; 2. 青岛海洋科学与技术国家实验室实验海洋生物学与生物技术实验室, 山东 青岛 266071; 3. 中国科学院大学, 北京 100049)
对海带(Saccharina japonica)转录组测序数据进行分析, 从70 497条Unigenes中共检测到9 237个简单序列重复(SSR)位点, 并对包含SSR序列的Unigenes进行功能注释。海带转录组中SSR的类型十分丰富, 其中单核苷酸和三核苷酸重复SSR的数量最多, 分别占SSR总数的40.9%和39.4%, 其次为四核苷酸、二核苷酸、五核苷酸和六核苷酸重复, 分别占SSR总数的9.8%, 6.1%, 3.1%和0.7%。SSR重复单元类型共有147种, 其重复次数的范围为5~70次。功能注释发现约50%含有SSR的Unigenes获得了注释信息, 并且大多数与已知蛋白有同源性。COG、GO功能分类结果均表明, 大量含有SSR序列的基因与多种生物功能有关, 其中与碳水化合物代谢及参与细胞组成相关的基因数量最多。本研究结果为深入开发功能性SSR标记奠定基础, 也为开展海带分子标记辅助选育提供支持。
微卫星; 转录组; 简单序列重复(SSR); 海带(Saccharina japonica)
海带(Saccharina japonica)是中国重要的养殖海藻之一, 广泛应用于食品、医药、化工等领域。全球海带养殖主要集中在中国、日本和韩国。中国作为海带养殖大国, 年产海带近500万t, 占全球海带产量的86%左右[1]。海带的重要性状均为典型的数量性状[2], 其遗传机制十分复杂。海带重要性状的分子遗传解析有赖于有效的分子标记的开发。近年来, 许多研究应用AFLP、RAPD、SSR等分子标记, 对海带进行种群遗传、数量性状作图等研究[2-6]。
相比于显性标记, 共显性的简单序列重复(Simple sequence repeat, SSR)标记(也称为微卫星标记)具有共显性、多等位, 变异高等优点, 在基因组中分布广泛, 常被应用于群体遗传分析、遗传连锁图谱构建、QTL作图等研究[7]。海带中运用SSR标记进行遗传研究的报道较少。Liu等[8]通过EST数据开发了15个海带EST-SSR并成功应用到QTL作图和海带群体遗传多样性分析中。由于受全基因组序列信息缺乏的限制, 海带中尚未开展高通量SSR标记的开发, 海带中开发的可用SSR标记还十分有限[8-11]。因此, 开发更多可用的SSR标记对海带的遗传研究具有重要意义。以往常用富集文库法和从数据库中已知的EST序列开发SSR, 但这些方法均耗时、费力, 且效率低。高通量测序的不断增加为SSR的开发提供了丰富资源, 如转录组和基因组的测序数据。SSR根据其开发来源的不同可以分为: 基因组SSR (genomic SSR)和转录组SSR(genic SSR)。基因组SSR常比转录组SSR更易呈现出多态性, 而转录组SSR比基因组的SSR更易与表型性状相关联[12]。
本文基于海带转录组数据, 对其SSR的组成和分布特征进行分析, 并对含有SSR序列的Unigenes进行功能注释与分类, 为从海带中开发具有功能的SSR标记并应用于数量性状遗传解析及分子标记辅助选育提供有利资源。
1 材料与方法
1.1 海带转录组中SSR序列的分析
实验数据来源于本实验室分别对黑暗处理和蓝光诱导下海带幼孢子体的转录组测序数据[13]。使用MISA脚本工具(http: //pgrc.ipk-gatersleben.de/misa/)对两个转录组一共得到的70 497条非冗余Unigenes (总长度为37 895 389 bp)进行了SSR序列的筛选。筛选条件为单核苷酸重复次数至少15次, 二核苷酸重复次数至少8次, 三核苷酸重复次数至少6次,四、五、六核苷酸重复次数至少5次, 复合型SSR的中间间隔小于10 bp。利用Primer3 version 4.0(http: // frodo.wi.mit.edu/primer3)对SSR进行引物设计。
1.2 含有SSR序列的Unigene的功能分析
使用Blastall程序分别将含有SSR序列的Unigene比对到Nr蛋白数据库、Swiss-Prot数据库、Kyoto Encyclopedia of Genes and Genomes (KEGG)数据库和Cluster of Orthologous Groups (COG) 数据库(E-value <1e-5)。使用Blast2go 和WEGO软件分别对Unigenes进行Gene ontology (GO) 注释信息分析和功能分类[14-15]。含有单核苷酸序列的Unigenes未进行功能分析。
2 结果
2.1 SSR序列的分布
利用MISA脚本工具在海带转录组70 497条Unigenes中检测到9 237个SSR位点。在检测的Unigenes中包含SSR的Unigenes共7 725条, 其中1 238条包含2个或2个以上的SSR位点。在检测到的SSR位点中, 185个位点以复合形式存在。海带转录组中SSR的平均分布频率为每4.1 kbp一个SSR位点。SSR类型分析显示其类型十分丰富, 且各种类型分布频率不均衡。单核苷酸和三核苷酸重复的SSR类型数量最多、所占比例最大, 分别为3 781个(40.9%)和3 642个(39.4%), 共占SSR总数的80.3%。而二、四、五和六核苷酸重复类型共占SSR总数的19.7%, 其数目和所占的比例分别为568(6.1%)、901(9.8%)、282(3.1%)、63(0.7%)。
一个SSR位点的重复次数是可变的, 这也是SSR呈现多态的主要原因。由于筛选条件的原因, 海带转录组SSR最小重复次数为5, 最大的重复次数为70,重复6次的SSR位点最多, 占总数的20.6%(1 901个) (图1)。以二、四、五和六核苷酸为重复单元的SSR重复次数主要集中在5~14次。长度超过20 bp的SSR序列共3 032个。二、四、五和六核苷酸重复单元的最大重复次数分别为70(GA)、40(ACA)、44(TAGA)、13(AAGCA)和10(CATCAC)。
2.2 SSR重复单元类型
本研究中, 海带转录组SSR重复单元的类型共有147, 其中将互补的序列归为一种类型。拥有重复单元类型最多的是五核苷酸重复(64种, 图2)。二核苷酸最丰富的重复单元类型为AG/CT型(53%), 而CG/GC类型最少, 在二核苷酸重复类型总数中所占比例不足1%(图3)。在三核苷酸重复单元中, AGC/CTG重复所占比例高达46%, 其次为ACC/GGT(14%) (图3)。四核苷酸、五核苷酸和六核苷酸重复单元类型较多, 所占比例较低, 有的类型仅出现1次。除单核苷酸重复外, 在所有重复单元类型中出现频率最多的是AGC/CTG。
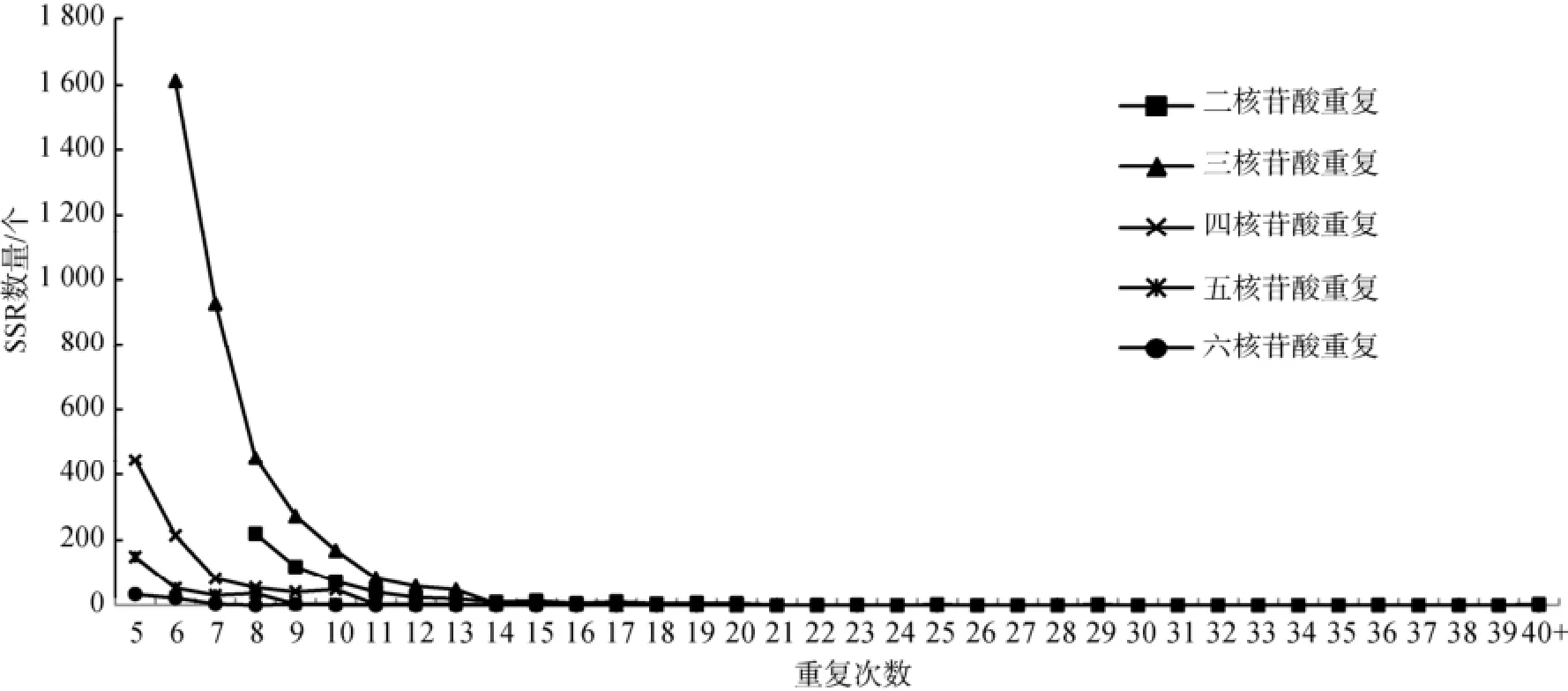
图1 海带转录组不同重复次数的SSR数量变化Fig. 1 Variations in SSR for different repeat times in the Saccharina japonica transcriptome

图2 海带转录组不同核苷酸类型SSR含有的重复单元类型数目Fig. 2 Number of repeat motif types in different SSR types in the Saccharina japonica transcriptome
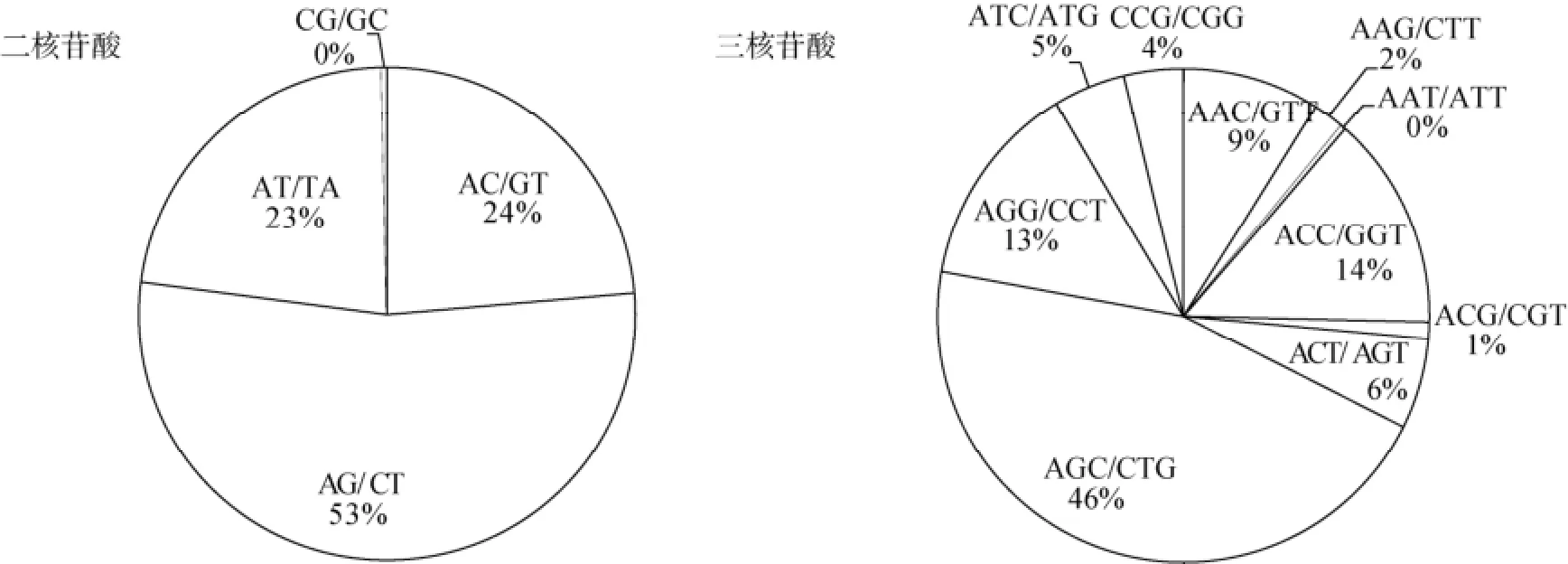
图3 海带转录组中二核苷酸和三核苷酸SSR的不同重复单元的比例Fig. 3 Proportion of different motifs in dinucleotide and thrinucleotide SSRs of the Saccharina japonica transcriptome
2.3 SSR相关Unigene的功能注释与分类
为了解所获得的SSR序列可能的生物学功能,我们对含有SSR序列的Unigene (共4 728条)进行了功能注释分析(单核苷酸除外)。同源比对分析结果表明2 240条(47.4%), 1 835条(38.8%), 1 384条(29.3%)和1 072条(22.7%)Unigenes分别与Nr、Swissport、KEGG和COG数据库中的已知序列具有高度同源性,其中2 842个SSR位于的2 366条(50.0%)Unigenes与至少一个数据库的蛋白序列具有高度同源性。大部分Unigenes与已知蛋白具有同源性, 464条与假定蛋白或未知蛋白具有较高同源性。
通过COG功能分类对这些注释的Unigenes进行功能归类。3 967条Unigene共归为24类(A: RNA加工和修饰; B: 染色质结构和动态; C: 能量生产和转换; D: 细胞周期调控, 细胞分裂, 染色体分裂; E: 氨基酸运输和代谢; F: 核苷酸运输和代谢; G: 碳水化合物运输和代谢; H: 辅酶运输和代谢; I: 脂质运输和代谢; J: 翻译, 核糖体结构和生物合成; K: 转录; L: 复制, 重组和修复; M: 细胞壁/膜/胞外被膜; N: 细胞运动; O: 翻译后修饰, 蛋白质折叠, 分子伴侣; P: 无机盐运输和代谢; Q: 次生代谢物的生物合成, 运输和分解代谢; R: 一般功能预测; S: 未知功能; T: 信号转导机制; U: 细胞内运输, 分泌和膜泡运输; V: 防御机制; W: 细胞外结构; Z: 细胞骨架)(图4), 其中“一般功能预测”是最大一类(占COG注释的Unigene总数的12.0%),其次是“碳水化合物运输和代谢”(9.3%)和“细胞壁/膜/胞外被膜”(9.2%); “细胞外结构”(0.2%)是最小的一类(图4)。可以看出含有SSR序列的这些基因功能丰富多样。GO分类分析将3 366个Unigenes分到3大类(生物学过程, 细胞组成, 分子功能)的31个功能类群中(图5)。其中, “细胞”、“细胞组分”、 “催化活性”以及 “代谢过程”是最大的几个功能类群。
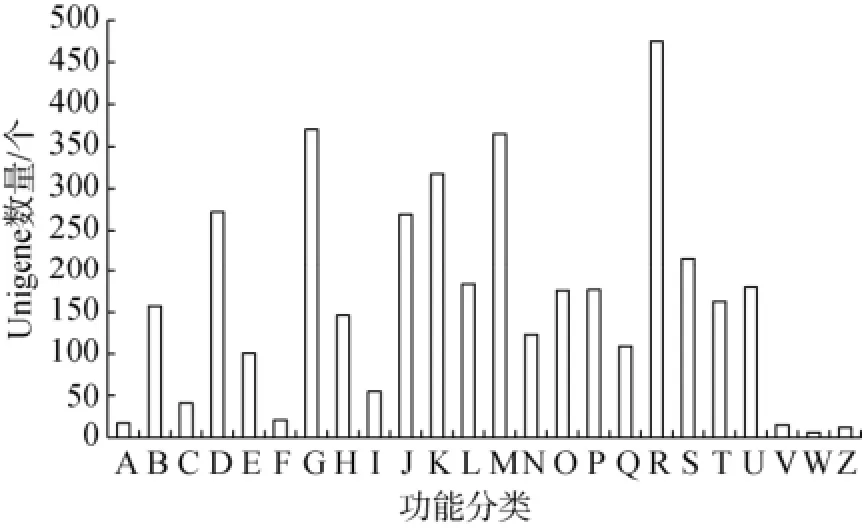
图4 海带转录组包含SSR序列的Unigene COG功能分类Fig. 4 COG functional classification of the unigenes containing SSR in the transcriptome of Saccharina japonica
3 讨论
3.1 海带转录组SSR序列特征
基于已知序列开发SSR标记是SSR标记开发最直接的、快捷的方法。目前已有多个物种通过对转录组数据的分析获得了大量的SSR标记[16-18]。我们的研究发现海带转录组中SSR标记资源也十分丰富,在70 497条Unigenes中共检测出9 237个SSR位点,包含各种核苷酸重复类型。在检测的SSR类型中, 单核苷酸重复类型是最多的, 但是由于转录组与基因组不同, 许多单核苷酸重复是由RNA聚合酶添加的polyA结构, 而不是存在于基因组DNA中的SSR,因此, 在做后续分析的时候并未对单核苷酸重复进行分析[19]。除了单核苷酸重复的SSR外, 最丰富的重复类型是三核苷酸重复, 这与Zhang等人[11]的研究结果一致。与高等植物相似, 海带中AT/TA重复是二碱基中较丰富的重复单元, 而CG/GC是最少的[7]。SSR重复次数的不同是引发其长度多态性的主要原因, 其突变机制主要有DNA聚合酶滑移错配、不平等重组、错配和逆转录转座[7]。而且SSR重复次数越多通常多态性也越高[20]。海带转录组中发现的SSR序列的重复次数较高, 最多重复次数为70。因此, 我们认为这些SSR具有高多态的潜力, 可在种群遗传多样性和种群结构研究中发挥重要作用。
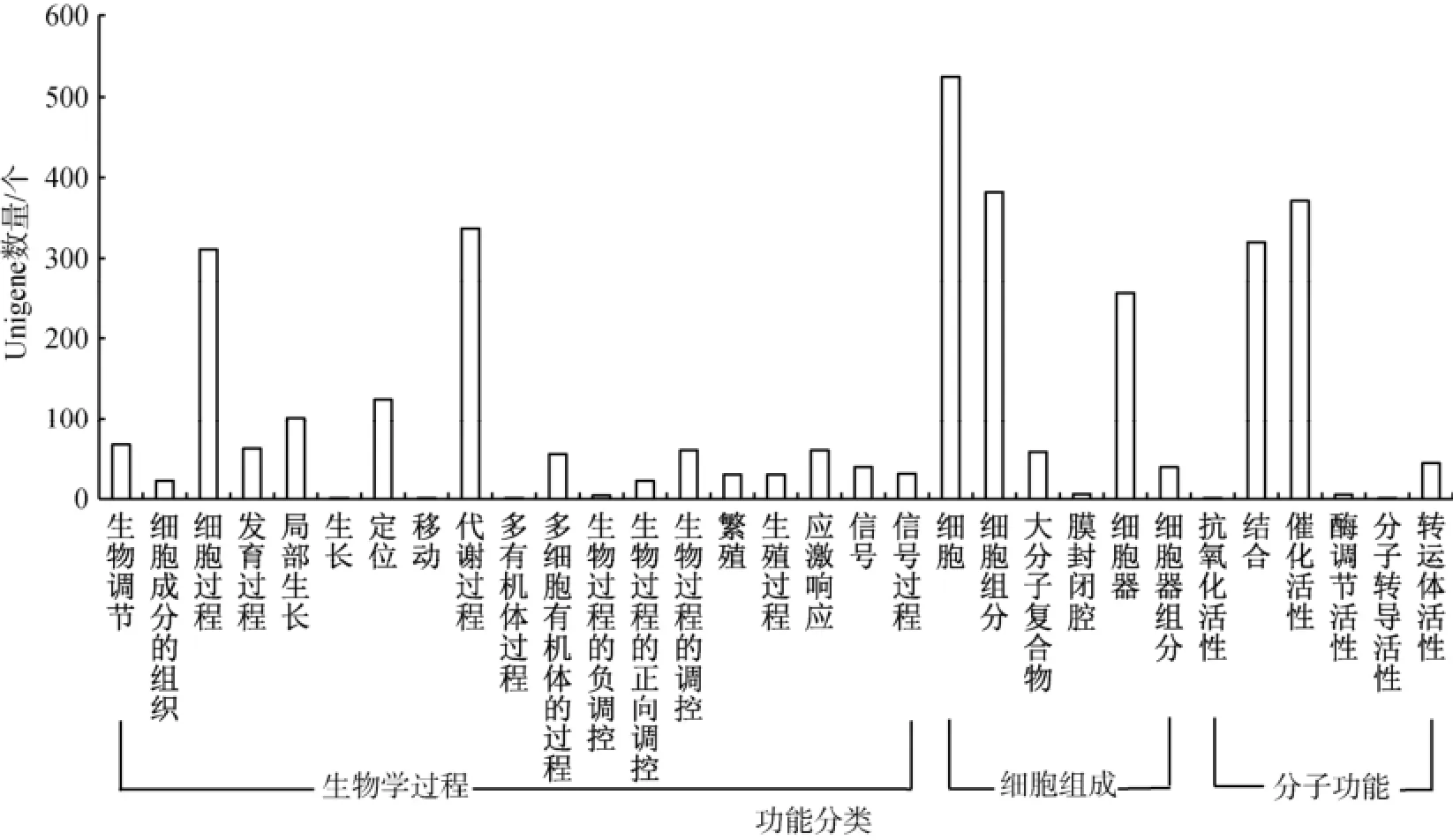
图5 海带转录组包含SSR序列的Unigene GO功能分类Fig. 5 GO functional classification of the unigenes containing SSR in the transcriptome of Saccharina japonica
3.2 海带SSR序列相关功能分析
海带转录组中SSR序列所在Unigenes的功能注释结果表明, 约50%的含有SSR的Unigenes具有注释信息, 并且大多数与已知蛋白具有高度同源性。COG、GO功能分类结果均表明, 大量的SSR序列分布在与碳水化合物代谢及细胞组成相关的基因中。其中一些基因可能与海带中重要的多糖, 甘露醇、褐藻胶和岩藻多糖的合成积累相关, 因为它们分别是海带中重要的储存多糖及细胞壁组成成分[21]。我们选取了含有SSR的几个参与淀粉和蔗糖代谢、三羧酸循环、丙酮酸代谢、果糖和甘露糖代谢的重要基因进行了引物设计(表1), 以便于后续应用研究。分布于这些基因中的SSR变异可能会与海带中这些多糖的合成和积累有关。并且碳水化合物代谢作为基础能量代谢, 与海带其他性状形成也相关。因此, 研究这些功能性SSR标记可为海带中相关多糖积累及细胞壁形成研究提供支撑, 也为将来海带的分子标记辅助选育提供依据。
4 结论
利用已知的海带转录组数据开发海带SSR标记是较为直接、快捷的方法。海带转录组中的SSR类型丰富且重复次数较高, 具有较高的多态性潜能,是开发新的SSR标记的重要资源。海带转录组中大量含有SSR序列的基因参与多种生物功能, 其中参与基础的碳水化合物代谢的基因最多, 这些功能性SSR标记更易于进行性状关联研究。因此, 本研究为进一步开发海带SSR标记奠定了基础, 对于后续海带遗传资源评价、遗传图谱绘制、分子标记辅助选育等研究具有重要意义。
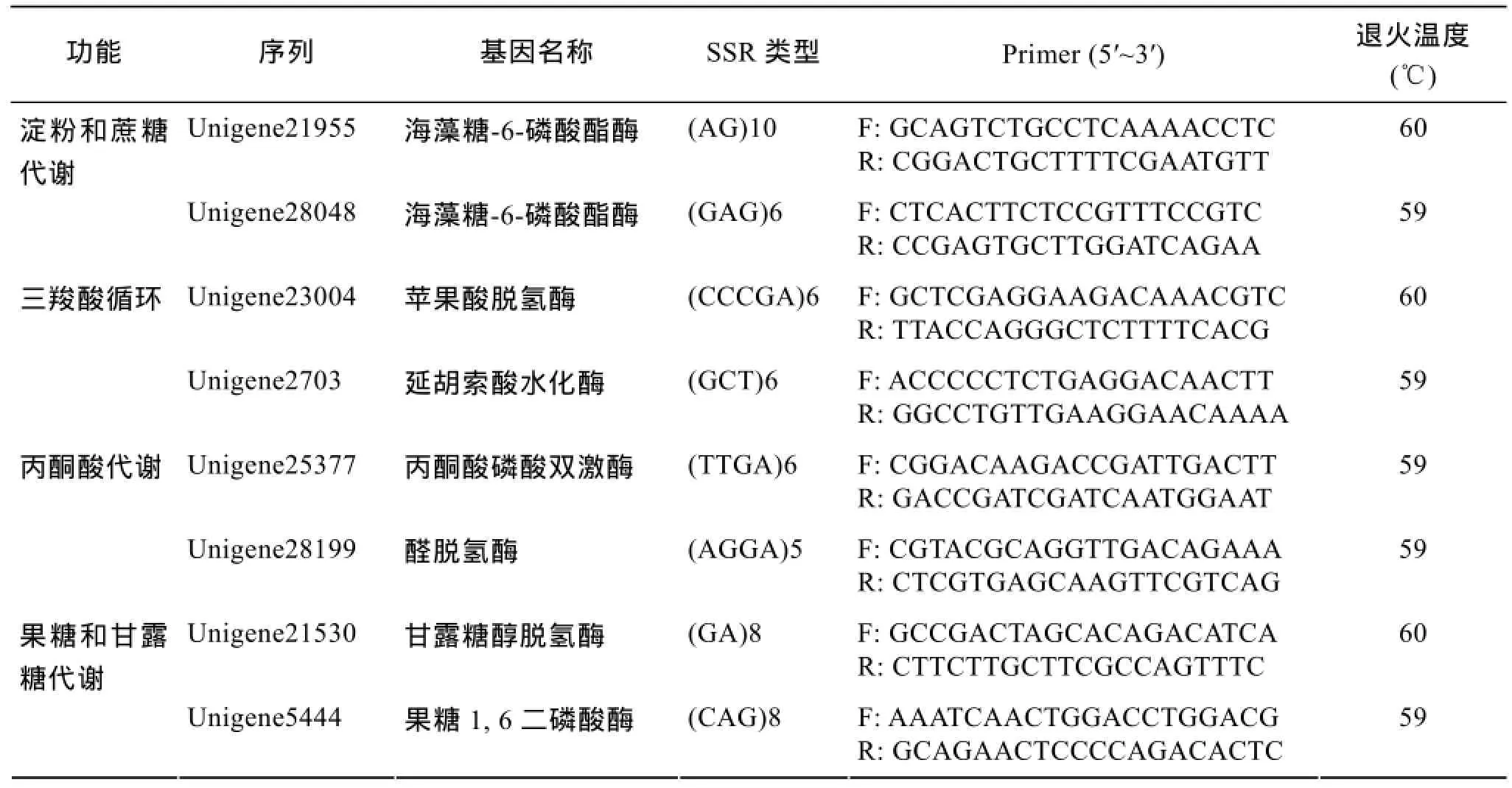
表1 海带中参与多糖代谢部分基因相关SSR及引物Tab. 1 SSR in the genes involved in carbohydrate metabolism and primer information in Saccharian japonica
[1] FAO. Fisheries and aquaculture information and statistics service [EB/OL].[2014-05-05]. http: //faostat3. fao.org/home/index.html#VISUALIZE_BY_DOMAIN.
[2] Liu Fuli, Yao Jianting, Wang Xiuliang, et al. Genetic diversity and structure within and between wild and cultivated Saccharina japonica (Laminariales, Phaeophyta) revealed by SSR markers[J]. Aquaculture, 2012, 358-359: 139-145.
[3] He Yingjun, Zou Yuping, Wang Xiaodong, et al. Assessing the germplasm of Laminaria (Phaeophyceae) with random amplified polymorphic DNA (RAPD) method[J]. Chinese Journal of Oceanology and Limnology, 2003, 21(2): 141-148.
[4] Liu Fuli, Shao Zhanru, Zhang Haining, et al. QTL mapping for frond length and width in Laminaria japonica aresch (Laminarales, Phaeophyta) using AFLP and SSR markers[J]. Marine Biotechnology, 2010, 12(4): 386-394.
[5] Liu Fuli, Wang Xiuliang, Liu Jidong, et al. Genetic mapping of the Laminaria japonica (Laminarales, Phaeophyta) using amplified fragment length polymorphism markers[J]. Journal of Phycology, 2009, 45(5): 1228-1233.
[6] Wang Xiuliang, Yang Yingxia, Cong Yizhou, et al. DNA fingerprinting of selected Laminaria (Phaeophyta) gametophytes by RAPD markers[J]. Aquaculture, 2004, 238: 143-153.
[7] Kalia R K, Rai M K, Kalia S, et al. Microsatellite markers: an overview of the recent progress in plants[J]. Euphytica, 2011, 177(3): 309-334.
[8] Liu Fuli, Wang Xiuliang, Yao Jianting, et al. Development of expressed sequence tag-derived microsatellite markers for Saccharina (Laminaria) japonica[J]. Journal of Applied Phycology, 2010, 22(1): 109-111.
[9] Shi Yuanyuan, Yang Guanpin, Liu Yongjian, et al. Development of 18 polymorphic microsatellite DNA markers of Laminaria japonica (Phaeophyceae)[J]. Molecular Ecology Notes, 2007, 7(4): 620-622.
[10] Zhang Jing, Li Wei, Qu Jieqiong, et al. Development and characterization of microsatellite markers from an enriched genomic library of Saccharina japonica[J]. Journal of Applied Phycology, 2015, 27(1): 479-487.
[11] Zhang Linan, Peng Jie, Li Xiaojie, et al. Development of 27 trinucleotide microsatellite markers for Saccharina japonica using next generation sequencing technology[J]. Conservation Genetics Resources, 2014, 6(2): 341- 344.
[12] Varshney R K, Graner A, Sorrells M E. Genic microsatellite markers in plants: features and applications[J]. Trends in Biotechnology, 2005, 23(1): 48-55.
[13] Deng Yunyan, Yao Jianting, Wang Xiuliang, et al. Transcriptome sequencing and comparative analysis of Saccharina japonica (Laminariales, Phaeophyceae) under blue light induction[J]. Plos One, 2012, 7(6): e39704.
[14] Conesa A, Gotz S, Garcia-Gomez J M, et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics,2005, 21(18): 3674-3676.
[15] Ye Jia, Fang Lin, Zheng Hongkun, et al. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research, 2006, 34: 293-297.
[16] Wei Wenliang, Qi Xiaoqiong, Wang Linhai, et al. Characterization of the sesame (Sesamum indicum L.) global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers[J]. BMC Genomics, 2011, 12: 451.
[17] Yue Xiaoyan, Liu Guoqin, Zong Yu, et al. Development of genic SSR markers from transcriptome sequencing of pear buds[J]. Journal of Zhejiang University-Science B, 2014, 15(4): 303-312.
[18] Dutta S, Kumawat G, Singh B P, et al. Development of genic-SSR markers by deep transcriptome sequencing in pigeonpea [Cajanus cajan (L.) Millspaugh][J]. BMC Plant Biology, 2011, 11: 17.
[19] La Rota M, Kantety R V, Yu J K, et al. Nonrandom distribution and frequencies of genomic and EST-derived microsatellite markers in rice, wheat, and barley[J]. BMC Genomics, 2005, 6: 23.
[20] Cai Ming, Pan Huitang, Wang Xuefeng, et al. Development of novel microsatellites in Lagerstroemia indica and DNA fingerprinting in Chinese Lagerstroemia cultivars[J]. Scientia Horticulturae, 2011, 131: 88-94.
[21] Michel G, Tonon T, Scornet D, et al. Central and storage carbon metabolism of the brown alga Ectocarpus siliculosus: insights into the origin and evolution of storage carbohydrates in Eukaryotes[J]. New Phytologist, 2010, 188(1): 67-81.
Received: Feb. 6, 2015
Characterization of SSR in Saccharina japonica transcriptome and functional analysis of SSR-containing unigenes
LI Qiu-ying1,2,3, ZHANG Jie1,2,3, YAO Jian-ting1,2, WANG Xiu-liang1,2, DUAN De-lin1,2
(1. Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 2. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China; 3. University of Chinese Academy of Sciences, Beijing 100049, China)
microsatellite; transcriptome; simple sequence repeat (SSR); Saccharina japonica
In this study, we identified a total of 9 237 SSR (simple sequence repeat) loci in 70 497 unigenes from the transcriptome data of Saccharina japonica and conducted a function analysis of unigenes containing SSR. SSRs were abundant in the transcriptome of S. japonica, and the most commonly repeating motifs were mononucleotides and trinucleotides (40.9% and 39.4% of the total SSRs, respectively), followed by tetranucleotides, dinucleotides, pentanucleotides, and hexanucleotides (9.8%, 6.1%, 3.1%, and 0.7% of the total SSRs, respectively). There were 147 repeated motif types ranging from 5 to 70 repetitions. Our analysis of the functional annotation revealed that nearly 50% of the unigenes containing SSRs contain annotation information and that most of them are homologous to known proteins. Our clusters of orthologous group (COG) classification and gene ontology (GO) assignment results indicate that genes containing SSR are involved in various biological functions, of which carbohydrate metabolism and cell components are the top two. These SSRs provide new resources for further developing functional SSR markers, which can play important roles in molecular marker-assisted selection while breeding S. japonica.
S917.3
A
1000-3096(2016)11-0001-06
10.11759/hykx20150206001
(本文编辑: 刘珊珊)
2015-02-06;
2015-03-17
国家自然科学基金项目(31272660); 国家科技支撑计划项目(2013BAB01B01); 国家海洋公益性行业科研专项(201405040)
[Foundation: National Natural Science Foundation of China, No. 31272660; National Key Technology Research and Development Program, No. 2013BAB01B01; Ocean Public Welfare Scientific Research Project, No. 201405040]
李秋莹(1986-), 女, 辽宁锦州人, 博士研究生, 研究方向为海藻遗传, 电话: 0532-82898554, E-mail: liqiuying21@163.com; 段德麟, 通信作者, 研究员, 电话: 0532-82898556, E-mail: dlduan@qdio.ac.cn;王秀良, 通信作者, 副研究员, 电话: 0532-82898554, E-mail: xlwang@ qdio.ac.cn

