不同产地黄硬皮马勃的红外光谱鉴别方法研究
吕伟奇,张 霁,赵艳丽,王元忠*
(1.云南中医学院 中药学院,云南 昆明 650500; 2.云南省农业科学院 药用植物研究所,云南 昆明 650200)
不同产地黄硬皮马勃的红外光谱鉴别方法研究
吕伟奇1,2,张 霁2,赵艳丽2,王元忠2*
(1.云南中医学院 中药学院,云南 昆明 650500; 2.云南省农业科学院 药用植物研究所,云南 昆明 650200)
黄硬皮马勃(SclerodermaflavidumEll.et EV.)主要生长在我国南方地区,有食用和药用价值。利用红外技术对其进行产地鉴别,为黄硬皮马勃的资源鉴别提供基础分析方法和理论依据。利用傅里叶变换红外光谱(Fourier transform infrared spectroscopy,FTIR)仪采集4个不同产地共40个黄硬皮马勃样本的红外光谱,每个样本平行扫描3次,取平均值。随机选择12个样本作为验证集,其余作为校正集,采用1 800~500 cm-1波段的光谱数据,对比多种预处理方法,选择最佳预处理方法建立模型并进行判别分析(discriminant analysis,DA)。结果表明,预处理方法ND(7∶3)+SD+MSC(ND为诺里斯导数平滑,SD为二阶导数,MSC为多元散射校正)构建的判别模型性能最佳,验证集样本的分类准确率和校正集样本的回判准确率均达到100%,模型鉴别效果良好,可靠性高。傅里叶变换红外光谱法结合判别分析,能有效鉴别不同产地黄硬皮马勃。
黄硬皮马勃; 产地鉴别; 傅里叶变换红外光谱; 判别分析
黄硬皮马勃(SclerodermaflavidumEll.et EV.)属于硬皮马勃目硬皮马勃科真菌,分布于云南、福建、广西、广东、香港、海南等地[1],夏秋季群生或单生于阔叶林地和林缘山坡上,与栎、马尾松形成外生菌根。此菌有轻微毒性,幼时可食用,味道鲜美,成熟后孢粉有较好的抑菌消炎作用[2],具有成为高效抗菌药物原料的潜力。
长久以来,人类都在与病原菌导致的疾病做抗争,抗菌药物数量和品质的需求不断提高。目前市场上抗菌类药物大多为抗生素[3]和化学合成药[4],这2类药物治疗效果明显,却易产生耐药性等副作用[5],给人体带来较大伤害[6],新型抗菌药研制引起广泛关注[7]。黄硬皮马勃等真菌含有天然抗菌效果的活性物质[8],对研制低副作用抗菌药物有重要意义。研究表明,大部分真菌物种的品质与其产地密切相关[9],进行黄硬皮马勃的产地鉴别,有助于筛选和开发优质黄硬皮马勃资源,为相关抗菌药物的研发提供选材方面的参考。
傅里叶变换红外光谱法作为常用的材料检测方法,通过红外光谱扫描,使样本分子中基团或化学键吸收能量产生振动,形成振动吸收光谱用于鉴别分析[10]。此方法具有灵敏、无损、样本用量少等特点[11],广泛应用于中药分析[12-13]、酒类分析[14-15]、农产品分析[16-17]等领域。
采自不同产地的黄硬皮马勃,外观无明显差异,不易判断产地来源,需要有效鉴别手段。目前,尚未见黄硬皮马勃产地鉴别方面的报道。为了探寻一种能够快速、准确鉴别黄硬皮马勃产地的方法,本研究采用傅里叶变换红外光谱法,采集云南4个地区黄硬皮马勃的红外图谱,结合判别分析[18]处理光谱数据,提取有效信息,辅助产地鉴别。
1 材料和方法
1.1 试验材料
试验所用的马勃样本于2014年采自云南省玉溪市(表1),经云南农业大学刘鸿高教授鉴定为黄硬皮马勃。

表1 材料的来源和数量
1.2 试验仪器及试剂
Frontier型傅里叶变换红外光谱仪(美国Perkin Elmer公司),配备DTGS检测器,扫描范围为4 000~400 cm-1,分辨率为4 cm-1,扫描信号累加16次;FW-100型高速万能粉碎机(天津市华鑫仪器厂);0.250 mm孔径标准筛(浙江上虞市道墟五四仪器厂);YP-2型压片机(上海市山岳科学仪器有限公司);XS125A型电子分析天平(瑞士Precisa公司);溴化钾(分析纯,天津市风船化学试剂科技有限公司)。
1.3 试验方法
1.3.1 样本预处理 将样本在60 ℃下干燥至恒定质量,粉碎后过0.250 mm孔径标准筛,放入自封袋储存备用,各产地样本随机编号见表1。
1.3.2 红外光谱采集 称取100 mg溴化钾压片,扫描后作为背景去除干扰信息。1~40号黄硬皮马勃样本在干燥箱中干燥24 h,依次精密称取1.0 mg放入玛瑙研钵中研细,按1∶100的比例加入溴化钾研磨均匀,压制成均匀透明的样本薄片,扫描薄片,采集样本的红外光谱,每个样本平行扫描3次,取平均值作为该样本原始光谱。
1.4 图谱优化处理
利用Omnic 8.0软件对样本光谱图进行吸光度转换、自动基线校正、纵坐标归一化等优化处理。
1.5 建立判别模型
图谱优化处理后,数据导入TQ 8.6软件,选择合适波段与最佳预处理方法建立判别模型进行分析。判别模型以每个产地材料中任意3个样本作为验证集,其余28个样本作为校正集(表1)。
2 结果与分析
2.1 平均光谱及其特征分析
由图1可以看出,黄硬皮马勃4个产地的平均红外光谱无明显差异,在3 347、2 926、1 637、1 393、1 248、1 076、1 041 cm-17个波长附近都有较强吸收峰,峰形基本一致。3 347 cm-1附近吸收峰峰形平缓,由阿洛糖醇等醇类化合物[19]中醇羟基的O-H键伸缩振动形成;2 926 cm-1附近吸收峰由烷烃结构中的C-H键反对称伸缩振动形成;1 637 cm-1附近吸收峰由烯烃结构中的C=C键伸缩振动形成;1 393 cm-1附近吸收峰由芳香环C=C键的伸缩振动形成;醇类化合物中C-OH键伸缩振动与COH面内弯曲振动发生耦合,形成1 248 cm-1附近吸收峰,同时,旋转异构体的存在导致1 076、1 041 cm-1附近双吸收峰[10]。上述分析表明黄硬皮马勃主要成分为多糖类物质。结合图1,显示4个产地黄硬皮马勃红外光谱图波形相似,直接观察无法达到产地鉴别的要求。
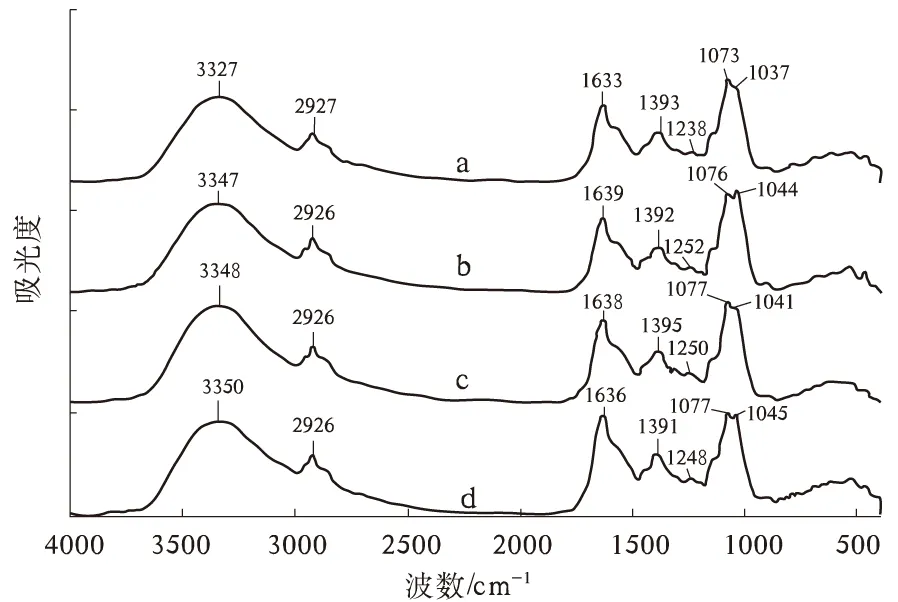
a.玉溪市红塔区; b.玉溪市九溪镇;c.玉溪市峨山双江镇; d.玉溪市大营街镇图1 不同产地黄硬皮马勃的光谱图
2.2 判别分析
判别分析利用已知样本的光谱数据,建立判别分析模型,可以对未知样本快速准确地进行分类鉴别。为建立高性能模型,需要筛选建模波段,对样本光谱进行预处理,以提取有效的建模信息。
样本红外图谱总体波段包括4 000~400 cm-1,含有较多复杂数据点,容易造成干扰,需要选择合适波段的数据进行分析[20]。由图1和图2可以看出,光谱图在波段1 800~500 cm-1波形起伏较大,图谱间差异表现明显,因此选择此波段光谱构建判别模型。

图2 一阶导数光谱图
将Omnic 8.0软件优化后的校正集样本图谱数据导入TQ 8.6软件,按不同预处理方法构建模型,比较模型性能以获得最佳的预处理方法(表2)。采用不同预处理方法构建的模型对验证集样本进行判别,结果显示,当建模主成分数量在一定范围时,各模型中验证集样本的分类准确率和校正集样本的回判准确率都达到100%。
注:ND为诺里斯导数平滑(norris derivative smoothing);FD为一阶导数(first derivative);SD为二阶导数(second derivative);SNV为标准归一化(standard normal variate);MSC为多元散射校正(multiplicative signal correction)。
相同预处理方法下,比较鉴别无误时各主成分数构建模型的性能,选择性能最佳的模型使用的主成分数作为最佳主成分数。不同预处理方法下,比较最佳主成分数构建模型的性能。结果显示,选用预处理方法ND(7∶3)+SD+MSC,以14个主成分建立模型,与其他方法相比构建的模型性能最佳,可靠性高。此处理方法下建模,前14个主成分累积贡献率为99.6%,可以解释大部分光谱特征信息。
判别分析建立的模型可以根据校正集中不同产地黄硬皮马勃样本的特征信息构建4个类群的距离判别函数式,计算验证集各样本与4个类群的马氏距离(mahalanobis distance,MD),马氏距离是通过数据协方差距离来计算的样本与类群的相似度,距离值越小,相似度越大[21]。表3为判别模型依据马氏距离得出的鉴别结果,所列样本均被准确鉴别。数据显示,大部分相同类群样本至同一类群的距离相近,说明相同产地黄硬皮马勃样本化学组分相似度高。第2类群中11、12号样本与13号样本到第1、2、3类群距离相差约为1,表明第2产地部分样本化学组分与同类群其他样本有差异,可能是第2产地内不同样本采集环境差异造成的。
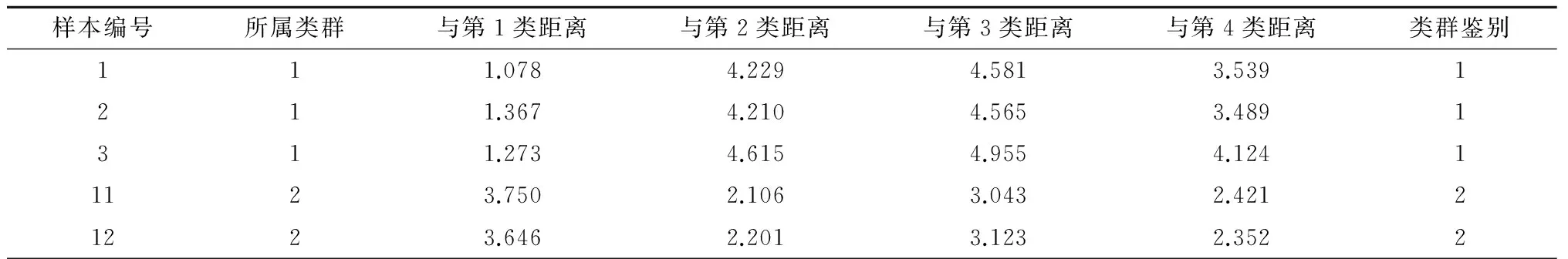
表3 验证集类别鉴定
3 结论与讨论
傅里叶变换红外光谱技术在20世纪后期的30 a发展较快,红外仪器迅速更新换代,性能大幅度提高,技术不断革新,且得到了大范围的推广,可有效应用于未知物的定量和定性分析[10]。红外技术通过读取微生物菌体中细胞壁、细胞膜及细胞内包括的蛋白质、水、脂肪、多糖等成分的化学键振动情况,提供整个微生物的光谱信息[22]。因此,傅里叶变换红外光谱技术可以提供含有真菌整体组分的指纹特征图谱,以不同产地黄硬皮马勃图谱为基础分析差异,可以实现传统分类方法无法实现的高效鉴别。
通过傅里叶变换红外光谱法采集4个不同产地黄硬皮马勃的红外光谱图,对全波段图谱数据进行特征分析,结果显示,不同产地样本图谱波形相似,难以直接辨别。为准确鉴别样本产地,采用TQ 8.6软件,选择1 800~500 cm-1光谱数据,构建判别分析模型。图谱预处理方法和建模使用的主成分数对模型性能有影响,需要筛选合适的预处理方法和主成分数。试验以诺里斯导数平滑、一阶导数、二阶导数、标准归一化、多元散射校正5种图谱处理方法组合为8种预处理方法,结合TQ软件选择不同预处理方法和主成分数构建多个模型并对比模型性能。对比发现,选择预处理方法ND(7∶3)+SD+MSC,以14个主成分建立判别分析模型性能最高,验证集样本的分类准确率和校正集样本的回判准确率均达到100%,鉴别效果良好。
本研究表明,判别分析通过提取红外图谱主要特征信息,可以有效分析不同样本图谱间的差异,准确鉴别不同产地黄硬皮马勃。傅里叶变换红外光谱法和判别分析的结合,以已知样本信息建立判别模型判断未知样本类群,为鉴别不同产地黄硬皮马勃提供了一种快速、准确的产地鉴别方法。此方法对黄硬皮马勃的产地鉴定具有重要意义,但无法明确不同产地样本品质的优劣,需要进一步研究。
[1] 黄年来.中国大型真菌原色图鉴[M].北京:中国农业出版社,1998:235.
[2] 卯晓岚.中国大型真菌[M].郑州:河南科学技术出版社,2000:555.
[3] Zilberberg M D,Shorr A F,Micek S T,etal.Multi-drug resistance,inappropriate initial antibiotic therapy and mortality in Gram-negative severe sepsis and septic shock:A retrospective cohort study[J].Critical Care,2014,18(6):596.
[4] Bruhn D F,Scherman M S,Woolhiser L K,etal.Pentacyclic nitrofurans withinvivoefficacy and activity against nonreplicatingMycobacteriumtuberculosis[J].Plos One,2014,9(2):e87909.
[5] Hill J A,Ammar R,Torti D,etal.Genetic and genomic architecture of the evolution of resistance to antifungal drug combinations[J].Plos Genetics,2013,9(4):269-271.
[6] Kalghatgi S,Spina C S,Costello J C,etal.Bactericidal antibiotics induce mitochondrial dysfunction and oxidative damage in mammalian cells[J].Science Translational Medicine,2013,5(192):192ra85.
[7] McPhillie M J,Cain R M,Narramore S,etal.Computational methods to identify new antibacterial targets[J].Chemical Biology & Drug Design,2015,85(1):22-29.
[8] Sibounnavong P,Charoenporn C,Kanokmedhakul S,etal.Antifungal metabolites from antagonistic fungi used to control tomato wilt fungusFusariumoxysporumf.sp.lycopersici[J].African Journal of Biotechnology,2011,10(85):19714-19722.
[9] Pena R,Lang C,Naumann A,etal.Ectomycorrhizal identification in environmental samples of tree roots by Fourier-transform infrared(FTIR) spectroscopy[J].Frontiers in Plant Science,2014,5:229.
[10] 翁诗甫.傅里叶变换红外光谱分析[M].北京:化学工业出版社,2010:1-33.
[11] Fernández-González A,Montejo-Bernardo J M,Rodríguez-Prieto H,etal.Easy-to-use analytical approach based on ATR-FTIR and chemometrics to identify apple varieties under Protected Designation of Origin(PDO)[J].Computers and Electronics in Agriculture,2014,108:166-172.
[12] Kwon Y K,Ahn M S,Park J S,etal.Discrimination of cultivation ages and cultivars of ginseng leaves using Fourier transform infrared spectroscopy combined with multivariate analysis[J].Journal of Ginseng Research,2014,38(1):52-58.
[13] Chuang Y K,Yang I C,Lo Y M,etal.Integration of independent component analysis with near-infrared spectroscopy for analysis of bioactive components in the medicinal plantGentianascabraBunge[J].Journal of Food and Drug Analysis,2014,22(3):336-344.
[14] Silva S D,Feliciano R P,Boas L V,etal.Application of FTIR-ATR to Moscatel dessert wines for prediction of total phenolic and flavonoid contents and antioxidant capacity[J].Food Chemistry,2014,150(2):489-493.
[15] Malacarne M,Bergamo L,Bertoldi D,etal.Use of Fourier transform infrared spectroscopy to create models forecasting the tartaric stability of wines[J].Talanta,2013,117(22):505-510.
[16] Lohumi S,Lee S,Lee W H,etal.Detection of starch adulteration in onion powder by FT-NIR and FT-IR spectroscopy[J].Journal of Agricultural and Food Chemistry,2014,62(38):9246-9251.
[17] Rohman A,Man Y B C,Yusof F M.The use of FTIR spectroscopy and chemometrics for rapid authentication of extra virgin olive oil[J].Journal of the American Oil Chemists Society,2014,91(2):207-213.
[18] 赵艳丽,张霁,袁天军,等.近红外光谱快速鉴别不同产地药用植物重楼的方法研究[J].光谱学与光谱分析,2014,34(11):2968-2972.
[19] 王晨英,高锦明,杨雪,等.黄硬皮马勃的化学成分[J].中草药,2002,33(9):778-780.
[20] Cao Z,Liu Y,Zhao J.Efficient discrimination of some moss species by Fourier transform infrared spectroscopy and chemometrics[J].Journal of Spectroscopy,2014,44(8):849-860.
[21] e Oliveira F S A S,Giana H E,Silveira L.Discrimination of selected species of pathogenic bacteria using near-infrared Raman spectroscopy and principal components analysis[J].Journal of Biomedical Optics,2012,17(10):2002-2009.
[22] Naumann D.Some ultrastructural information on intact,living bacterial cells and related cell-wall fragments as given by FTIR[J].Infrared Physics,1984,24(2):233-238.
Discrimination ofSclerodermaflavidumfrom Different Geographical Origins by Infrared Spectroscopy
LÜ Weiqi1,2,ZHANG Ji2,ZHAO Yanli2,WANG Yuanzhong2*
(1.College of Traditional Chinese Medicine,Yunnan University of Traditional Chinese Medicine,Kunming 650500,China; 2.Institute of Medicinal Plants,Yunnan Academy of Agricultural Sciences,Kunming 650200,China)
SclerodermaflavidumEll.et EV.which has both medicinal and edible value,is mainly distributed in south China.Discrimination ofS.flavidumfrom different geographical origins by infrared spectroscopy could provide an basic analysis method and theoretical basis for distinguishing its resource.Fourier transform infrared(FTIR) spectroscopy method was used to study 40S.flavidumsamples from four different producing areas.Every sample was scanned for three times to get the average spectrum.Twelve samples were chosen randomly to form the validation set and others were used to build calibration set.According to the contrastive results of different pretreatment methods for the spectra with the range of 1 800—500 cm-1,the best pretreatment method was used to build the discriminant analysis model.The results indicated that the model with pretreatment method ND(7∶3)+SD+MSC was the best.The prediction accuracy of the validation sets and the correct rate of returned classification of calibration set all reached 100%.The model had good effect and high reliability.FTIR combined with discriminant analysis could provide a fast and high-efficiency way to distinguish theS.flavidumwith different geographical origins,effectively.
SclerodermaflavidumEll.et EV; discrimination of geographical origins; fourier transform infrared(FTIR) spectroscopy; discriminant analysis
2015-06-20
国家自然科学基金项目(31460538)
吕伟奇(1991-),男,山东聊城人,在读硕士研究生,研究方向:中药资源开发与利用。 E-mail:lvweiqi1991@126.com
*通讯作者:王元忠(1981-),男,云南怒江人,助理研究员,硕士,主要从事药用真菌研究。E-mail:boletus@126.com
S567
A
1004-3268(2016)01-0104-05
——马勃

