鼠伤寒沙门氏菌ompc基因PCR的检测方法
宫 强,李战丽,牛明福,王 辉,刘永康,秦翠丽,孙晓菲
(河南科技大学食品与生物工程学院,河南 洛阳 471023)
鼠伤寒沙门氏菌ompc基因PCR的检测方法
宫 强,李战丽,牛明福,王 辉,刘永康,秦翠丽,孙晓菲
(河南科技大学食品与生物工程学院,河南 洛阳 471023)
建立一种鼠伤寒沙门氏菌的聚合酶链式反应检测方法。根据GenBank中已发表的鼠伤寒沙门氏菌的ompc的基因 序列,设计和合成了一对特异性引物,并优化了反应条件,检测了该方法的敏感性和特异性。结果成功扩增出了鼠伤寒沙门氏菌470 bp的ompc特异性基因片段,而对致病性大肠杆菌、金黄色葡萄球菌、绿脓杆菌和志贺氏菌4 种食品中常见的病原菌均未扩增出相应片段。敏感性实验结果表明本方法可检测到最低1 pg/μL的鼠伤寒沙门氏菌基因组DNA。
鼠伤寒沙门氏菌;ompc基因;聚合酶链式反应;检测方法
沙门氏菌是常见的肠道致病菌之一,其中部分沙门氏菌具有人畜共患性,也是重要的食源性病原微生物,可通过食品感染人从而引发食物中毒。据统计在全球范围内的细菌性食物中毒中,沙门氏菌引起的食物中毒名列榜首[1],我国内陆地区也以沙门氏菌为首。在我国目前流行的众多血清型中,鼠伤寒沙门氏菌是最为常见的一种,也是引起沙门氏菌食物中毒的主要血清型之一。据Helms等[2]的统计结果显示,鼠伤寒沙门氏菌引起的感染性疾病接近 沙门氏菌感染总数的五分之一。为预防和控制鼠伤寒沙门氏菌引起的食物中毒,除做好常规的消毒卫生等环节外,对食品中该菌的快速检测无疑对预防该菌引起的食物中毒具有重要的意义。然而,目前对该菌的检测方法仍然是以传统的病原分离和生化鉴定为主,虽然该方法能取得可靠的鉴定结果,但存在操作繁琐、耗时长等许多不足之处,因此急需开发一种快速、敏感且特异的检测方法。本研究以鼠伤寒沙门氏菌的ompc基因为基础,建立了用于检测此菌的聚合酶链式反应(polymerase chain reaction,PCR)方法,以期能为鼠伤寒沙门氏菌PCR快速检测试剂盒的研制提供参考依据。
1 材料与方法
1.1 菌株与试剂
鼠伤寒沙门氏菌和志贺氏菌 广东省微生物菌种保藏中心;致病性大肠杆菌、金黄色葡萄球菌和绿脓杆菌为本实验室分离、鉴定和保存;DL-5000 Marker、dNTPs、TaqDNA聚合酶 大连宝生物工程有限公司;琼脂原 北京鼎国生物工程有限公司。
1.2 方法
1.2.1 鼠伤寒沙门氏菌基因组DNA的提取
由保存的菌种中无菌挑取鼠伤寒沙门氏菌单个菌落,接种于LB培养基中,37 ℃振荡培养过夜,取50 mL菌液以十六烷基三甲基溴化铵(hexadecyltrimethylammonium bromide,CTAB)法提取鼠伤寒沙门氏菌的基因组。
1.2.2 引物设计
根据GenBank上已发表的沙门氏菌ompc基因的序列设计合成用于扩增该基因470 bp片段的特异性引物,用无菌去离子水将上下游引物均稀释至浓度为25 μmol/L,-20 ℃冻存备用,引物序列如下所示:
PU:5’-AGTTAAAGTACTGTCCCTCC-3’;PL:5’-CGAAGAAGTCGGTGTTAC-3’。
1.2.3 ompc基因的PCR扩增
以鼠伤寒沙门氏菌基因组DNA为模板,以PU和PL引物进行PCR扩增目的基因,并设立无菌去离子水为模板的阴性对照。反应体系中含模板1 μL,PU和PL引物各0.5 μL,10×PCR buffer 3 μL,dNTPs 1.5 μL,Taq DNA聚合酶0.4 μL,以无菌去离子水补足至总体积25 μL。反应程序如下:95 ℃预变性5 min;94 ℃变形性45 s;52 ℃退火25 s;72 ℃延伸45 s,共30 个循环;72 ℃终延伸7 min。PCR结束后取3 μL产物进行0.8%的琼脂原凝胶电泳以检测PCR扩增片段的大小,随后送测序公司进行测序。
1.2.4 ompc基因PCR反应体系与反应条件的优化
为探索最佳的PCR反应条件,本实验对反应过程中的多个条件包括引物浓度、退火温度、dNTPs浓度和循环次数进行优化,具体参数如表1所示。PCR反应结束后各取2 μL产物进行0.8%的琼脂原凝胶电泳,并以Image LabTM软件对PCR产物的亮度进行分析。
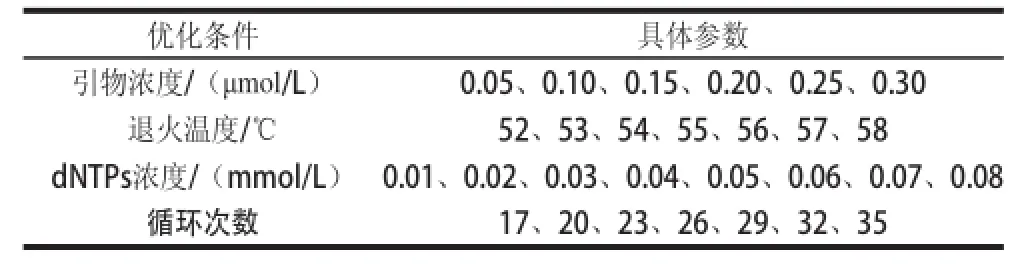
表1 PCR反应条件的优化Table 1 Optimization of PCR reaction conditions
1.2.5 敏感性实验
以紫外分光光度计测定本实验所提取的鼠伤寒沙门氏菌的基因组质质浓度,以无菌去离子水稀释至模板质质浓度为1 ng/μL,随后按照10 倍的比例进行梯度稀释,直至模板质质浓度为0.1 pg/μL。随后按上述所测得的最佳反应条件进行扩增,同时设置无菌去离子水为模板的阴性对照,从而检测出本实验所建立的PCR反应的敏感性。
1.2.6 特异性实验
以CTAB法分别提取绿脓杆菌、致病性大肠杆菌、金黄色葡萄球菌和志贺氏菌的基因组DNA,以鼠伤寒沙门氏菌基因组DNA为阳性对照,以无菌去离子水模板做阴性对照,以本实验所设计合成的引物,对上述各菌的基因组DNA进行PCR扩增以鉴定该PCR检测法的特异性。PCR反应条件如下:模板1 μL,PU和PL引物各0.5 μL(终浓度为0.25 μmol/L),10×PCR buffer 3 μL,dNTPs 1.5 μL(终浓度为0.08 mmol/L),Taq DNA聚合酶0.4 μL,以无菌去离子水补足至总体积25 μL。反应程序如下:95 ℃预变性5 min;94 ℃变形性45 s;56 ℃退火25 s;72 ℃延伸45 s,共30 个循环;72 ℃终延伸7 min。PCR结束后取1.5 μL产物进行0.8%的琼脂原凝胶电泳以检测是否有目的基因的产生,从而检测特异性。
2 结果与分析
2.1 鼠伤寒沙门氏菌ompc基因PCR扩增结果
以合成的ompc基因的特异性引物鼠伤寒沙门氏菌的基因组DNA进行PCR扩增,琼脂原凝胶电泳结果如图1所示,在470 bp处出现与预期大小一致目的条带,PCR产物经序列测定与GenBank中登陆的的ompc基因序列完全一致,说明PCR扩增成功得到目的基因片段。

图1 1 ompcompc基因PCR产物电泳图Fig.1 Electrophoretogram of PCR amplified products of ompc gene
2.2 PCR反应条件的优化结果
2.2.1 引物浓度的优化
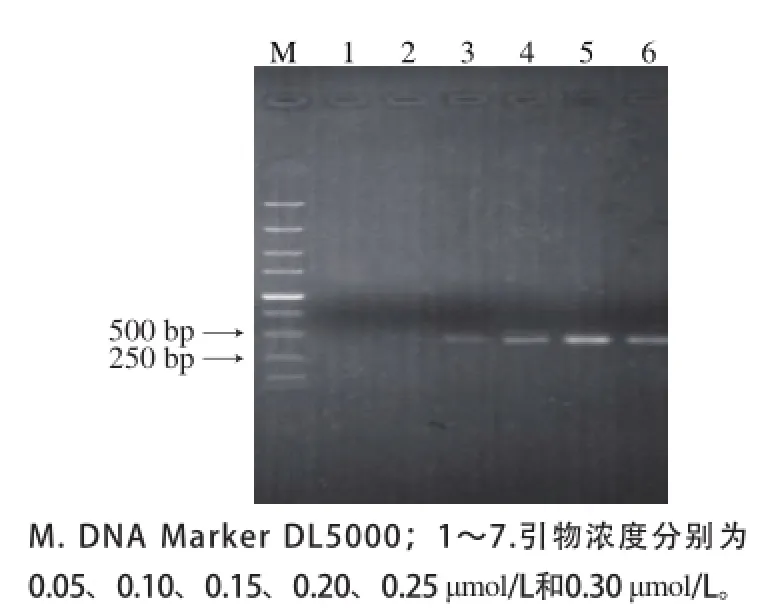
图2 引物浓度的优化结果Fig.2 Optimization of primer concentration
对PCR反应过程中的引物浓度进行优化,结果如图2所示。在本实验所检测的引物浓度中,当引物浓度为0.25 μmol/L时,目的条带最明亮,因此选取每条引物浓度为0.25 μmol/L作为最佳引物浓度。
2.2.2 退火温度的优化
对PCR反应过程中的退火温度进行优化,结果如图3所示,当退火温度在56 ℃时,PCR产物条带最亮,因此选择56 ℃作为ompc基因PCR反应的最佳退火温度。

图3 退火温度的优化结果Fig.3 Optimization of annealing temperature
2.2.3 dNTPs浓度的优化

图4 dNTPs浓度的优化结果Fig.4 Optimization of dNTP concentration
dNTPs浓度的优化结果如图4所示。利用Image LabTM软件分析结果显示,在本实验所设定的dNTPs浓度中,当dNTPs浓度为0.08 mmol/L时,目的条带最明亮,因此选择0.08 mmol/L为反应的最佳dNTPs浓度。
2.2.4 循环次数的优化
对PCR反应过程中的循环次数进行优化,结果如图5所示,在循环次数为23 次时可以观察到有目的条带出现,在循环29 次时即可出现较为明亮的条带,之后PCR产物的亮度并未随循环次数的增加而明显升高,因此本研究选取29 次循环为最佳循环次数。
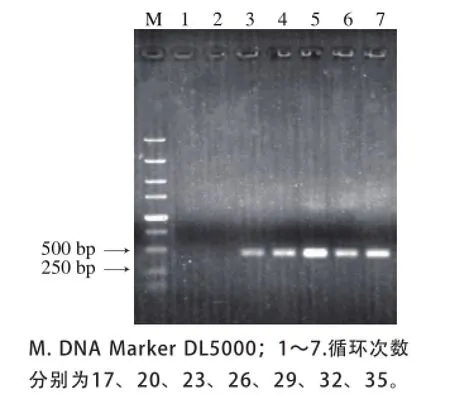
图5 PCR反应循环次数优化结果Fig.5 Optimization of PCR cycle number
2.3 PCR反应敏感性实验结果
以倍比稀释后的鼠伤寒沙门氏菌基因组DNA为模板进行PCR敏感性实验,结果如图6所示,在模板DNA的质低至1 pg/μL时仍能扩增出目的基因片段,而在0.1 pg/μL时则扩增不出目的条带,由此可见本实验所建立的PCR检测方法的敏感度为1 pg/μL。

图6 敏感性实验结果Fig.6 Results of sensitivity tests
2.4 PCR扩增特异性实验结果

图7 特异性实验结果Fig.7 Results of specificity tests
以本实验所用引物,按优化获得的最佳反应条件对食品中常见的致病性大肠杆菌、金黄色葡萄球菌、绿脓杆菌和志贺氏菌的基因组DNA进行PCR扩增。经Image LabTM软件分析实验结果,如图7所示,以鼠伤寒沙门氏菌基因组为模板可扩增出470 bp大小的的ompc DNA片段,而在同样的实验条件下以致病性大肠杆菌、金黄色葡萄球菌、绿脓杆菌和志贺氏菌的基因组为模板则扩增不出该DNA片段,表明本实验的PCR检测方法具有良好的特异性。
3 讨 论
鼠伤寒沙门氏菌作为重要的食源性病原微生物之一,因其传播途径广,侵染宿主多,流行较为严重,加之人们对食品安全的关注,使该菌引起的食物中毒越来越受到人们的关注。因此,寻求一种快速、特异且灵敏的检测方法对于该菌进行早期检测无疑具有重要的现实意义。目前,对于该菌检测方法的研究主要是传统的病原菌分离培养、生化鉴定等方法,该方法虽然较为准确,但操作费时[3],而该菌引起的食物中毒来势较急,因此该传统方法不适于临床的早期检测。
PCR检测技术是目前较为成熟的食品微生物检测技术[4],由于其在微生物检测诊断方面具有快速、敏感等优势而受到人们的青睐。目前与其相关的检测技术已成为科研人员研究的热点之一,包括常规PCR、多重PCR、套式PCR、荧光定质PCR技术和免疫PCR技术等[5]。该技术已应用于多种食源性病原微生物如志贺氏菌、李氏杆菌、金黄色葡萄球菌、霍乱弧菌、大肠杆菌等的早期检测中[6-10]。
本研究建立了一种基于鼠伤寒沙门氏菌ompc基因的PCR检测方法,成功扩增出目的基因,同时针对食品中较为常见的4 类病原菌包括致病性大肠杆菌、金黄色葡萄球菌、绿脓杆菌和志贺氏菌进行了PCR的特异性实验,结果证实本实验具有较好的特异性。目前对于各类沙门氏菌PCR检测方法的研究也较为多见,有只针对一种沙门氏菌的检测方法[11-14],也有检测多种沙门氏菌的PCR检测方法[15-16],亦有同时检测沙门氏菌和食品中其他常见菌的多重PCR检测方法[17-19]。目前针对鼠伤寒沙门氏菌的也有所报道,但不同研究建立的各类PCR检测方法的敏感性差异较大,而且表示方法有所不同。如陈晓玲等[20]建立的鼠伤寒沙门氏菌PCR检测方法的敏感性为129 pg/μL。欧阳本等[21]利用该菌的mgtC和sopB基因建立了虫重PCR检测方法,该方法的敏感性为10 CFU/mL。荣策等[22]建立了实时荧光定质PCR检测方法,其敏感性为300 CFU/mL。冯飞等[23]建立的多重PCR检测方法的敏感性为630 CFU/mL。而本实验建立的鼠伤寒沙门氏菌ompc基因的PCR检测方法的敏感性可达1 pg/μL,目前鲜有以ompc基因建立PCR检测方法的相关报道。而且本实验对PCR反应的退火温度、循环次数等反应参数进行了优化,进一步缩短了反应时间,节约了成本,从而为鼠伤寒沙门氏菌的快速诊断提供了一定的参考。
[1] KUNWAR R, SINGH H, MANGLA V, et al. Outbreak investigation:Salmonella food poisoning[J]. Medical Journal of Armed Forces India,2013, 69(4): 388-391.
[2] HELMS M, ETHELBERG S, MФLBAK K, et al. International Salmonella typhimurium DT104 infections, 1992—2001[J]. Emerging Infectious Diseases, 2005, 11(6): 859-867.
[3] 曹冬梅, 徐杨, 袁慕云, 等. 焦磷酸测序检测食品中鼠伤寒沙门氏菌[J].微生物学杂志, 2013, 33(6): 101-105.
[4] 王华, 刘斌. PCR技术在食品微生物检测中的应用[J]. 生物技术通报, 2010(2): 63-67.
[5] UYTTENDAELE M, RAJKOVIC A, CEUPPENS S, et al. PCR applications in food microbiology[J]. Encyclopedia Food Microbiology, 2014, 2: 1033-1041.
[6] BINET R, DEER D M, UHLFELDER S J. Rapid detection of Shigella and enteroinvasive Escherichia coli in produce enrichments by a conventional multiplex PCR assay[J]. Food Microbiology, 2014,40(6): 48-54.
[7] GARRIDO-MAESTU A, CHAPELA M J, VIEITES J M, et al. Application of real-time PCR to detect Listeria monocytogenes in a mussel processing industry: impact on control[J]. Food Control, 2014,46(12): 319-323.
[8] CHEN Xingxing, GAN Min, XU Hong, et al. Development of a rapid and sensitive quantum dot-based immunochromatographic strip by double labeling PCR products for detection of Staphylococcus aureus in food[J]. Food Control, 2014, 46(12): 225-232.
[9] IMANI F A A, IMAN I D, HOSSEINI D R, et al. Design of a multiplex PCR met hod for detection of toxigenic-pathogenic in vibrio cholera[J]. Asian Pacific Journal of Tropical Medicine, 2013, 6(2): 115-118.
[10] GORDILLO R, RODR☒GUEZ A, WERNING M L, et al. Quantification of viable Escherichia coli O157:H7 in meat products by duplex real-time PCR assays[J]. Meat Science, 2014, 96(2): 964-970.
[11] 许会会, 雷连成, 谢芳, 等. 沙门氏菌PCR检测方法的建立[J]. 中国畜牧兽医, 2010, 37(4): 94-97.
[12] 陈丽萍, 刘忠民, 陈芸, 等. 鸡白痢沙门氏菌PCR检测技术的建立与应用[J]. 畜牧兽医杂志, 2011, 30(6): 23-25; 27.
[13] MALORNY B, BUNGE C, HELMUTH R, et al. A real-time PCR for the detection of Salmonella enteritidis in poultry meat and consumption eggs[J]. Journal of Microbiological Methods, 2007,70(2): 245-251.
[14] RANI N, VAJPAYEE P, BHATTI S, et al. Quantification of Salmonella typhi in water and sediments by molecular-beacon based qPCR[J]. Ecotoxicology and Environmental Safety, 2014, 108(10): 58-64.
[15] FREITAS C G D, SANTANA A P, SILVA P H C D, et al. PCR multiplex for detection of Salmonella enteritidis, Typhi and Typhimurium and occurrence in poultry meat[J]. International Journal of Food Microbiology, 2010, 139(1/2): 15-22.
[16] NGAN G J Y, NG L M, LIN R T P, et al. Development of a novel multiplex PCR for the detection and differentiation of Salmonella enterica serovars Typhi and Paratyphi A[J]. Rersearch in Microbiology, 2010, 161(4): 243-248.
[17] 张亮, 刘磊, 豆艳丽, 等. 浓缩苹果汁中3 种污染菌多重PCR检测方法的建立[J]. 甘肃农业大学学报, 2012, 47(3): 124-128.
[18] CHANDRA M, CHENG P, RONDEAU G, et al. A single step multiplex PCR for identification of six diarrheagenic E. coli pathotypes and Salmonella[J]. International Journal of Medical Microbiology,2013, 303(4): 210-216.
[19] 牟恺, 陈智, 王春民, 等. 同时检测四种病原茵的PCR方法研究[J].山东农业大学学报: 自然科学版, 2010, 41(2): 253-257.
[20] 陈晓玲, 周玲艳, 温仕杰, 等. 沙门氏菌PCR快速检测技术研究[J].湖北农业科学, 2009, 48(3): 527-529.
[21] 欧阳本, 孙振, 祁克宗, 等. 鼠伤寒沙门氏菌毒力岛基因mgtC和sopB虫重PCR检测的研究[J]. 华北农学报, 2013, 28(3): 48-52.
[22] 荣策, 赵彤彤, 许龙岩, 等. 实时荧光PCR法检测鼠伤寒沙门氏菌[J].食品安全质质检测学报, 2012, 3(4): 300-305.
[23] 冯飞, 谢振文, 曾慕衡. 鼠伤寒沙门氏菌多重PCR检测方法的研究[J].中国生物工程杂志, 2011, 31(1): 65-69.
Establishment of PCR Detection Method for Salmonella typhimurium Based on ompc Gene
GONG Qiang, LI Zhanli, NIU Mingfu, WANG Hui, LIU Yongkang, QIN Cuili, SUN Xiaofei
(College of Food and Bioengineering, Henan University of Science and Technology, Luoyang 471023, China)
To establish a PCR detection method for Salmonella typhimurium, the ompc gene was amplified by PCR using the specific primers designed according to the sequence published in GenBank. The PCR reaction conditions were optimized based on the sensitivity and specificity of this method. The results showed that the 470 bp DNA frag ment was specifically amplified from Salmonella typhimurium and no DNA fragment was obtained from other common pathogenic bacteria in foods incl uding Escherichia coli, Staphylococcus aureus, Pseu domonas aeruginosa and Shigella. The sensitivity of this PCR method was 1 pg/μL. In conclusion, this experiment can lay a foundation for further exploration of rapid detection methods for Salmonella typhimurium in foods.
Salmonel l a typhimurium; omp c gene; PCR; detection method
Q939.93
A
1002-6630(2015)16-0251-04
10.7506/spkx1002-6630-201516048
2014-11-06
河南科技大学青年科学基金项目(2012QN002);河南科技大学SRTP项目(2014115)
宫强(1979—),男,副教授,博士,研究方向为病原微生物检测与防治。E-mail:gongqiang79@126.com

