转录调控研究的方法学进展
王俊阳,王为善,李肖,赵华,杨克迁
转录调控研究的方法学进展
王俊阳1,王为善2,李肖2,赵华1,杨克迁2
1 天津科技大学生物工程学院,天津 300457 2 中国科学院微生物研究所微生物资源前期开发国家重点实验室,北京 100101

王俊阳, 王为善, 李肖, 等. 转录调控研究的方法学进展. 生物工程学报, 2015, 31(8): 1141–1150.Wang JY, Wang WS, Li X, et al. Progress in transcriptional studies. Chin J Biotech, 2015, 31(8): 1141–1150.
在多个层次受到严格的控制,其中转录水平的调控是控制基因表达的重要环节之一。近几年,随着分子生物学研究方法的不断发展,转录调控的研究方法学也出现了新的变化。本文综述了基于作者所在课题组对链霉菌次级代谢调控研究过程中优化和改进的转录调控研究方法,如基于SYBR Gold的凝胶阻滞实验,基于荧光标记和毛细管电泳检测的足迹法,基于报告基因的直接调控关系分析等;同时也对研究调控蛋白和靶启动子相互作用的新方法进行了阐述,如表面等离子共振技术和等温滴定量热测定技术等。这些转录调控研究方法的优化和总结,能够帮助研究者开展相关研究。
转录调控,启动子,调控蛋白,相互作用
基因表达调控是现代分子生物学研究的中心课题之一。要了解生物的生长、分化规律,形态结构特征及代谢功能,就必须对基因表达调控的“适时”、“适量”控制机制进行深入研究,掌握了基因调控机制,就等于掌握了一把揭示生物学奥秘的钥匙。基因表达调控主要表现在转录水平、转录后水平、翻译水平和翻译后水平几个层次;其中,转录水平的调控是基因表达调控的关键,尤其在原核生物中[1],这也充分体现了基因表达调控的最经济控制原则。
反式作用因子和顺式作用元件的相互作用是转录水平基因表达调控的分子基础。反式作用因子主要指调控蛋白、sigma因子等;顺式作用元件主要指转录因子结合位点 (TFBS)、核心启动子等[2]。因此,转录水平调控的主要研究内容是发现目标启动子的反式作用因子、发现反式作用因子在目标启动子结合的顺式作用元件以及两者之间的相互作用。其主要步骤是:首先通过体内、体外实验确定直接调控关系;然后在分子层面上阐明相互作用的顺式元件在启动子上所处的位置[3];最后分析反式作用因子及其顺式作用元件相互作用的动力学特征。本实验室一直从事链霉菌次级代谢调控的研究,深刻认识到研究方法的进步能够直接加速研究的进展。因此,本文将主要综述本实验室对传统的转录调控研究方法进行的优化和改进,以期帮助研究者采用新的方法更好地开展相关研究。
1 体外相互作用研究
研究一个调控蛋白的调控作用,一般首先对其进行敲除和过表达,观察哪些基因的表达可能受到该调控蛋白的影响;然后进一步分析这一影响是敲除和过表达的直接效应还是间接效应。主要研究策略是用体外试验证明调控蛋白和受调控基因启动子之间是否有直接相互作用。采取的研究方法为凝胶阻滞实验 (Electrophoretic mobility shift assay,EMSA)[4]、表面等离子共振实验 (Surface plasmon resonance,SPR)[5]和等温滴定量热测定 (Isothermal titration calorimetry,ITC) 实验[6]等证实二者可以在体外特异结合。下面将分别对这些方法进行阐述。
1.1 基于SYBR Gold的凝胶阻滞 (EMSA) 实验
凝胶阻滞分析是用来研究启动子区域与调控蛋白的相互作用的经典方法,通常首先将放射性32P标记的DNA探针与纯化蛋白,或提取物中的蛋白混合物一起孵育,然后在非变性凝胶中分析该结合反应产物。与游离DNA探针相比,蛋白-DNA探针复合物的迁移率将降低,因此,将会在曝光后的电泳胶中观察到DNA条带的“阻滞”[4,7]。但是,由于很多实验室不具备使用放射性同位素的条件,且同位素对实验人员也可能会造成一定的伤害,因此该技术目前只能在国内条件比较成熟的实验室使用。为了避免同位素使用的这些不利因素,我们实验室开发了基于SYBR Gold (Life technologies) 染色的EMSA实验,并取得了良好的效果[8]。即DNA探针扩增后无需标记,直接和蛋白进行相互作用,进行非变性凝胶电泳分析,然后再将胶置于1/10 000的SYBR Gold稀释液中染色,SYBR Gold一旦与DNA探针结合,可以使荧光增强 1 000倍以上,其量子产率为0.6,最后用常规的300 nm的紫外成像仪,可清晰观察到金色条带 (图1A)。由于EMSA实验本身不需要昂贵的设备,我们又解决了它对同位素的依赖问题,目前该实验能够在常规实验室开展。
1.2 表面等离子共振 (SPR) 实验
表面等离子共振是利用入射光以临界角入射到两种不同折射率的介质界面(如玻璃表面的金属镀层) 时产生渐次波,渐次波与金属的自由电子耦合则发生表面等离子共振,从而使反射光在一定角度内减弱甚至完全消失的物理现象 (反射光完全消失时的入射光角度为共振角,也称SPR角)[9]。因此可以通过获取生物反应过程中样品的共振角的动态变化,得到DNA探针和调控蛋白之间相互作用的特异性信号[10],并且可以通过不同浓度的DNA探针和调控蛋白之间相互作用,得到它们相互结合的动力学参数。本实验室将委内瑞拉链霉菌启动子通过生物素固定在芯片上,用Biacore 3000分析了非典型应答调控蛋白JadR1对启动子的结合参数,并进一步分析了非典型应答调控蛋白的效应物杰多霉素 (Jadomycin) 对JadR1结合启动子的影响 (图1B)[11]。同时,SPR实验还能够方便地分析调控蛋白和小分子效应物的结合参数,我们用Biacore 3000分析了委内瑞拉链霉菌调控的蛋白JadR*对不同杰多霉素及其生物合成中间体的结合能力,最终发现中间体DHR是JadR*的最佳效应物[12]。SPR技术具有取代EMSA的显著优势,如目前GE推出的Biacore T200,具有EMSA无可比拟的检测灵敏度,能在极低样品量的情况下完成DNA探针和调控蛋白之间相互作用的动力学分析。
1.3 等温滴定量热测定 (ITC) 实验
当物质相互结合时,会产生热量或吸收热量。因此确定相互作用反应过程中热力学参数的变化可以对相互作用的动态过程进行定量的描述。等温滴定量热测定(Isothermal titration calorimetry,ITC) 技术是一种监测由结合成分的添加而起始的化学反应的热力学技术,目前,该技术已经成为鉴定生物分子间相互作用的金标准。由于它通过高灵敏度、高自动化的微量量热仪连续、准确地监测和记录一个变化过程的量热曲线,所以可以得到结合常数 (a)、结合位点数 (),结合焓 (Δ)、熵 (Δ)、恒压热容 (Δp) 等相关参数[13]。这些信息为我们提供了生物分子相互作用的真实写照[6]。本实验室用NANO-ITC 2G (TA instruments) 对一些调控蛋白结合靶启动子的结合参数进行了详细的表征[14];另外我们还用该设备对调控蛋白结合效应物小分子的相关参数进行了分析 (图1C),展示了外源抗生素JdB对天蓝色链霉菌ScbR2的结合情况,数据表明,它们近似1∶1的结合[14]。这充分表明ITC技术在相互作用相关参数的获取方面明显强于SPR和EMSA。但是ITC和SPR实验都需要昂贵的相关设备和专业的技术人员,另外ITC实验还需较高浓度的目的蛋白来获得可检测的反应热,这些在一定程度上限制了它们在转录调控研究中的使用。

图1 体外验证调控蛋白和DNA探针相互作用
2 体内调控关系研究
上述的体外研究方法,能够充分确定调控蛋白对靶启动子是直接调控还是间接调控。但是,展开以上体外实验的瓶颈是很多调控蛋白在大肠杆菌中不能以可溶的形式重组表达、纯化,或者表达量不足以开展体外实验。为了解决这一问题,我们建立了确定调控蛋白和靶启动子直接或者间接调控关系的体内研究方法。因为在体内环境中,细菌存在热激蛋白等能够保证重组蛋白的部分可溶性;同时,在异源宿主,还能够有效避免间接调控的影响。我们建立的在异源宿主中探索调控蛋白和靶启动子调控关系的方法见图2A[15],首先构建用靶启动子启动报告基因的质粒 (图2A的pOkasOlux),转入大肠杆菌后能够检测到发光 (图2B);然后用另一个与之兼容的质粒表达调控蛋白转入大肠杆菌后 (图2A的pScbR2),如果调控关系是直接效应,便能够观察到发光受到明显的激活或者抑制 (图2B)。另外,如果该调控蛋白能够响应效应物,还可以进一步添加效应物,分析添加不同浓度的效应物后,调控蛋白对启动子产生的不同调控效应。本实验室用这一表征方法分析天蓝色链霉菌ScbR2蛋白和JadR2蛋白对启动子的抑制情况 (图2B)[15],并且通过进一步添加效应物JdB,分析了JdB诱导ScbR2在启动子去除抑制的过程 (图2C)[14]。通过这种在异源宿主体内研究调控蛋白对靶启动子活性的影响,可分析出调控蛋白对靶启动子的直接效应,充分排除了其他间接的扰动。

图2 异源宿主体内调控蛋白和启动子之间的调控关系研究
3 确定作用位点的研究
确定了调控蛋白和靶启动子的直接调控关系后,需要进一步在分子层面详细阐释调控蛋白在靶启动子上的结合序列和该序列在启动子上所处的具体位置。而研究结合序列在启动子上的位置的必要前提是确定该启动子的转录起始位点。其中确定调控蛋白在靶启动子上的结合序列的常规技术为足迹法 (Footprinting)[4];确定转录起始位点的常规技术为S1 mapping实验、引物延伸实验和5¢-RACE实验[4]。由于5¢-RACE实验有常规的试剂盒,下面就本实验室改进的基于荧光标记和毛细管电泳分析的足迹法和S1 mapping进行阐述。
3.1 足迹法
Footprinting是确定DNA结合蛋白在DNA 分子上的结合序列的经典遗传学方法。其原理是:将调控蛋白结合的双链DNA探针片段进行单链单末端标记,与纯化的DNA结合蛋白在体外进行结合,然后加入DNaseⅠ对DNA-蛋白质复合物进行随机切割,产生一系列不同长度的DNA片段,其相邻片段只相差一个核苷酸 (图3A)。其中被DNA结合蛋白特异结合的序列由于空间位阻等多种效应,不能被DNaseⅠ切割,因此在放射自显影图谱上,DNA梯度条带在相应于DNA结合蛋白的结合区中断,从而形成一空白区,如果同时将Sanger ddNTP终止法测序反应产物进行PAGE和同位素曝光后,便可读出调控蛋白在DNA的结合序列[4,16-17]。
传统的经典同位素Footprinting实验需要经历标记同位素、做PAGE胶、电泳、显影和曝光等流程,需要大约两周甚至更长的时间;不仅实验周期长,且同位素的放射性危害比较大,因此限制了此方法的应用。目前,我们在国内首先对基于毛细管的Footprinting技术进行了探索:其原理和步骤与传统方法大致相同,只是将5¢端32P同位素标记替换为HEX、FAM等荧光末端标记;酶切后,将PAGE胶分离替换为毛细管分离;将显影曝光等步骤通过毛细管电泳的荧光检测器来实现。用ABI 3730xl毛细管电泳,只需要2 h就完成了结果的输出;另外由于ABI 3730xl是96通道批量模式,可以同时完成更多的结合条件的平行实验,增加了实验的成功率,同时也扩大了该方法的使用范围。
为了建立基于荧光标记和毛细管检测的Footprinting方法并验证该方法的可靠性,首先我们用该方法对已报道的γ-丁酸内酯受体ScbR对启动子的结合序列进行了分析。Takano等通过传统的同位素标记的Footprinting实验证实,ScbR能够特异结合在启动子的A位 (Site A) 和R位 (Site R) (图3B)[18]。我们异源表达了ScbR蛋白,合成了末端FAM荧光标记的探针,通过PCR得到321 bp的启动子区域。然后进行结合、酶切等实验,通过ABI3730xl测序仪的毛细管电泳分离和荧光检测器监测分析,结果如图3C所示,可以清晰地看到ScbR在启动子区域有两个结合位点;我们进一步通过毛细管电泳用内标法和ddNTP终止法测序确定ScbR结合的具体序列,内标采用ROX500 (ABI),结果如图4A所示,可以看出ScbR的结合位置R位和A位分别应该在22−52 bp和155−185 bp;图4B为ddNTP终止法对A位进行的末端定位,具体末端为155−185 bp,和ROX500内标标记的一致。以上结果也和文献报道的一致,所以可以得出结论:基于荧光标记和毛细管电泳的Footprinting结果和使用同位素标记得到的结果具有一样的可靠性。近几年,我们用该技术对天蓝色链霉菌ScbR2蛋白和委内瑞拉链霉菌JadR2蛋白分别在相应靶启动子的结合位点进行了成功解析[19];同时也将该技术提供给国内同行使用,获得了较好的研究结果[20-22]。
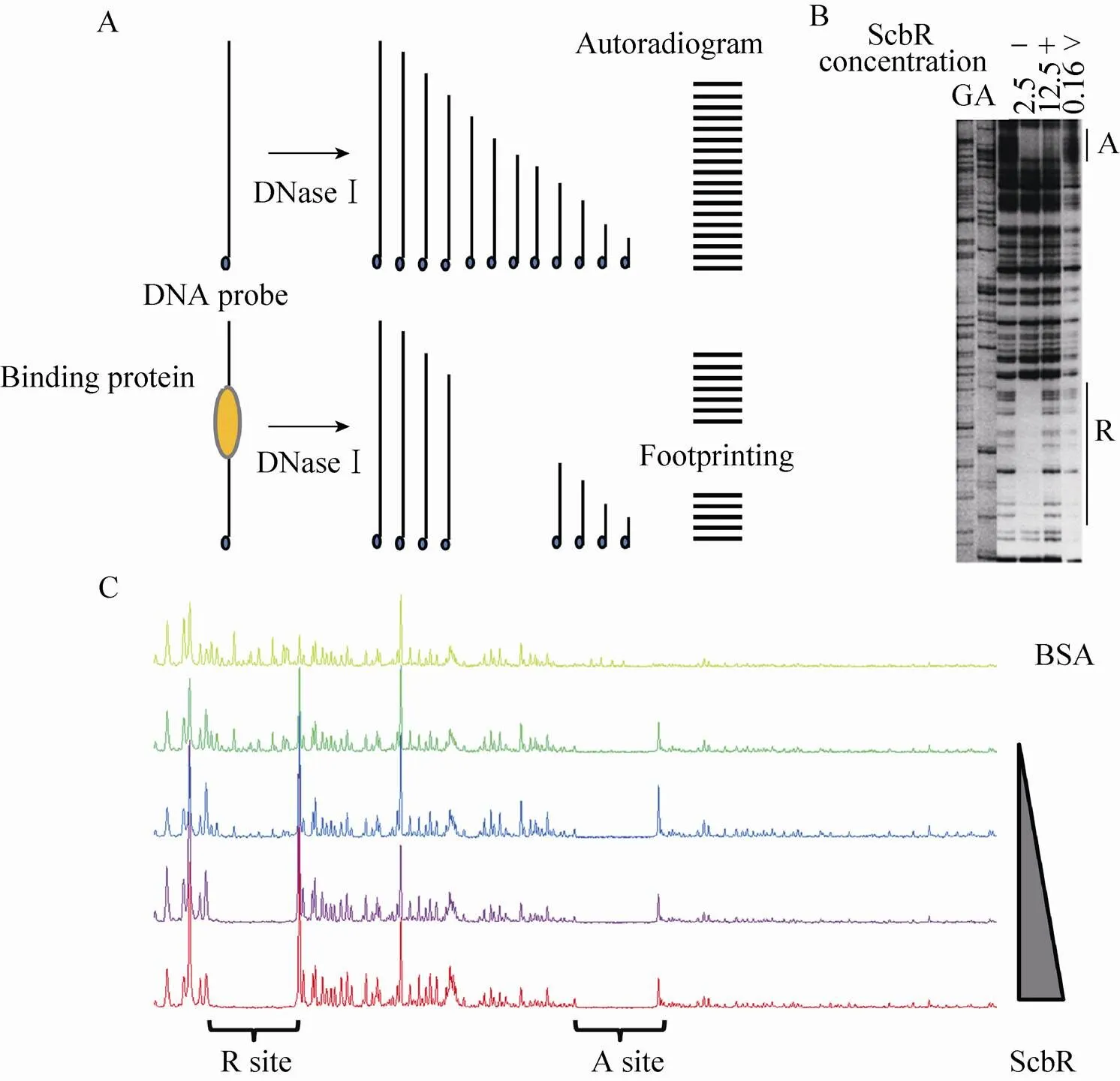
图3 足迹法确定DNA的结合序列
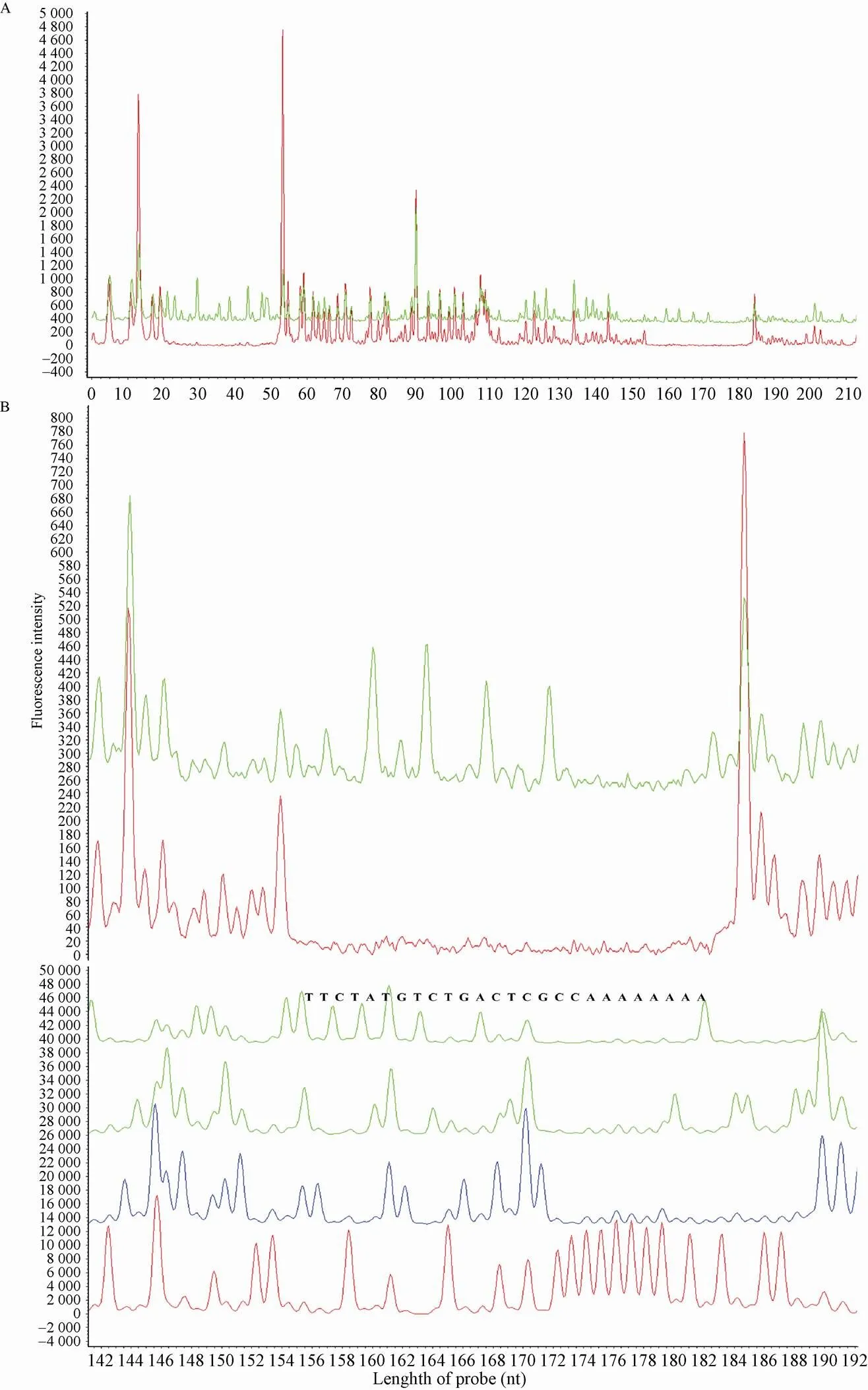
图4 毛细管电泳确定结合序列的具体位置
3.2 S1 mapping技术
确定启动子转录起始位点 (TSS) 最精确的方法是S1 mapping和引物延伸 (Primer extention) 实验,这两种方法都用到PAGE胶分离和同位素显影曝光[4,16]。同Footprinting实验一样,我们尝试了用毛细管的检测方法替换PAGE胶分离,用荧光标记替换同位素标记来改进S1 mapping实验,成功完成了对启动子的S1 mapping分析。首先制备启动子区域荧光探针,然后将其与委内瑞拉链霉菌总的RNA杂交、酶切后,用ABI 3730xl进行检测,毛细管电泳结果见图5,可以确定启动子的转录起始位点为基因的翻译起始密码子ATG上游114 bp的G[19];这一结果和我们用5¢-RACE试剂盒确定的转录起始位点完全一致。用荧光标记和毛细管电泳的方法进行的S1 mapping试验周期和灵敏性都明显优于5¢-RACE。目前,Chen等利用该技术完成了相关的工作[22]。

图5 基于毛细管的S1 mapping分析jadW1启动子的转录起始位点[19]
4 展望
转录水平的基因调控是一个极为复杂的过程,但主要需要在两个方面进行深入研究。一方面是同一层次之间相互联系与作用,如调控蛋白与靶启动子之间的关系,主要研究手段也就是我们文中提到的一些技术,这些技术协同应用能够充分阐明调控蛋白的调控机理。相信本实验室对这些技术的改进能够加速研究者对调控机理的认识;另一方面不同层次之间相互联系和动态变化,即用动态的调控网络重新阐释生理现象,相关研究手段有ChIP-chip/seq、genomic SELEX、细菌单杂交技术等[23-25]。本文主要对第一方面的调控蛋白与靶启动子的调控机理解析展开了系统综述,以期在国内对相关研究有所帮助。第二方面主要是在系统的组学水平对调控全局展开研究,目前我们也正在努力地开发方便高效的能够在实验水平上阐明调控网络的研究手段,即分别建立鉴定特定转录因子的全局调控靶点方法和发现调控特定启动子的所有转录因子的方法,以期促进在转录调控这一前沿领域开展系统研究工作。
[1] Ishihama A. Prokaryotic genome regulation: multifactor promoters, multitarget regulators and hierarchic networks. FEMS Microbiol Rev, 2010, 34(5): 628–645.
[2] Rodionov DA. Comparative genomic reconstruction of transcriptional regulatory networks in bacteria. Chem Rev, 2007, 107(8): 3467–3497.
[3] Liu G, Chater KF, Chandra G, et al. Molecular regulation of antibiotic biosynthesis inMicrobiol Mol Biol Rev, 2013, 77(1): 112–143.
[4] Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press, 1989: 20–85.
[5] Jayarajah CN, Thompson M. Signaling of transcriptional chemistry in the on-line detection Format. Biosens Bioelectron, 2002, 17: 159–171.
[6] Draczkowski P, Matosiuk D, Jozwiak K. Isothermal titration calorimetry in membrane protein research. J Pharm Biomed Anal, 2014, 87(18): 313–325.
[7] Weinmann AS, Bartley SM, Zhang T, et al. Use of chromatin immunoprecipitation to clone novel E2F target promoters. Mol Cell Biol, 2001, 21(20): 6820–6832.
[8] Wang J, Wang W, Wang L, et al. A novel role of ‘pseudo’ γ-butyrolactone receptors in controlling γ-butyrolactone biosynthesis in. Mol Microbiol, 2011 , 82(1): 236–250.
[9] Brockman JM, Nelson BP, Corn RM. Surface plasmon resonance imaging measurements of ultrathin organic films. Annu Rev Phys Chem, 2000, 51: 41–63.
[10] Forcada-Nadal A, Forchhammer K, Rubio V. SPR analysis of promoter binding ofPCC6803 transcription factors NtcA and CRP suggests cross-talk and sheds light on regulation by effector molecules. FEBS Lett, 2014, 588(14): 2270–2276.
[11] Wang L, Tian X, Wang J, et al. Autoregulation of antibiotic biosynthesis by binding of the end product to an atypical response regulator. Proc Natl Acad Sci USA, 2009, 106(21): 8617–8622.
[12] Zhang Y, Pan G, Zou Z, et al. JadR*-mediated feed-forward regulation of cofactor supply in jadomycin biosynthesis. Mol Microbiol, 2013, 90(4): 884–897.
[13] Shen JL. Application of differential scanning calorimetry for the studying of biological macromolecules. J Baoding Teachers College, 1999, 12(2): 34–42 (in Chinese).沈菊林. 差示扫描量热技术在生物大分子体系研究中的应用. 保定师专学校, 1999, 12(2): 34–42.
[14] Wang W, Ji J, Li X, et al. Angucyclines as signals modulate the behaviors of. Proc Natl Acad Sci USA, 2014, 111(15): 5688–5693.
[15] Xu G, Wang J, Wang L, et al. "Pseudo" gamma-butyrolactone receptors respond to antibiotic signals to coordinate antibiotic biosynthesis.J Biol Chem, 2010, 285(35): 27440–27448.
[16] Li R, Liu G, Xie Z, et al. PolY, a transcriptional regulator with ATPase activity, directly activates transcription ofR in polyoxin biosynthesis in. Mol Microbiol,2010, 75(2): 349–364.
[17] Pan Y, Liu G, Yang H, et al. The pleiotropic regulator AdpA-L directly controls the pathway-specific activator of nikkomycin biosynthesis inMol Microbiol,2009, 72(3): 710–723.
[18] Takano E, Chakraburtty R, Nihira T, et al. A complex role for the gamma-butyrolactone SCB1 In regulating antibiotic production inA3(2). Mol Microbiol, 2001, 41(5): 1015–1028.
[19] Wang J, Wang W, Wang L, et al. A novel role of pseudo'gamma-butyrolactone receptors in controlling gamma-butyrolactone biosynthesis inMol Microbiol, 2011, 82(1): 236–250.
[20] Guo J, Zhang X, Chen Z, et al. Two adjacent and similar TetR family transcriptional regulator genes, SAV577 and SAV576, co-regulate avermectin production in. PLoS ONE, 2014, 9(6): e99224.
[21] Luo S, Sun D, Zhu J, et al. An extracytoplasmic function sigma factor, sigma (25), differentially regulates avermectin and oligomycin biosynthesis in. Appl Microbiol Biotechnol, 2014, 98(16): 7097–7112.
[22] Chen DW, Zhang Y, Jiang CY, et al. Benzoate metabolism intermediate benzoyl coenzyme A affects gentisate pathway regulation inoni. Appl Environ Microbiol, 2014, 80(13): 4051–4062.
[23] Bouvet P. Determination of nucleic acid recognition sequences by SELEX. Methods Mol Biol, 2001, 148: 603–610.
[24] Johannes F, Wardenaar R, Colome-Tatche M, et al. Comparing genome-wide chromatin profiles using ChIP-chip or ChIP-seq. Bioinformatics, 2010, 26(8): 1000–1006.
[25] Meng X, Brodsky MH, Wolfe SA. A bacterial one-hybrid system for determining the DNA-binding specificity of transcription factors. Nat Biotechnol, 2005, 23(8): 988–994.
(本文责编 郝丽芳)
Progress in transcriptional studies
Junyang Wang1, Weishan Wang2, Xiao Li2, Hua Zhao1, and Keqian Yang2
1,,300457,2,,,100101,
Gene expression exhibits temporal and spatial patterns to response environmental changes and growth cycle. Gene expression is under strict control at different levels among which control at transcription level is the predominant mode, especially in prokaryotes. In this review, we summarized the new developments of methods used in transcriptional studies, including modifications and improvements of the classic methods, such as gel-shift assay, DNA foot printing, andreporter system. In addition, we introduced examples to apply new methods, such as surface plasmon resonance (SPR) and isothermal titration calorimetry (ITC) to characterize protein-DNA, ligand-protein, and ligand-protein-DNA interactions. The collection of these methods and their application could guide and accelerate relevant studies.
transcription regulation, promoter, regulatory proteins, interaction
10.13345/j.cjb.140486
October 15, 2014; Accepted:December 26, 2014
National Natural Science Foundation of China (Nos. 31400034, 31130001), National Basic Research Program of China (973 Program) (No. 2013CB734001).
Keqian Yang. Tel/Fax: +86-10-64807459; E-mail: yangkq@im.ac.cn Hua Zhao. Tel/Fax: +86-22-60601396; E-mail: zhaohua@tust.edu.cn
国家自然科学基金 (Nos. 31400034, 31130001), 国家重点基础研究发展计划 (973计划) (No. 2013CB734001) 资助。
2015-02-03
http://www.cnki.net/kcms/detail/11.1998.q.20150203.1625.004.html

