集胞藻PCC 6803中脂肪酸激活酶Slr1609互作蛋白的鉴定
徐乐,巫琴,晋虎,陈磊,张卫文
集胞藻PCC 6803中脂肪酸激活酶Slr1609互作蛋白的鉴定
徐乐*,巫琴*,晋虎,陈磊,张卫文
天津大学化工学院合成微生物学实验室,天津 300072

徐乐, 巫琴, 晋虎, 等. 集胞藻PCC 6803中脂肪酸激活酶Slr1609互作蛋白的鉴定. 生物工程学报, 2015, 31(8): 1194–1202.Xu L, Wu Q, Jin H, et al. Identification and characterization of partner proteins interacting with fatty acid activation enzyme Slr1609 in Synechocystis sp. PCC 6803. Chin J Biotech, 2015, 31(8): 1194–1202.
集胞藻中是编码脂肪酸激活酶的基因,对与其相关的重要功能伴侣蛋白进行研究,可以完善对脂肪酸合成模块的认识,为进一步通过合成生物学技术改造蓝细菌提供理论支持。本研究在集胞藻PCC 6803中建立了蛋白质复合体分析及鉴定技术:利用氯霉素抗性基因筛选,构建带有3×FLAG 标签的Slr1609突变株,通过RT−PCR优化重组蛋白表达条件;同时对突变株进行了Western blotting鉴定,以及利用Native-PAGE验证了蛋白质复合体的存在。最后,LC-MS/MS质谱鉴定获得了Slr1609蛋白复合体中的可能伴侣蛋白。
,脂肪酸,蛋白质复合体,蓝细菌
蓝细菌Cyanobacteria为最古老的光合自养型原核生物[1],分布广泛,适应能力非常强,可以在冰冻、缺氧、高温、干涸、高盐及强辐射等极端环境中生长[2-3]。近年来,通过合成生物学改造蓝细菌生产生物燃料,正受到来自学术界和企业界越来越多的关注;利用蓝细菌生产氢气、乙醇、乙烯[4]、异戊二烯、正丁醇[5]、脂肪酸和脂肪醇等多种生物燃料分子已取得了一定的成功[6]。Atsumi 等[7]通过定向工程改造聚球藻PCC 7942从CO2直接生产异丁醛和异丁醇,并过表达二磷酸核酮糖羧化酶(Rubisco) 大幅度提高了产物的产量。Lindberg等[8]用光诱导型启动子P在集胞藻PCC 6803中异源表达葛根 (kudzu) 的基因,使之可以产生异戊二烯。Dexter等[9]通过同源双交换的方法,将运动发酵单胞菌中的和基因整合到集胞藻PCC 6803染色体上,突变株可稳定生产生物乙醇,且730为1的菌量日产乙醇5.2 mmol/L。集胞藻sp. PCC 6803是一种单细胞嗜中温蓝细菌,于1996年完成基因组测序[10]。其具有遗传背景清楚、生长快速、光合作用效率高[11]、培养条件简单、不产毒素、具有天然外源DNA转化系统[12]等特点,是研究光合作用以及合成生物学改造研究的重要模式菌株和底盘细胞[13]。
蓝细菌的脂肪酸族生物燃料合成离不开其脂肪酸的代谢途径。脂肪酸合成途径属于Ⅱ型脂肪酸合成途径[14]。集胞藻PCC 6803在正常生理状态下,细胞内存在大量自由脂肪酸,而这些自由脂肪酸需要被脂肪酸激活酶激活成为脂酰CoA或脂酰ACP,才能被下游的膜脂合成途径、脂肪醇和脂肪烃等代谢途径所利用[15-16]。在近期的工作中,Liu等[17]使用基因工程和代谢工程技术改造集胞藻PCC 6803使其生产C10−C18脂肪酸,最高产量达 (197±14) mg/L。同时使用CO2诱导型启动子,构建CO2诱导融膜系统,突变株SD239回收的脂肪酸产量达到36.1×10–12mg/cell[18]。这一研究工作表明工程蓝细菌生产脂肪族类液体生物燃料的产量提高还有很大的空间。
蛋白质是细胞代谢活动的主要承担者,它们通常通过形成蛋白质复合体来行使功能,所以脂肪酸合成相关酶复合体的研究对于了解细胞代谢调控机理等具有重要的意义。复合体的纯化分离现阶段主要有免疫共沉淀、标签标记亲和纯化、GST沉降技术和串联亲和纯化等[19]。Boehm等[20]用BN-PAFE验证了FPLC对集胞藻PCC 6803中光合系统Ⅱ的分离效果。Ma等[21]先用Native-PAGE分离出NDH-1复合体,再通过第二维的SDS-PAGE分离出8个条带。Brown 等[22]利用串联亲和纯化技术(TAP) 在集胞藻PCC 6803纯化出少量的脂肪酸合成酶(FAS) 复合体。以上研究成果证明通过蛋白质复合体纯化分离及鉴定技术可以为蓝细菌研究提供重要的生物学发现。
在早期研究中,Von Berlepsch等[15]将基因敲除,利用同位素示踪法对集胞藻PCC 6803的脂肪酸代谢途径进行详细分析,最后推测是编码蓝细菌中唯一的脂肪酸激活酶的基因。近期Gao等[23]通过对这一基因进行亚细胞定位,发现既存在于细胞膜系统中,也存在于细胞质中。本研究针对编码脂肪酸激活酶的基因[23]展开互作蛋白的鉴定和分析研究,以期完善对脂肪酸合成模块的认识,为进一步通过合成生物学技术改造蓝细菌提供理论支持。
1 材料与方法
1.1 菌株、质粒和培养条件
集胞藻sp. PCC 6803从American Type Culture Collection购买,培养于BG11培养基,30 ℃,50 µmol/(m2·s) 和130 r/min条件摇瓶培养[24]。
含氯霉素抗性质粒pACYC184的大肠杆菌培养于LB培养基,37 ℃、200 r/min摇瓶中培养12−24 h。
1.2 突变株的构建
在的C端插入3×FLAG标签,并在其后插入氯霉素抗性基因。从Cyanobase数据库获取基因序列(http://genome.microbedb. jp/cyanobase/Synechocystis),并用Primer 5设计引物,其中R1含有3×FLAG标签(表1)。首先用表1中的引物分别PCR扩增出目标基因C端插入3×FLAG标签的上游片段,氯霉素抗性基因的中游片段和基因的下游片段,3条片段的长度都约为1 kb。再以F1和R3为引物,融合PCR扩增出大小约为3 kb的线性DNA片段(图1)。PCR反应条件如下:98 ℃预变性30 s;98 ℃变性10 s,72 ℃退火10 s,72 ℃延伸1.5 min,30个循环;最后72 ℃延伸 5 min,4 ℃保存。将3 kb线性DNA片段转化到集胞藻PCC 6803中,筛选抗性菌落,同时进行菌落PCR验证。
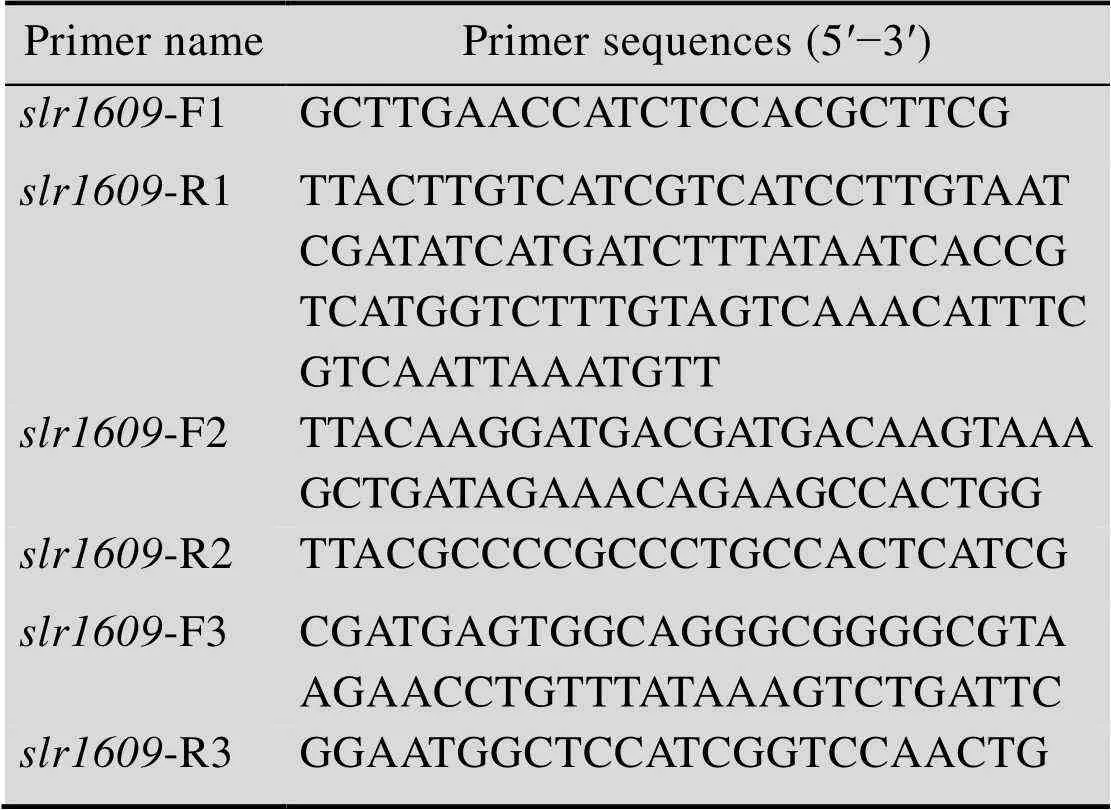
表1 融合PCR所用引物

图1 用融合PCR方法插入3×FLAG标签原理
1.3 RT-qPCR (Reverse transcription quantitative PCR) 分析
用RT-PCR在转录水平上检测不同培养时间下的基因表达,用作为内参基因进行校正。引物见表2。取3 d和5 d两个时间点,每个时间点做3个平行样,用ZR RNA MicroPrep™ RNA Kit (ZYMO) 提取总RNA,再用SuperScript®VILO™ cDNA Synthesis Kit (Invitrogen) 将总RNA逆转录为cDNA,最后用Power SYBR®Green RT−PCR Reagents Kit (ABI)配制实时荧光定量PCR反应体系进行反应,PCR程序如下:95 ℃预变性10 min;95 ℃变性15 s,60 ℃退火/延伸1 min,40个循环。
1.4 蛋白质的提取及纯化
1.4.1 蛋白质的提取
采用珠磨法破碎细胞,取1 L培养5 d的集胞藻PCC 6803菌液,4 ℃、7 500 r/min离心 15 min,弃上清,并均匀分别转入10个1.5 mL离心管中。加入与菌体等体积的玻璃珠(直径0.1 mm),再加入100 µL裂解液(20 mmol/L 4-羟乙基哌嗪乙磺酸(HEPES),pH 7.4,10 mmol/L MgCl2,5 mmol/L CaCl2,20%甘油,1% β-DM),PMSF终浓度为1 mmol/L[25]重悬。放入样本快速破碎匀浆仪(天津欧诺) 中进行破碎(30 s,2 min冰浴,4 500 r/min,6个循环),离心取上清混在一起记为蛋白粗提液。
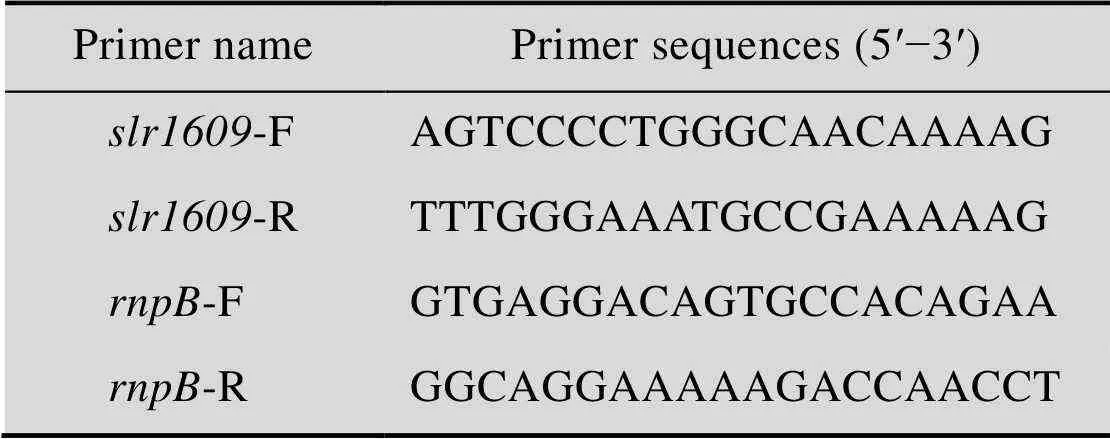
表2 RT-PCR引物
1.4.2 蛋白质的纯化
Gel预处理:1) 将ANTI-FLAG M2 Affinity Gel置于冰上融化后,取10 μL于离心管中,4 ℃、8 000×离心30 s,去上清;2) 向离心管中加入1 mL裂解液,轻轻颠倒摇动清洗Gel,4 ℃、 8 000×离心30 s,去上清;3) 重复清洗1次。
蛋白质结合:1) 向Gel中加入1 mL蛋白质粗提物;2) 4 ℃轻柔振荡过夜,然后4 ℃、8 000×离心30 s,去上清。
Gel清洗:1) 向Gel中加入1 mL 裂解液,轻轻颠倒摇动清洗Gel,4 ℃、8 000×离心30 s,去上清;2) 重复清洗2次。
蛋白质洗脱:1) 配制洗脱液:100 μL 蛋白纯化洗脱缓冲液 (20 mmol/L HEPES,pH 7.4,10 mmol/L MgCl2,5 mmol/L CaCl2,150 mmol/L NaCl,20%甘油) 中加入6 μL 5 μg/μL 3×FLAG peptide溶液,终浓度为300 ng/μL。2) 将洗脱液加入Gel中,在冰上轻柔振荡1 h,在4 ℃下,8 000×离心30 s,上清即为纯化的蛋白。
以上过程均在冰上进行以防抗体失效。
1.5 Western blotting检测纯化产物
根据标准的方法[26]进行Western blotting。蛋白样品的上样量为20 μL (约300 ng),用SDS-PAGE (12%) 或Native-PAGE (5%) 进行分离,通过电转移方法将蛋白转移至PVDF膜上,然后进行免疫反应。
用鼠抗FLAG抗体和辣根过氧化物酶标记的羊抗鼠IgG分别作为一抗和二抗进行免疫印迹检测,ECL检测试剂盒显色后用Chemi Doc XRS+凝胶成像仪曝光成像。
1.6 蛋白质复合体成分的质谱鉴定
取20 µL纯化后蛋白质溶液,进行Native-PAGE并进行银染(Sigma)。切下所对应条带即为全细胞中目标蛋白质复合体样品。将蛋白样品酶解处理,进行质谱检测(华大蛋白质研发中心有限公司)。对质谱数据进行分析,利用数据分析软件即Data Analysis Software软件标峰后,将原始文件转换成mgf格式,再用Mascot search engine version 2.3.01将数据与已建立的集胞藻PCC 6803全蛋白数据库(来自于NCBI) 进行数据搜索比对。其中,Mascot程序使用基于概率的MOWSE算法、前体肽离子/信息和MS-MS肽片段离子数据来鉴定蛋白质。
搜索的参数设置如下:Database: PCC 6803;Taxonomy: All entries;Enzyme: Trypsin;Missed missed cleavage: 1;Fixed modification: Carbamidomethyl(C);Quantitation method: None;Varied modification: Oxidation(M), Gln->pyro-Glu(N-term Q);Peptide mass tolerance: ±0.1 Da;Fragment Mass Tolerance: ±0.1 Da;Mass values: Monoisotopic;Instrument type: ESI-QUAD-TOF;
进行数据搜索时,蛋白鉴定的Mascot score值大于14 (<0.05),序列覆盖度(Sequence coverage) >1%则认为是阳性匹配。
2 结果与分析
2.1 slr1609突变株的构建
集胞藻PCC 6803经过转化后,从平板上挑取单菌落进行菌落PCR验证,由于集胞藻PCC 6803含有多条染色体,所以需要检验是否单菌落的细胞中所有染色体均带上3×FLAG标签以及氯霉素抗性基因。菌落PCR利用扩增上、下游同源臂的特异性引物F1和R3进行,分别以突变株和野生株为扩增模板进行PCR,然后通过琼脂糖凝胶电泳来检测扩增片段大小是否正确。由于突变株中插入了3×FLAG标签以及氯霉素抗性基因,与集胞藻PCC 6803野生株相比大了1 kb左右,若替换完全,突变株菌落PCR应当只得到一条3 kb的条带,而野生株只有2 kb左右的片段,琼脂糖凝胶电泳检测结果见图2,可知突变型菌株已经完全替换。
2.2 突变株培养时间的优化
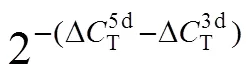

图2 菌落PCR

表3 slr1609和rnpB的RT-PCR CT值
RNA preparation, reverse transcription and real-time PCR were performed as described in section 1.3.

图3 不同培养时间点slr1609的相对表达量
2.3 带3×FLAG标签目的蛋白的表达、纯化及Western blotting分析
根据UniProt数据库(http://www.uniprot.org),编码的脂肪酸激活酶的蛋白质分子量为77.7 kDa,而3×FLAG标签的大小为2.7 kDa,可以计算出融合蛋白大小约为80.4 kDa。取 200 mL培养5 d的突变株和野生株,用BugBuster Master Mix (Novagen) 提取蛋白质,并通过Western blotting (12%分离胶) 初步验证3×FLAG标签的表达。从图4可以看出,突变株中的C端插入3×FLAG标签后仍能够正常表达,并且蛋白分子量大小也正确,而在集胞藻PCC 6803野生株检测不到3×FLAG信号,在蛋白水平表明突变株的正确构建和3×FLAG标签具有较强的特异性,可以用于蛋白质复合体的分析。
2.4 检测蛋白质复合体纯化效果
取1 L培养5 d的突变株培养液,按照上述方法纯化突变株中的蛋白质。取20 μL突变株纯化后蛋白质溶液进行Native-PAGE并进行银染(图5A),同时蛋白溶液稀释10倍后等量进行Western blotting分析,以检测融合蛋白是否以蛋白质复合体的形式被纯化出来,分离出的复合体以备质谱分析。图5B中的Western blotting结果可以看出分子量大于170 kDa的位置显示明显的FLAG信号;图5A中可以看出,突变株纯化后的蛋白质在分子量大于170 kDa的位置同样有蛋白质存在,表明细胞中Slr1609融合蛋白以蛋白质复合体的形式被纯化和分离出来,并且可能因为一些蛋白的离体产生拖尾现象。

图4 Western blotting验证3×FLAG标签的插入与表达
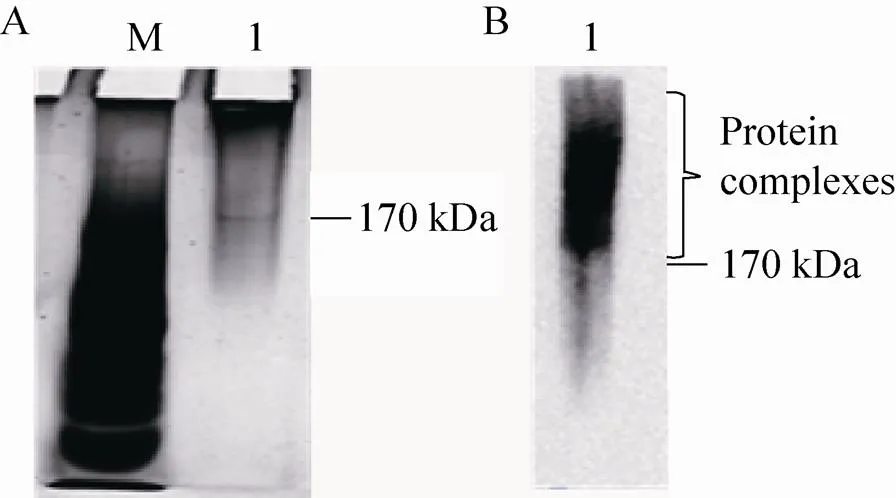
图5 Native-PAGE检测蛋白质复合体纯化效果
2.5 质谱结果分析
将上述银染后的蛋白带切下后进行质谱检测,再把NanoLC-MS/MS结果提交至集胞藻PCC 6803全蛋白数据库检索,在肽段容差 ±0.1 Da、<0.05的检索条件下进行数据搜索比对,根据蛋白质丰度、序列匹配程度与覆盖率、检测到肽段数量及可信度、同一肽段检测次数等指标,对鉴定出的结果进行综合评分,共鉴定到5个阳性结果 (表4)。其中,Slr1834鉴定得分为30,有3个肽段归属于同一蛋白质,2个肽段可信度超过阈值分数 (<0.05),覆盖率为4%;Slr1783鉴定得分为21,鉴定到1个可信度超过阈值分数 (<0.05) 的肽段,覆盖率为6%;Slr0415鉴定得分为19,有3个肽段归属于同一蛋白质,1个肽段可信度超过阈值分数 (<0.05),覆盖率为3%;Sll0350鉴定得分为19,有3个肽段归属于同一蛋白质,1个肽段可信度超过阈值分数 (<0.05),覆盖率为1%;Slr1609鉴定得分为18,鉴定到1个可信度超过阈值分数 (<0.05) 的肽段,覆盖率为2%。
从表中可以看出LC-MS/MS鉴定得到的各个蛋白质的功能。Slr1834是光系统Ⅰ中P700-叶绿素a复合体上的脱辅基蛋白亚基,属于能量代谢中光合作用的功能蛋白;Slr1783是双组分信号系统的应答调控子NarL的亚基,属于信号转导调控功能蛋白;Slr0415是ATP结合盒转运蛋白,属于运载功能蛋白;Sll0350是未知功能蛋白;Slr1609是我们的目标融合蛋白,即脂肪酸激活酶。此结果进一步确认了我们的纯化和Native-PAGE系统正确地将Slr1609及其蛋白质复合体的蛋白进行了成功分离。
通过对上述蛋白质的功能分析,Slr1834、Slr1783和Slr0415 3个蛋白均为细胞膜中常见的参与光合作用、信号转导、运载的功能蛋白,可能是由于这些蛋白的丰度较高从而非特异性结合在纯化柱上,造成亲和纯化不可避免的假阳性。质谱还鉴定出一个未知功能蛋白Sll0350,利用SMART (http://smart.embl-heidelberg.de) 工具对该蛋白的结构域进行分析,并未发现跨膜结构域,可以排除膜蛋白的可能性,而鉴于此蛋白可能与脂肪酸激活酶Slr1609形成蛋白质复合体,因此我们认为该蛋白有可能与脂肪酸代谢有关,但仍需进一步的实验(例如,基因敲除等) 验证来提供更有力的证据。

表4 LC-ESI-MS/MS检测蛋白质复合体结果
3 结论
目前对多不饱和脂肪酸的代谢和调控途径的认识十分有限,限制了将这类高效代谢和调控模块用于大幅度提高生物产品。集胞藻PCC 6803中脂肪酸合成相关的酶复合物研究,对大规模生成多不饱和脂肪酸这种重要生物产品有重要意义。迄今为止,我们建立并优化了集胞藻PCC 6803中高通量快速的基因突变体系:经过优化过的融合PCR方法,从拿到引物到获得基因突变所需DNA片段并进行转化,仅需半天时间。利用RT-PCR技术通过鉴定目标基因转录水平,优化了重组蛋白表达条件。对3×FLAG标记的突变株进行了Western blotting鉴定。利用LC-MS/MS鉴定了重组突变株目标蛋白质复合体成分,质谱结果表明,蛋白质复合体中能够检测到目标蛋白Slr1609,证明3×FLAG标签构建正确且能正常表达纯化,整个重组突变-亲和纯化技术是可行的。质谱结果还鉴定出一个未知功能蛋白Sll0350,可能是与脂肪酸代谢有关的蛋白。下一步实验中我们将利用分子生物学(基因敲除、基因过表达等) 和组学手段(基因组、转录组和代谢组等) 对有效候选伴侣蛋白进行功能研究与鉴定,进而解析集胞藻PCC 6803中多不饱和脂肪酸相关代谢和调控途径。
[1] Badger MR, Price GD. CO2concentrating mechanisms in cyanobacteria: molecular components, their diversity and evolution. J Exp Bot, 2003, 54(383): 609–622.
[2] Kehr JC, Gatte Picchi D, Dittmann E. Natural product biosyntheses in cyanobacteria: a treasure trove of unique enzymes. Beilstein J Org Chem, 2011, 7: 1622–1635.
[3] Dantas EW, Moura AN, Bittencourt-Oliveira MC. Cyanobacterial blooms in stratified and destratified eutrophic reservoirs in semi-arid region of Brazil. An Acad Bras Cienc, 2011, 83(4): 1327–1338.
[4] Ungerer J, Tao L, Davis M, et al. Sustained photosynthetic conversion of CO2to ethylene in recombinant cyanobacterium6803. Energ Environ Sci, 2012, 5(10): 8998–9006.
[5] Lan EI, Liao JC. ATP drives direct photosynthetic production of 1-butanol in cyanobacteria. Proc Natl Acad Sci USA, 2012, 109(16): 6018–6023.
[6] Lu X. A perspective: photosynthetic production of fatty acid-based biofuels in genetically engineered cyanobacteria. Biotechnol Adv, 2010, 28(6): 742–746.
[7] Atsumi S, Higashide W, Liao JC. Direct photosynthetic recycling of carbon dioxide to isobutyraldehyde. Nat Biotechnol, 2009, 27 (12): 1177–1180.
[8] Lindberg P, Park S, Melis A. Engineering a platform for photosynthetic isoprene production in cyanobacteria, usingas the model organism. Metab Eng, 2010, 12(1): 70–79.
[9] Dexter J, Fu PC. Metabolic engineering of cyanobacteria for ethanol production. Energ Environ Sci, 2009, 2(8): 857–864.
[10] Ikeuchi M. Complete genome sequence of a cyanobacteriumsp. PCC 6803, the oxygenic photosynthetic prokaryote. Tanpakushitsu Kakusan Koso, 1996, 41(16): 2579–2583.
[11] Wu Q, Chen L, Wang JX, et al. Engineering photosynthetic cyanobacterial chassis: a review. Chin J Biotech, 2013, 29(8): 1086–1099 (in Chinese). 巫琴, 陈磊, 王江新, 等. 应用合成生物学策略优化光合蓝细菌底盘.生物工程学报, 2013, 29(8): 1086–1099.
[12] Kufryk Gl, Sachet M, Schmetterer G, et al. Transformation of the cyanobacteriumsp. PCC 6803 as a tool for genetic mapping: optimization of efficiency. FEMS Microbiol Lett, 2002, 206(2): 215–219.
[13] Lü J, Sun HL, He H, et al. Synthetic biology: its applications in biotechnology. Prog Biochem Biophys, 2012, 39(2): 105–118 (in Chinese). 吕静, 孙洪磊, 何皓, 等. 合成生物学及其在生物技术中的应用进展. 生物化学与生物物理进展, 2012, 39(2): 105–118.
[14] White SW, Zheng J, Zhang YM, et al. The structural biology of type II fatty acid biosynthesis. Annu Rev Biochem, 2005, 74: 791–831.
[15] Von Berlepsch S, Kunz HH, Brodesser S, et al. The Acyl-acyl carrier protein synthetase fromsp. PCC 6803 mediates fatty acid import. Plant Physiol, 2012, 159(2): 606–617.
[16] Kaczmarzyk D, Fulda M. Fatty acid activation in cyanobacteria mediated by acyl-acyl carrier protein synthetase enables fatty acid recycling. Plant Physiol, 2010, 152(3): 1598–1610.
[17] Liu X, Sheng J, Curtiss R 3rd. Fatty acid production in genetically modified cyanobacteria. Proc Natl Acad Sci USA, 2011, 108 (17): 6899–6904.
[18] Liu X, Fallon S, Sheng J, et al. CO2-limitation-inducible green recovery of fatty acids from cyanobacterial biomass. Proc Natl Acad Sci USA, 2011, 108(17): 6905–6908.
[19] Dziembowski A, Séraphin B. Recent developments in the analysis of protein complexes. FEBS Lett, 2004, 556(1/3): 1–6.
[20] Boehm M, Romero E, Reisinger V, et al. Investigating the early stages of photosystem II assembly insp. PCC 6803: isolation of CP47 and CP43 complexes. J Biol Chem, 2011, 286(17): 14812–14819.
[21] Ma W, Deng Y, Ogawa T, et al. Active NDH-1 complexes from the cyanobacteriumsp.strain PCC 6803. Plant Cell Physiol, 2006, 47(10): 1432–1436.
[22] Brown AP, Affleck V, Fawcett T, et al. Tandem affinity purification tagging of fatty acid biosynthetic enzymes insp. PCC 6803 and. J Exp Bot, 2006, 57(7): 1563–1571.
[23] Gao Q, Tan X, Lu X. Enzymatic and physiological characterization of fatty acid activation insp. PCC 6803. J Basic Microb, 2013, 53(10): 848–855.
[24] Stanier, RY, Kunisawa R, Mandel M, et al. Purification and properties of unicellular blue-green algae (order). Bacteriol Rev, 1971, 35(2): 171–205.
[25] Sobotka R, Tichy M, Wilde A, et al. Functional assignments for the carboxyl-terminal domains of the ferrochelatase fromsp. PCC 6803: the CAB domain plays a regulatory role, and region II is essential for catalysis. Plant Physiol, 2011, 155(4): 1735–1747.
[26] Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. 3rd ed. New York: Cold Spring Harbor Laboratory Press, 2001.
[27] Tian X, Chen L, Wang J, et al. Quantitative proteomics reveals dynamic responses ofsp. PCC 6803 to next-generation biofuel butanol. J Proteomics, 2013, 78: 326–345.
(本文责编 郝丽芳)
Identification and characterization of partner proteins interacting with fatty acid activation enzyme Slr1609 insp. PCC 6803
Le Xu*, Qin Wu*, Hu Jin, Lei Chen, and Weiwen Zhang
Laboratory of Synthetic Microbiology, School of Chemical Engineering and Technology, Tianjin University, Tianjin 300072, China
To understand molecular modules related to polyunsaturated fatty acids (PUFA) synthesis and eventually produce PUFA at high efficiency, we developed a protein complex analysis technology insp. PCC 6803, and applied it to identify possible partner proteins interacting with the key enzymes that catalyze PUFA biosynthesis. We first constructed a recombinant expression of protein ofencoding the fatty acid activation enzyme, by fusing 3×FLAG tag with the target protein. Then we verified its expression by Western blotting targeting 3×FLAG tag. To maximize purification of Slr1609 protein complex, we optimized the protein expression conditions of Slr1609 inin a 5 L fermenter by monitoring itsgene expression using RT-qPCR. The purification of the Slr1609 protein complexes was demonstrated by a Native-PAGE analysis. Finally, LC-MS/MS proteomic analysis allowed identification of the possible partner proteins interacting with Slr1609.
, fatty acid, protein complex, cyanobacterium
10.13345/j.cjb.140492
October 20, 2014;Accepted:January 22, 2015
National Basic Research Program of China (973 Program) (Nos. 2012CB721101, 2011CBA00803).
Lei Chen. Tel: +82-22-27406364; Fax: +86-22-27403389; E-mail: lchen@tju.edu.cn
*These authors contributed equally to this study.
国家重点基础研究发展计划 (973计划) (Nos. 2012CB721101, 2011CBA00803) 资助。
2015-03-25
http://www.cnki.net/kcms/detail/11.1998.Q.20150325.1122.003.html

