传统发酵食品中乳酸菌生态演替研究进展*
缪璐欢,白凤翎,励建荣
(渤海大学食品科学研究院,辽宁省食品安全重点实验室,“食品贮藏加工及质量安全控制工程技术研究中心”辽宁省高校重大科技平台,辽宁锦州,121013)
传统乳酸发酵食品是以天然乳酸菌为主体微生物发酵原料中的碳水化合物和蛋白质等大分子物质,形成以乳酸为主要代谢产物的具有酸、香、鲜风味的食品,主要包括酸奶、干酪和开菲尔等发酵乳制品,泡菜、酸菜、橄榄等发酵果蔬制品,火腿、香肠等发酵肉制品以及酵头发酵面制品等。传统乳酸发酵食品中具有丰富而庞杂的微生物区系,通过微生物种群与基质的相互作用,不仅使食品获得了良好的风味,改善食品的品质,提高食品的营养价值,而且提升了食品的安全性,延长产品的保藏期。
乳酸菌是传统乳酸发酵食品的主体微生物类群,分为同型和异型乳酸发酵乳酸菌,前者降解碳水化合物后产物为乳酸,后者除产生乳酸外,产生醇类、酮类、醛类等挥发性化合物,赋予发酵食品特有的口味和香味。同时,乳酸菌通过生态位竞争,形成酸性环境和产生拮抗性代谢产物,较好地控制了产品中的腐败和致病微生物。因此,传统乳酸发酵食品中乳酸菌对产品的品质和安全性具有十分突出的作用。本文对传统乳酸发酵食品发酵过程中乳酸菌的发生、发展和演替过程和分析方法进行综述,分析各种传统乳酸发酵食品中乳酸菌与基质之间、乳酸菌与他种微生物之间、乳酸菌与乳酸菌之间的相互作用关系,进而阐明乳酸菌在传统发酵食品中的地位和作用,为实现传统乳酸发酵食品的技术改造,开发乳酸菌资源提供借鉴与参考。
1 传统发酵食品中乳酸菌的生态演替
传统乳酸发酵食品充分体现了乳酸菌多样性和多变性的生态学特点,乳酸菌的丰度(种群数量)和均匀度(种群量的大小)与演替过程主要受发酵原料、时间、温度、地理环境以及其他微生物等因素的影响。
1.1 韩国泡菜
传统韩国泡菜(kimchi)是以蔬菜为主要原料经天然微生物发酵而成的食品,蔬菜中可溶性物质和部分浸出物为乳酸菌为主体的微生物生长提供营养需求,乳酸菌的生长较好地抑制了腐败菌的繁殖。根据酸度变化,一般可将发酵过程分为初始阶段(酸度<0.2)、未成熟阶段(酸度0.2~0.4)、成熟阶段(酸度0.4 ~0.9)和过熟阶段(酸度 >0.9)[1]。
Lee等[2]应用PCR-DGGE技术研究发现,传统韩国泡菜中主要乳酸菌类群为Weissella confusa、Leuconostoc citreum、Lactobacillus sakei和 Lb.curvatus。Cho等[3]应用多重 PCR技术研究发现,Leu.mesenteroides是泡菜未成熟期和成熟期的优势菌,Lb.sakei从初始发酵阶段到过发酵阶段一直存在。Park等[4]研究泡菜整个发酵过程中乳酸菌菌群为Weissella、Lactobacillus、Pediococcus 和 Leuconostoc。Jung等[5]应用焦磷酸测序技术分析表明,在不考虑泡菜类型和发酵剂的条件下,泡菜的主要乳酸菌类群为 Leuconostoc、Lactobacillus 和 Weissella,其 中 Leu-conostoc数量最多,Lactobacillus和Weissella次之。综上研究结果表明,虽然不同品种和地域的传统韩国泡菜产品中的乳酸菌类群构成不同,但主要乳酸菌类群为 Leuconostoc、Lactobacillus、Weissella 和 Pediococcus。
从韩国泡菜发酵过程中乳酸菌的菌相演替来看,发酵早期的优势菌群为Leuconostoc和 Weissella,随后Lb.sakei替代 Leuconostoc和Weissella占优势,到发酵后期,主体乳酸菌为Lb.plantarum。韩国泡菜中乳酸菌来源于附着在蔬菜表面上的天然微生物类群,在发酵早期,pH在5.64~4.27,相对较高,乳酸菌主要为Leuconostoc、Lactobacillus和 Weissella[6-7]。Jeong 等研究发现,Leuconostoc、Lactobacillus和 Weissella是发酵开始阶段的乳酸菌类群,可能由于 Leuconostoc和Weissella是原料葱和蒜中的优势乳酸菌[8]。Cho等[9]研究发现,15℃预发酵2 d,然后在-1℃下进行主发酵,第一发酵阶段泡菜中主要优势菌为 Leu.citreum 和 Leu.gasicomitatum,第 二 发 酵 阶 段W.koreensis占主要优势。到发酵后期,当pH低于4.0 时,Hong 等[6]研 究 发 现,乳 酸 菌 主 要 为Lb.plantarum,可能是由于泡菜中Leuconostoc、Weissella和Pediococcus耐酸性较弱的缘故。
1.2 酵头
酵头是东西方各种发酵面制品(面包和馒头等)的天然母发酵剂,按工艺酵头分为I、II和III三种类型。I型为传统酵头,需经每天翻新保持微生物处于活性状态,II型为高温条件下制作含有多种微生物的酵头,III型酵头为含有乳酸菌的干燥粉状物[10]。酵头中具有丰富多样的微生态体系,酵母和乳酸菌是主要的微生物类群。其中,乳酸菌对面团结构和风味形成具有十分重要的作用,同时还具有提高营养价值和延长保质期的作用。
面团是营养丰富而全面的生态系统,微生物发酵作用将蛋白质、糖类等大分子物质水解成氨基酸、单糖等容易被乳酸菌生长吸收的小分子代谢产物,因此酵头中乳酸菌呈现种类丰富而多样的特点。Vuyst等[11]研究表明,酵头中乳酸菌包括 Lactobacillus、Leuconostoc、Pediococcus和Weissella等菌属。其中,Lactobacillus是传统酵头中最常见的乳酸菌类群,主要包括 Lb.brevis、 Lb.plantarum、 Lb.paralimentarius、Lb.rossiae 和 Lb.sanfranciscensis 等[12]。Robert 等[13]研究发现,Lactobacillus(39%)、Pediococcus(38%)、Leuconostoc(17%)、Weissella(4%)、Lactococcus(1%)和Enterococcus(<1%)是法国传统酵头中的乳酸菌菌群。Corsetti等[12]分析酵头中乳酸菌菌群的构成为 Enterococcus、Lactococcus、Leuconostoc、Pediococcus、Streptococcus和 Weissella。Moroni等[14]研究低温持续自主发酵的buckwheat和teff传统I型酵头中乳酸菌组成,结果表明乳酸菌主要为Lactobacillus、Pediococcus、Leuconostoc。
总之,传统酵头具有丰富的乳酸菌微生态系统,最常见的是 Lactobacillus,Lactococcus、Enterococcus、Leuconostoc和Weissella广泛存在于谷物和面粉中,但其耐受酸化环境的能力弱而在酵头中处于弱势地位[12]。与其他发酵食品相比,酵头中发挥显著作用的主要是专性异型发酵乳酸菌,这是由于它们能够发酵糖类产生乙酸以及乳酸等代谢产物[15]。酵头发酵过程中乳酸菌的演替主要与菌株对酸的适应机制和耐受性以及碳水化合物和氮代谢有关。Moroni等[14]应用PCR-DGGE技术研究buckwheat和teff酵头中乳酸菌的演替过程,结果表明,初始发酵12 h主要有Lb.sakei、Lb.graminis 和 P.pentosaceus,12h 后 为Lb.plantarum、W.cibaria、Leu.holzapfelii,最后阶段Lb.plantarum和W.cibaria的数量减少。Weckx等[16]研究发现黑麦酵头发酵经过3个主要阶段,初期乳酸菌主要为 Lactococcus和 Enterococcus,中期被 Leuconostoc和 Weissella所取代,第 5天后主要为Lb.fermentum和Lb.plantarum。
1.3 发酵香肠
发酵香肠是经乳酸菌、霉菌、酵母、微球菌等多种微生物共同发酵的肉制品,乳酸菌对产品的风味形成、营养价值提升和安全性保障都具有十分重要的作用。Urso等[17]研究意大利东北部 Friuli-Venezia-Giulia地区3种自然发酵香肠乳酸菌组成,发现Lb.curvatus(14.4%)和Lb.sakei(75.9%)数量最多。Bonomo等[18]对意大利南部Basilicata地区传统发酵香肠进行研究,发现Lb.sakei(67%)是优势乳酸菌,其次是P.pentosaceus(16%)和Leu.carnosum(8%),然后是 Lb.plantarum(4%)、Lb.brevis(2%)和 Leu.pseudomesenteroides(2%)等。Cocolin 等[19]对意大利北部不同省份的3种发酵香肠乳酸菌生态学进行研究,发现优势乳酸菌主要为 Lb.sakei和Lb.curvatus。
从乳酸菌在发酵香肠的菌群演变来看,发酵早期主要为Lb.sakei和Lb.curvatus,成熟阶段的优势乳酸菌为 Lb.sakei。Danilovi等[20]分析了传统 Petrovac香肠整个发酵过程的乳酸菌菌相演替情况,结果表明,E.caseliflavus在开始第2天占主要优势地位,从第2天至第 90天 Leu.mesenteroides、P.pentosaceus和Lb.sakei共存,并形成优势菌群。Greco等[21]分析意大利干发酵香肠的乳酸菌菌相,结果表明主要由Lb.sakei、Lb.plantarum 和 Lb.curvatus构成。Tran等研究发现,Lb.plantarum和P.pentosaceus是nem chua香肠成熟阶段中的优势菌群,这两种菌产酸和耐酸能力较强,随着pH减低,代替了初始发酵时期的耐酸性较弱的异型发酵乳酸菌[22]。
1.4 开菲尔
开菲尔(Kefir)是源于高加索地区由多种微生物协同作用而成的复合型发酵乳,其中开菲尔粒(Kefir grains,KG)是富含天然乳酸菌、酵母和醋酸菌等微生物的共发酵体系。乳酸菌是KG的主体微生物类群,数量约为108~109CFU/g,主要包括 Lactococcus、Lactobacillus和 Leuconostoc。Simova 等[23]对保加利亚地区开菲尔粒中菌相研究发现,乳酸菌占总微生物的83% ~ 90%,主 要 为 Lc.lactis subsp.lactis、Streptococcus thermophilus、Lb.delbrueckii subsp.bulgaricus、Lb.helveticus、Lb.casei subsp.pseudoplantarum和Lb.brevis;酵母占10% ~17%,主要为 Kluyveromyces marxianus var.lactis、Saccharomyce cerevisiae、Candida inconspicua 和 Candida maris。Nalbantoglu等[24]分析了土耳其开菲尔粒的乳酸菌菌相构成,结果表明,Lactobacillus的数量最多,包括Lb.kefiranofaciens、 Lb.buchneri 和 Lb.helveticus。ZHOU等[25]对我国西藏开菲尔粒的细菌菌相分析,Leu.mesenteroides、 Lb.helveticus、 Lb.kefiranofaciens、Lc.lactis、Lb.kefiri和Lb.casei为优势菌种。
图1是 Witthuhn等[26]分析传统 kefir发酵过程中微生物群落分布状况,A、B、C分别为开菲尔发酵20、25和30 d时微生物菌群组成。从图中可以看出,发酵20 d时 Leu.mesenteroides subsp.mesenteroides占26.9%,Lc.lactis subsp.lactis占32.4%,此外还含有40.4% 的 Zygosaccharomyces sp.。 到 25d 时,Lb.fermentum占80.5%,成为优势菌,还有15.3%的Lc.lactis subsp.lactis和少量的Cryptococcus humicolus(4.0%)。随着发酵时间的延长,发酵 30 d时Lb.fermentum达到98.2%,占有绝对优势。Magalhães等[27]将开菲尔粒接种于糖水中在25℃发酵24 h,发现 Lb.paracasei、Lb.parabuchneri和 Lb.kefiri在一直存在于发酵过程,Lb.casei和Lc.lactis在发酵初期存在,分别在12 h和18 h后又被重新检测到。Leu.citreum在发酵12 h后才发现,到24 h后检测到Lb.paracasei subsp.tolerans和 Lb.buchneri。
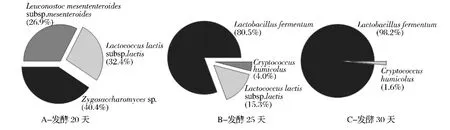
图1 传统kefir发酵过程中微生物群落分布状况Fig.1 The average distribution frequency of the microbial population of traditional kefir production
2 食品乳酸菌生态学研究方法
食品微生态学研究方法主要包括以培养和鉴定技术获取特征微生物的传统微生物学方法,以微生物细胞结构成分或代谢特征差异进行鉴别的非培养的生理生化方法,基于核酸为微生物信息载体的分子生物学技术和以微生物代谢产物为信息对象的现代分析技术等。
2.1 传统微生物学方法
传统微生物学方法依据乳酸菌的生物学特征应用选择性培养基从传统乳酸发酵食品中分离各种乳酸菌菌落,再经过形态学和生理生化特征进行分类鉴定,获取乳酸菌菌相信息。Drosinos等[28]用MRS培养基从发酵香肠中分离出288株乳酸菌,通过形态学和生理生化鉴定 255株为 Lactobacillus,15株 Leuconostoc,18株为 Lc.lactis subsp.lactis和 E.faecium。Iacumin等[29]利用平板法分析意大利北部酵头中微生物菌相构成,结果发现乳酸菌数量在103~109CFU/g之间,酵母数量在102~107CFU/g范围内,乳酸菌显著高于酵母为优势菌群。
2.2 磷脂脂肪酸(PLFA)/脂肪酸甲酯(FAME)分析法
PLFA/FAME分析法是基于细胞组分水平研究微生物的方法,以微生物细胞膜中磷脂中脂肪酸的结构多样性和生物特异性为基础,作为标记物揭示微生物的数量和菌群组成。目前应用于发酵食品的研究相对较少,Lee等[30]利用微生物鉴定系统构建了韩国泡菜中230株乳酸菌、8个种群的FAMEs指纹数据库,并对79株Leuconostoc进行鉴定。
2.3 Biolog微平板分析法
Biolog微平板法是美国Biolog公司研发的自动化鉴定微生物群落的方法,基于微生物对碳源底物利用的差异性比较分析微生物的群落构成。Lee等[31]利用该方法分析韩国泡菜中乳酸菌的菌群构成,结果表明获得的75菌株均为Leuconostoc。
2.4 分子生物学技术
分子生物学技术主要包括特异性片段测序及分析技术、遗传指纹图谱分析技术、宏基因组分析技术等。
特异性片段测序及分析技术是利用通用引物扩增特定基因片段后直接测序,主要是针对16S rRNA进行序列分析。Kim等[32]将样品用磷酸缓冲溶液稀释,通过涡流和声波处理等步骤,直接提取样品中的DNA,并通过16S rRNA基因序列分析泡菜中细菌群落结构,发现 Lactobacillus、Leuconostoc和 Weissella等乳酸菌为优势菌群。
遗传指纹图谱分析技术是采用凝胶电泳等方法对代表微生物群落结构的核酸分子进行分离,依据其迁移的差异构建遗传指纹图谱。经PCR对特定基因片段扩增后,分析样品中微生物的菌相构成,如变性梯度凝胶电泳技术(PCR-DGGE)、基因组重复序列PCR技术(rep-PCR)、随机扩增多态性DNA(RAPD)技术和脉冲场凝胶电泳(PFGE)技术等。ZHANG等[33]应用PCR-DGGE技术分析内蒙古西部酵头中乳酸菌群落构成,通过条带比对,发现Lb.plantarum和 Lb.sanfranciscensis为优势菌群。Tran等[22]利用rep-PCR和PFGE技术获取发酵香肠中乳酸菌的分子指纹图谱,乳酸菌构成分别为 Lb.plantarum(67.6%)、 P.pentosaceus (21.6%)、 Lb.brevis(9.5%)和 Lb.farciminis(1.4%)。Urso等[17]从意大利3种发酵香肠分离的乳酸菌中提取100 ng DNA,经过RAPD-PCR分析,Lb.curvatus和Lb.sakei是这些香肠中共同菌株。
宏基因组分析技术选择性提取环境中样品的总DNA/RNA,通过构建宏基因组文库进行高通量测序分析或筛选新的活性物质。Jeong等[34]使用DNA快速抽提盒提取样品中的DNA,经过16S rRNA序列扩增,通过焦磷酸测序技术分析泡菜中的微生物群落,结果发现加入红辣椒粉的泡菜在发酵初期生物多样性比未加红辣椒粉的减少速度缓慢。Leite等[35]通过PCR-DGGE技术和焦磷酸测序技术分析巴西不同地区的3种开菲尔粒的微生物多样性和群落结构,结果表明 Lb.kefiranofaciens和 Lb.kefiri为优势菌群。Jung等[36]在泡菜发酵期间,通过热酚法提取泡菜中的全部RNA并分离出mRNA,采用Illumina高通量得到泡菜发酵期间的转录文本,结果发现 Leu.mesenteroides在早期发酵时期最为活跃,而Lb.sakei和W.koreensis在后发酵时期数量较多。
2.5 基于代谢产物的现代分析技术
代谢产物可间接反映食品中微生物的分布与演替情况,通过代谢产物分析获取微生物种类及数量的相关信息。通常以色谱技术分离食品中微生物的代谢产物,以质谱和核磁共振等技术进行产物鉴定,实际工作中多采用分离与鉴定联用技术。Weckx等[16]应用GC-MS和HPLC-MS分析4种黑麦酵头发酵过程中糖类、氨基酸及其相关代谢产物变化情况,来推测发酵微生物的演变过程。Jeong等[8]通过核磁共振技术分析韩国水泡菜中的游离糖变化,发现随着游离糖的快速消耗,丙三醇和乙醇等产物迅速增加,以此判断泡菜的质量和产品成熟期。
由于食品中存在大量活的不可培养的微生物,传统微生物学方法不能获取全面真实微生物信息,非培养的生理生化方法受环境的影响,准确率有待于提高。因此,目前分子生物学技术是全面获取食品微生态学研究的主要手段。在食品微生态学研究中,应在传统微生物学和现代分子生物学获取微生物菌群信息的基础上,结合现代分析技术分析微生物的代谢产物获取微生物及其代谢产物的全面信息。
3 展望
微生态学是一门新的学科主要用于土壤、海洋环境及人类和动物肠道中微生物多样性分析,用于传统发酵食品处于起步阶段,已用于传统韩国泡菜、欧洲酵头和发酵香肠、开菲尔等食品。研究方法从传统的分离培养技术、化学成分分析方法到应用现代分子生物学技术和基于代谢组学的分析技术。本文以乳酸菌为主线分析传统发酵食品中乳酸菌菌群的发生、发展与演替过程,阐明传统乳酸发酵食品乳酸菌与原料之间、乳酸菌与他种微生物之间、乳酸菌菌群之间的相互作用关系,旨在探究发酵过程乳酸菌与产品品质形成的内在联系,为提高我国传统发酵食品的研究水平,提升应用现代技术对传统发酵食品的改造水平,达到缩短发酵周期、提高产品品质和经济效益的目的。
[1] Codex Alimentarius Commission.Codex standard for kimchi.Codex Stan[M].Rome:Food and Agriculture Organization of the United Nations,2001:223.
[2] Lee J S,Heo G Y,Lee J W,et al.Analysis of kimchi microflora using denaturing gradient gel electrophoresis[J].International Journal of Food Microbiology,2005,102(2):143-150.
[3] Cho K M,Math R K,Islam S M,et al.Novel multiplex PCR for the detection of lactic acid bacteria during kimchi fermentation [J].Molecular and Cellular Probes,2009,23(2):90-94.
[4] Park J M,Shin J H,Lee D W,et al.Identification of the lactic acid bacteria in kimchi according to initial and overripened fermentation using PCR and 16S rRNA gene sequence analysis[J].Food Science and Biotechnology,2010,19(2):541-546.
[5] Jung J Y,Lee SH,Lee H J,et al.Effects of Leuconostoc mesenteroides starter cultures on microbial communities and metabolites during kimchi fermentation[J].International Journal of Food Microbiology,2012,153(3):378-387.
[6] HONG Y,YANG H S,CHANG H C,et al.Comparison of bacterial community changes in fermenting kimchi at two different temperatures using a denaturing gradient gel electrophoresis analysis[J].Journal of Microbiology and Biotechnology,2013,23(1):76-84.
[7] Jung H J,Hong Y,Yang H S,et al.Distribution of lactic acid bacteria in garlic(Allium sativum)and green onion(Allium fistulosum)using SDS-PAGE whole cell protein pattern comparison and 16S rRNA gene sequence analysis[J].Food Science and Biotechnology,2012,21(5):1 457-1 462.
[8] Jeong S H,Jung J Y,Lee S H,et al.Microbial succession and metabolite changes during fermentation of dongchimi,traditional Korean watery kimchi[J].International Journal of Food Microbiology,2013,164(1):46-53.
[9] Cho J,Lee D,Yang C,et al.Microbial population dynamics of kimchi,a fermented cabbage product[J].FEMS Microbiology Letters,2006,257(2):262-267.
[10] De Vuyst L,Neysens P.The sourdough microflora:biodiversity and metabolic interactions[J].Trends in Food Science & Technology,2005,16(1):43-56.
[11] De Vuyst L,Vancanneyt M.Biodiversity and identification of sourdough lactic acid bacteria[J].Food Microbiology,2007,24(2):120-127.
[12] Corsetti A,Settanni L,Chaves López C,et al.A taxonomic survey of lactic acid bacteria isolated from wheat kernels and non-conventional flours[J].Systematic and Applied Microbiology,2007,30(7):561-571.
[13] Robert H,Gabriel V,Fontagné-Faucher C.Biodiversity of lactic acid bacteria in French wheat sourdough as determined by molecular characterization using species-specific PCR[J].International Journal of Food Microbiology,2009,135(1):53-59.
[14] Moroni A V,Arendt E K,Bello F D.Biodiversity of lactic acid bacteria and yeasts in spontaneously-fermented buckwheat and teff sourdoughs[J].Food Microbiology,2011,28(3):497-502
[15] Settanni L,Ventimiglia G,Alfonzo A,et al.An integrated technological approach to the selection of lactic acid bacteria of flour origin for sourdough production[J].Food Research International,2013,54(2):1 569-1 578.
[16] Weckx S,Vander Meulen R,Maes D,et al.Lactic acid bacteria community dynamics and metabolite production of rye sourdough fermentations share characteristics of wheat and spelt sourdough fermentations[J].Food Microbiology,2010,27(8):1 000-1 008.
[17] Urso R,Comi G,Cocolin L.Ecology of lactic acid bacteria in Italian fermented sausages:isolation,identification and molecular characterization[J].Systematic and Applied Microbiology,2006,29(8):671-680.
[18] Bonomo M G,Ricciardi A,Zotta T,et al.Molecular and technological characterization of lactic acid bacteria from traditional fermented sausages of Basilicata region(Southern Italy)[J].Meat Science,2008,80(4):1 238-1 248.
[19] Cocolin L,Dolci P,Rantsiou K,et al.Lactic acid bacteria ecology of three traditional fermented sausages produced in the North of Italy as determined by molecular methods[J].Meat Science,2009,82(1):125-132.
[20]DanilovicˊB,JokovicˊN,PetrovicˊL,et al.The characterisation of lactic acid bacteria during the fermentation of an artisan Serbian sausage(Petrovská Klobása)[J].Meat Science,2011,88(4):668-674.
[21] Greco M,Mazzette R,De Santis E P L,et al.Evolution and identification of lactic acid bacteria isolated during the ripening of Sardinian sausages[J].Meat Science,2005,69(4):733-739.
[22] Tran K,May B K,Smooker P M,et al.Distribution and ge-netic diversity of lactic acid bacteria from traditional fermented sausage[J].Food Research International,2011,44(1):338-344.
[23] Simova E,Beshkova D,Angelov A,et al.Lactic acid bacteria and yeasts in kefir grains and kefir made from them[J].Journal of Industrial Microbiology and Biotechnology,2002,28(1):1-6.
[24] Nalbantoglu U,Cakar A,Dogan H,et al.Metagenomic analysis of the microbial community in kefir grains[J].Food Microbiology,2014,41:42-51.
[25] ZHOU J,LIU X,JIANG H,et al.Analysis of the microflora in Tibetan kefir grains using denaturing gradient gel electrophoresis[J].Food Microbiology,2009,26(8):770-775.
[26] Witthuhn R C,Schoeman T,Britz T J.Characterisation of the microbial population at different stages of Kefir production and Kefir grain mass cultivation[J].International Dairy Journal,2005,15(4):383-389.
[27] Magalhães K T,Pereira G V M,Dias D R,et al.Microbial communities and chemical changes during fermentation of sugary Brazilian kefir[J].World Journal of Microbiology and Biotechnology,2010,26(7):1 241-1 250.
[28] Drosinos E H,Mataragas M,Xiraphi N,et al.Characterization of the microbial flora from a traditional Greek fermented sausage[J].Meat Science,2005,69(2):307-317.
[29] Iacumin L,Cecchini F,Manzano M,et al.Description of the microflora of sourdoughs by culture-dependent and culture-independent methods[J].Food Microbiology,2009,26(2):128-135.
[30] Lee J S,Chun C O,Kim H J,et al.Analysis of cellular fatty acid methyl esters(FAMEs)for the identification of Leuconostoc strains isolated from kimchi[J].Journal of Microbiology,1996,34(3):225-228.
[31] Lee J S,Chun C O,Hector M,et al.Identification of Leuconostoc strains isolated from kimchi using carbon-source utilization patterns[J].Journal of Microbiology,1997,35(1):10-14.
[32] Kim M,Chun J.Bacterial community structure in kimchi,a Korean fermented vegetable food,as revealed by 16S rRNA gene analysis[J].International Journal of Food Microbiology,2005,103(1):91-96.
[33] ZHANG J,LIU W,SUN Z,et al.Diversity of lactic acid bacteria and yeasts in traditional sourdoughs collected from western region in Inner Mongolia of China[J].Food Control,2011,22(5):767-774.
[34] Jeong S H,Lee H J,Jung J Y,et al.Effects of red pepper powder on microbial communities and metabolites during kimchi fermentation[J].International Journal of Food Microbiology,2013,160(3):252-259.
[35] Leite A M O,Mayo B,Rachid C,et al.Assessment of the microbial diversity of Brazilian kefir grains by PCRDGGE and pyrosequencing analysis[J].Food Microbiology,2012,31(2):215-221.
[36] Jung J Y,Lee S H,Jin H M,et al.Metatranscriptomic analysis of lactic acid bacterial gene expression during kimchi fermentation[J].International Journal of Food Microbiology,2013,163(2):171-179.

