罗田迟熟板栗品种SSR分子标记初步鉴定
李琳玲,程华,陈小玲,余海洋 经济林木种质改良与资源综合利用湖北省重点实验室,湖北 黄冈438000;
黄冈师范学院生命科学学院,湖北 黄冈438000
程水源 (武汉轻工大学生物与制药工程学院,湖北 武汉430023)
SSR(Simple Sequence Repeat)又称微卫星序列,是重复序列的主要组成成分之一。SSR是一类由1~6个核苷酸为重复单位序列组成的串联重复序列[1]。这些简单重复序列绝大部分随机、均匀、广泛地分布于真核生物的基因组上,并且由于重复次数不同而造成简单序列长度多态性(Simple Sequence Length Polymorphism,SSLP)。不同物种的SSR序列在长度、组成、重复次数、突变率和在染色体上的分布情况高度多态,反映出高度的等位基因多样性。SSR标记由Moore等于1991年创立,SSR分子标记技术具有共显性、涵盖范围广、揭示多态性高等优点,已在育种、指纹数据库的构建、品种鉴定及种子纯度等方面得到广泛应用,是目前应用最广泛的分子标记技术之一[2,3]。
我国板栗栽培历史悠久,种质资源丰富,在复杂的地理条件和气候条件下,形成多种生态类型和地方品种[4]。湖北省罗田县是我国重要的板栗主产区,栽培面积4.8万hm2,年产板栗约3.0万t左右,板栗产业系列产品年产值达4.8亿元,在当地经济发展中起着不可替代的作用[5]。板栗品种资源丰富,但对这些品种的划分传统方法主要是依据板栗的形态特征、生物学特性等来鉴别,而在实际生产中对形态相似的品种仅依靠营养器官的形态特征难以在苗木期就准确鉴别,且木本植物结实周期长,因而不利于优良品种的生产、推广和种质资源保存。如何准确鉴别品种,及时对新品种予以审定、注册与保护,建立准确可靠的品种特异性检测方法,对提高板栗的经济效益具有非常重要的实际价值[6]。
本研究以罗田迟熟板栗品种的叶片总DNA为材料,利用前期开发的SSR引物进行PCR扩增,筛选出多态性较高的引物用于8个板栗品种的遗传差异性分析。在NTSYS 2.10e分析软件中,根据SM相似系数法求得品种间的遗传相似性矩阵,再用UPGMA进行聚类分析,构建系统聚类分析树状图,探讨了罗田板栗种质资源概况,以期能够更快速、准确、简便地获得板栗种质资源的遗传信息,从分子水平上为板栗种质资源的开发和保存提供参考。
1 材料与方法
1.1 试验材料
试验材料为湖北省罗田县板栗产区的8个栽培品种,每个品种随机选取10~20片幼嫩叶片装入做好标记的自封袋中,于-40℃冰箱中冷冻保存。材料来源和编号详见表1。
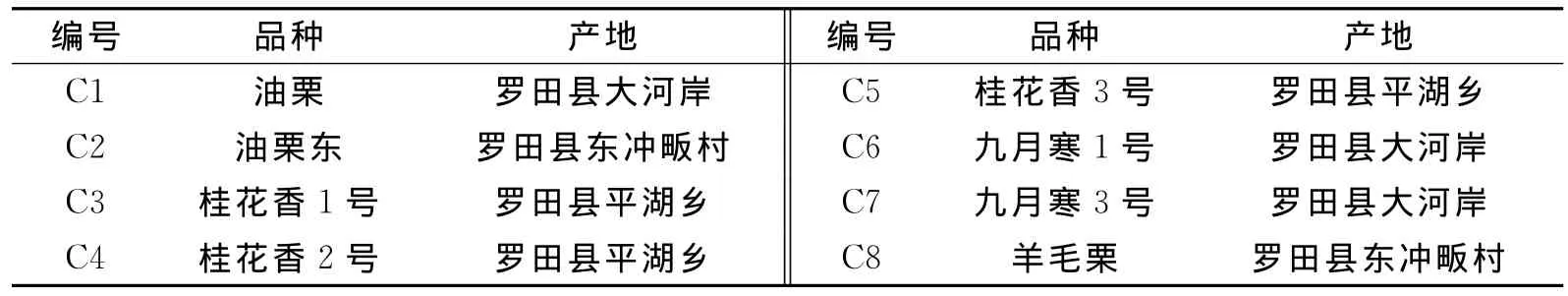
表1 供试板栗来源及编号
主要试剂琼脂糖、5×TBE缓冲液、2×Taq PCR MasterMix购自北京天根生化科技有限公司;Maker DL2000购自大连宝生物工程有限公司;CTAB提取DNA试剂、30%聚丙烯酰胺、过硫酸胺、TEMED、硝酸银、氢氧化钠、甲醛、无水乙醇、冰醋酸等购自武汉贝尔生物技术有限公司。
1.2 试验方法
1.2.1 板栗基因组DNA的提取与检测
参考魏春红等[7]、程丽莉等[8]的CTAB法略做改进,提取8个板栗品种叶片的总基因组DNA。0.8%琼脂糖凝胶电泳检测提取的DNA质量,紫外分光光度计检测DNA浓度,提取的总DNA保存于-20℃冰箱。
1.2.2 引物来源及SSR-PCR扩增反应体系的建立
所用引物依据NCBI板栗EST文库,筛选出SSR序列,参照网站公布的SSR引物序列,共设计出8条扩增引物(表2),由生工生物工程有限公司合成。SSR-PCR扩增反应体系设为30μL,包括 2×Taq PCR MasterMix 反应液(0.1UTag Polymerase/μL、500μmol/L dNTP each、20mmol/L Tris-HCl、100mmol/L KCl、3mmol/L MgCl2及其他稳定剂和增强剂)15μL,引物3μL,DNA模板50ng,ddH2O 9μL。扩增程序设为94℃预变性4min;然后94℃变性30s,41~55℃退火40s,72℃3min,共35个循环;最后72℃延伸3min。
1.2.3 引物筛选
选用基因组DNA纯度较高的板栗样品为模板,分别对8个引物采用以上扩增程序进行PCR扩增。扩增产物先用0.8%的琼脂糖凝胶电泳初步检测,再用6%的聚丙烯酰胺凝胶电泳进一步分析,筛选出多态性较高的引物用于8个板栗样品的鉴定。
1.2.4 数据统计与分析
将选扩的PCR产物全部上样6%的聚丙烯酰胺凝胶电泳分离,在90V电压下电泳约50min,经硝酸银染色,氢氧化钠显影,所获得的扩增条带以0、1统计建立数据库。条带统计以其清晰可重复为基本原则,采用人工读带法,在相同迁移位置,有带记为1,无带记为0。在NTSYS 2.10e分析软件中,根据SM相似系数法求得品种间的遗传相似性矩阵,再用UPGMA进行聚类分析,构建系统聚类分析树状图[9]。
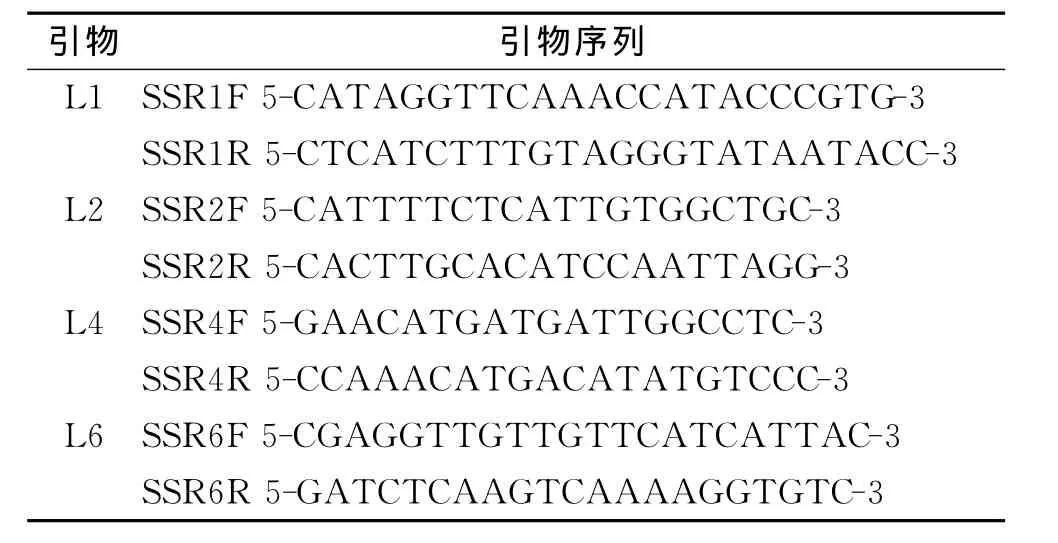
表2 SSR分析所用引物序列
2 结果与分析
2.1 植物总DNA琼脂糖凝胶电泳图
由图1可以看出:8份板栗叶片总DNA凝胶电泳的带型显示,DNA纯度较高,质量较好,可以进行下一步的SSR-PCR扩增实验。
2.2 引物筛选琼脂糖凝胶电泳图
随机选取油栗的DNA为模板,分别对8条SSR扩增引物进行筛选,淘汰无扩增产物或多态性较差的引物,选留扩增条带清晰且有明显多态性片段的引物。根据琼脂糖电泳得到的初步检测结果(图2),从中筛选出了L1、L2、L4、L6 4个引物(表3)。这些引物均能在供试板栗中扩增出清晰、稳定、重复性和多态性较高的条带。

图1 总DNA琼脂糖凝胶电泳结果

图2 引物筛选琼脂糖凝胶电泳结果

表3 SSR分析所用引物序列扩增结果
2.3 PCR扩增产物PAGE电泳图
4条引物分别对罗田8个板栗栽培品种DNA进行PCR扩增,经统计共扩增出36条DNA带,其中多态性条带31条,平均每个引物扩增出9条带,平均多态性位点百分率为86.1%,具体扩增结果见表3。不同引物扩增出的条带数差异小。引物多态性达80%以上的 共3个,其中引物6的多态性最高,为100%。4条引物分别建立了相应的DNA指纹图谱,由各引物扩增效果所反应出的DNA条带多态性,能较好地区分各板栗品种。

图3 SSR-PCR扩增产物PAGE电泳结果
2.4 聚类分析
表4为NTSYS 2.10e软件计算出的板栗品种间的Dice遗传相似系数。由表4可知,8个板栗品种间的遗传相似系数范围在0.22~0.89之间,平均遗传相似性系数为0.65。

表4 板栗品种间Dice遗传相似系数
在获得两两不同品种间的Dice遗传相似系数基础上,以36个位点的谱带为原矩阵,采用UPGMA法构建了板栗品种间的遗传关系聚类图(图4)。树状图与品种间的遗传相似系数反应的情况基本一致,即遗传相似系数越高,亲缘关系越近,品种间差异就越小。从树状图可以看出,在相似性系数0.35处,8个品种资源划分成A、B 2支,B支只有羊毛栗。其余7个品种聚为A群,在相似系数0.69处A群分为A1和A2 2个亚群。A1亚群包括油栗、油栗东,从8个品种的4个SSR指纹图谱中也可以明显看出,这2个品种的指纹图谱非常接近。A2亚群包括桂花香1号、桂花香2号、桂花香3号、九月寒1号、九月寒3号5个板栗品种,其中桂花香1号、桂花香2号、桂花香3号遗传相似系数在0.89以上,九月寒1号、九月寒3号遗传相似系数也在0.89以上,品种间的亲缘关系很近。总体来看,8个板栗品种之间的遗传多样性比较丰富,SSR分子标记可基本将其区分为2大类,羊毛栗单独一类,其余7个品种为第二类。
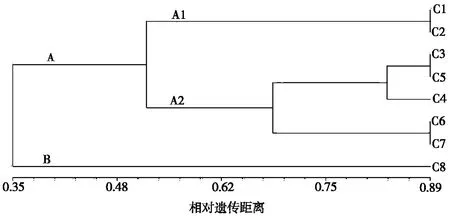
图4 基于SSR标记以UPGMA法构建的8个板栗品种的分子系统树
3 讨论
SSR标记作为一种有效的基因型鉴技术,能够产生足够多的等位多态性,因而已成为育种系谱分析的有效手段。植物基因组中,各个SSR位点除重复次数不同外,其碱基组成及结构是相似的,而且在基因组中能检测多个位点,因个体、品种(系)或群体的差异,可通过电泳、染色或分子杂交显示出目的条带的有无,即产生DNA指纹图。同时,由于SSR在基因组中广泛分布,因此SSR指纹图能代表一定的基因组特征。研究DNA的差异来分析群体的遗传结构及遗传多样性更为直接,对正确评价植物种质资源的遗传多样性、制定育种策略十分重要。指纹图谱在作物品种鉴定、亲子分析、品种注册、良种质量监测、分辨真假杂种、控制种子纯度以及知识产权保护等方面具有重要作用[10]。研究表明,在目前使用的绝大多数分子标记系统中,SSR标记是变异率最高的,即能检测到的遗传多态性较多,SSR标记还有重复性好、特异位点扩增、发生频率高、共显性遗传等优点[11],因而成为林木遗传多样性研究、亲缘关系分析、遗传连锁图谱构建和数量性状基因定位以及群体遗传结构研究的重要方法[12]。从本研究所构建的指纹图可以看出:共扩增出36条DNA带,其中多态性条带31条,平均每个引物扩增出9条带,平均多态性位点百分率为86.1%。不同引物扩增出的条带数差异小。4条引物分别建立了相应的DNA指纹图谱,由各引物扩增效果所反应出的DNA条带多态性,能较好地区分开各迟熟板栗品种。
随着分子生物学的发展,新兴的分子标记技术不断涌现,SSR分子标记技术也不断得到改进和完善[13]。SSR标记因同时具有多态性百分率高,重复性好,共显性等特点,在作物种质资源遗传多样性和基因分子标记等遗传育种领域中已较广泛应用[10,14]。SSR标记聚类结果为种质资源的分类、利用和亲缘关系的管理,提供了重要依据[15]。这就意味着板栗育种方面,可能会从传统育种转向基因育种方向,并且从表型选择向基因型选择方向发展。关于板栗种质资源鉴定和管理,可能会有地域或是环境影响因素,或是地理相关趋势,这就需要利用SSR-PCR或ISSR-PCR构建更大范围的板栗地理聚类结果和DNA指纹库。
[1]Hamada H,Petrina M G,Kakunaga T.A novel repeated element with Z-DNA-forming potential is widely found in evolutionarily diverse eukaryotic genomes[J].Proc Natl Acad Sci,USA,1982,79:6465~6569.
[2]罗兵.分子标记研究进展 [J].安徽农业科学,2013,41(12):5210~5212,5226.
[3]Moore S S,Sargeant L L,King T J,etal.The conservation of dinucleotide microsatellites among mammalian genomes allows the use of heterologous PCR primer pairs in closely related species[J].Genomics,1991,10:654~660.
[4]丁向阳.分子标记技术在板栗研究中的应用进展 [J].福建林业科技,2007,34(1):4~8.
[5]姜德志,程水源,王燕,等.罗田3个板栗栽培品种主要营养成分分析 [J].湖北农业科学,2012,50(23):4882~4884.
[6]周连第,兰彦平,韩振海.板栗品种资源分子水平遗传多样性研究 [J].华北农学报,2006,21(3):81~85.
[7]魏春红,李毅.植物总DNA的提取.现代分子生物学实验 [M].北京:高等教育出版社,2006.
[8]程丽莉,苏淑钗,秦岭,等.板栗叶片DNA的提取及AFLP反应体系的建立 [J].北京农学院学报,2005,20(2):5~9.
[9]Yong L,De-Chun L,Bo W,etal.Genetic diversity of pummelo(CitrusgrandisOsbeck)and its relatives based on simple sequence repeat markers [J].Chinese Journal of Agricultural Biotechnology,2006,3:119~126.
[10]郭瑞星,刘小红,荣廷昭,等.植物SSR标记的发展及其在遗传育种中的应用 [J].玉米科学,2005,13(2):8~11.
[11]黄秦军,苏晓华,张香华.SSR分子标记与林木遗传育种 [J].世界林业研究,2002,15(3):4~20.
[12]王小国,梁红艳,张薇.82份春小麦种质资源遗传多样性的SSR分析 [J].西北农业学报.2013,22(1):34~40.
[13]曹士亮,曹靖生,王成波,等.玉米SSR分子标记技术操作流程研究进展 [J].中国农学通报,2012,28(15):1~4.
[14]宪立杰,刘兴菊,李雪雁,等.葡萄种质遗传多样性的SSR分析及指纹库构建 [J].北方园艺,2013,(11):87~90.
[15]欧阳磊,陈金慧,郑仁华,等.杉木育种群体SSR分子标记遗传多样性分析 [J].南京林业大学学报(自然科学版),2014,38(1):21~26.

