第三代DNA测序及其相关生物信息学技术发展概况
杨悦,杜欣军,梁彬,郭季冬,程晓真,王硕
(天津科技大学食品营养与安全重点实验室,天津300457)
第三代DNA测序及其相关生物信息学技术发展概况
杨悦,杜欣军,梁彬,郭季冬,程晓真,王硕*
(天津科技大学食品营养与安全重点实验室,天津300457)
本文介绍了第三代DNA测序的技术原理及应用现状,并对相关的生物信息学技术进行了综述。第三代测序技术以单分子测序为主要特点,目前已广泛应用于食品科学及生命科学研究的各个领域,其代表有Heliscope BioScience公司的SMS技术、Pacific BioSciences公司的SMRT技术等。本文同时归纳总结了基因组学相关的生物信息学发展状况及常用的数据库。
基因组学;第三代DNA测序技术;生物信息学;数据库
1986年美国科学家Thomas Roderick首次提出基因组学的概念,基因组学包括核苷酸测序及序列分析、基因定位、基因功能分析等内容[1]。基因组学始于人类基因组图谱绘制和测序的提出,这一伟大的理想在2004年完成,使基因组学成为生命科学领域中最重要和最基础的研究领域之一[2],如今也广泛于食品科学、环境科学等众多研究领域。
基因组学的迅速发展离不开DNA测序技术与生物数据处理手段-生物信息学。从上世纪六、七十年代开始,由最初的人工DNA测序到现在的第三代测序技术-单分子实时测序技术,DNA测序技术经历了翻天覆地的变化,同时,DNA测序获得的大量数据促进了生物信息学的产生和发展,利用生物信息学的方法分析和处理序列数据对认识和揭示基因组序列中蕴含的信息至关重要。本文旨在阐述第三代DNA测序技术的技术原理及应用情况,同时介绍了与之相关的生物信息学的研究内容及一些常用的数据库,为基因组测序及后续分析工作提供参考。
1第三代测序技术
目前正在兴起的第三代测序是单分子测序[3-6],这种技术无需PCR扩增,这种方法测序通量更高,操作过程更简单,成本更低。另外它还具有3个显著的特点:第一,单分子测序技术可以直接对RNA进行序列,这样大幅度降低体外逆转录产生的系统误差;第二,可以直接检测甲基化的DNA序列,为表观遗传学研究奠定了基础;第三,可以对特定序列的SNP进行检测,实现对稀有突变及其频率的测定。目前市面上单分子测序平台有Heliscope BioScience公司的SMS(true single molecular sequencing)技术[7-8],Pacific BioSciences公司的SMRT(single molecule real-time)技术[9],VisiGen Biotechnologies公司的FRET(fluorescence resonance energy transfer)技术[10]以及Oxford Nanopore Nechnologies公司的纳米孔技术[11]。
1.1SMS测序平台
SMS技术仍然建立在合成测序的基础之上,只是检测方法更加灵敏。它是利用电场的作用以采集与聚合酶结合的标记核苷酸的荧光特征进行测序[12]。其原理如图1所示[13]。
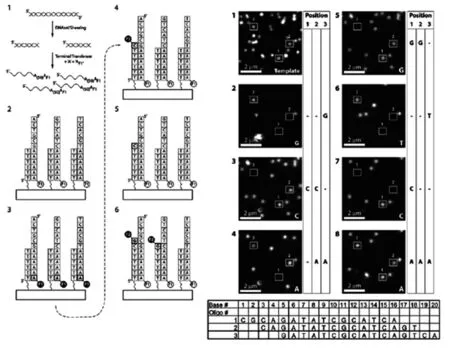
图1 SMS技术原理Fig.1The principle of SMS technology
(1)将待测的DNA序列随机打断并在3'末端加上polyA,利用末端转移酶进行荧光标记和阻断,阻断的目的是防止在测序过程中核苷酸在模板的3'末端进行延伸;(2)将这些标记好的小片段与带有polyT引物的平板杂交并精确定位;(3)逐一加入A、C、G、T4种荧光修饰的dNTP及聚合酶,当碱基互补延伸后,利用全内反射显微镜(total internal reflection microscopy,TIRM)进行单色成像,之后切开荧光染料和抑制基团,洗涤,加帽,允许下一个核苷酸的掺入;(4)如此反复循环,就可以实现实时测序采集荧光信号获得碱基信息。数十个循环后,将测得的DNA序列拼接,即得到完整的基因序列,目前已有所应用[14-15]。SMS测序技术的优点是:文库制备简单,不需要PCR扩增或连接酶,尤其适合RNA直接测序,无需传统的cDNA合成步骤,从而避免了体外逆转录产生的错误;缺点是初始读长较短,仅有35 bp,准确率较低,同时单分子测序成本较高,阻碍着这项技术的推广应用。
1.2SMRT测序平台
SMRT测序技术的单分子荧光检测设备采用零模式波导技术,以SMRT芯片为载体进行测序反应,其原理如图2所示[16-17]。

图2 SMRT技术原理Fig.2The principle of SMRT technology
测序的大致流程如下:(1)将待测的DNA样品随机打断,制成液滴后将其分散到SMRT芯片中;(2)MRT芯片是包含成千上万的纳米孔(Zero-Mode Waveguides,ZMWs)的金属片,这些纳米孔的直径短于激光的单个波长并且内部锚定有DNA聚合酶,测序时待测的DNA单链进入ZMW被DNA聚合酶捕获后,四种不同荧光标记的dNTP加在反应孔的上端,当dNTP与待测的DNA模板互补延伸时,DNA聚合酶首先捕获与模板匹配的dNTP,在荧光检测区被激光束激发出荧光,进而识别核苷酸的种类;(3)在荧光脉冲结束后,被标记的磷酸集团被切割并释放,DNA聚合酶转移到下一个位置,下一个待测的碱基连接到位点上开始释放荧光脉冲,进行下一个循环。SMRT测序技术是实际意义上的实时测序,完全依靠DNA聚合酶的作用,使测序速度明显提高,同时DNA聚合酶自身的延续性也能够保证了测序的读长,降低了测序的时间及费用;但是不足之处是会由于碱基掺入速度过快而出现插入和缺失错误,从而影响测序的准确性。
1.3FRET测序平台
FRET技术基本原理是利用荧光共振能量转移(fluorescence resonance energy transfer)现象,具体是指在进行测序时被荧光受体标记的4种脱氧核苷酸分子随着测序引物的延伸会发出特异性的微光,以达到对DNA的碱基序列进行连续、快速检测的目的[10]。其测序原理如图3[18-19]。
图3 FRET技术原理Fig.3The principle of FRET technology
测序流程如下:(1)将被供体荧光基团修饰的DNA聚合酶及待测的DNA模板分子固定在载玻片上;(2)向其加入含引物、4种dNTP(其磷酸上标记特异的荧光受体基团)测序缓冲液,测序过程中,当dNTP靠近含荧光供体基团的聚合酶时,后者就能释放能量激光并发出特异的荧光(即FRET信号),从而识别相应的碱基类型;(3)当dNTP被识别后,荧光基团就会随着磷酸离开,保证下一个dNTP能继续反应,从而达到测序的目的。FRET测序技术最明显的优势是测序过程简单直接,速度较快,如同看电影一般[20],其测序速度有望达到1百万碱基/秒;但是缺乏相应的技术参数从而限制了其广泛应用。
1.4纳米孔测序平台
纳米孔技术是一种纯物理学的方法,是利用不同的碱基通过纳米孔时产生的电信号变化来对其进行测序[21]。其技术原理类似于电泳,如图4所示[22-24]。
一线教师的付出是辛劳的,不仅仅是脑力劳动,同时还有体力劳动,费心费力。备课、上课、批改作业和试卷,还要反思教学,针对个别学生补缺补差,很少有休息时间。可是,作为一名教师,只有用心教学才能赢得家长的尊重和理解,在工作中,只有家长和学校相互配合,教师和家长之间互相理解和尊重,才能共同的把孩子教育好。应该说,我们需要家长的配合,理解和支持,让学生学好是共同的期待,这需要我们用心与家长沟通、交流,做家长的朋友。得到了家长的理解和支持,我们才能教出成绩,从而收获快乐,提升幸福感。
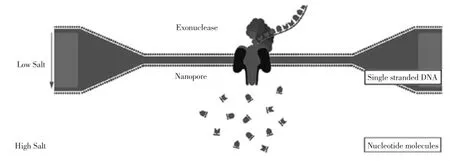
图4 纳米孔技术原理Fig.4The principle of Oxford Nanopore technology
大致过程为:待测的DNA序列在核酸外切酶的作用下迅速的逐一切割其脱氧核糖核苷酸分子,切下的核苷酸落入直径非常小的纳米孔(Nanopore)中,由于这种孔的直径只允许单一的核苷酸通过,当其通过纳米孔时,就会产生不同的电流变化幅度,从而区分不同的碱基,进而推测出待测DNA的序列信息。纳米孔单分子测序技术相对于其他的单分子测序技术而言,无需传统的DNA聚合酶、连接酶或者dNTPs,样本处理简单,同时也不需要复杂的光学探测系统(如激光发射器和CCD信号采集系统等),因此大大降低了测序成本,另外由于其测序的对象为单个核苷酸,所以这种技术有很好的持续性和准确性,还可以直接对RNA样品进行测序;缺点就是单个核苷酸通过纳米孔的速度及纳米孔的厚度可能引起电流差异特征性的不明显,从而降低测序的精确度。
测序技术不断的更新换代,与前代产品相比,第三代测序技术具有比不可比拟的优点,不仅大幅度降低了高昂的测序费用,而且使得对更多的物种进行测序成为可能,这些都将对分子生物学、基因组学和进化生物学的研究产生深远的影响。另一方面,第二代测序和第三代测序技术并驾齐驱使基因组测序的成本迅速下降,这将会给食品、医疗卫生等行业带来里程碑式的变革。
2基因组测序相关生物信息学技术及常用数据库
2.1生物信息学
生物信息学(Bioinformatics)是以计算机为工具对生物遗传信息进行加工处理以获得所需信息的科学[25]。这一门新兴的交叉学科以信息学、统计学、生物学、计算机为主要研究手段,在当今的生命科学和自然科学领域应用十分广泛。生物信息学起源于20世纪70年代,各种生物信息学的基本理论逐渐诞生,其中最重要的突破是Kimura提出的分子钟假说[26]。生物信息学发展成为一门独立的学科是在80年代,在这期间逐渐形成自己独特的理论体系和解决问题的方法,例如序列比对中的经典算法和FASTA家族的数据库搜索算法[27]。
2.2基因组测序相关生物信息学技术
过去30年,基因组DNA测序技术发展迅猛,应用领域也不断扩展,各种物种基因组测序的完成只是基因组计划的第一步,从基因组序列中提取有用信息,进而揭示其蕴含的全部意义,才是这些基因组计划的最终目标。在各种物种基因组被逐步破解的过程中,生物信息学能够通过信息学、统计学、计算机等手段对基因组测序所产生的海量数据进行科学的处理及分析,因此其在基因组及后基因组时代逐渐承担起越来越重要的角色。本文将生物信息学在基因组学方面的应用归纳为以下几个方面:
第二,基因区域及功能预测。经过序列拼接后能够得到完整的基因组序列信息,但是如果想要研究每个基因的功能就需要分析和解读核酸序列中所表达的结构与功能的生物信息。在真核生物中,并不是所有的基因都能够行使功能,例如在人类的基因组中,编码基因仅占总序列的3%~5%。所谓基因区域的预测,一般是指预测DNA序列中编码蛋白质的部分,即外显子部分。预测外显子的基本算法有ORF(open reading frame)法、核苷酸语汇(nucleotide words)及线性判别分析(Linear Discriminant Analysis,LDA)等。找到这些编码基因后,就要进行基因功能的预测,基本方法是序列同源比较,寻找蛋白质家族保守顺序,常用的算法有Smit-Waterman算法,FASTA算法和BLAST算法。
第三,代谢网络建模的分析。将分析得到的某种生物的基因组序列根据功能进行分类及其代谢组学的研究是近几年的研究前沿方向,将基因定位到代谢网络中(其涉及到生化反应途径,基因调控,信号转导过程等),这种后基因组时代的研究涉及到大规模网络的生命过程,又叫做“网络生物研究”[30]。如今,利用生物信息学技术开发专门软件工具来自动分析大规模网络系统的物理属性,提供路径导航、模式搜索、图形简化等分析手段以及基于代谢控制分析原理,使用常微分方程来求解反应速率,已经成为一种研究热点。
第四,数据库的建设及整合。生物数据库是进行生物信息学研究的基础,尽管目前已有许多公共的数据库可供使用,如Genbank等,这些都凝聚了大量生物信息学的工作。但我们进行专项研究时,往往需要根据具体分析内容构建新的数据库。要建立自己的数据库,就必须分析数据库的储存形式和复杂程度,设计相应的分析程序及算法,实现并行计算和先进的内存管理以提高数据库的速度等,这些都需要通过生物信息学来实现。另外,生物信息学技术还可以将多个数据库整合在一起提供综合服务,实现数据库的一体化和集成环境,能够使用户共享不同数据库,达到资源共享。
2.3生物信息学的常用数据库
随着第一代测序仪的全面推广,基因组测序数据量快速增加,使数据库的容量逐渐扩大,因此基因的预测和比对将生物信息学带入了一个崭新的时期,加速了各种数据库的诞生。
2.3.1生物信息学数据库的分类
根据建库方式,生物信息学中的数据库大致分为四类[31]:一级数据库、二级数据库、专家库及整合数据库。一级数据库最基础,一般是由国家或国际组织建设和维护,例如GenBank、EMBL及DDBJ等;二级数据库是在一级数据库的基础上,结合特殊的需要将部分数据从一级数据库中取出,经过重新组合(包括一定的修正或调整)而成的数据库,其专一性很强,数据量相对较少,如KEGG、CAZY及COG等;专家库是一种特殊的二级数据库,它是通过有经验的专家经过人工校对标识之后建立的,这类数据库的优点是质量高,使用方便可靠,但是更新和发展比较缓慢,如Unipro-Swiss-Prot等。整合数据库是将不同数据库的内容按照一定的要求整合而成,如商业及内部数据库。
2.3.2常用生物信息学数据库
熟练掌握常用数据库及软件对基因组拼接和分析至关重要,下面简要介绍几个常用的数据库。
三个一级核酸数据库GenBank、EMBL和DDBJ在生命科学中占据着不可动摇的重要地位,是生物信息学中不可或缺的数据资源与分析工具。GenBank由美国国立卫生研究院下属的国立生物技术信息中心(national center for biotechnology information,NCBI)建立[32-33],这个数据库汇集并注释了所有公开的核酸序列,Genbank的数据可以从NCBI的FTP服务器上免费下载完整的库,或下载积累的新数据,NCBI还提供广泛的数据查询、序列相似性搜索以及其它分析服务,官方网址为http://www.ncbi.nlm.nih.gov/genbank。EMBL全称为European molecular biology laboratory,是由欧洲生物信息研究所创建的欧洲分子生物学实验室核苷酸数据库,该数据库由Oracal数据库系统管理维护,查询检索可以通过因特网上的序列提取系统(SRS)服务完成[34],官方网址为http://www.embl.org/。DDBJ的英文全称为DNA data bank of Japan,是日本DNA数据库系统,人们可以使用其主页上提供的SAS工具进行数据检索和分析[35],官方网址为http://www. ddbj.nig.ac.jp/。这三个数据库都是国际核苷酸序列数据库合作的成员,他们定期进行数据交换,互通有无,同步更新。
重要的二级数据库有KEGG、CAZY和COG等。KEGG即Kyto Encyclopeida of Genes and Genomes,译为京都基因与基因组百科全书,是全面破译基因组的数据库,将基因组序列信息、化学、药物和基因的功能信息有机地结合起来,其特色是代谢途径的分析,对于获得全基因组序列的物种,只要输入其全部的蛋白质序列,通过计算机化处理,就可以预测出该物种的代谢网络途径。该数据库的官方网址是http://www. genome.jp/kegg/,更新版本为Release 69.0,最近更新日期是2014年1月1日。CAZY是Carbohydrate-Active enzymes Database的缩写,是有关碳水化合物酶类的数据库,依据对糖苷键的作用将其分类,这些作用包括形成、降解及修饰,该数据库对物种的初级代谢研究具有重要的意义,其官方网址为http://www.cazy.org/,最近更新日期为2014年1月14日。COG全称为Clusters of Orthologous Groups of proteins,是直系同源蛋白质聚类数据库,可以根据系统进化关系将测序完成的各种生物中的编码蛋白进行分类,每个COG都有功能注释,对于预测单个蛋白质的功能或者新物种的功能都非常有用,该数据库的官方网址为http://www. ncbi.nlm.nih.gov/COG/。
专家库Unipro-Swiss-Prot是目前世界上规模较大的蛋白质数据库,由欧洲生物信息研究所和瑞士生物信息研究所共同维护的,这个数据库尽可能减少了冗余序列,并与其它30多个数据建立了交叉引用,功能比较强大,官方网址为http://www.uniprot.org/[36],更新版本为UniProt release 2014_01,最近更新日期是2014年1月22日。
3结论与展望
近十几年来,随着高通量DNA自动测序技术的广泛应用,越来越多的物种包括动物、植物及微生物的基因组测序完成,DNA的数据量也以指数速度增长。但是常用的高通量测序仪如Solexa和SOLiD平台测出的序列读长都较短,需要进行拼接和注释才能得到完整的基因组信息,此刻生物信息学技术就显得尤为关键。相信在不久的将来,成本不断降低的高通量测序技术与数据处理能力不断提高的生物信息学技术能够更好的结合并成为一项常规的实验手段,成为促进整个生物学发展的强大动力。
[1]Peakall D,Shugar L.The human genome Projeet(HGP)[J].Ecotoxicology,2002,11(1):7-9
[2]International Human Genome Sequencing Consortium.Finishing the euchromatic sequence of the human genome[J].Nature,2004,431(7011):931-945
[3]Chan E Y.Advances in sequencing technology[J].Mutat Res,2005,573(1/2):13-40
[4]Mardis E R.Next-generation DNA sequencing methods[J].Annu RevGenomics Hum Genet,2008,9:387-402
[5]Schuster S C.Next-generation sequencing transforms todaypsbiology[J].Nat methods,2008,5:16-18
[6]解增言,林俊华,谭军,等.DNA测序技术的发展历史与最新进展[J].生物技术通报,2010(8):64-71
[7]Bowers J,Mitchell J,Beer E,et al.Virtual terminator nucleotides for next-generation DNA sequencing[J].Nat Methods,2009,6:593-595
[8]TesslerL A,Reifenberger J G,Mitra R D.Protein quantification in complex mixtures by solid phase single-molecule counting[J].Anal Chem,2009,81:7141-7148
[9]Pacific B,PacBio M.USA on World Wide Web URL:http://www. pacificbiosciences.com
[10]Roy R,Hohng S,Ha T.A practical guide to single-molecule FRET[J].Nat methods,2008,5(6):507-516
[11]Clarke J,Wu H C,Jayasinghe L,et al.Continuous base identification for single-molecule nanopore DNA sequencing[J].Nat Nanotechnol,2009,4:265-270
[12]Ashkenasy N,Sanchez-Quesada J,Bayley H,et al.Recognizing a single base in an individual DNA strand:a step toward DNA sequencing in nanopores[J].Angew Chem Int Ed Engl,2005,44(9):1401-1404
[13]Harris T D,Buzby P R,Babcock H,et al.Single-molecule DNA sequencing of a viral genome[J].Science,2008,320(5872):106-109
[14]Pastor W A,Pape U J,Huang Y,et al.Genome-wide mapping of 5-hydroxymethylcytosine in embryonic stem cells[J].Nature,2011,473(7347):394-397
[15]Goren A,Ozsolak F,Shoresh N,et al.Chromatin profiling by directly sequencing small quantities of immunoprecipitated DNA[J].Nat Methods,2010,7(1):47-49
[16]Astier Y,Braha O,Bayley H.Toward single molecule DNA sequencing:direct identification of ribonucleoside and deoxyribonucleoside 5'-monophosphates by using an engineered protein nanopore equipped with a molecular adapter[J].J Am Chem Soc,2006,128(5):1705-1710
[17]Eid J,Fehr A,Gray J,et al.Real-time DNA sequencing from single polymerase molecules[J].Science,2009,323(5910):133-138
[18]Flusberg B A,Webster D R,Lee J H,et al.Direct detection of DNA methylation during single-molecule,real-time sequencing[J].Nat. Methods,2010,7(6):461-465
[19]Hardin S,Gao X L,Briggs J,et al.Methods for real-time single molecule sequence determination[P].US Patent 7329492,2008
[20]Gupta P K.Single-molecule DNA sequencing technologies for future genomics research[J].Trends Biotechnol,2008,26(11):602-611
[21]Rhee M,Burns M.Nanopore sequencing technology:research trends and applications[J].Trends Biotechnol,2006,24(12):580-586
[22]Clarke J,Wu H C,Jayasinghe L,et al.Continuous base identification for single-molecule nanopore DNA sequencing[J].Nat Nanotechnol.,2009,4(4):265-270
[23]Stoddart D,Heron A J,Mikhailova E,et al.Single-nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore[J].Proc Natl Acad Sci U S A,2009,106(19):7702-7707
[24]Schadt E E,Turner S,Kasarskis A.A window into third-generation sequencing[J].Hum Mol Genet,2010,19(R2):R227-R240
[25]Cantor C R,Lim H A.Electrophoresis,Supercomputing and the Human genomes[M].World Scientific Publishing Co,1991
[26]Motoo K,Tomoko O.On Some Principles Governing Molecular Evolution[J].Proc Natl Acad Sci U S A,1974,71(7):2848-2852
[27]Wilbur W J,Lipman D J.Rapid similarity searches of nucleic acid and protein data banks[J].Proc Natl Acad Sci U S A,1983,80(3):726-730
[28]张春霆.生物信息学的现状与展望[J].世界科技研究与发展,2000,22(6):17-20
[29]Rudert F G,Ilag L.Functional genomicswith protein-protein interactions[J].Biotechnol Annul Rev,2000,5:45-86
[30]Barabasi A-L,Oltvai Z N.Network Biology:Understanding The Cell’s Function Organization[J].Nat Rev Genet,2004.5:101-113
[31]姜鑫.生物信息学数据库及其利用方法[J].现代情报,2005,25(6):185-187
[32]维斯特海德,帕里什,特怀曼.生物信息学(中译本)[M].北京:科学出版社,2004
[33]蒋彦,王小行,曹毅,等.基础生物信息学及应用[M].北京:清华大学出版社,2003
[34]钟杨,张亮,赵琼.简明生物信息学[M].北京:高等教育出版社,2001
[35]张晓东,张传富,彭科峰,等.生物信息学数据库研究进展[J].生物信息学,2006,4(3):143-145
[36]Berman H M,Westbrook J,Feng Z,et al.The Protein Data Bank[J].Nucleic Acids Res,2000,28(1):235-242
Development of the Third Generation Sequencing Technologies and Related Bioinformatics
YANG Yue,DU Xin-jun,LIANG Bin,GUO Ji-dong,CHENG Xiao-zhen,WANG Shuo*
(Key Laboratory of Food Nutrition and Safety,Tianjin University of Science and Technology,Tianjin 300457,China)
In the present study,the principles and applications of the third generation of DNA sequencing technology were summerized,as well as the progresses of bioinformatics involved genome sequencing.The third generation DNA sequencing technology was characterized by single DNA molecular and had been used in many fields of food science and life science research,for instance,SMS from Heliscope BioScience and SMRT from Pacific BioSciences.Meanwhile,the developement of bioinformatics and the main bioinformatics databases were summarized in the paper.
genomics;the third DNA sequencing technology;bioinformatics;database
10.3969/j.issn.1005-6521.2015.10.038
2014-01-28
杨悦(1984—),女(汉),博士研究生,研究方向:食品微生物。
王硕,男,教授,博士,研究方向:食品安全与食品微生物。

