新疆野生羊肚菌物种多样性研究
武冬梅,许文涛,谢宗铭,李全胜,祝建波,沈海涛,罗云波,*(.新疆农垦科学院生物技术研究所/作物种质创新与基因资源利用兵团重点实验室,新疆石河子832000;2.中国农业大学食品科学与营养工程学院,北京00083;3.农业部转基因生物食用安全监督检验测试中心(北京),北京00083;.石河子大学生命科学学院,新疆石河子832000)
新疆野生羊肚菌物种多样性研究
武冬梅1,2,许文涛2,3,谢宗铭1,李全胜1,祝建波4,沈海涛4,罗云波2,3,*
(1.新疆农垦科学院生物技术研究所/作物种质创新与基因资源利用兵团重点实验室,新疆石河子832000;2.中国农业大学食品科学与营养工程学院,北京100083;3.农业部转基因生物食用安全监督检验测试中心(北京),北京100083;4.石河子大学生命科学学院,新疆石河子832000)
根据子实体形态特征并结合rDNA ITS(Internal Transcribed Spacer)序列分析技术分析了根据形态特征挑选的、采自新疆伊犁、塔城、石河子、乌鲁木齐、阿勒泰五地区的31株野生羊肚菌子实体。rDNA ITS序列分析表明,采自新疆兵团185团且形态学分类为半开羊肚菌(Morchella semilibera)的2株菌株实为羊肚菌科常见属钟菌属的皱盖钟菌(Verpa bohemica)。以2株皱盖钟菌作为外类群,29株样品与来自GenBank的羊肚菌属各物种进行ITS序列聚类,可以分为四大聚类群,其中25株菌株与Morchella sp.Mel-17/19/20/34、Morchella sp.Mel-23/24/31/32、Morchella sp.Mel-13/26聚为第I类群;半开羊肚菌单独聚为第II类群;1株菌株与Morchella frustrata聚为第III类群;3株菌株与Morchella sp.Mes-6、Morchella sp.Mes-7、Morchella sp.Mes-17共同聚为第四类群。结合形态学特征,初步认为,上述五地区羊肚菌属至少由7个物种构成。根据国内最新研究资料,其中2种为中国新纪录种,分别为Morchella sp.Mes-17和Morchella frustrata(中立羊肚菌)。
新疆,野生羊肚菌,物种多样性,内转录间隔区(ITS)
羊肚菌属,隶属子囊菌门,盘菌纲,盘菌目,羊肚菌科[1],该属模式种是羊肚菌[2]。因其典型特征为具有蜂窝状或羊肚状的菌盖,故名羊肚菌。羊肚菌子实体肉质脆嫩,味道鲜美,是一种世界公认的珍稀、野生食药用真菌[3-4]。
新疆特殊的地理环境孕育了丰富的野生羊肚菌资源,赵震宇[5]记载新疆常见的羊肚菌有5种,分别是黑脉羊肚菌、圆锥羊肚菌、羊肚菌、小羊肚菌、皱柄羊肚菌;卯晓岚[6]记载新疆的5种羊肚菌分别是黑脉羊肚菌、尖顶羊肚菌、羊肚菌、粗腿羊肚菌、高羊肚菌;已有相关研究文献报道,新疆野生羊肚菌有小顶羊肚菌、高羊肚菌、小羊肚菌、尖顶羊肚菌、粗柄羊肚菌、黑脉羊肚菌、羊肚菌、离柄羊肚菌等[7-10]。
传统的羊肚菌属物种分类主要依据子实体的外观形状、菌丝、子囊、子囊孢子及侧丝的形态特征[11-12],如,根据菌盖的形状和颜色,菌盖与菌柄是否分离,菌盖表面棱纹的排列和颜色深浅,菌物学家将羊肚菌属分为3大类,即黑色羊肚菌类、黄色羊肚菌类和半开羊肚菌类[13-14]。在上述三个广义物种类群的基础上,根据成熟子囊果的子实体和菌柄是否变红提出了第4个类群,即变红羊肚菌类群,包括产于美洲热带和亚热带的3个种[2]。
有关新疆野生羊肚菌属的物种构成,无论5种,还是7种的分类记述,都是基于这种形态学分类界定的形态学物种。但因野生羊肚菌发育过程中具有形态特征随环境条件和个体发育年龄发生一定变化的复杂性和不稳定性,导致形态学分类具有不确定性,很容易导致出现同菌异名及同名异菌现象。
近年来,分子生物学技术的飞速发展带领了真菌分类和系统发育研究的革命性变革[15-16],DNA序列分析,特别是ITS序列分析技术,因其能实质性地反映出属间、种间及菌株直接的碱基对差异,已被广泛应用于真菌属内不同种间或近似属间的分类研究[17-25]。新疆的科研工作者也开展了基于ITS序列的野生羊肚菌分类研究,如苏俊等[26]对新疆伊犁霍城县大西沟采集的两种形态不同的野生羊肚菌进行分子水平的分类学鉴定,结果与传统形态学鉴定结果不一致;刘伟等[27]的研究结果也表明,两地区羊肚菌形态学上的差异是由于环境或发育不同阶段的差异造成的;冯丽等[28]利用ITS序列分析技术确定采自新疆哈密松树塘的野生羊肚菌为肋脉羊肚菌。
上述这种分子水平的研究对确定新疆不同地区羊肚菌种类并正确认识和利用野生羊肚菌具有重要的理论意义。本研究以2011~2013年间自新疆野生羊肚菌主产区伊犁、塔城、石河子、乌鲁木齐、阿勒泰等地区采集到的部分野生羊肚菌为材料,提取子实体基因组DNA进行ITS序列比对分析,构建进化树,进一步探讨新疆野生羊肚菌的物种多样性,旨在较为准确地确定各地区分布的物种,进而为野生羊肚菌人工栽培及进一步的开发利用提供科学依据。
1 材料与方法
1.1材料与仪器
野生羊肚菌子实体分别于2011~2013年采自新疆伊犁、塔城、石河子、乌鲁木齐、阿勒泰等地区,共197份样品,分别命名为M1~M197,根据Bunyard[29]的分类方法,按照子实体形态差异、色泽等特征将样品分为3个大类群,黑色羊肚菌(黑脉羊肚菌、尖顶羊肚菌、高羊肚菌)、半开羊肚菌、黄色羊肚菌(羊肚菌、粗柄羊肚菌)。按照不同地区及形态特征的典型性分别从中选取2~3份样品作为ITS序列分析样品,共计31份,样品编号、来源、形态学分类鉴定结果见表1; 2×Easy Taq Mix、液氮、2×CTAB DNA提取液、裂解液北京康为世纪生物公司;SanPrep柱式DNA胶回收试剂盒、蛋白酶K溶液(20mg/mL)、β-巯基乙醇(100mg/mL)、RNA酶(10mg/mL) 上海生工生物工程有限公司。
MM400组织破碎仪德国莱弛,用于样品的破碎;DK-S24水浴锅上海精宏;德国IKA涡旋振荡器MS1 minishaker,用于样品的混匀;Z-16KC冷冻离心机德国Sigma,用于DNA提取时冷冻离心;PCR仪Bio-Rad,Thermal cycle,用于DNA扩增;T2A电泳仪凝胶成像系统美国Bio-Rad,用于电泳凝胶图像的分析。
1.2实验方法
1.2.1野生羊肚菌子实体基因组DNA提取及琼脂糖凝胶电泳检测CTAB法提取野生羊肚菌子实体基因组DNA:分别称取0.12~0.15g野生羊肚菌子实体样品于2.0mL离心管中,于液氮中冷冻2min,置于组织破碎仪中研磨45s至粉末状;迅速加入1000μL 2×CTAB DNA提取缓冲液、β-巯基乙醇20μL,蛋白酶K10μL,涡旋混匀,冰浴保存10min后4℃,12000r/min离心10min;弃上清,沉淀中加2×CTAB DNA裂解液(65℃预热)500μL,β-巯基乙醇20μL,蛋白酶K10μL,混匀后置65℃水浴抽提30min,每10min翻转一次,以保证充分裂解;冷却,加入500μL氯仿∶异戊醇(24∶1)混合液,12000r/min离心10min;取上清,冷却,加入500μL氯仿∶异戊醇(24∶1)混合液,12000r/min离心10min;取上清,加入0.6倍体积预冷的异丙醇,混匀,静置10min,12000r/min离心10min;弃上清,沉淀用75%的乙醇洗涤两次,10000r/min离心10min;弃上清,通风干燥,40μL ddH2O溶解DNA,加入3μL RNA酶,37℃水浴消化30min后用1.0%的琼脂糖凝胶电泳检测。以ddH2O将获得的DNA稀释10倍备用。
1.2.2ITS区扩增及克隆测序采用White[21]设计的真菌核糖体基因间隔区(ITS)通用引物ITS1/ITS4分别对31株供试材料rDNA ITS序列进行PCR扩增。引物序列如下:ITS1:TCCGTAGGTGAACCTGCGG;ITS4:TCCTCCGCTTATTGATATGC。引物由上海生工生物工程技术服务有限公司合成。
ITS-PCR扩增反应体系(20μL)为:2×Easy Taq Mix 10μL,ITS1(10μmol/L)0.8μL,ITS4(10μmol/L)0.8μL,模板DNA 0.6μL,ddH2O 7.8μL。反应程序为:95℃预变性4min,95℃变性30s,57℃复性30s,72℃延伸60s,31个循环后72℃延伸7min结束反应。
PCR产物经1.0%琼脂糖凝胶电泳检测,分别用DNA凝胶回收试剂盒分别回收纯化目的片段,将扩增产物连接到pUCm-T载体上,转化大肠杆菌,转化子经PCR鉴定后委托上海生工生物工程技术服务有限公司完成测序。
汉中职业技术学院肖宁的《学前教育专业校企合作共建机制的探索与实践》一文中提出“共建人才培养方案、共建实践教学基地、共建课程、共建课堂”四建的全新观点。在这当中校企、校园要共同参与人才培养的测评及教学管理的工作,共同制定考核标准与行业准入标准,设定相似的教学环境与工作环境,统一教学内容和工作任务,了解教师与岗位工作者的要求,切实落实完善“校企共育,全程实践”的人才培养模式。
1.2.3序列分析与系统进化树的构建31株供试材料测序后分别利用DNAStar Lasergene.v 7.1软件包中的Editseq进行处理,得到ITS片段全序列(18s+ITS1+ 5.8S+ITS2+28s)信息。将获得的ITS全序列剔除两端的载体序列、引物序列及18S、28S序列,使序列信息尽可能一致。利用上述软件计算各序列间的相似性、序列趋异度,MEGA 5.0软件计算样品遗传距离。利用NCBI数据库中BLAST程序进行序列同源性比较。
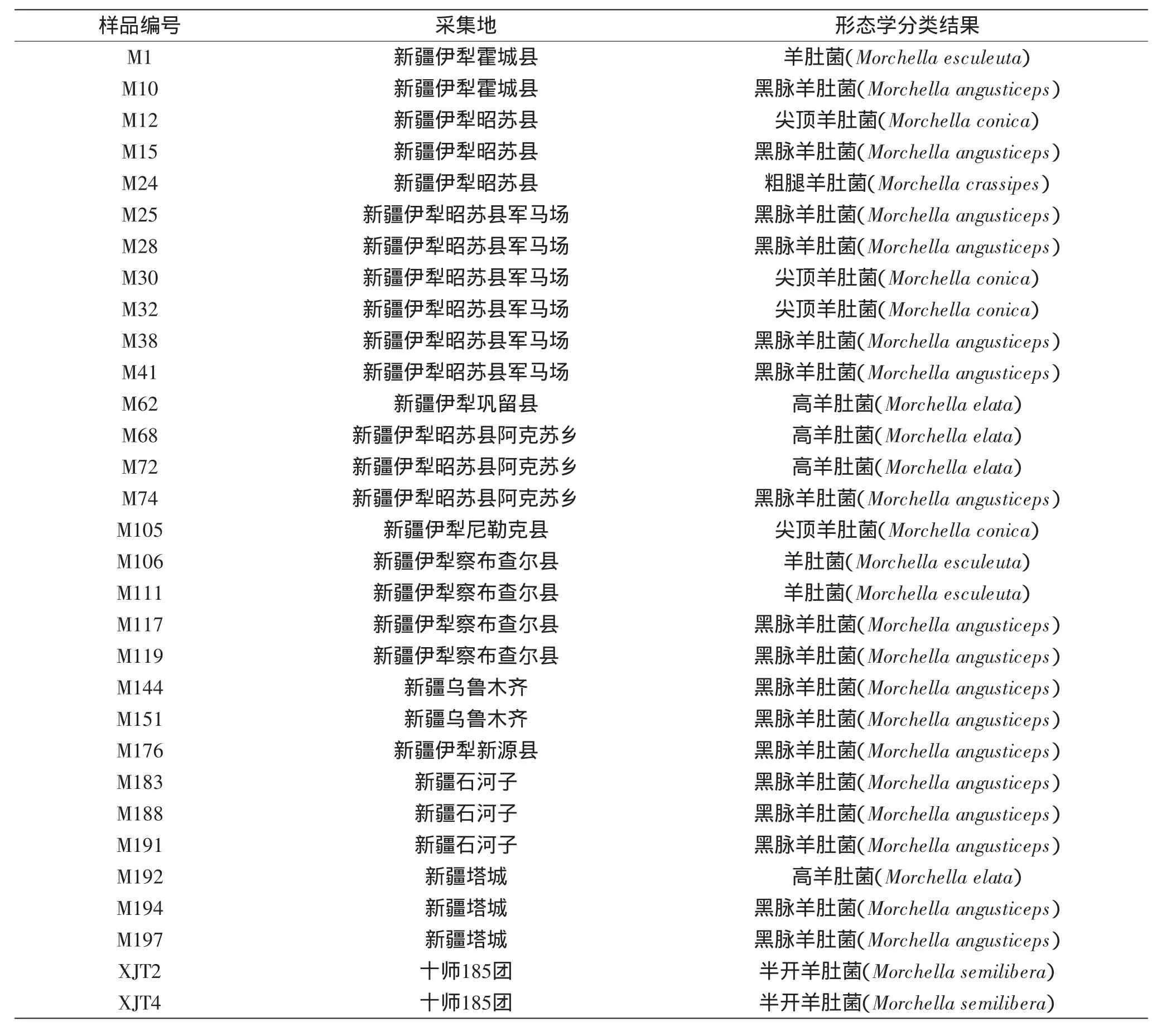
表1 材料及来源Table 1 Materials and source
鉴于相关的研究报道[30],Genbank内至少有66%的羊肚菌ITS序列的物种名称是错误的,同物异名和异物同名的现象非常严重,不能以GenBank中的ITS序列作为判定标准,本研究所用比对分析序列全部为杜习慧等[30]提交的正确序列。用MEGA 5.0中的Alignment程序对所测得的ITS序列进行多重对位排列,并手动删除对位排列结果中的非对位排列序列。对位排列结果中的空位或缺失数据作pair-wise处理,分析时所用参数均为系统默认值。以采自新疆生产建设兵团十师185团的皱盖钟菌XJT2、XJT4分别作为构建系统进化树时外类群菌株,采用邻位相连法[31]构建系统发育树,利用自展法(Bootstrap,1000次重复)检验各分支的置信度[32]。
2 结果与分析
2.1野生羊肚菌子实体基因组DNA提取及ITS序列PCR扩增测序
由于野生羊肚菌中多糖、胶质等次生代谢物含量较高,容易与DNA吸附,给基因组DNA的提取和纯化带来一定困难。研究中采用改进的CTAB法,电泳结果显示该法可以顺利提取出羊肚菌子实体基因组DNA(图1、图2)。
分别以31株供试样品基因组DNA为模板,采用通用引物ITS1/ITS4进行ITS序列扩增,均得到清晰的、与预期大小一致的特异性条带。其中,有25个样品的ITS序列在750bp左右,1个样品的ITS序列在1000bp左右,3个样品的ITS序列在1200bp左右,2个样品的ITS序列在800bp左右(图3~图4)。该结果与Wipf等[13]的研究结果一致,即黑色羊肚菌的ITS序列长度在740~750bp之间;黄色羊肚菌的ITS序列在1150~1220bp之间。
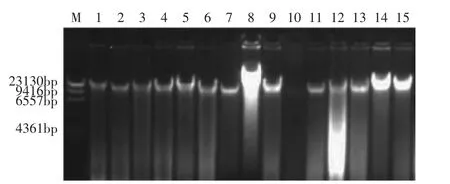
图1 野生羊肚菌基因组DNAFig.1 Wild Morchella genome DNA
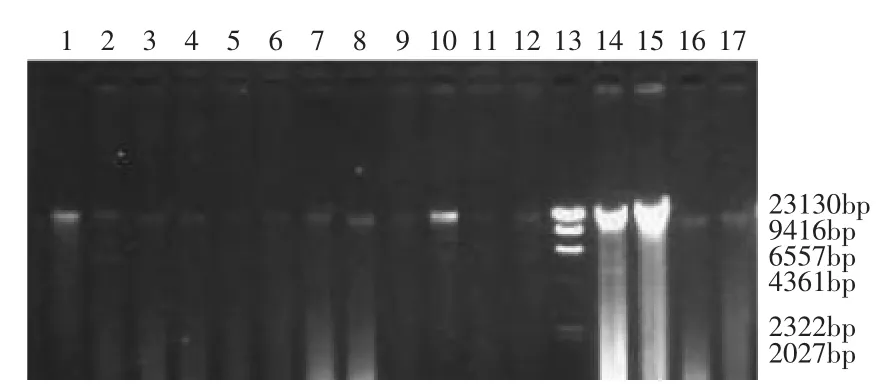
图2 野生羊肚菌基因组DNAFig.2 Wild Morchella genome DNA
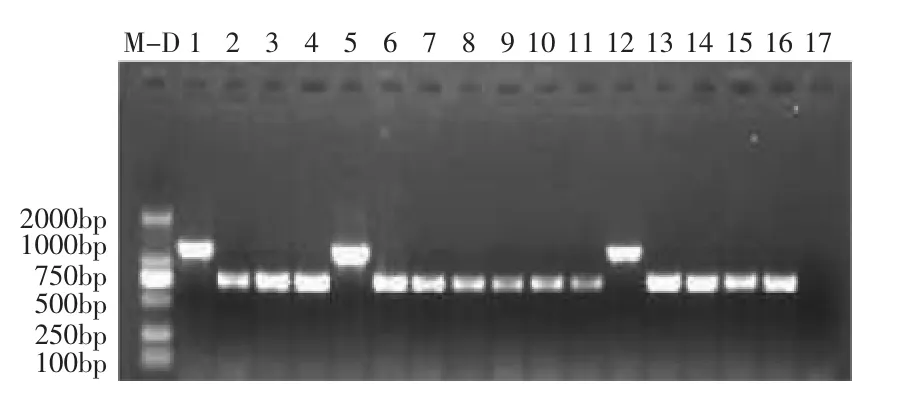
图3 基因组DNA ITS序列扩增Fig.3 Amplification of ITS from genome

图4 基因组DNA ITS序列扩增Fig.4 Amplification of ITS from genome
PCR扩增产物分别经SanPrep柱式DNA胶回收试剂盒回收纯化后连接至pUCm-T载体,转化子经PCR鉴定,均得到与预期大小一致的特异性目的片段。结果表明,已分别克隆了31份样品ITS序列。所得转化子经测序后均全部获得ITS区全序列信息。
2.2ITS序列分析
31份样品中,种内趋异度较小,为0.1~0.3;种间趋异度较大,如M1和M194序列趋异度达66.8。M1、M24、M62、M194、XJT2、XJT4 6份样品的序列相似率为56.1%~70.5%,其余样品序列相似率在97.1%~100.0%之间。31份样品的遗传距离为0.0000~0.5067,其中26份遗传距离为0.0000~0.0255;XJT2、XJT4为钟菌属,与其他材料遗传距离较大,为0.5067;M1、M24和M62、M194与其他材料的最大遗传距离分别为0.3507、0.3353、0.3166。遗传距离参数能够反映种间的亲缘关系。
利用NCBI数据库中BLAST程序进行序列同源性比较,结果显示,31份供试材料中,M10、M12、M25、M30等16份材料ITS序列与数据库中收录的Morchella sp.Mel-17/19/20/34序列相似率达99%~100%;M15、M72、M111、M151等7份材料ITS序列与Morchella sp. Mel-23/24/31/32序列相似率达99%;M188、M106与Morchella sp.Mel-13/26序列相似率达99%~100%;M24、M62与Morchella sp.Mes-6、Morchella sp.Mes-7序列相似率99%;M194与Morchella frustrata(中立羊肚菌)序列相似率达99%;M1与Morchella sp.Mes-17序列相似率达99%;XJT2、XJT4则与Verpa bohemica(皱盖钟菌)序列相似率达99%。
2.3基于ITS序列的系统发育树构建与分析
31份供试材料ITS序列、自NCBI数据库下载的同源性较高的序列及实验材料中没有涉及的羊肚菌属其他物种ITS序列,以皱盖钟菌Verpa bohemica作为外类群,采用邻位相连法分别构建系统发育树,所显示的进化树如图5所示。
从图5系统发育树可以看出,以采集的皱盖钟菌为外类群,其余29份样品与下载自NCBI数据库中的尖顶羊肚菌、粗柄羊肚菌、半开羊肚菌、羊肚菌进行聚类,可以分为四大聚类群。M25、M30等25份样品序列差异较小,亲缘关系较近,与来自数据库的Morchella sp.Mel-17/19/20/34、Morchella sp.Mel-23/ 24/31/32、Morchella sp.Mel-13/26所代表的不同物种,以99%的Bootstrap值共同聚在第I类群;半开羊肚菌单独聚为第II类群;样品M194以99%的Bootstrap值与Morchella frustrata聚为第III类群;来自NCBI的羊肚菌,粗柄羊肚菌,海绵羊肚菌、Morchella sp.Mes-17与供试样品M1、M24、M62共同聚类在第IV类群,但样品M1与Mes-17亲缘关系近,M24、M62则与Morchella sp.Mes-6亲缘关系更近。
3 结论
综合利用形态学分类方法、rDNA ITS序列分析技术分析确定采集自新疆伊犁、塔城、石河子、乌鲁木齐、阿勒泰等地区的野生羊肚菌的归属,探讨新疆野生羊肚菌物种多样性,为新疆野生羊肚菌保护及开发利用提供帮助。
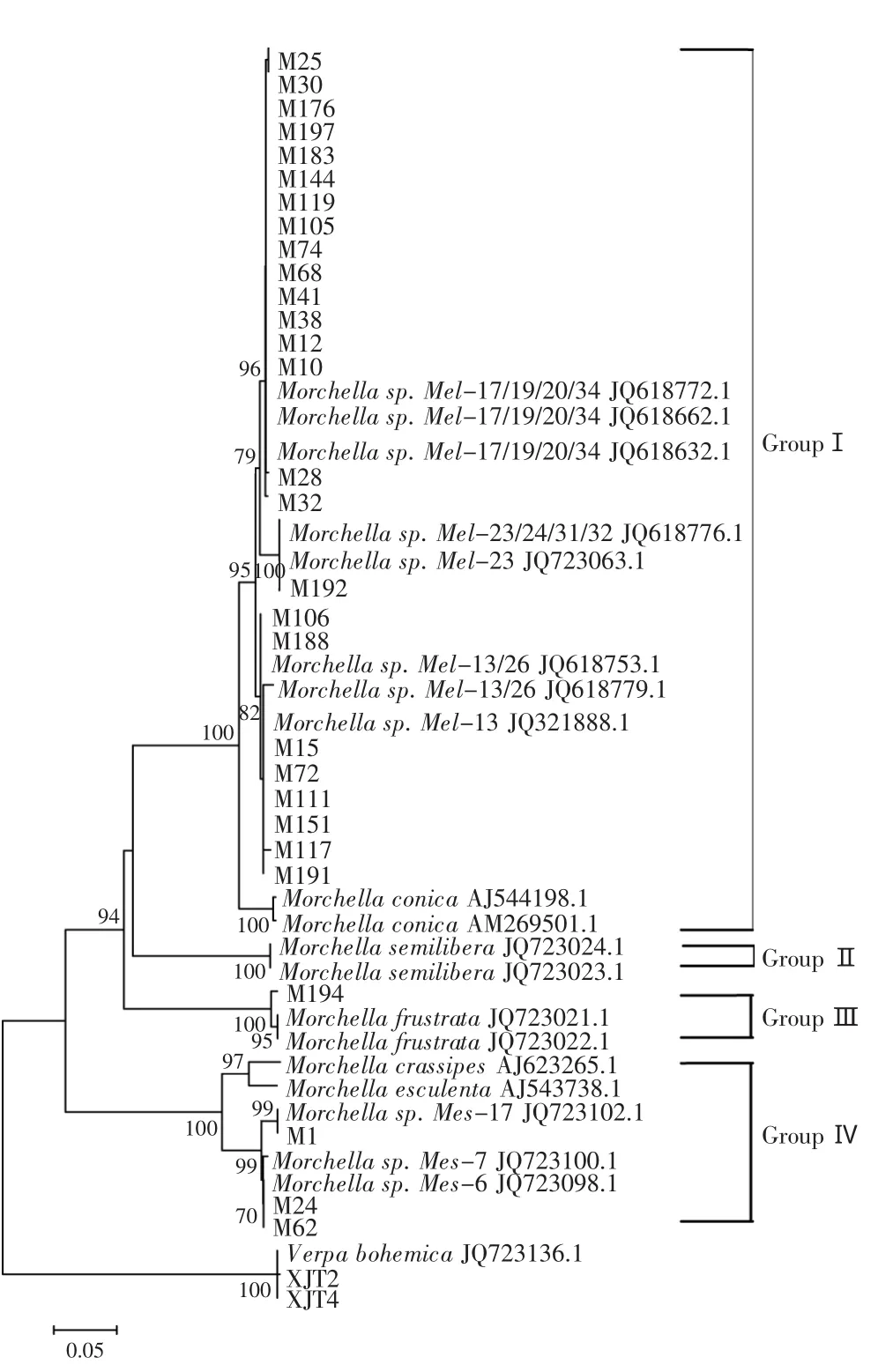
图5 基于ITS序列构建的羊肚菌系统发育树Fig.5 Phylogenetic trees based on rDNA ITS sequence
根据分析结果初步判定上述五地区的野生羊肚菌由黑色羊肚菌和黄色羊肚菌两大类群构成,黑色羊肚菌物种构成为Morchella sp.Mel-17/19/20/34、Morchella sp.Mel-23/24/31/32、Morchella sp.Mel-13/ 26、Morchella frustrata(中立羊肚菌);黄色羊肚菌物种构成为Morchella sp.Mes-17、Morchella sp.Mes-6、Morchella sp.Mes-7。研究显示,迄今为止,羊肚菌属系统发育种中只有20个系统发育种有各自的独特形态特征,可以用形态特征区分,并有相应的中文名及拉丁名[2],其他物种都有待进一步研究和命名,因此,本研究根据rDNA ITS序列对新疆野生羊肚菌归属仅进行了初步的分析,还不能界定到具体的物种并正确命名。但结合形态学特征,可以认为,上述五地区至少分布有7种野生羊肚菌。且根据最新研究资料[33],7个物种中,有5种在前人的研究中有过报道,Morchella frustrata(中立羊肚菌)及Morchella sp.Mes-17在中国尚未有报道,初步判定为新种,正在做进一步的鉴定。
另外,31份样品中,采集自新疆兵团185团的样品,根据形态学鉴定为半开羊肚菌,分子鉴定结果显示,该菌株实为羊肚菌科常见的钟菌属物种皱盖钟菌。
4 讨论
野生羊肚菌发育过程中形态特征随环境条件和个体发育年龄发生一定程度的变化,根据传统的分类方法很难对其进行准确的分类鉴定。目前主要采用ITS序列分析技术进行鉴定。
尽管目前ITS序列分析技术已经成为真菌界广泛应用的一个分子标记,已经成功应用于对松茸、松乳菇等珍稀食用菌纯培养菌种的鉴定[34-36],为菌种的鉴定及系统发育分析提供了重要依据。但是,近年来,对于Genbank中序列的可靠性,部分学者持怀疑态度,认为至少有66%的羊肚菌ITS序列的物种名称是错误的,同物异名和异物同名的现象非常严重[33,37-41]。本研究中进行同源序列聚类时也发现存在聚类混乱现象,因此,仅仅依靠ITS序列的序列分析就略显不够严谨。
鉴于以上原因,在运用ITS序列分析技术的同时,如果再辅以RPB1、RPB2、EF1a等基因片段,可能会更准确、全面。因此,本研究将在物种多样性研究的基础上,进一步结合RPB1、RPB2、EF1a等基因片段做进一步的分析,以使结果更加完善,这不仅可以丰富新疆野生羊肚菌多样性的菌种资源库信息,还可以为野生羊肚菌的人工栽培及进一步开发利用提供科学依据。新疆地大物博,物种多样性丰富,羊肚菌属的分类研究任重而道远。
[1]Hibbett D,Binder M,Bischoff JF,et al.A higher-level phylogenetic classification of the Fungi[J].Mycological Research,2007,111(5):509-547.
[2]杜习慧,赵琪,杨祝良.羊肚菌的多样性、演化历史及栽培研究进展[J].菌物学报,2014,33(2):1-16.
[3]Royse DJ,May B.Interspecific allozyme variation among Morchella spp and its inferences for systematics within the genus[J].Biochemical Systematics and Ecology,1990,18(7-8):475-479.
[4]戴玉成,杨祝良.中国药用真菌名录及部分名称的修订[J].菌物学报,2008,27:801-824.
[5]赵震宇.新疆食用菌志[M].乌鲁木齐:新疆农业出版社,1982.
[6]卯晓岚.中国大型真菌[M].郑州:河南科学技术出版社,2000:595-600.
[7]谢占玲,谢占青.羊肚菌研究综述[J].青海大学学报:自然科学版,2007,25(2):36-40.
[8]兰进,曹文芩,徐锦堂.中国羊肚菌属真菌资源[J].资源科学,1999,21(2):56-61.
[9]王纯利,努尔古丽.新疆羊肚菌研究开发进展及其前景[J].新疆农业科技,2004(4):39.
[10]张广伦,肖正春,赵培忠.新疆野生粗柄羊肚菌的化学成分分析[J].江苏食用菌,1992(5):21-22.
[11]戴芳澜.中国真菌总汇[M].北京:科学出版社,1979:238-239.
[12]沈洪,陈明杰,赵永昌,等.云南羊肚菌rDNA的ITS序列与亲缘关系分析[J].食用菌学报,2007,14(2):15-18.
[13]Wipf D,Munch J C,Botton B.DNA phylogenies in morels:complete sequences of the internal transcribed spacer of genes coding for rRNA in Morchella esculent(yellow morel)and Morchella conia(black morel)[J].Applied and Environmental Microbiology,1996,62(9):3541-3543.
[14]Guzmán G,Tapia F.The known morels in Mexico,a description of a new blushing species,Morchella rufobrunnea,and new data on M.guatemalesis[J].Mycologia,1998,90(4):705-714.
[15]Yang ZL.Molecular techniques revolutionize knowledge of basidiomycete evolution[J].Fungal Diversity,2011,50:47-58.
[16]杨祝良.基因组学时代的真菌分类学:机遇与挑战[J].菌物学报,2013,32(6):931-946.
[17]陈剑山,郑服从.ITS序列分析在真菌分类鉴定中的应用[J].安徽农业科学,2007,35(13):3785-3786.
[18]李海波,吴学谦,王立武,等.青藏高原黄绿蜜环菌纯培养菌种的分离培养及分子鉴定[J].菌物学报,2008,27(6):873-883.
[19]林晓民,李振岐,王少先.真菌rDNA的特点及在外生菌根菌鉴定中的应用[J].西南农业学报,2005,14(2):120-125.
[20]刘文丛,刘颖,郭相,等.滇西北地区羊肚菌的分子鉴定及ITS序列分析[J].江苏农业科学,2012,40(2):31-34.
[21]White T J,Bruns TD,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics In PCR Protocols:A guide to methods and applications[J].New York:Academic Press,1990,315-322.
[22]Bruns TD,White TJ,Taylor JW.Fungal molecular systematics[J].Annual Review of Ecology and Systematics,1991,22:525-564.
[23]Geml J,Laursen GA,Timling I,et al.Molecular phylogenetic biodiversity assessment of arctic and boreal ectomycorrhizal Lactarius Pers.(Russulales;Basidiomycota)in Alaska,based on soil and sporocarp DNA[J].Molecular Ecology,2009,18(10):2213-2227.
[24]Li HB,Wu XQ,Wang LW,et al.Pure culture isolation,cultivation and molecular identification of Armillaria luteovirensfrom Tibet Plateau[J].Mycosystema,2008,27(6):873-883.
[25]Tedersoo L,Suvi T,Jairus T,et al.Revisiting ectomycorrhizal fungi of the genus Alnus:differential host specificity,diversity and determinants of the fungal community[J].The New Phytologist,2009,182(3):727-735.
[26]苏俊,孜来古丽·米吉提,艾尔肯·热合曼,等.两种新疆羊肚菌的分类学鉴定[J].新疆农业科学,2011,48(9):1704-1710.
[27]刘伟,冯丽,张亚平.新疆羊肚菌rDNA的ITS序列分析[J].石河子大学学报:自然科学版,2009,27(5):584-587.
[28]冯丽,刘伟,黄建峰,等.松树塘羊肚菌生境调查及分子鉴定[J].中国食用菌,2009,28(5):34-36.
[29]Bunyard B A,Nicholson M S,Royse D J.Phylogeny of the genus Agaricus inferred from restriction analysis of enzymatically amplified ribosomal DNA[J].Fungal Genetics and Biology,1996,20(4):243-253.
[30]Du XH.How well do ITS rDNA sequences differentiate species of true morels(Morchella)[J].Mycologia,2012b,104:1351-1368.
[31]Saitou N,Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Molecular Biology and Evolution,1987,4(4):406-425.
[32]Felsenstein J.Confidence limits on phylogenies:an approach using the bootstrap[J].Evolution,1985,39:783-791.
[33]杜习慧.羊肚菌属分子系统学和生物地理学研究-兼论该属两个物种的群体遗传学[D].北京:中国科学院,2012.
[34]张徽思,罗孝坤,张丽英,等.松茸菌丝体的纯培养及其鉴定[J].中国食用菌,2010,29(3):34-36.
[35]熊涛,肖满,曾哲灵,等.松乳菇组织分离菌株rDNAITS序列分子鉴定[J].微生物学通报,2006,33(4):1-4.
[36]孙晓琳,商圆圆,张恩奎,等.基于ITS序列分析鉴定栓孔菌属物种[J].食品与发酵科技,2013,49(2):85-88.
[37]Brock PM,Döring H,Bidartondo MI.How to know unknown fungi:the role of a herbarium[J].New Phytologist,2009,181:719-724.
[38]Harris DJ.Can you bank on GenBank[J].Trends in Ecology and Evolution,2003,18:317-319.
[39]Xi-Hui Du,Qi Zhao,Kerry O’Donnell,et al.Multigene molecular phylogenetics reveals true morels(Morchella) are especially species-rich in China[J].Fungal Genetics and Biology,2012,49:455-469.
[40]Vilgalys R.Taxonomic misidentification in public DNA databases[J].New Phytologist,2003,160:4-5.
[41]Nilsson RH,Kristiansson E,Ryberg M,et al.Intraspecific ITS variability in the kingdom fungi as expressed in the international sequence databases and its implication for molecular species identification[J].Evol Bioinfor,2008,4:193-201.
Study on species diversity of wild Morchella in Xinjiang
WU Dong-mei1,2,XU Wen-tao2,3,XIE Zong-ming1,LI Quan-sheng1,ZHU Jian-bo4,SHEN Hai-tao4,LUO Yun-bo2,3,*
(1.Biotechnology Research Institue,Xinjiang Academy of Agricultural and Reclamation Sciences/Xinjiang Production& Construction Group Key Laboratory of Crop Germplasm Enhancement and Gene Resources Utilization,Shihezi 832000,China;2.College of Food Science and Nutrition Engineering,China Agricultural University,Beijing 100083,China;3.TheSupervision,Inspection&TestingCenterofGeneticallyModifiedFoodSafety,MinistryofAgriculture,Beijing100083,China;4.College of Life Sciences of Shihezi University,Shihezi 832000,China)
Thirty-one wild Morchella specimens,which were collected from five different areas in Xinjiang and selected on the basis differences in fruit body morphology,then they were identified by morphological characteristics and internal transcribed spacer sequence analysis respectively.The results showed that two strains belonged to Verpa bohemica,not Morchella semilibera,which collected from 185 regiment in Xinjiang. When the two Verpa bohemica strains were taken as outgroup,phylogenetic trees were produced by the ITS sequences of the twenty-nine strains and other species of Morchella from GenBank.Phylogenetic analysis indicated that the twenty-five strains and Morchella sp.Mel-17/19/20/34、Morchella sp.Mel-23/24/31/32、Morchella sp.Mel-13/26 formed the first cluster.Morchella semilibera belonged to the second cluster.One strains and Morchella frustrata formed the third cluster.The fourth cluster included three strains and Morchella sp.Mes-6、Morchella sp.Mes-7 and Morchella sp.Mes-17.Morphological characteristics and phylogenetic analysis indicated that there were at least seven species of Morchella in these five area of Xinjiang.According to recent research,Morchella sp.Mes-17 and Morchella frustrate were two new species of Morchella in China. Key words:Xinjiang;wild Morchella;species diversity;internal transcribed spacer sequence
TS201.1
A
1002-0306(2015)02-0167-06
10.13386/j.issn1002-0306.2015.02.027
2014-05-06
武冬梅(1976-),女,硕士研究生,副研究员,主要从事应用微生物学及生物技术应用方面的研究。
罗云波(1960-),男,博士,教授,主要从事食品安全、食品生物技术、果蔬贮藏保鲜方面的研究。
国家自然科学基金资助项目(31160011);国家星火计划项目(2013GA.891001);石河子大学科学技术研究发展计划(2012ZRKXYQ-YD15)。

