植物凝集素结构多样性与药物设计
赵蕴杰,刘志超1222,刘琴,陈高,陈禅友,曾辰
1.乔治华盛顿大学物理系,美国华盛顿哥伦比亚特区20052 2.江汉大学生命科学学院,武汉430056
*论著——生物信息技术*
植物凝集素结构多样性与药物设计
赵蕴杰1,2,刘志超12221,2,刘琴,陈高,陈禅友,曾辰
1.乔治华盛顿大学物理系,美国华盛顿哥伦比亚特区20052 2.江汉大学生命科学学院,武汉430056
植物凝集素蛋白最早发现于19世纪,可特异性结合碳水化合物与糖分子。植物凝集素蛋白结合碳水化合物的高度特异性使我们可以将其作为生物工具应用到不同领域。例如,植物凝集素的重要特征之一就是可以特异性结合昆虫消化道表面的糖分子,从而起到抗虫的作用。植物凝集素也可以特异性结合癌细胞膜表面的糖分子,从而导致细胞凋亡或抑制肿瘤的生长。研究表明,仅豆科植物的凝集素分子表面就有大约40万个潜在的结合位点可以特异性结合不同的糖分子。因此,研究植物凝集素的结构特征信息对更好地理解植物凝集素的结构功能关系与应用有重要的意义。近年来,计算生物学的飞速发展使我们可以在分子层次上研究植物凝集素。本文主要从计算生物学角度综述植物凝集素的结构,序列保守性与动力学等研究现状,并以菜豆凝集素为例讨论植物凝集素的药物设计和医学应用。
植物凝集素;三级结构;进化;药物设计;医学应用
植物凝集素最早发现于19世纪80年代,至今已有一百多年的历史,因其凝集红细胞与特异性结合碳水化合物的能力,具有非常广泛的用途[1-4]。例如,昆虫和高等动物的消化道表面有很多细胞膜糖蛋白,植物凝集素与其特异性结合会引起昆虫和高等动物的不适,从而起到保护植物的作用。植物凝集素也会与器官暴露在上皮细胞的糖分子特异性结合,利用植物凝集素特异性结合碳水化合物的特性,可以将其作为生物粘附剂输送药物到特定靶标[5-7]。另外,植物凝集素还可以刺激细胞有丝分裂。研究表明,仅豆科植物的凝集素分子表面就有大约40万个潜在的结合位点,植物凝集素具有非常广泛的应用前景[8,9]。因此,研究植物凝集素的结构和功能关系,如何利用植物凝集素的结构和生物学特性逐渐成为了热点研究问题。
2013年诺贝尔化学奖授予了Martin Kaplus, Michael Levitt和AriehWarshel以表彰他们在发展多尺度复杂化学系统模型方面所做的贡献[10,11]。近年来,计算生物学领域的飞速发展给我们提供了一种可以理解和捕捉生物实验中涉及几千甚至几百万原子协同发生化学反应的工具,尤其是分子力场和分子动力学模拟的不断发展让我们可以捕捉生物分子在原子层次上的动态行为[12-17]。计算生物学在植物凝集素领域也有非常广泛的应用,例如Surolia等用分子动力学模拟研究香蕉凝集素二聚体在不同温度下的结构稳定性情况[18,19]。香蕉凝集素是一种同源二聚体的非糖基化蛋白,高温动力学模拟结果表明,香蕉凝集素二聚体接触面的三级结构相互作用对二聚体结构的稳定至关重要。在400~500K的高温动力学模拟中,该二聚体结构仍然十分稳定,仅仅在无规卷曲有较大的柔性变化。Surolia等进一步模拟了糖基化劲直刺桐凝集素,模拟结果表明糖基化劲直刺桐凝集素二聚体接触面上三级相互作用的动力学特征和静态结构中观察到的三级结构相互作用特征有较大不同[20]。因此,综合考虑植物凝集素的静态与动力学特征对深刻理解植物凝集素的结构与生物功能关系,设计生物粘附剂输送药物分子和设计抑制剂降低植物凝集素毒性等应用均有非常大的帮助。
本文主要从计算生物学的角度综述植物凝集素的现状及其应用,主要内容有:(1)总结蛋白质结构数据库(Protein Data Bank,简称PDB)中植物凝集素的三级结构特征;(2)以菜豆凝集素为例分析植物凝集素的序列保守性,静态结构与动力学特征;(3)以菜豆凝集素为例讨论如何设计抑制剂降低植物凝集素的凝血毒性。
1 植物凝集素三级结构特征
植物凝集素需要折叠成特定的三级结构才能实现其生物学功能,因此充分了解植物凝集素的三级结构特征对理解其生物学功能至关重要。蛋白质三级结构数据库是目前最主要的收集生物大分子三级结构的数据库,通过X射线晶体衍射、核磁共振、电子衍射和冷冻电镜等实验手段测定蛋白质、核酸等生物大分子的三级结构[21]。目前,蛋白质三级结构数据库共收集了106710个生物分子结构数据。本章节我们将结合蛋白质三级结构数据库中测定的植物凝集素实验结构讨论植物凝集素的三级结构特征。
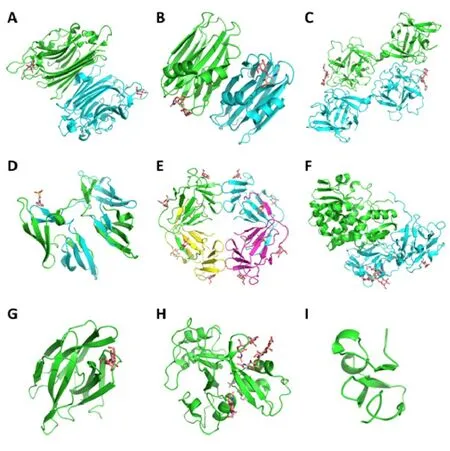
图1 植物凝集素蛋白质三级结构示意图不同颜色分别表示二聚体或多聚体中不同的蛋白质链或糖分子Fig.1 Cartoon representations of lectin proteins from different fold families. Dimer structures are colored in blue and green. Sugars are colored in orange. (A) Legume lectin (PDB code: 3WCR). (B) Agaricusbisporus agglutinin homologs (PDB code: 1Y2V). (C) Amaranthins (PDB code: 1JLX). (D) Cyanovirin family (PDB code: 1L5B). (E) Galanthusnivalis agglutinin (PDB code: 1MSA). (F) Ricin-B (PDB code: 1HWM). (G) Jacalin (PDB code: 1C3M). (H) LysM (PDB code: 4EBY). (I) Hevein (PDB code: 1HEV).
研究表明,植物凝集素可根据序列和糖分子结合位点结构分为12类结构[22]。豆科植物凝集素(Legume lectin)广泛分布在如菜豆,刀豆,黄豆,豌豆,扁豆,花生等植物中,可特异性识别半乳糖,甘露糖,葡萄糖等糖分子。图1A为菜豆凝集素的结构,该单体结构为一个7条β折叠片形成的β折叠层和6条β折叠片形成的β折叠层通过β转角连接组成,单体长度一般为250个氨基酸左右,并进一步形成稳定的二聚体结构(PDB code:3WCR)[23]。ABA(Agaricusbisporus agglutinin homologs)植物凝集素单体一般由140个氨基酸左右的序列组成,可特异性识别T antigen和N-glycans等分子。图1B为ABA植物凝集素结构(PDB code: 1Y2V)[24],该凝集素单体为一个4条β折叠片形成的β折叠层和6条β折叠片形成的β折叠层通过螺旋-环-螺旋的模体结构连接组成,并进一步形成二聚体结构,T antigen结合在螺旋-环-螺旋模体结构区域。Amaranthins植物凝集素主要分布在苋属植物中,可特异性结合T antigen和GalNAc等分子。图1C为Amaranthins植物凝集素结构,该单体结构由两个150个氨基酸左右的β-trefoil通过α螺旋串联组成,并进一步通过头-尾相互作用形成稳定的二聚体结构(PDB code: 1JLX)[25]。CVN(Cyanovirin family)植物凝集素最早在蓝藻中发现,因其与人类免疫缺陷病毒(HIV)表面的糖分子有较高亲和性,是潜在的抗病毒分子工具。图1D为CV-N凝集素结构,该单体结构主要由4个β折叠层连接组成(101个氨基酸左右),并进一步通过头-尾相互作用形成稳定的二聚体结构(PDB code:1L5B)[26]。GNA(Galanthusnivalis agglutinin)植物凝集素最早从雪莲花中发现,可特异性结合甘露糖分子。实验研究表明,GNA凝集素的空间拓扑结构非常保守,主要由3个β折叠层(每个β折叠层均有一个甘露糖结合位点)形成β-prism结构,凝集素单体一端插入另外一个单体形成β折叠层进而形成二聚体,并进一步形成稳定的四聚体结构(图1E,PDB code:1MSA)[27]。Ricin-B植物凝集素最早在蓖麻中发现,为最早发现的植物凝集素,该凝集素可以特异性结合GalNAc等分子。晶体结构研究表明,典型的Ricin-B凝集素单体为两个串联的重复结构域组成,每个结构域为三对称的β-trefoil结构(图1F,PDB code:1HWM)[28]。Jacalin植物凝集素可特异性结合半乳糖和甘露糖分子,图1G为菊科植物的Jacalin凝集素结构(实验仅解析出单体结构,PDB code:1C3M)[29],该结构域由12个β折叠片形成3个重复的β折叠结构,并进一步形成二聚体和四聚体结构。LysM植物凝集素可以特异性结合甘露糖和氨基葡萄糖等分子,空间拓扑结构不十分保守,典型的LysM单体结构为40个氨基酸组成的β-α-α-β三对称结构,并形成多聚体结构(图1H,实验仅解析出单体结构,PDB code:4EBY)[30]。Hevein是1975年在橡胶树中发现的植物凝集素类型,其序列长度为43个氨基酸左右,空间拓扑结构并不十分保守。图1I中橡胶树Hevein凝集素N端为2个β折叠片段组成的β折叠结构,C端为一个β折叠片段,两个β折叠结构间由两个较短的螺旋结构连接组成,并进一步形成四聚体结构(PDB code:1HEV)[31]。CRA(Class V chitinase homologs)植物凝集素最早由刺槐中提取[32],单体长度为337个氨基酸左右。CRA植物凝集素目前还没有解析出晶体结构,同源建模研究表明该结构主要由TIM‐barrel组成,并进一步形成二聚体结构。Nic(Nictaba family)和EEA(Euonymus europaeuslectin)植物凝集素可以特异性结合甘露糖和聚糖等分子,并形成二聚体结构,目前没有实验结构。
植物凝集素多为二聚体或多聚体结构,形成二聚体主要有3种方式:(1)通过单体β折叠结构之间的相互作用形成二聚体结构;(2)通过单体头-尾相互作用形成二聚体结构;(3)通过单体一端插入另一单体形成二聚体结构。植物凝集素二聚体结构的稳定性对其发挥生物学功能十分关键。
2 菜豆凝集素保守性与动力学分析
菜豆是我们日常生活中的常用食材,我们以菜豆凝集素(PDB code: 3WCR)为例对凝集素二聚体结构的序列保守性和动力学特征进行分析(比对方法为CSBLAST,序列库为UNIREF90)[33-35]。图2A为序列保守性投影到其三级结构的结果。序列保守性共分为9个等级,1-3为不保守氨基酸序列(偏蓝),7-9为保守氨基酸序列(偏红)。总体来说,45.4%的氨基酸序列较为保守(7-9),25.8%的氨基酸序列不保守(1-3),大部分β片结构均非常保守,其中4个β片结构(LEU67-TYR72,ASP107-PRO115,HIS160-VAL165和TRP225-THR234)组成了菜豆凝集素三级结构的拓扑骨架,6个β片结构(SER23-PHE29,SER88-ASN96,ALA184-ASP191,LEU196-TYR203,THR208-THR215和ASP245-LEU255)与另一单体的相应结构部分形成三级相互作用,从而形成稳定的二聚体结构。糖结合位点区域的两个β片结构(THR44-VAL45和LEU51-ARG52)的氨基酸序列不保守,有较多突变发生。菜豆凝集素要形成稳定的二聚体结构才能发挥生物学功能,因此菜豆凝集素三级结构拓扑骨架上的氨基酸序列和二聚体接触面上的氨基酸序列均非常保守。不同的凝集素蛋白可以特异性结合不同的糖分子,因此糖结合位点附近的氨基酸序列不保守,有较多突变。
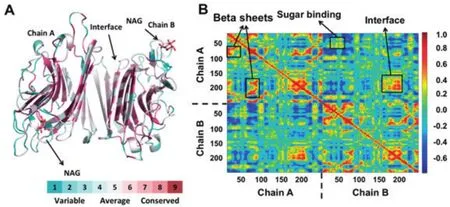
图2 菜豆凝集素序列保守性和动力学关联系数分析Fig.2 Lectin sequence conservation and dynamical correlation analysis. (A) Cartoon representation of the lectin dimer structure colored by sequence conservation scores. (B) Dynamical correlation analysis of the motions of lectin dimer obtained from MD simulations.
生物分子静态结构分析只能反映该分子某一时刻的结构特征,其动力学特征对全面了解结构功能关系有非常重要的帮助。为了更加全面地描述菜豆凝集素分子的生物学特性,我们对菜豆凝集素的二聚体结构进行了分子动力学模拟分析(模拟软件为GROMACS,力场为Amber03,水模型为TIP3P,模拟时间为50ns,初始结构为菜豆凝集素二聚体结构PDB code:3WCR)[36-38]。动力学网络中的关联分析(数值区间为-1~1)可以描述分子在动力学模拟过程中不同结构域或氨基酸残基之间的协同运动情况,动力学关联分析为正值表示两个结构域(或氨基酸残基)之间有较为紧密的协同运动关系(红色区域),动力学关联分析为负值表示两个结构域之间的运动特征相反(蓝色区域),动力学关联分析为0则表示两个结构域之间没有关系(浅绿色区域)。图2B为动力学关联分析的结果,数值大于0.7表明氨基酸残基之间有非常高的运动协同性,主要为同一个二级结构单元中的氨基酸残基。数值在0.5-0.6之间表明氨基酸残基之间有较高的运动协同性,例如菜豆凝集素三级结构拓扑骨架的β片结构(LEU37-ARG41,LEU67-TYR72,SER88-ASN96,LEU196-TYR203和THR208-THR215)之间有较高的运动协同性(图2B),二聚体接触面上的β片结构(SER23-PHE29,SER88-ASN96,ALA184-ASP191,LEU196-TYR203,THR208-THR215和ASP245-LEU255)之间也有较高的运动协同性(图2B)。菜豆凝集素需要形成稳定的二聚体结构才能发挥生物学功能,因此动力学模拟中接触面氨基酸残基之间的三级相互作用使两个凝集素单体紧密结合,并协同运动。

图3 菜豆凝集素糖分子结合位点间的主要动力学通路Fig.3 Optimal pathway that enforces the correlation between the two sugar binding sitesis shown as the red spheres. These spheres represent the CA atoms of amino acids along the optimal pathway. The two monomers are colored in green and blue. The sugar molecules are colored in orange.
特异性结合糖分子是菜豆凝集素的重要生物学特征,因此我们也着重分析了菜豆凝集素二聚体两个单体中糖结合位点之间的运动协同性。结果表明,不同单体的糖分子结合位点之间也有较高的运动协同性(图2B)。我们进一步利用动力学网络模型分析了不同单体糖分子结合位点之间的动力学通信路径,两个结合位点间通过15个氨基酸连接(A:ASN33, A:ARG31, A:PHE29, A:TRP249, A:SER92, A:LEU187, A:ALA199, A:ILE211, B:SER200, B:LEU187, B:SER92, B:TRP249, B:PHE29, B:ARG31, B:ASN33)(图3)。糖结合位点附近的氨基酸残基保守性较低,如ASN33(1)和ARG31(4)。动力学通信路径上的其它氨基酸残基均较为保守,如A:PHE29(7),A:TRP249(9),A:ALA199(8),B:TRP249(9), B:PHE29(7)等。菜豆凝集素不同单体的糖结合位点氨基酸残基通过保守的氨基酸通路来保持协同作用,从而发挥相应的生物学功能。
3 植物凝集素药物设计与医学应用
植物凝集素可以特异性结合不同的碳水化合物分子,在生物医学中有广泛的应用。然而部分含有植物凝集素的食物被食用后会结合器官表皮上的糖分子,从而导致胃肠或肝脏无法正常地代谢蛋白质,并制造有毒副产品。如果食用过多与血型不兼容的植物凝集素,体内毒素会急剧增加。因此,如何设计抑制剂来降低植物凝集素的毒性,进而缓解或者消除植物凝集素引发的症状也是近年来研究食品安全等问题的热点之一。在此,我们也以日常食用的菜豆凝集素为例讨论如何设计菜豆凝集素抑制剂来缓解菜豆凝集素产生的毒性症状。方法的主要思路为:(1)在目标蛋白质结构中寻找并分析潜在的药物靶点;(2)结合潜在药物靶点的结构信息(药物靶点的空腔体积,表面积,关键氨基酸等)筛选或设计药物。针对菜豆凝集素,我们首先计算分析了单体结构上所有的潜在药物靶点。图4中球形结构区域为菜豆凝集素单体中空腔体积最大的三个潜在药物靶点分布情况,其中蓝色区域的空腔体积最大(体积为263.87 Å3),表面积为345.51 Å2;紫色区域的空腔体积次大(体积为230.21Å3),表面积为369.47Å2;黄色区域的空腔体积为194.62Å3,表面积为449.84Å2。抑制剂分子若结合在蓝色或者紫色区域会影响该区域的局部结构,可能进一步改变菜豆凝集素单体结构,从而降低其二聚体的结合能力。黄色靶点区域在菜豆凝集素二聚体的结合面,抑制剂如果结合在黄色靶点区域,将直接和菜豆凝集素单体发生竞争,从而直接降低菜豆凝集素二聚体的形成效率,降低菜豆凝集素的凝血毒性。目前,我们已根据该黄色区域的结构特征筛选并设计了短肽来抑制菜豆凝集素形成二聚体结构,并已进行了相应的凝血毒性实验,有较好效果。

图4 菜豆凝集素潜在药物靶点预测结果Fig.4 Sphere representation of the top three pockets of lectin monomer (PDB: 3WCR). The volumes of pocket 1 (colored in blue), pocket 2 (colored in purple), and pocket 3 (colored in yellow) are 263.87 Å3,230.21Å3, and 194.62 Å3, respectively. The surface areas of the three pockets are 345.51 Å2, 369.47 Å2, 449.84 Å2, respectively.
4 结 论
本文以计算生物学的视角并结合菜豆凝集素实例,从植物凝集素的三级结构、序列保守性、动力学特征和抑制剂设计等方面讨论了植物凝集素的研究进展及药物设计的可能性。
1)目前蛋白质结构数据库中的植物凝集素主要可分为12个较大的结构类型,大多数植物凝集素都形成二聚体或者多聚体结构。
2)菜豆凝集素序列保守性分析表明二聚体接触面上的氨基酸较为保守(形成稳定的二聚体结构),糖结合位点的氨基酸差异性较大(特异性结合糖分子)。动力学关联分析表明二聚体接触面氨基酸,不同糖分子结合位点的氨基酸均有较好的运动协同性。
3)以菜豆凝集素为例设计短肽抑制剂来降低菜豆凝集素的凝血毒性,讨论了植物凝集素药物设计的可行性与可操作性。
随着计算生物学的不断发展,利用植物凝集素的生物学特性并结合计算生物学的优势,将极大促进植物凝集素抗虫,抗凝血,抗病等的应用推广,为植物凝集素应用研究开辟新的思路。
(References)
[1] BanerjeeN, Sengupta S, Roy A, et al.Functional alteration of a dimeric insecticidal lectin to a monomeric antifungal protein correlated to its oligomericstatus[J]. PLoS One, 2011, 6(4): e18593.
[2]MalikA, Lee J, Lee J. Community-based network study of protein-carbohydrate interactions in plant lectins using glycan array data[J]. PLoS One, 2014, 9(4): p. e95480.
[3]Elgavish S, ShaananB. Chemical characteristics of dimer interfaces in the legume lectinfamily[J]. Protein Sci, 2001, 10(4): 753-761.
[4]Loris R, Hamelryck T, BouckaertJ ,et al.Legume lectin structure[J]. BiochimBiophysActa, 1998, 1383(1): 9-36.
[5]Plattner VE, Wagner M, Ratzinger G,et al. Targeted drug delivery: binding and uptake of plant lectins using human 5637 bladder cancer cells[J]. Eur J Pharm Biopharm, 2008, 70(2):572-576.
[6]Wirth M, Gerhardt K, Wurm C,et al. Lectin-mediated drug delivery: influence of mucin on cytoadhesion of plant lectins in vitro[J]. J Control Release, 2002, 79(1-3):183-191.
[7]Wang XY, Koller R, Wirth M,et al. Lectin-Grafted PLGA Microcarriers Loaded with Fluorescent Model Drugs: Characteristics, Release Profi les, and CytoadhesionStudies[J]. Sci Pharm, 2014, 82(1): 193-205.
[8] Badari N AR, Sivaramakrishna A, Marimuthu KM, et al.A comparative study of phytohaemagglutinin and extract of Phaseolus vulgaris seeds by characterization and cytogenetics[J]. SpectrochimActa A MolBiomolSpectrosc, 2015, 134: 143-137.
[9]JaffÉWG. Hemagglutinins, in Toxic Constituents of Plant Foodstuffs[M]. Academic Press, 1969: 69-101.
[10]Jorgensen, WL. Foundations of biomolecularmodeling[J]. Cell, 2013, 155(6): 1199-1202.
[11] CuiQ, NussinovR. Making biomolecular simulations accessible in the post-Nobel Prize era[J]. PLoSComputBiol, 2014. 10(8):e1003786.
[12]ZhaoY, GongZ, Xiao Y.Improvements of the hierarchical approach for predicting RNA tertiary structure[J]. J BiomolStructDyn, 2011, 28(5): 815-826.
[13]Zhao Y, Huang Y, Gong Z, et al. Automated and fast building of three-dimensional RNA structures[J]. Sci Rep, 2012, 2: 734.
[14]Zhao Y, Zeng C, Tarasova NI, et al. A new role for STAT3as a regulator of chromatintopology[J]. Transcription, 2013, 4(5): 227-231.
[15]Chen H, Zhao Y, Li H,et al. Break CDK2/Cyclin E1 interface allosterically with small peptides[J]. PLoS One,2014, 9(10): e109154.
[16]Du C, Chen H, Zhao Y, et al.How Far and How Fast Can One Move on Neutral Network?[J]. Journal of Theoretical & Computational Chemistry, 2013, 12(8): 1341010.
[17]Wang J, Zhao Y, Zhu C, et al.3dRNAscore: a distance and torsion angle dependent evaluation function of 3D RNA structures[J]. Nucleic Acids Res, 2015, 2: gkv141.
[18] GuptaGS. Vishveshwara, SuroliaA. Stability of dimeric interface in banana lectin: Insight from molecular dynamics simulations[J]. IUBMB Life, 2009, 61(3): 252-260.
[19] GuptaGS. Sinha, SuroliaA. Unfolding energetics and stability of banana lectin[J]. Proteins, 2008, 72(2): 754-760.
[20]KaushikS, Mohanty D, Surolia A.Role of glycosylation in structure and stability of Erythrinacorallodendronlectin (EcorL): a molecular dynamics study[J]. Protein Sci, 2011, 20(3): 465-481.
[21]Berman HM, Westbrook J, FengZ et al., The Protein Data Bank[J]. Nucleic Acids Res, 2000. 28(1): 235-42.
[22] Van DEJ. History of plant lectin research, in Lectins[M]. Springer, 2014:3-13.
[23] Nagae M, Soga K, Morita-Matsumoto K, et al., Phytohemagglutinin from Phaseolus vulgaris (PHA-E) displays a novel glycan recognition mode using a common legume lectinfold[J]. Glycobiology, 2014. 24(4): 368-78.
[24] Carrizo ME, Capaldi S, Perduca M, et al., The Antineoplastic Lectin of the Common Edible Mushroom (Agaricusbisporus) Has Two Binding Sites, Each Specific for a Different Configuration at a Single EpimericHydroxyl[J]. Journal of Biological Chemistry, 2005, 280(11): 10614-10623.
[25]Transue TR, Smith AK, Mo H, et al., Structure of benzyl T-antigen disaccharide bound to Amaranthuscaudatusaggl utinin[J]. Nat StructBiol, 1997. 4(10): 779-83.
[26] Barrientos LG, Louis JM, Botos I, et al., The Domain-Swapped Dimer of Cyanovirin-N Is in a Metastable Folded State[J]. Structure. 10(5): 673-686.
[27] Hester G, Kaku H, Goldstein JI, et al., Structure of mannose-specific snowdrop (Galanthusnivalis) lectin is representative of a new plant lectinfamily[J]. Nature Structural Biology, 1995.2: 472-479.
[28] Pascal JM, Day PJ, Monzingo AF, et al., 2.8-A crystal structure of a nontoxic type-II ribosome-inactivating protein, ebulinl[J]. Proteins, 2001. 43(3): 319-26.
[29] Bourne Y, Zamboni V, Barre A, et al., Helianthus tuberosuslectin reveals a widespread scaffold for mannosebinding lectins[J]. Structure, 1999. 7(12): 1473-82.
[30] Liu T, Liu Z, Song C, et al., Chitin-induced dimerization activates a plant immune receptor[J]. Science, 2012. 336(6085): 1160-4.
[31]Andersen NH, Cao B, Rodriguez-Romero A, et al., Hevein: NMR assignment and assessment of solution-state folding for the agglutinin-toxin motif[J]. Biochemistry, 1993. 32(6): 1407-22.
[32] Van DEJ, Culerrier R, Barre A, et al., A Novel Family of Lectins Evolutionarily Related to Class V Chitinases: An Example of Neofunctionalization in Legumes[J]. Plant Physiology, 2007. 144(2): 662-672.
[33] UniProt, C., The Universal Protein Resource (UniProt) in 2010[J]. Nucleic Acids Res, 2010. 38(Database issue): D142-8.
[34]Angermuller, C, A. Biegert, and J. Soding, Discriminative modelling of context-specific amino acid substitution probabilities[J]. Bioinformatics, 2012. 28(24): 3240-7.
[35]Biegert, A. and J. Soding, Sequence context-specific profiles for homology searching[J]. ProcNatlAcadSci U S A, 2009. 106(10): 3770-5.
[36] Duan Y, Wu C, Chowdhury S, et al., A point-charge force field for molecular mechanics simulations of proteins based on condensed-phase quantum mechanical calculations[J]. J ComputChem, 2003. 24(16): 1999-2012.
[37]Mahoney, M.W. and W.L. Jorgensen, A five-site model for liquid water and the reproduction of the density anomaly by rigid, nonpolarizable potential functions[J]. Journal of Chemical Physics, 2000. 112(20): 8910-8922.
[38] Pronk S, Pall S, Schulz R, et al., GROMACS 4.5: a highthroughput and highly parallel open source molecular simulation toolkit[J]. Bioinformatics, 2013. 29(7): p. 845-54.
Plant lectin protein structural classifi cation and drug design
ZHAO Yunjie1,2, LIU Zhichao1, LIU Qin2, CHEN Gao2, CHEN Chanyou2, ZENG Chen1,2
1 Department of Physics, The George Washington University, Washington DC, 20052, USA 2 School of Life Sciences, Jianghan University, Wuhan 430056, China
Plant lectinis a specifi c carbohydrate-bindingor sugar-binding protein which wasfi rst discoveredin the 19th century.Itshigh specifi city in carbohydratebindingmakes it a useful molecular tool for different biological applications. For example,plant lectin’s ability to target sugar or carbohydrate in the gastrointestinaltract of insects provides a defense mechanism against pests. Lectin protein can also bind to the membrane of cancer cellsinducing apoptosis or inhibiting growth.Previous studies showed that there are over 400,000 estimated binding sites for kidney bean lectins alone. Structural information of plant lectinsisthus essentialfor understanding their biological functions and applications.Here we report some of the most prevalenttertiary structures of plant lectins, and further probed the dynamical properties of kidney bean lectin via molecular dynamics simulations. Thestructural and dynamical information may shed some insights in lectin protein engineering and drug design for industrial and medical applications.
Plant lectin; tertiary structure; evolution; drug design; medical application
R730.58
A doi 10.11966/j.issn.2095-994X.2015.01.01.12
2015-02-24;
2015-03-21
武汉市黄鹤英才项目
赵蕴杰,博士后研究员,研究方向为生物物理学,电子信箱:yjzhao.wh@gmail.com;陈禅友(通信作者),教授,研究方向为植物遗传改良和利用,电子邮箱:ccy@jhun.edu.cn;曾辰(通信作者),教授,研究方向为生物物理学,电子信箱:chenz@gwu.edu
引用格式:赵蕴杰,刘志超,刘琴,等.大数据癌症风险预测系统[J].世界复合医学, 2015 , 1(1): 68-73.