黄鳝主要组织相容性复合体Iα 基因的克隆及多态性分析
李伟,江翱,吴辉
(长江大学生命科学学院,湖北 荆州 434025)
主要组织相容性复合体(MHC)在机体获得性免疫中具有重要作用[1]。MHC I 类和II 类分子分别负责内、外源性抗原的加工和呈递[2]。MHC I 类分子是由α 链和β 链(由β2 微球蛋白编码)组成的异二聚体。经典的MHC Iα 分子包含连接肽、跨膜区、胞质区和胞外区等结构域。MHC 分子不仅在群体内存在着大量等位基因,而且等位基因间也存在较多变异[3]。II 类分子的多态性主要集中于外显子2,而I 类分子的多态性主要集中于外显子2 和外显子3[4]。这些区域包含众多抗原结合位点(PBR)。PBR位点的氨基酸发生变异会极大地影响MHC 识别病原并呈递给T 细胞受体的能力[5]。MHC 分子的多态性和哺乳动物、家禽及鱼类的抗病性密切相关[6–8]。MHC 作为用分子标记辅助育种的重要候选基因倍受重视。近年来有多种经济鱼类的MHC 分子被陆续克隆[9–15],鱼类MHC Iα 基因的多态性研究已较为深入[16–18]。黄鳝MHC IIB 基因具有较丰富的多态性[4],但关于其MHC Iα 基因的研究尚少。
黄鳝是一种重要的淡水养殖鱼类,具有很高的食用和药用价值。克隆和发掘具有重要开发潜力的抗病基因,并应用于分子标记辅助育种对黄鳝的健康养殖具有重要意义。笔者克隆黄鳝MHC Iα 基因的全长cDNA 序列,探究其基因结构,分析该基因的组织表达特异性和病原菌感染对基因表达的影响,探讨黄鳝MHC Iα 基因的多态性,以期探明MHC I 类分子多态性与鱼体抗病力的关系。
1 材料与方法
1.1 试验用鱼及病原菌感染
健康鳝鱼约200 条,每条质量60~70g,均购买于市场。室温条件下,于水箱中暂养1 周后提取总RNA 和进行病原菌感染试验。取心脏、肝脏、脾脏、胃、肾脏、血细胞、皮肤、肠、肌肉和脑等10 种组织,在液氮中速冻后提取总RNA。
细菌感染试验按文献[4]报道的方法进行。病原细菌Aeromonas hydrohilia 在液体LB 中 28℃培养到对数生长期后,4 500 r/min 离心收集,用灭菌生理盐水(0.9%)将菌液稀释至约2.2×107CFU/mL。随机取45 尾鱼用MS222 处理麻醉后进行腹腔注射。每条鱼注射20 μL。对照鱼注射生理盐水。分别在感染后4、12、24、48、72 h 随机选3个个体,取其肝脏、脾脏和肾脏3 种组织液氮速冻后提取总RNA。
1.2 DNA 的提取和cDNA 的合成
黄鳝肝脏DNA提取按照DNA分离试剂盒说明书进行。各组织总RNA 提取按照Trizol 试剂说明书进行。cDNA 合成按照PrimeScript RT regent 试剂盒说明书进行。
1.3 黄鳝MHC Iα 基因的克隆
根据已报道的鱼类MHC Iα 基因序列,设计了一对简并引物de–IA–F(5′–CAAACTTYCCAGAGT TTGT–3′)和de–IA–FR(5′–CCAGAGAGCTGRAAC ACACA–3′),用来扩增黄鳝MHC Iα 基因的cDNA片段。根据上述cDNA 片段设计基因特异性引物IA–GSP5(5′–TGGACACCTCCACTTTGGTTGAAG CGC–3′)和IA–GSP3(5′–CCCAACAACGATGGGAC CTTCCAG–3′),分别用于5′和3′ RACE 扩增。RACE扩增按照Smart RACE cDNA 扩增试剂盒说明书进行。Touchdown 扩增反应程序:94℃预变性3min;94℃变性1min,70℃退火45 s,72℃延伸2min,5个循环;94℃预变性1min,65℃退火50 s,72℃延伸1min,30个循环;延伸10min。
PCR 产物回收、纯化后,连接至pMD–19T 载体,克隆至Top10 感受态细胞,挑取阳性克隆进行序列测定。根据cDNA 全长设计特异性引物,分离黄鳝MHC Iα 基因的内含子,将各序列拼接后得到gDNA 结构。
1.4 序列分析
利用网站https://www.predictprotein.org/对黄鳝MHC Iα 基因推断的氨基酸序列进行二级结构预测。保守结构利用在线网站http://www.ncbi.nlm.nih.gov/structure 进行分析。
1.5 黄鳝MHC Iα 基因的表达分析
采用荧光定量PCR 技术,检测MHC Iα组织表达特异性和病原菌感染对该基因表达的影响。以不添加模板的反应作为空白对照,以β–actin 的表达作为内参。设计的actin 引物为sense(5′–GCTGTGCT GTCCCTGTA–3′)和antisense(5′–GAGTAGCCACG CTCTGTC–3′)。设计的基因特异性引物为RT–F (5′–CAGATGTGATGGAGATTGTGTG–3′)和 RT–R (5′–CACCTACATTTGCTCCACTAC–3′)。荧光定量PCR 在7 500 RT–PCR 系统(ABI)上进行。20 μL 反应体系中包含1 μL 模板,10 μL SYBR Premix Ex Taq,0.4 μL 引物,7.8 μL ddH2O。反应程序:95℃预变性30 s;95℃变性30 s,54℃退火25 s,72℃延伸1min,40个循环。
1.6 黄鳝MHC Iα 基因外显子3 的多态性分析
利用引物IA–e3–F(5′–CTCAGGTGTCCACDT TTTACAG–3′)和IA–e3–R(5′–CATACCTGTTCTCA GCAGAGAGCT–3′)进行外显子3 多态性分析。42个个体被用来检测多态性。每个个体测定其5~7个阳性克隆的序列。等位基因按照文献[19]的规则进行命名;PBR 位点的确定根据文献[20]的规则进行。利用MEGA 4.0 软件分析变异位点,运用Poisson- corrected 算法计算等位基因氨基酸间的遗传距离。同义替代率(dS)和非同义替代率(dN)按照MEGA4.0中的Nei–Gojobori 算法进行,用Jukes–Cantor 算法进行校正。P 值和标准误差的确定通过1 000次重复抽样支持率的Z 检验获得。
2 结果与分析
2.1 黄鳝MHC Iα 基因的序列分析
黄鳝MHC Iα 基因全长5 037 bp,5′UTR 长78 bp,3′UTR 长600 bp,编码1 条350个氨基酸的多肽链。其gDNA 序列包括4个外显子和3个内含子;内含子1 长1 440 bp,内含子2 长1 147 bp,内含子3 长719 bp;连接肽(CP)、跨膜区(TM)、胞质区(CT)之间没有内含子序列存在(图1)。

图1 黄鳝MHC Iα 基因的结构及其推断的氨基酸序列 Fig.1 Genomic structure of swamp eel MHC Iα and deduced amino acid sequence
2.2 黄鳝MHC Iα 基因的表达
黄鳝MHC Iα 基因在各组织的表达结果表明:健康黄鳝的肝脏等10 种组织MHC Iα 基因表达量存在着很大差异,血细胞的表达量最高,肝脏、心脏、脾脏和胃中的表达量中等,而皮肤、脑、肌肉、肾脏和小肠中的表达量较低(图2)。病原细菌感染会显著影响肝脏、脾脏和肾脏中MHC Iα 基因的表达。感染后4 h 内肝脏、脾脏和肾脏中MHC Iα 基因的表达量基本保持不变。4 h 后肾脏中的基因表达量急剧上升,24 h 达到峰值,然后逐渐下降(图3);肝脏中的表达在感染4 h 后呈下降趋势,72 h 后有所恢复(图4)。感染4 h 后脾脏中MHC Iα 基因的表达量先下降,24 h恢复至正常水平后又下降,呈波动趋势(图5)。
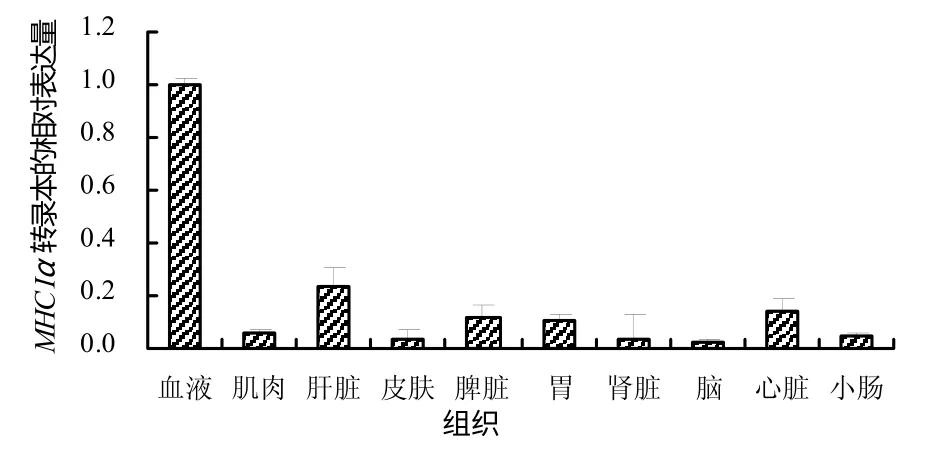
图2 黄鳝MHC Iα 基因在不同组织中的相对表达量 Fig.2 Relative expression analysis of swamp eel MHC Iαgene in different tissues
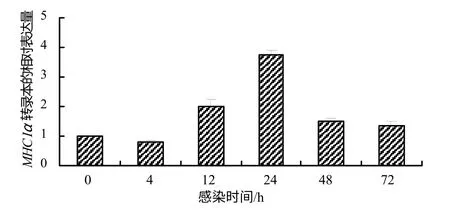
图 3 病原菌感染后肾脏MHC Iα 基因的相对表达量 Fig. 3 Relative expression analysis of MHC Iαgene in kidney after infection with A. hydrophilia

图4 病原菌感染后肝脏MHC Iα 基因的相对表达量 Fig.4 Relative expression analysis of MHC Iαgene in liver after infection with A. hydrophilia

图5 病原菌感染后脾脏MHC Iα 基因的相对表达量 Fig. 5 Relative expression analysis of MHC Iαgene in spleen after infection with A. hydrophilia
2.3 黄鳝MHC Iα 基因的多态性
42个健康个体共得到有效等位基因50个,每个个体包含1~6个等位基因。据此推测,黄鳝基因组上可能至少含有3个MHC Iα 基因座位。分析等位基因PBR 位点和非PBR 位点的遗传变异后发现:PBR 位点氨基酸水平的平均遗传距离为0.393;非PBR 位点氨基酸水平的平均遗传距离为0.178。各等位基因的PBR 位点和非PBR 的同义替代率( dS) 和非同义替代率( dN)计算结果表明:无论是PBR 位点还是非PBR 位点,其dN/dS均大于1。采用单尾的Z 检验进行正选择(dN>dS) 检验,P 值<0.05,说明黄鳝MHC Iα 基因在进化过程中受到正向选择的影响(表1)。

表1 等位基因间的遗传距离及同义替代率与非同义替代率在外显子3 的分布 Table 1 Pairwisegenetic distance and condon–based evolutionary divergence in all sites and PBR
3 结论与讨论
通过同源克隆,结合RACE 技术,克隆了黄鳝MHC Iα 基因。经序列分析,发现黄鳝MHC Iα 基因是“4 外显子–3 内含子”结构。这与其他物种的MHC Iα 基因结构不同[15,21–24]。黄鳝的MHC Iα 基因在10 种被检测组织里的表达量存在着显著差异;病原细菌A. hydrophilia 感染可以显著影响肝脏、脾脏和小肠等组织里MHC Iα 基因的表达水平,这与许多鱼类MHC 分子的研究报道[4,10—11,14–15,25]相似。黄鳝MHC Iα 基因具有丰富的多态性,50个不同的等位基因间非同义替代率(dN)与同义替代率(dS)的比值大于1,差异达到了显著水平(P<0.05),说明黄鳝MHC Iα 基因在进化过程中受到正向选择的影响[12,26]。
[1] Klein J , Figueroa F . Evolution of the major histocompatibility complex[J] . Critical Reviews in Immunology,1986,6:295–386.
[2] Rothbard J B,Gefter M L.Interactions between immunogenic peptides and MHC proteins[J].Annual Review of Immunology,1991,9:527–565.
[3] Kjoeglum S,Larsen S,Bakke H G.How specific MHC class I and class II combinations affect disease resistance against infectious salmon anaemia in Atlantic salmon (Salmo salar)[J].Fish Shellfish Immunol,2006,21:431–441.
[4] Li W,Sun W X,Hu J F,et al.Molecular characterization,polymorphism and expression analysis of swamp eel major histocompatibility complex class IIBgene,after infection by Aeromonas hydrophilia[J].The Journal of Animal & Plant Sciences,2014,24(2):481–491.
[5] Sommer S.The importance of immunegene variability (MHC) in evolutionary ecology and conservation[J]. Front Zool,2005,2:16–34.
[6] Zhang Y X,Chen S L,Liu Y G, et al.Major histocompatibility complex II B allele polymorphism and its association with resistance/susceptibility to Vibrio anguillarum in Japanese Flounder (Paralichthys olivaceus)[J]. Marine Biotechnology,2006,8,600–610.
[7] Du M,Chen S L,Liu Y H,et al.MHC polymorphism and disease resistance to Vibrio anguillarum in 8 families of half-smooth tongue sole (Cynoglossus semilaevis)[J]. BMC Genetics,2011,12:78.
[8] Tang J,Zhou C,Zhang Z J,et al.Association of polymorphisms in non-classic MHCgenes with susceptibility to autoimmune hepatitis[J].Hepatobiliary & Pancreatic Diseases International,2012,11:125–131.
[9] 苏建明,肖调义,张学文,等.草鱼MHCⅡA 基因cDNA 的克隆与组织表达分析[J].湖南农业大学学报:自然科学版,2008,34(6):673–679.
[10] Pang J,Gao F,Lu M,et al.Major histocompatibility complex class IIA and IIBgenes of Nile tilapia Oreochromis niloticus,genomic structure,molecular polymorphism and expression patterns[J].Fish Shellfish Immunol,2013,34:486–496.
[11] Zhou F,Dong Z,Fu Y,et al.Molecular cloning,genomic structure,polymorphism and expression analysis of major histocompatibility complex class II Bgene of Nile tilapia (Oreochromis niloticus)[J].Aquaculture,2013,372–375:149–157.
[12] 李同明,周芬娜,崔志峰,等.尼罗罗非鱼主要组织相容性复合体IA 基因的克隆与多态分析[J].山东大学学报:理学版,2013,48(11):14–22.
[13] 张新中,鲁义善,吴灶和,等.红笛鲷主要组织相容性复合物ⅠA 抗原基因的克隆与表达分析[J].水产学报,2012,36 (10):1482–1492.
[14] L i H J,Jiang L X,Han J B,et al.Major histocompatibility complex class IIA and IIBgenes of the spotted halibut Verasper variegatus,genomic structure,molecular polymorphism,and expression analysis[J]. Fish Physiol Biochem,2011,37:167–180.
[15] Xu T J,Chen S L.Molecular cloning,genomic structure and expression analysis of major histocompatibility complex class IAgene of half-smooth tongue sole (Cynoglossus semilaevis)[J].Fish Physiol Biochem,2011,37:85–90.
[16] Grimholt U,Drablos F,Jorgensen S M,et al.The major histocompatibility class I locus in Atlantic salmon (Salmo salar L.):Polymorphism,linkage analysis and protein modeling[J].Immunogenetics,2002,54:570–581.
[17] 夏春,徐广贤,林常有,等.草鱼MHC class I 等位基因克隆及其多态性分析[J].自然科学进展,2004,14(1) :51–58.
[18] Fujiki K,Booman M,Chin-Dixon E,et al.Cloning and characterization of cDNA clones encoding membrane- bound and potentially secreted major histocompatibility class IA receptors from walleye (Stizostedion vitreum)[J]. Immunogenetics,2001,53(9):760–769.
[19] Klein J,Bontrop R E,Dawkins R L,et al.Nomenclature for the major histocompatibility complexes of different species,a proposal[J].Immunogenetics,1990,31:217–219.
[20] Brown J H,Jardetzky T S,Gorga J C,et al. Three- dimensional structure of the human class II histocompatibility antigen HLADR1[J].Nature,1993,364:33–39.
[21] Srisapoome P,Ohira T,Hirono I,et al.Cloning,characterization and expression of cDNA containing major histocompatibility complex class I,IIA and IIBgenes of Japanese flounder Paralichthys olivaceus[J]. Fish Sci,2004,70:264–276.
[22] Lukacs M F,Harstad H,Grimholt U,et al.Genomic organization of duplicated major histocompatibility complex class I regions in Atlantic salmon (Salmo salar)[J]. BMC Genomics,2007,8:251.
[23] Antao A B,Wilson M,Wang J,et al.Genomic organization and differential expression of channel catfish MHC class Igenes[J].Dev Comp Immunol,2001,25(7):579–595.
[24] Lunney J K,Ho C S,Wysocki M,et al.Moleculargenetics of the swine major histocompatibility complex,the SLA complex[J].Dev Comp Immunol,2009,33:362–374.
[25] 马晓茜,刘至治,李思发,等.团头鲂主要组织相容性复合体Ⅰ类基因全长cDNA 的克隆及组织表达分析[J].上海海洋大学学报,2011,20(1):34–43.
[26] Matsuo M Y,Aaskawa S,Shimizu N,et al.Nucleotide sequence of the MHC class Igenomic region of a teleost,the medaka(Oryzias latipes)[J].Immunogenetics,2002,53:930–940.

