猪心肌和骨骼肌miRNA转录组比较分析
谭 娅,马继登,刘一辉,沈林園,李学伟,李明洲,朱 砺
(四川农业大学 动物遗传育种与繁殖研究所,四川 雅安 625014)
猪心肌和骨骼肌miRNA转录组比较分析
谭 娅,马继登,刘一辉,沈林園,李学伟,李明洲,朱 砺
(四川农业大学 动物遗传育种与繁殖研究所,四川 雅安 625014)
【目的】 研究microRNA (miRNA)在猪不同类型肌肉生长发育中的作用。【方法】 以猪心肌以及2种骨骼肌(背最长肌和腰大肌)为材料,采用Illumina高通量测序技术鉴定其miRNA转录组,并进行差异表达分析和靶基因富集分析。【结果】 从猪心肌、背最长肌和腰大肌组织的3个小RNA测序文库中共获得约56M的序列,通过生物信息分析共鉴定出907个非冗余miRNA,其中441个miRNA在3个文库共同表达,123个miRNA在文库间表现为显著的差异表达(P<10-6)。对3个文库差异表达miRNA的相关性分析发现,背最长肌与腰大肌的相关性最高(r=0.94),证明2种骨骼肌的表达模式十分相近,而心肌与2种骨骼肌的表达模式不同。通过对各文库高表达的miRNA分析发现,表达量前十的miRNA中有6个miRNA的表达量在心肌中极显著上调。对在心肌内上调的6个miRNA进行的靶基因预测和生物学功能预测发现,这些差异miRNA主要靶向作用于钙信号通路和胰岛素信号通路。荧光定量PCR (qRT-PCR)验证结果表明高通量测序结果准确可靠。【结论】 从miRNA层面揭示了猪心肌和骨骼肌的差异,这种差异在一定程度上印证了心肌和骨骼肌之间迥异的生理学特征。
microRNA;心肌;骨骼肌;高通量测序;猪
心肌和骨骼肌均是来源于脊椎动物的中胚层横纹肌,其中心肌的线粒体和肌红蛋白含量丰富,氧化能力和抗疲劳能力均高于骨骼肌。相比之下,骨骼肌为异质性很高的组织,根据肌纤维类型可将骨骼肌分为氧化型(腰大肌)和酵解型(背最长肌),这2种类型的骨骼肌在氧化酵解和抗疲劳能力方面存在显著差异[1-2]。心肌和骨骼肌在生物体内发挥着不同的生物学功能,同时其细胞结构也呈现出明显差异[3]。哺乳动物的大多数心肌细胞是具有分支结构的双核细胞,心肌细胞间具有闰盘结构,而骨骼肌细胞为长梭形的多核细胞。肌细胞结构上的这种差异与心肌和骨骼肌的功能差异息息相关。
miRNA(microRNA)是一类长18~24 nt的内源性非编码小RNA,它通过与靶mRNA的3′UTR区完全或不完全互补配对实现对靶向mRNA的降解或翻译抑制[4]。前人研究表明,miRNA在生物个体的生理和病理过程中都发挥着重要作用[5-6],血清应答因子(Serum response factor,SRF)/心肌素(Myocardin)和肌肉分化因子(Myogenic differentiation,MyoD)/肌肉增强因子(Myocyte enhancer factor,MEF)分别是心肌和骨骼肌发育过程中关键性的转录调控因子,miRNA可通过正反馈或负反馈调控这些转录因子,从而调节心肌和骨骼肌的生长发育[7]。
猪在解剖学和生理学上与人具有很高的相似性,因此通常被作为一种生物模型进行生物医学研究[8]。目前,已有研究比较了氧化型和酵解型肌肉的miRNA转录组[9],而骨骼肌与心肌miRNA转录组的比较还未见报道。因此,本试验利用高通量测序技术鉴定和对比了猪心肌和骨骼肌的miRNA转录组,试图从miRNA层面探讨和解释两者的本质差异,并为后续肌肉相关的研究提供基础数据。
1 材料与方法
1.1 样品采集
3头210日龄的雌性长白猪,按照NY/T 65-2004猪饲养标准饲养于重庆市畜牧科学研究院。试猪屠宰后,迅速分离心肌(CM)、背最长肌(LDM,酵解型骨骼肌的典型代表)和腰大肌(PMM,氧化型骨骼肌的典型代表)组织,通过液氮瞬间冷冻后转移至-80 ℃冰箱保存备用。
1.2 试验方法
1.2.1 RNA的提取 采用Ambion公司的mirVanaTMmiRNA Isolation试剂盒提取样品的RNA。所有RNA样品进行1%甲醛变性凝胶电泳、核酸蛋白仪和毛细管电泳检测。
1.2.2 小RNA文库的构建及高通量测序 分别取3头试验猪心肌、背最长肌和腰大肌组织的总RNA各5 μg,将相同组织的总RNA混合即得该组织的小RNA文库。高通量测序文库构建的主要步骤如下:利用聚丙烯酰胺凝胶电泳从总RNA中纯化出14~40 nt的小RNA,用T4连接酶(Promega,美国)连接上特异的接头序列;利用M-MLV逆转录酶体系(Invitrogen公司,美国)对加接头后的小RNA进行反转录和扩增;利用LC Sciences公司的Illumina GA-2(36 nts)测序平台对扩增后的cDNA进行测序。
1.2.3 miRNA测序结果的生物信息学分析 原始的测序结果通过Illumina GA-2测序平台自带的质量控制程序(Pipeline software)进行预处理,参照Li等[10]的方法对数据做进一步处理。去除接头序列后,测序读段(reads)经过长度(14~27 nt)过滤、序列组成(A、C、G 或 T 含量<80%)过滤以及拷贝数(拷贝数>3)过滤,剩下的读段再通过与NCBI[11]、Rfam[12]以及Repbase database[13]数据库的比对去除猪已知的RNA序列,最终得到高质量读段(High-quality reads)。将高质量读段与miRBase中猪以及其他24种哺乳动物已知的前体miRNA序列进行比对,鉴定出猪已知、保守以及推定的miRNA序列。
1.2.4 miRNA差异表达分析及生物学功能预测 利用IDEG6[14]在线软件对3个文库miRNA表达量进行标准化和显著性检验(包括Audic-Claver test、Fisher exact test 以及 Chi-square 2×2 test 3种方法)。在3种检验方法下同时达到P<10-6(Bonferroni correction)的miRNA定义为差异表达miRNA。
采用PicTar[15]和TargetScan human 5.1[16]2种软件对组织间差异表达的miRNA进行靶基因预测,并取这2个软件预测结果的交集作为最终预测的靶基因。将预测得到的靶基因通过DAVID在线软件(http://david.abcc.ncifcrf.gov/)进行基因注解(GOterm)和KEGG通路(KEGG pathway)分析,获得miRNA靶基因富集的生物学通路。
1.2.5 荧光定量PCR分析 随机选取5个miRNA,采用伯乐公司的FX96实时荧光定量系统对其在3种肌肉组织中的表达量进行荧光定量PCR(qRT-PCR)检测。荧光定量PCR试剂盒采用High-Specificity miRNA qRT-PCR Detection Kit(Stratagene公司,美国)。定量上、下游引物由miRNA序列本身和通用引物组成,定量试验选用 U6 snRNA,18S rRNA 和Met-tRNA 作为内参基因[17],采用2-ΔΔCt法对定量试验结果进行分析处理,采用单因素方差分析对差异显著性进行检验,P<0.05为差异显著,P<0.01为差异极显著。
2 结果与分析
2.1 RNA的质量检测
1%甲醛变性凝胶电泳检测RNA有26S和18S 2条清晰亮带,且26S条带亮度约为18S条带亮度的2倍,几乎不见5S条带;核酸蛋白仪检测结果显示,RNA样品的A260/A280约为1.95;毛细管电泳检测结果也表明RNA的完整性和纯度都能达到小RNA测序的要求。
2.2 小RNA测序数据概述
利用高通量测序技术从猪心肌(CM)、背最长肌(LDM)和腰大肌(PMM)组织的小RNA测序文库中共获得约56 M(million)原始测序数据。过滤掉已知的小RNA(如rRNA、tRNA、snRNA、snoRNA)、mRNA以及重复序列片段(图1-A),得到可参与后续分析的高质量读段。其中从心肌、背最长肌和腰大肌测序文库分别获得20.31 M,15.87 M和17.93 M的高质量读段,这些测序读段分别占原始数据的94.12%,95.48%和97.14%。
如图1-B所示,3个文库的高质量读段长度分布(length distribution)基本一致,绝大部分高质量读段长度在21~23 nt,该长度分布与典型的miRNA长度分布相符[18],从侧面反映了本研究中小RNA测序数据的可靠性。
2.3 miRNA表达谱鉴定
根据前体和成熟体的特征,本研究共鉴定出3类miRNA:第1类为猪已知 miRNA,共包括321个成熟miRNA,此类miRNA能被定位到271个猪已知的miRNA前体上。目前,从miRBase 19.0数据库(2012-08)中已知猪有271个miRNA前体和306个miRNA成熟体,本研究中所鉴定出的miRNA覆盖了所有已报道的猪miRNA序列;第2类为猪保守的miRNA,包括72个成熟miRNA,这些miRNA能定位到猪的基因组以及miRBase数据库中65个其他已知哺乳动物的前体miRNA;第3类为猪推定的miRNA,该组包括512个成熟miRNA,对应了505个前体miRNA序列,这些前体miRNA的基因组序列经UNAFold[19]软件预测能形成稳定的发卡结构,同时这些序列的长度为18~26 nt,并且在miRBase数据库中不能与任何哺乳动物的前体miRNAs比对上。由于不同前体miRNA可以编码相同的成熟miRNA,最终在3个文库中总共鉴定得到907个非冗余的成熟miRNA序列(Unique miRNA)。
本研究发现,猪已知miRNA的数量仅占CM、LDM和PMM 3个文库miRNA总数的42.78%,39.36%和41.01%,但其拷贝数总和占所有miRNA拷贝数总和的99.39%,96.45%和97.19%,另外2类miRNA的拷贝数仅占到所有miRNA拷贝数总和的1.59%,3.53%和2.80%(图1-C),这说明测序得到的大部分序列为猪已知miRNA的表达序列,同时也证明了低拷贝数的miRNA可以通过测序技术进行有效的鉴定和分析。
2.4 miRNA在3个文库中的差异表达分析
本研究对所鉴定的907个非冗余miRNA进行了组织特异性表达分析,结果如图2所示。
由图2可知,共有441(48.62%)个非冗余 miRNA在3个文库中共表达,另有161(17.75%),68(7.51%)和158(17.42%)个非冗余miRNA分别特异性表达于CM、LDM和PMM组织中。为了进一步检测3个组织中miRNA的差异表达情况,综合运用IDEG6在线分析软件中3种不同的统计方法(Audic-Claverie test、Fisher exact test 以及Chi-square 2×2 test)对miRNA的表达进行了差异显著性检验。结果表明,在907个非冗余miRNA中,有123个非冗余miRNA在3个文库间呈现出显著的差异表达模式(P<10-6)。
对上述123个差异表达的非冗余miRNA进行表达相关性分析发现,背最长肌与腰大肌的相关系数(r=0.94,P<0.01)明显高于心肌与背最长肌(r=0.77,P<0.01)以及心肌与腰大肌(r=0.81,P<0.01)的相关系数(图3),该结果说明同为骨骼肌的背最长肌和腰大肌的miRNA表达谱更为接近,而心肌的miRNA表达谱与2种骨骼肌明显不同。该结果证明在miRNA表达谱层面即能很好地将猪的心肌和骨骼肌加以区分。

图3 猪CM、LDM和PMM 3个文库123个差异表达miRNA的相关性分析CM.心肌;LDM.背最长肌;PMM.腰大肌;lg CM、lg LDM和lg PMM分别为CM、LDM和PMM文库miRNA拷贝数的常用对数Fig.3 Correlation analysis of 123 differentially expressed miRNAs among three libraries of pigCM.Cardiac muscle;LDM.Longissimus dorsi muscle;PMM.Psoas major muscle;All reads of miRNAs from CM,LDM and PMM libraries were calculated as lg
2.5 差异表达miRNA的生物学功能预测
猪心肌内表达量前十的miRNAs及其分布比例见图4。

图4 猪心肌内表达量前十的miRNAs及其分布比例Fig.4 Top ten miRNAs and their ratios of high-quality reads in CM of pig
本研究鉴定出的猪miRNA拷贝数呈现出一种较大的动态变化,表达量较低的miRNA其测序拷贝数仅有3个,但是表达量高的miRNA其测序拷贝数能达到上百万个,而3个文库中表达量前十的miRNA(miR-143-3p、miR-378-1&-2-3p、miR-1-3p、miR-133a-1&-2-3p、miR-27b-3p、miR-148a-3p、let-7f-1&-2-5p、miR-30e-5p、miR-101-1&-2-3p和let-7a-1-5p)占整个文库拷贝数的绝大部分。在心肌中,表达量前十的miRNA拷贝数占高质量读段的82.24%(图4),如此高丰度的表达可能暗示它们在心肌中有重要功能。此外,本研究发现在心肌内表达量前十的miRNA在其他2个文库也高表达,并且其中有8个miRNAs(miR-143-3p、miR-378-1&-2-3p、miR-1-3p、miR-133a-1&-2-3p、miR-27b-3p、miR-30e-5p、miR-101-1&-2-3p和let-7a-1-5p)在3个文库呈现出差异表达模式(P<10-6),这8个miRNA中有6个miRNA(miR-143-3p、miR-378-1&-2-3p、miR-27b-3p、miR-30e-5p、miR-101-1&-2-3p和let-7a-1-5p)在心肌中的表达量同时极显著高于背最长肌和腰大肌(图5)。

图5 猪心肌内高表达的前十个miRNAs在3个文库的相对表达量**表示与心肌相比差异极显著(P<0.01);CM.心肌;LDM.背最长肌;PMM.腰大肌Fig.5 Relative expression levels of the ten most highly expressed miRNAs in cardiac muscle across the three libraries of pig** indicates highly significant difference (P<0.01) when compared with CM;CM.Cardiac muscle;LDM.Longissimus dorsi muscle;PMM.Psoas major muscle
综合考虑目前猪基因组注释工作的缺陷以及已鉴定的猪miRNA种类的局限,本研究对这6个在猪心肌内显著上调的miRNA进行了靶基因的预测以及KEGG通路分析,并将预测得到的靶基因在DAVID在线分析软件中对其功能进行分类,结果如图6所示。由图6可知,这些miRNA靶基因显著地富集于扩张型心肌病、肥厚型心肌病、钙信号通路、胰岛素信号通路和Ⅱ型糖尿病等生物学通路,其中钙离子反应、负调控钙离子转运等生物学过程与钙信号通路有关,而胰岛素刺激反应、胰岛素刺激的细胞反应、胰岛素受体信号通路以及调控胰岛素受体信号通路等生物学过程与胰岛素信号通路有关。
2.6 高通量数据验证
随机选取5个miRNAs(miR-378-1&-2-3p、miR-139-5p、miR-499-5p、miR-206-3p和miR-208b-3p)在心肌、背最长肌和腰大肌组织中进行qRT-PCR分析,并与miRNA高通量测序结果进行皮尔森相关性分析,结果(表1)表明,高通量测序结果和qRT-PCR结果呈极显著的正相关(r=0.98±0.02,P<0.01),表明本研究高通量测序结果准确可靠。
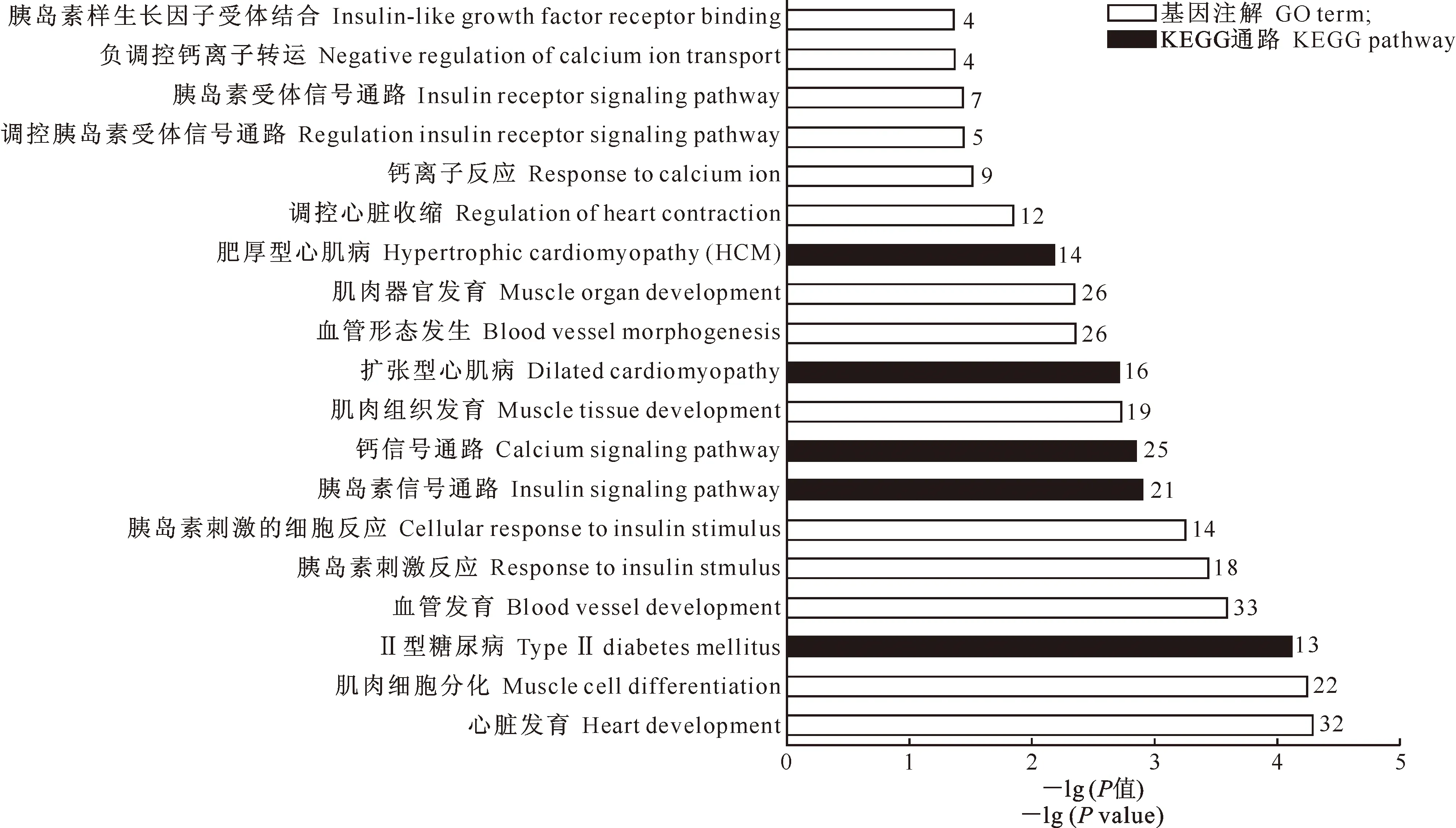
图6 猪心肌内上调的6个miRNA靶基因的生物学功能分析图柱旁边的数字为富集在该通路的基因数目Fig.6 Gene function analysis on targets of the six up-regulated miRNAs in pig CM Number behind each bar is the involved genes

表1 猪miRNA高通量测序结果与qRT-PCR结果的皮尔森相关性分析Table 1 Pearson’s correlation analysis between deep sequencing results and qRT-PCR results of pig miRNAs
3 讨 论
从本研究猪心肌、背最长肌和腰大肌组织的3个文库高质量读段长度分布上看,21~23 nt读段最多,该长度分布符合miRNA长度分布模式,这是Dicer酶切割的典型产物长度[18]。Illumina GA-2测序平台的测序长度在18~24 nt,符合miRNAs成熟体的长度范围,并且随机选取的5个miRNA的qRT-PCR结果与高通量测序结果相关性也极高,证明本研究高通量测序结果的可靠性。
miR-1和miR-133是肌肉特异的miRNA,不仅参与肌细胞的增殖和分化的全过程,同时也参与心肌细胞的传导性功能以及肌肉的肥大等过程,因此其在猪心肌和骨骼肌里都高表达;miR-143是心血管特异性的miRNA,其对血管平滑肌有重要调控作用[20]。本研究表明,miR-143在猪心肌内表达量最高;miR-378在猪心肌和骨骼肌内均高表达,其可抑制靶基因肌细胞分化抑制因子(myoR)的表达,在肌细胞分化过程中,miR-378的表达量上升,从而促进了肌肉的分化[21];let-7a在心肌和骨骼肌中广泛表达;而关于miR-27b、miR-30e和miR-101与肌肉相关的报道很少,这些高表达的miRNAs预示了其在猪心肌和骨骼肌中均有重要的生物学功能。
本研究对在猪心肌内上调的6个miRNAs的靶基因进行了基因注解(GOterm)和KEGG通路分析,发现了一些重要的生物学通路,比如钙信号通路和胰岛素信号通路。钙信号与肌肉的收缩及代谢、细胞的增殖以及其他一些信号通路密切相关;而胰岛素信号参与了葡萄糖和脂质稳态的调节、细胞增殖与分化及抗凋亡等重要过程。结合前人的研究报道,本研究找到了心肌内富集钙信号通路和胰岛素信号通路的证据。Ikeda等[22]和Dong等[23]通过荧光素酶报告系统验证miR-1的靶基因为钙调蛋白(CaM),而miR-133的靶基因为钙调磷酸酶(CaN),CaM和CaN都是钙信号通路的关键性因子。本研究发现,miR-1和miR-133在2种骨骼肌里的表达水平均高于心肌,说明骨骼肌内CaM和CaN的表达较心肌低,则钙信号通路活性也相应较低,这与前面预测的钙信号通路在心肌内富集的结果相符。Pandey 等[24]发现,miR-29a能够通过调控 PI3Kp85亚基活性直接减弱下游的胰岛素信号;并且也有报道表明miR-29a可间接抑制Akt的表达[25],从而负调控胰岛素信号通路。本研究中,miR-29a(本次测序结果中的一个miRNA)在心肌内的表达量同时低于2种骨骼肌(结果未列出),可能导致PI3K和Akt在心肌的表达量升高,使胰岛素信号增强,这也与笔者预测的胰岛素信号在心肌内富集的结果相一致。
本研究中,猪心肌和骨骼肌的miRNA存在着差异表达,且表达量排名前十的miRNAs差异十分明显。心肌上调的miRNAs靶基因主要富集在钙信号通路和胰岛素信号通路,这些差异可能部分揭示了猪心肌和骨骼肌功能的差异,同时也为今后肌肉相关的研究提供基础数据。
[1] Essen B,Jansson E,Henriksson J,et al.Metabolic characteristics of fibre types in human skeletal muscle [J].Acta Physiologica Scandinavica,1975,95(2):153-165.
[2] Bottinelli R,Reggiani C.Human skeletal muscle fibres:Molecular and functional diversity [J].Progress in Biophysics and Molecular Biology,2000,73(2):195-262.
[3] Fawcett D W,McNutt N S.The ultrastructure of the cat myocardium:Ⅰ.Ventricular papillary muscle [J].The Journal of Cell Biology,1969,42(1):1-45.
[4] Bartel D P.MicroRNAs:Genomics,biogenesis,mechanism and function [J].Cell,2004,116(2):281-297.
[5] Ambros V.MicroRNA pathways in flies and worms:Growth,death,fat,stress and timing [J].Cell,2003,113(6):673-676.
[6] McManus M T.MicroRNAs and cancer [J].Seminars in Cancer Biology,2003,13(4):253-258.
[7] van Rooij E,Quiat D,Johnson B A,et al.A family of microRNAs encoded by myosin genes governs myosin expression and muscle performance [J].Developmental Cell,2009,17(5):662-673.
[8] Rocha D,Plastow G.Commercial pigs:An untapped resource for human obesity research [J].Drug Discovery Today,2006,11(11):475-477.
[9] Liu Y,Li M,Ma J,et al.Identification of differences in microRNA transcriptomes between porcine oxidative and glycolytic skeletal muscles [J].BMC Molecular Biology,2013,14(1):7.
[10] Li M,Xia Y,Gu Y,et al.MicroRNAome of porcine pre-and postnatal development [J].PLoS One,2010,5(7):e11541.
[11] Pruitt K D,Tatusova T,Klimke W,et al.NCBI reference sequences:Current status,policy and new initiatives [J].Nucleic Acids Res,2009,37:D32-D36.
[12] Gardner P P,Daub J,Tate J G,et al.Rfam:Updates to the RNA families database [J].Nucleic Acids Res,2009,37:D136-D140.
[13] Kohany O,Gentles A J,Hankus L,et al.Annotation,submission and screening of repetitive elements in Repbase:Repbase submitter and censor [J].BMC Bioinformatics,2006,7:474.
[14] Romualdi C,Bortoluzzi S,D′Alessi F,et al.IDEG6:A web tool for detection of differentially expressed genes in multiple tag sampling experiments [J].Physiol Genomics,2003,12(2):159-162.
[15] Krek A,Grün D,Poy M N,et al.Combinatorial microRNA target predictions [J].Nature Genetics,2005,37(5):495-500.
[16] Lewis B P,Burge C B,Bartel D P.Conserved seed pairing,often flanked by adenosines,indicates that thousands of human genes are microRNA targets [J].Cell,2005,120(1):15-20.
[17] Li M,Xia Y,Gu Y,et al.MicroRNAome of porcine pre- and postnatal development [J].PLoS One,2010,5(7):e11541.
[18] Blaszczyk J,Tropea J E,Bubunenko M,et al.Crystallographic and modeling studies of RNase Ⅲ suggest a mechanism for double-stranded RNA cleavage [J].Structure,2001,9(12):1225-1236.
[19] Markham N R,Zuker M.UNAFold:Software for nucleic acid folding and hybridization [J].Methods Molecular Biology,2008,453(2):3-31.
[20] Cordes K R,Sheehy N T,White M P,et al.miR-145 and miR-143 regulate smooth muscle cell fate and plasticity [J].Nature,2009,460(7256):705-710.
[21] Gagan J,Dey B K,Layer R,et al.MicroRNA-378 targets the myogenic repressor MyoR during myoblast differentiation [J].Journal of Biological Chemistry,2011,286(22):19431-19438.
[22] Ikeda S,He A,Kong S W,et al.MicroRNA-1 negatively regulates expression of the hypertrophy-associated calmodulin andMef2agenes [J].Molecular and Cellular Biology,2009,29(8):2193-2204.
[23] Dong D L,Chen C,Huo R,et al.Reciprocal repression between microRNA-133 and calcineurin regulatescardiac hypertrophy anovel mechanism for progressive cardiac hypertrophy [J].Hypertension,2010,55(4):946-952.
[24] Pandey A K,Verma G,Vig S,et al.miR-29a levels are elevated in the db/db mice liver and its overexpression leads to attenuation of insulin action onPEPCKgene expression in HepG2 cells [J].Molecular and Cellular Endocrinology,2011,332(1):125-133.
[25] He A,Zhu L,Gupta N,et al.Overexpression of micro ribonucleic acid 29 highly up-regulated in diabetic rats leads to insulin resistance in 3T3-L1 adipocytes [J].Molecular Endocrinology,2007,21(11):2785-2794.
Comparative analysis on miRNA transcriptomes of cardiac muscle and skeletal muscle in pigs
TAN Ya,MA Ji-deng,LIU Yi-hui,SHEN Lin-yuan, LI Xue-wei,LI Ming-zhou,ZHU Li
(InstituteofAnimalGeneticsandBreeding,SichuanAgriculturalUniversity,Ya’an,Sichuan625014,China)
【Objective】 This study aimed to understand the function of miRNA in development of distinct muscles in pigs.【Method】 Illumina deep sequencing approach was applied for identification of miRNA transcriptomes of cardiac muscle and two skeletal muscles (longissimusdorsimuscle andpsoasmajor muscle) in pigs,and their differential expressions and target genes were analyzed.【Result】 A total of 56 million sequences were obtained from three small RNA sequencing libraries and 907 miRNAs were identified,of which 441 were co-expressed and 123 were differentially expressed (P<10-6) across the three libraries.The correlation analysis of these differentially expressed miRNAs showed thatlongissimusdorsimuscle andpsoasmajor muscle had a similar expression pattern (r=0.94),while cardiac muscle had a different expression pattern.By ranking analyses of highly expressed miRNAs in three libraries,six out of the top ten miRNAs in cardiac muscle were up-regulated simultaneously compared tolongissimusdorsimuscle andpsoasmajor muscle.GO and KEGG analyses on potentially function of target genes of the six up-regulated miRNAs in cardiac muscle showed that the involved target genes mainly enriched in calcium signaling pathway and insulin signaling pathway.qRT-PCR results also validated the high confidence of the deep sequencing data.【Conclusion】 The differences of miRNA transcriptomes in pigs,to some extent,highlighted the dramatically physiological differences between cardiac muscle and skeletal muscles.
microRNA;cardiac muscle;skeletal muscle;deep sequencing;pig
2014-01-27
国家高技术研究发展计划(“863”计划)项目(2013AA102502);国家转基因生物新品种培育重大专项(2014ZX0800950B,2011ZX08006-003);教育部长江学者和创新团队发展计划项目(IRT13083)
谭 娅(1989-),女,重庆开县人,在读硕士,主要从事猪microRNA相关研究。
朱 砺(1975-),男,四川广元人,教授,主要从事猪的遗传育种研究。E-mail:zhuli7508@163.com
时间:2015-06-30 13:47
10.13207/j.cnki.jnwafu.2015.08.023
Q78;S828.8
A
1671-9387(2015)08-0019-08
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20150630.1347.023.html

