燕麦盐胁迫响应基因的差异表达与生理响应的关系
高彩婷,刘景辉,徐寿军,张玉芹,于华荣
(1内蒙古农业大学农学院,呼和浩特010019;2内蒙古民族大学农学院,内蒙古通辽028000)
燕麦盐胁迫响应基因的差异表达与生理响应的关系
高彩婷1,2,刘景辉1*,徐寿军2,张玉芹2,于华荣2
(1内蒙古农业大学农学院,呼和浩特010019;2内蒙古民族大学农学院,内蒙古通辽028000)
以耐盐燕麦品种VAO-9为材料,通过Illumina测序与数字基因表达谱技术对300mmol/L NaCl处理前后的叶片cDNA文库进行RNA-Seq与DGE分析,同时测定0(CK)、100、200、250、300mmol/L NaCl胁迫下VAO-9幼苗叶片的相对电导率、丙二醛含量和脯氨酸含量,探讨燕麦盐胁迫响应基因的差异表达与生理响应的关系。结果表明:(1)RNA-Seq分析得到Unigenes 65 801条,其基因表达呈现高度的不均一性和冗余性;若差异基因表达谱鉴定分析以log2Ratio≥2且FDR值≤0.001为选择标准,则发现上调和下调表达基因在胁迫0.5h时分别有306和64个,在胁迫3h时分别有639和290个,胁迫24h时分别有1 488和882个。(2)KEGG代谢分析显示,有23 652条Unigenes比对到KEGG中的128条代谢途径,包括与逆境胁迫相关的植物激素信号转导途径、ABC转运蛋白途径、肌醇磷酸代谢途径、渗透调节途径等。(3)在300mmol/L NaCl处理下燕麦叶片的相对电导率、丙二醛含量、脯氨酸含量等生理指标的变化与相关差异表达基因的变化趋势基本一致,说明基因差异表达量与生理反应密切相关。研究认为,在相同的栽培及胁迫处理条件下,可根据植物盐响应生理指标的变化判断耐盐基因的表达情况。
燕麦;盐胁迫;Illumina测序;表达谱;生理响应
土壤盐渍化是世界土壤的主要问题之一,据联合国粮农组织统计,全球陆地的6%以上已经盐渍化,不合理的耕作灌溉造成现有的可耕地不断次生盐渍化。种植耐盐植物,改良植物耐盐性是修复盐渍土最经济有效的途径。新一代高通量测序技术为挖掘耐盐基因、研究植物对盐胁迫的响应机制、揭示其耐盐机理提供了更简单、经济的方法,为改良植物耐盐性、培育耐盐新品种提供理论依据。
燕麦是禾本科(Gramineae)燕麦属(Avena)一年生草本植物,具有耐旱、耐盐碱、耐瘠薄等特点,可作为盐碱地改良的替代作物[1]。国内对于燕麦耐盐性的研究主要集中在栽培耕作措施[2-3]和生理生化指标[4]两方面,对燕麦耐盐分子机制的研究鲜见报道。植物耐盐分子机制的研究在其它植物上取得了一定进展,找到了部分耐盐相关基因,如钾离子转运蛋白、钠离子转运体、H+-ATPase、Na+/H+逆向运输蛋白以及脯氨酸合成途径的关键酶——Δ1-吡咯啉-5-羧酸合成酶(P5CS)基因等[5-8]。有研究发现,拟南芥钾离子转运蛋白基因AtHKT1;1可以通过与sos3的作用抑制根部Na+的过度积累[9];水稻中的Na+转运体OsHKT1;1和OsHKT1;5可通过控制Na+的移动来调节水稻中K+/Na+的比率,在盐胁迫条件下维持根中高的K+浓度和低的Na+浓度[1012];小麦Na+/H+反转运蛋白基因TaNHX1在幼苗经400mmol/L NaCl处理1h后,其转录水平有所提高[13]。盐胁迫等胁迫条件可诱导P5CS基因表达,促进脯氨酸积累,调节细胞的渗透平衡[14]。盐芥幼苗用200mmol/L NaCl处理2h后,其中P5CS基因表达量是对照的2倍[15]。
不同植物或同一植物的不同品种耐盐性不同,不同盐浓度处理下,基因表达量与表达时间不同,植物所表现的生理生化反应也不同。因此在没有燕麦基因组信息的条件下,可利用高通量测序技术查找燕麦盐胁迫响应基因,研究其差异表达情况,并通过对盐胁迫下短期内燕麦的生理响应的研究探索二者之间的关系。本研究以筛选出的耐盐燕麦品种VAO-9为材料,用Illumina测序,得到燕麦盐胁迫的转录组及表达谱信息,比较分析燕麦盐胁迫处理前后转录水平上的表达谱变化,为发现燕麦耐盐基因、深入研究其耐盐机制奠定基础;同时,利用水培法研究盐胁迫下24h内燕麦的生理生化变化,旨在为燕麦耐盐分子机理的研究提供生理参考依据,并依据转录组与表达谱实验结果进一步探讨盐胁迫下燕麦的生理响应与基因差异表达的关系。
1 材料和方法
1.1 试验材料
选用加拿大综合性状好、耐盐性强的燕麦品种VAO-9为试验材料。浸种催芽后,分3组于20℃光照下培养2周,一叶期水培,之后用Hongland营养液培养。14d后,一组正常培养作对照;一组用300mmol/L NaCl胁迫处理,分别取未处理(CK)和处理后0.5、3、24h相同部位叶片,依次定为A、B、C、D样,混样提取总RNA,用于转录组测序,分样用于表达谱分析;一组用0、100、200、250、300 mmol/L NaCl处理0.5、3、8、24h,取相同部位叶片作生理测试。
1.2 RNA提取及测序文库的构建
TRIzol法提取叶片总RNA后[16],用带有Oligo(dT)的磁珠富集纯化mRNA,加入fragmentation buffer将mRNA打断成短片段,并以mRNA为模板,反转录合成cDNA链,经QiaQuick PCR试剂盒纯化、EB缓冲液洗脱后做末端修复、加A并连接测序接头,用琼脂糖凝胶电泳进行片段大小选择,最后进行PCR扩增,构建测序文库,用Illumina HiSeqTM2000进行测序[17]。
1.3 生理指标测定
用抽气法[18]测定叶片相对电导率,茚三酮比色法[18]测叶片游离脯氨酸含量,硫代巴比妥酸法[19]测叶片丙二醛(MDA)含量。
1.4 数据处理及生物信息学分析
使用Excel及SPSS(18.0版)处理分析3个生理指标数据,用HiSeq control software分析测序数据,参考Audic等[20]的方法筛选差异表达基因,进行表达谱(DGE)[21]分析。
2 结果与分析
2.1 测序结果及质量分析
转录组测序得到77 524 148原始短读长序列,去掉接头、低质量的序列得65 424 436条干净的短读长序列,且Q20达94.88%,不确定碱基比例为0,GC含量52.95%(表1),拼接后得到平均长度为645bp的Unigenes 65 801条,说明转录组测序质量较高,可以进行表达谱测序。通过与NR(nonredundant nucleotides)、SwissPro、KEGG(kyoto encyclopedia of genes and genomes)、COG(clusters of orthologous groups of proteins)和GO(gene ontology)等数据库进行比对注释,发现燕麦叶片转录组中有大量响应盐胁迫、干旱胁迫、温度胁迫等逆境胁迫的基因(表2)。当测序量达到30M以上时,基因覆盖率基本达到饱和(图1)。本研究中燕麦表达谱测序量达到147 M,且A、B、C、D等4个样本测序所得Clean Tags比例相近,说明测序质量好,结果可靠。但与参考基因组相比,4个样本测序标签分别有55.98%、54.28%、52.85%、52.12%能被参考标签注释,可能是因为燕麦基因组与参考基因组的序列差异较大所致。

表1 测序数据量统计Table 1 Sequencing output summary

表2 燕麦响应盐胁迫的相关基因Table 2 Genes in response to salt stress in oat
2.2 燕麦测序标签拷贝数和表达量的分布
表3显示,正常培养和盐处理(A、B、C、D)燕麦叶片基因表达谱分析表明,虽然4个样本拷贝数>100的高表达标签种类很少,分别为3.70%、3.67%、3.46%、4.63%,但其表达量分别占mRNA总表达量的72.67%、70.73%、66.31%、68.06%;而拷贝数<5的标签虽然表达量小,分别只占mRNA总量3.69%、3.88%、4.43%、4.35%,但种类非常丰富,分别占mRNA种类的54.20%、54.59%、54.24%、54.26%。这说明细胞中基因表达表现出高度的不均一性和部分冗余性,少数基因呈现高丰度表达,而多数基因表达水平很低(表3)。

续表2 Continued Table 2
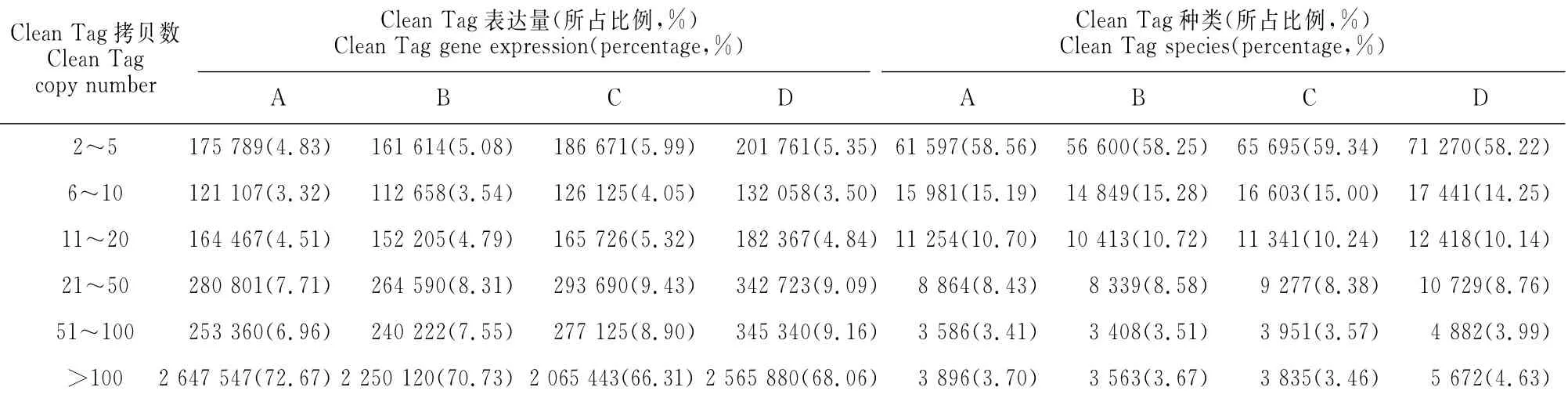
表3 燕麦叶片基因表达谱Clean Tag表达量和种类分布Table 3 Expression level and classes of Clean Tags with different copies expressed in oat leaves

图1 燕麦Illumina测序饱和度分析Fig.1 Saturation analysis of sequencing in oat
2.3 燕麦盐胁迫响应基因的差异表达分析
差异表达基因分析时,选取了65 801个参考基因,其中可被检测的(含有CATG位点)有效参考基因55 061个,占参考基因总数的83.68%(图2)。以log2Ratio(胁迫处理与正常条件下基因差异表达倍数)≥2,且FDR值≤0.001为差异表达基因的选择标准,结果如图3所示:上调和下调表达基因在胁迫0.5h时分别有306个和64个,其中表达上(下)调2~5倍的基因占62.4%(79.7%),上(下)调5~10倍的基因占27.1%(18.8%),上(下)调10倍以上的占10.5%(1.6%);在胁迫3h时分别有639和290个,其中上(下)调2~5倍的基因占73.2%(70.7%),上(下)调5~10倍的基因占20.2%(21.4%),上(下)调10倍以上的占6.6%(7.9%);胁迫24h时分别有1 488个和882个,其中上(下)调2~5倍的基因占64.2%(63.2%),5~10倍的基因占25.3%(30.4%),10倍以上的占10.5%(6.5%)。可见,随着盐胁迫时间的延长,燕麦叶片盐胁迫基因差异表达水平变化主要集中在5倍以内,表现出高度的不均一性。
对差异表达基因功能进行注释,发现有29个上调表达的基因(log2Ratio≥2且FDR<0.001),12个下调表达的基因(log2Ratio≤-1且FDR<0.001)与NR、SwissPro、KEGG、COG、GO等数据库均比对不上(表4和表5),这些基因可能是燕麦特有的与盐胁迫相关的基因,其作用机理需要进一步研究。
图2 燕麦正常和盐胁迫条件下差异表达基因分布Fig.2 Distribution of differentially expressed genes in oat under control and salt stress
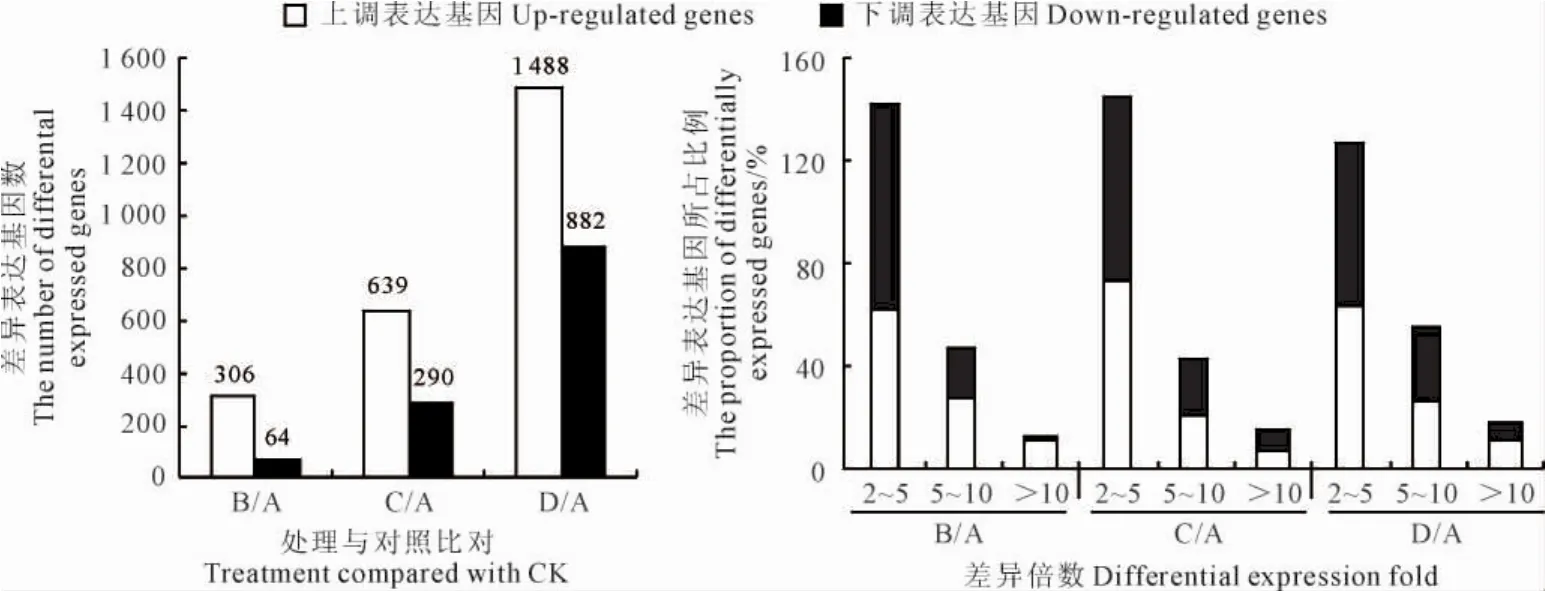
图3 燕麦盐响应基因的差异表达量及差异表达倍数分析Fig.3 Analysis of the amount and fold of differentially expressed genes of oat responding to salt stress
2.4 盐胁迫下燕麦叶片的生理变化特征
如图4所示,在100mmol/L NaCl胁迫下,燕麦叶片的相对电导率和丙二醛含量随胁迫时间持续上升,但与对照相比的增长幅度不大;在200、250、300mmol/L NaCl胁迫24h内,燕麦叶片的相对电导率、丙二醛含量、脯氨酸含量总体上呈现出先上升,后下降,随后又上升的趋势。说明低浓度盐胁迫对燕麦影响较小,未能诱导燕麦耐盐基因的表达,而高浓度盐胁迫下,不同响应速度及表达时间的耐盐相关基因差异表达,使相关的生理指标出现了以上变化趋势。相对电导率与丙二醛的变化趋势可能与测序中发现的H+-ATPase基因、钠离子转运蛋白基因HKT、钾离子转运蛋白基因、Na+/H+逆向转运蛋白基因等在不同胁迫时间的差异表达有关(表6),这些基因表达合成膜转运蛋白、保护性蛋白等为Na+、H+、K+的转运提供能量与通道,保护自身细胞组织免受伤害。300mmol/L盐胁迫3h时,叶片
脯氨酸含量增加较快,可能是高盐胁迫激发燕麦迅速启动P5CS基因编码合成脯氨酸;而胁迫24h时,相关基因上调表达量加大,使脯氨酸的合成大于分解,脯氨酸含量迅速积累,但是生理反应略滞后于基因表达,所以脯氨酸的生理变化趋势与测序结果相符(表6),说明可以根据燕麦对盐胁迫的生理响应判断其相关基因的表达情况,并作为测序结果的验证依据。

表4 不同胁迫时间均上调表达的新基因(log2Ratio≥2且FDR<0.001)Table 4 The unigenes all showed up-regulation in different treatments
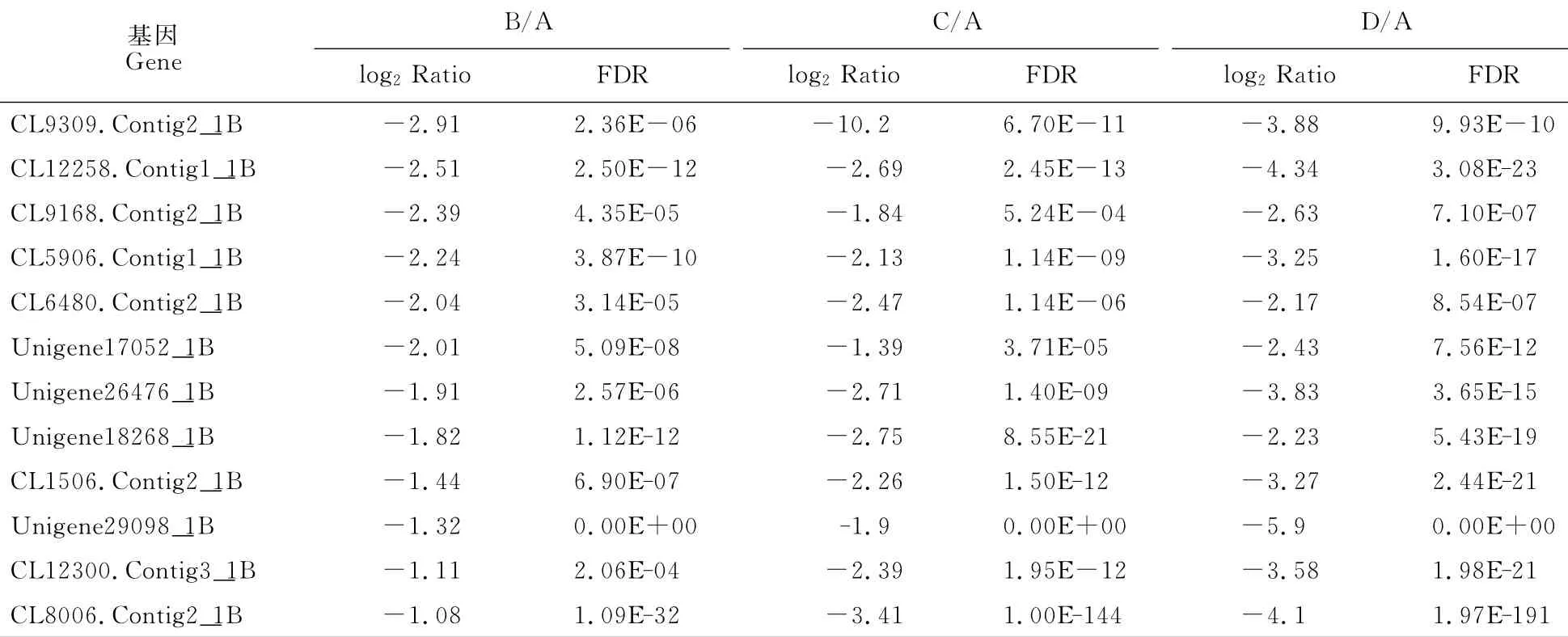
表5 不同胁迫时间均下调表达的新基因(log2Ratio≤-1且FDR<0.001)Table 5 The unigenes all showed down-regulation in different treatments
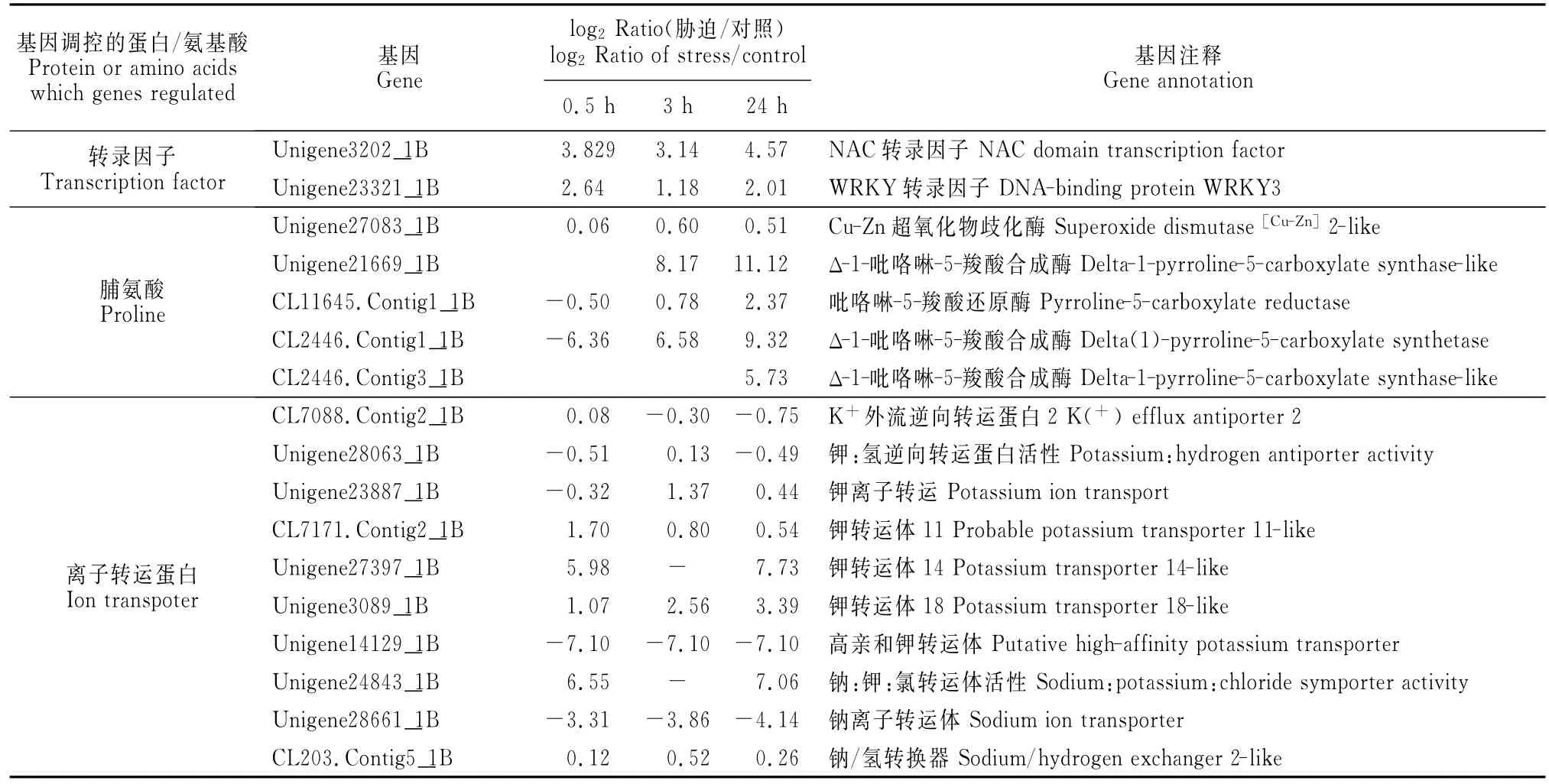
表6 部分盐响应基因的差异表达情况Table 6 Differential expression of part genes response to salt stress

图4 盐胁迫下燕麦叶片相对电导率、脯氨酸、丙二醛含量变化Fig.4 The changes of relative conductivity,proline content and malondialdehyde content in leaves of oat under salt stress
3 讨 论
盐胁迫下,植物通过改变基因的表达来调控代谢与信号转导途径等,从转录、翻译水平响应逆境。研究人员已经利用基因芯片,采用基因突变、基因过表达等技术从拟南芥、水稻等植物中鉴定了许多盐胁迫响应基因[22-23],但植物响应盐胁迫的分子机制复杂,不同的植物存在自身独特的响应方式,且基因芯片只适用于具有全基因组信息的生物。采用高通量测序技术,从转录水平上研究燕麦的表达谱差异,有助于燕麦响应盐胁迫的分子机制及其它禾本科植物耐盐机理的研究。本试验通过对燕麦叶片进行Illumina测序结果表明,基因覆盖率基本接近饱和,不同盐处理时间测序结果可靠,能满足表达谱分析的需要。但由于燕麦基因组信息缺乏,增加了实验中发现的未知基因和新基因研究的难度,限制了表达谱数据的深入挖掘与分析。
本研究通过对耐盐性强的燕麦品种VAO-9的高通量测序产生的数字基因表达谱分析发现,虽然胁迫只引起2%左右的测序标签表达量发生了变化,但其差异表达基因的功能多样化,影响燕麦大多数生理生化代谢途径。说明植物转录系统是相对稳定的,同时又是高度灵敏的一个开放式表达体系[24]。盐胁迫下渗透调节物质、抗氧化蛋白的修复和离子平衡等相关基因的诱导表达表明,燕麦对盐胁迫的保护性响应是多种代谢途径和信号通路共同作用的结果。脯氨酸等渗透调节物质的迅速积累也是燕麦抵御逆境的方式,本研究发现吡咯啉-5-羧酸还原酶(P5CR)和吡咯啉-5-羧酸合成酶(P5CS)基因的上调表达量变化与脯氨酸含量变化趋势基本一致,说明盐胁迫的生理响应可作为判断相关基因表达调控的依据;本实验中还发现中高浓度盐处理8h,脯氨酸含量略有下降,可能是脯氨酸脱氢酶(ProDH)合成基因在胁迫3至8h内上调表达,使ProDH活性升高,抑制脯氨酸的合成。
耐盐植物可通过胞质膜和液泡膜上的各种离子泵调节离子的吸收和区域化以适应盐胁迫,有研究发现用400mmol/L NaCl处理小麦苗1h,Na+/H+反转录基因TaNHx1转录水平有所提高[25],Shi等[26]克隆并证明了Na+/H+反转录蛋白基因SOS 1是Na+和K+在液泡中富集所必须的。本研究中发现与离子平衡有关的Na+/H+、K+/H+逆向转动蛋白基因、K+运输蛋白基因等差异表达,调节燕麦的离子吸收以适应盐胁迫。
除以上发现外,燕麦表达谱分析还发现有响应逆境胁迫的转录因子如MYB、NAC、AP2/EREBP、DREB等上调表达。MYB是植物最大的转录因子家族之一,从转录水平上调控下游基因的表达水平[27],响应逆境胁迫;NAC转录因子是陆生植物特有的逆境胁迫响应因子,水稻SNAC基因在小麦中超表达也可以提高其抗旱和抗盐性[28];AP2/EREBP家族转录因子受生物和非生物胁迫诱导表达,在低温、干旱、盐等胁迫抗性中发挥重要作用,如棉花GhDREB基因过量表达可以提高小麦的干旱、高盐和冻害等多种胁迫抗性[29]。本研究发现NAC、WRKY转录因子与部分钾、钠离子转运蛋白基因的表达量变化趋势相同,可能是这两个转录因子对这些基因的表达有调控作用。离子转运蛋白对于植物响应逆境胁迫,避免离子毒害,调节渗透平衡具有重要作用。本研究中发现一个高亲和性钾离子转运蛋白基因(Unigene14129_1B)在3个胁迫处理中皆下调表达,且表达量相同,这与拟南芥、水稻等植物的研究结果不同[30-31],该基因在燕麦响应盐胁迫中的作用及其代谢过程需要进一步研究。其它离子转运蛋白基因的差异表达证实燕麦可通过选择性吸收K+、Na+来调节地上部分K+/Na+,维持细胞内离子的动态平衡[32]。
燕麦对盐胁迫的代谢响应说明要通过克隆某一个基因达到改良盐敏感品种的耐盐性难度很大。通过对燕麦的转录组表达谱分析,可以较全面的理解燕麦盐胁迫下各代谢途径的相互作用关系,为挖掘植物耐盐新基因,筛选主效基因,阐明燕麦耐盐的分子机制,指导耐盐育种提供了理论基础。
本研究中燕麦叶片的生理变化趋势与表达谱结果一致,但除已知的基因,其他大量差异表达的基因具体调控哪些相关的生理过程需要进一步研究。水稻高盐胁迫下基因的表达结果显示,有的基因在胁迫20min即开始表达,3h后趋于未胁迫水平;大部分基因属于中期上调表达,即胁迫3h开始表达,还有部分基因胁迫24h开始表达[33]。本研究发现,随着盐胁迫时间的延长,燕麦大部分基因差异表达量依次升高,且胁迫24h上调表达的基因所占比例较大(61.2%);少部分基因胁迫0.5h后迅速表达,3h后与对照相近,24h表达量再度上升,这与林鸿宣等的研究结果存在差异。差异产生的原因可能是VAO-9能在24h内承受更高浓度盐的胁迫,300 mmol/L NaCl胁迫3h未能最大程度激发耐盐基因的表达,但随着胁迫时间的延长,燕麦耐盐基因逐渐被诱导表达,24h时差异表达量达到最高值。
综上所述,本研究通过转录组测序所得原始序列数据去除杂质后得到65 424 436条Clean reads,拼接组装后得到平均长度为645bp的Unigenes 65 801条。表达谱分析鉴定发现:胁迫0.5h,306个基因上调表达,64个基因下调表达;胁迫3h,上、下调表达基因分别为639和290个;胁迫24h,上调表达的基因达1 488个,下调表达的有882个,3种比对结果均显示上调2~5倍的基因占60%以上。差异表达基因涉及逆境胁迫相关的植物激素信号转导代谢途径、ABC转运蛋白代谢、肌醇磷酸代谢途径、渗透调节途径等。同时,相对电导率、丙二醛含量、脯氨酸等3个指标的变化趋势基本上与表达谱中相关的差异表达基因量的变化相同,说明以上3个生理指标的变化可以间接反映出燕麦在不同盐胁迫浓度下耐盐基因的表达情况,可作为测序结果的生理验证。
参考文献:
[1] 刘景辉,胡跃高.燕麦抗逆性研究[M].北京:中国农业出版社,2010:1-6.
[2] WU J Y(武俊英),LIU J H(刘景辉),LI Q(李 倩).Effect of tillage techniques and planting depth on saline-alkali tolerance of oats in Inner Mongolia[J].Agricultural Research in the Arid Areas(干旱地区农业研究),2009,27(2):138-141,147(in Chinese).
[3] LI Q(李 倩),LIU J H(刘景辉),WU J Y(武俊英).Effect of different planting patterns on growth and development of oat in saline-alkaline field[J].Journal of Triticeae Crops(麦类作物学报),2008,28(4):669-673(in Chinese).
[4] WANG B(王 波),ZHANG J C(张金才),SONG F B(宋凤斌).Physiological responses to saline-alkali in oats[J].Institute of Soil and Water Conservation(水土保持学报),2007,21(3):86-89(in Chinese).
[5] HU C A,DELAUNEY A J,VERMA D P.A bifunctional enzyme(delta 1-pyrroline-5-carboxylate synthesase)catalyzes the first two steps in proline biosynthesis in plants[J].Proc.Natl.Acad.Sci.USA,1992,89(19):9 354-9 358.
[6] HORIE T,YOSHIDA K,NAKAYAMA H,et al.Two types of HKT transporters with different properties of Na+and K+transport in Oryza sativa[J].Plant J.,2001,27(2):129-138.
[7] SHI H Z,LEE B H,WU S J,et al.Overexpression of a plasma membrane Na+/H+antiporter gene improves salt tolerance in Arabidopsis thaliana[J].Nat.Biotechnol.,2003,21:81-85.
[8] MICH ELET B,BOUTRY M.The plasma membrane H+-ATPase:a highly regulated enzyme with mutiple physiological function[J].Plant Physiol.,1995,108:1-6.
[9] HORIE T,HORIE R,et al.Calcium regulation of sodium hypersensitivities of sos3and athkt1mutants[J].Plant Cell Physiol.,2006,47(5):622-633.
[10] HORIE T,YOSHIDA K,NAKAYAMA H,et al.Two types of HKT transporters with different properties of Na+and K+transport in Oryza sativa[J].Plant J.,2001,27(2):129-138.
[11] GARCIADEBLAS B,SENN M E,et al.Sodium transport and HKT transporters:the rice model[J].Plant J.,2003,34(6):788-801.
[12] REN Z H,GAO J P,et al.A rice quantitative trait locus for salt tolerance encodes a sodium transporter[J].Nature Genet.,2005,37(10):1 141-1 146.
[13] WANG Z N,ZHANG J S,GUO B H,et al.Cloning and characterization of the Na+/H+antiport genes fromTriticum aestivum[J].Acta Botanica Sinica,2002,44(10):1 203-1 208.
[14] RANA M.Genes and salt tolerance:bringing them together[J].New Phytologist,2005,167(3):645-663.
[15] 张 荃.盐芥(Thellungiella halophila)作为耐盐模式植物的耐盐机理研究[D].济南:山东师范大学,2004.
[16] SIMMOL/LS D,CIZDZIEL P E,et al.TRIzolTM:a new reagent for optimal single-step isolation of RNA[J].Focus,1993,4:99-102.
[17] QIU Q,MA T,HU Q J.Genome-scale transcriptome analysis of the desert poplar,Populus euphratica[J].Tree Physiology,2011,31:452-461.
[18] 陈建勋,王晓峰.植物生理学实验指导(第2版)[M].广州:华南理工大学出版社,2006:64-66.
[19] 李合生.植物生理生化实验原理和技术[M].北京:高等教育出版社,2000,184:192-196.
[20] AUDIC S,CLAVERIE J M.The significance of digital gene expression profiles[J].Genome Res.,1997,7(10):986-95.
[21] WANG X W,LUAN J B,LI J M,et al.S S:De novo characterization of a whitefly transcriptome and analysis of its gene expression during development[J].BMC Genomics,2010,11(1):400-412.
[22] JENSEN M K,LINDEMOSE S,DE MASI F,et al.ATAF1transcription factor directly regulates abscisic acid biosynthetic gene NCED3 in Arabidopsis thaliana[J].FEBS Open Bio.,2013,29(3):321-327.
[23] ZENG T,KOU Y J,LIU H B,et al.OsWRKY45alleles play different roles in abscisic acid signalling and salt stress tolerance but similar roles in drought and cold tolerance in rice[J].J.Exp.Bot.,2011,62(14):4 863-4 874.
[24] SUN A Q,ZHANG J D,WAN Y S,et al.In silico expression profile of genes in response to drought in peanut[J].Acta Agronomica Sinica,2013,39(6):1 045-1 053.
[25] WANG Z N,ZHANG J S,GUO B H,et al.Cloning and characterization of the Na+/H+antiport genes fromTriticum aestivum[J].Acta Botanica Sinica,2002,44(10):1 203-1 208.
[26] SHI H,ISHITAN I M,KIM C,et al.The Arabidopsis thaliana salt tolerance gene SOS 1encodes a putative Na+/H+antiporter[J].Proc.Natl.Acad.Sci.USA,2000,97(12):6 896-6 901.
[27] RAHAIE M,XUE G P,NAGHAVI M R,et al.A MYB gene from wheat(Triticum aestivumL.)is up-regulated during salt and drought stresses and differentially regulated between salt-tolerant and sensitive genotypes[J].Plant Cell Rep.,2010,29:835-844.
[28] NAKASHIMA K,TAKASAKI H,MIZOI J,et al.Yamaguchi-Shinozaki K.NAC transcription factors in plant abiotic stressresponses[J].Biochim Biophys Acta,2012,1 819:97-103.
[29] GAO S Q,CHEN M,XIA L Q,et al.A cotton(Gossypium hirsutum)DRE-binding transcription factor gene,GhDREB,confers enhanced tolerance to drought,high salt,and freezing stresses in transgenic wheat[J].Plant Cell Rep.,2009,28:301-311.
[30] KIM E J,KWAK J M,et al.AtKUP1:an Arabidopsisgene encoding high-affinity potassium transport activity[J].Plant Cell,1998,10(1):51-62.
[31] MADHUR G,QIU X H,WANG L,et al.KT/HAK/KUP potassium transporters gene family and their whole-life cycle expression profile in rice(Oryza sativa)[J].Molecular Genetics and Genomics,2008,280(5):437-452.
[32] SA R L(萨如拉),LIU J H(刘景辉),LIU W(刘 伟),et al.Cation-responsive mechanisms of oats to alkali stress[J].Acta Agronomica Sinica(作物学报),2014,40(2):362-368(in Chinese).
[33] 晁代印.水稻高盐胁迫下的转录谱及耐盐相关基因分析[D].上海:中国科学院上海生命科学院,2007.
(编辑:裴阿卫)
Exploring the Relationship of Differentially Expressed Genes and Physiological of Oats in Response to Salt Stress
GAO Caiting1,2,LIU Jinghui1*,XU Shoujun2,ZHANG Yuqin2,YU Huarong2
(1Agricultural College,Inner Mongolia Agricultural University,Huhhot 010019,China;2Agronomy College,Inner Mongolia U-niversity for The Nationlities Tongliao,Inner Mongolia 028000,China)
In this study,a salt tolerant variety VAO-9was used to analyse leaf cDNA library of oat treated with 300mmol/L NaCl by Illumina sequencing and in silico expression profile.At the same time,relative conductivity,MDA,and proline content of leaves under 0(CK),100,200,250and 300mmol/L NaCl stressed were determined,probed the relationship between the differentially expressed of genes and physiological response to salt stress.The results showed that:(1)transcriptome sequencing get 65 801Unigenes,and the gene expression in oat transcriptome showed high nonhomogeneity and redundancy.FDR value≤0.001and log2Ratio≥2as the selection criteria when identify and analyze differentially expressed genes(DEGs),compared with the control,there were 306and 64genes up-and down-regulated expressed respectively coercion 0.5h;coercion 3h,there were 639genes up-regulated,290genes down-regulated;stressed 24h,1488genes up-regulated and 882genes down-regulated.(2)The KEGG pathway analysis indicatedthere are 23 652unigenes comparison to the 128KEGG metabolic pathways,including abiotic stress-related pathways,such as plant hormone signaling pathways,ABC transporter protein pathway,inositol phosphate pathway,osmotic adjustment,and so on.The changing trends of the physiological indicators(relative conductivity,MDA,and proline contents in oat leaves)were the same as the differentially expressed genes under 300mmol/L NaCl stressed,which indicated that the amount of differentially expressed genes is closely related to the physiological responses.Thus,salt tolerance genes expression can be judged according to the physiological indicators of plant salt response.
Oat;salt stress;Illumina sequencing;digital expression profiling;physiological response
Q945.78;Q789
A
10.7606/j.issn.1000-4025.2015.07.1385
1000-4025(2015)07-1385-09
2014-11-18;修改稿收到日期:2015-05-07
国家自然科学基金(31060174,30660084);内蒙古自然科学基金(2010Zd07,200607010301);国家现代农业产业技术体系建设专项(CARS-08-B-5);内蒙古农业大学科技创新团队项目(NDTD2010-8)
高彩婷(1977-),女,讲师,在读博士研究生,主要从事耕作制度与农业生态系统研究。E-mail:bao79@163.com
*通信作者:刘景辉,教授,博士生导师,主要从事耕作制度与农业生态系统研究。E-mail:cauljh@163.com

