伶鼬榧螺(Olivamustelina)的分子鉴定及其形态变异
李海涛,何薇,周鹏,陈凯彪,董燕红
(1.国家海洋局 南海环境监测中心,广东 广州 510300)
伶鼬榧螺(Olivamustelina)的分子鉴定及其形态变异
李海涛1,何薇1,周鹏1,陈凯彪1,董燕红1
(1.国家海洋局 南海环境监测中心,广东 广州 510300)
根据榧螺螺旋部的差异可将其分为两种不同的形态类型,类型Ⅰ(Morphotype Ⅰ)的个体螺旋部陷入体螺层中,其壳顶与前部数螺层愈合;类型Ⅱ(Morphotype Ⅱ)的个体螺旋部高出壳顶,缝合线明显,此类型可明确鉴定为伶鼬榧螺(Olivamustelina)。本研究通过线粒体COⅠ和16S rRNA基因序列的分析对两种形态类型的榧螺进行了分子鉴定。结果表明:两种形态类型的榧螺含有共享的单倍型;不同形态类型间的遗传距离和同一形态类型内的遗传距离重叠;两者的单系性在系统发育分析中没有得到显现,而是共同构成一个单系。因此,两种形态类型的个体均为伶鼬榧螺。类型Ⅰ可能是伶鼬榧螺中一种少见的有别于典型个体的形态变异类型。
伶鼬榧螺;COⅠ基因;16S rRNA基因;形态变异
1 引言
榧螺属(Oliva)隶属于新腹足目(Neogastropoda)榧螺科(Olividae),是一类具有美丽外壳的贝类。榧螺属的贝壳具有一定的药用价值,有清燥润肺、平胆潜阳之功效,主治高血压、青盲内障、骨蒸劳热等[1]。近年来,由于野生资源衰竭,部分榧螺种类已处于濒危状态[2]。
伶鼬榧螺(Olivamustelina)是南海沿岸水域较为常见的种类,也是榧螺属最主要的入药种类。典型的伶鼬榧螺其螺旋部高出壳顶,且缝合线明显。但在自然界中也存在另外一类个体,其贝壳花纹与伶鼬榧螺相似,但螺旋部陷入体螺层中,壳顶与前部螺层愈合。这两种形态类型的差别属于种内还是种间变异,尚未得到其他研究的证实。
由于缺乏肋纹等特征,贝壳的颜色和花纹目前仍是榧螺属分类最重要的依据。但这类特征也常发生变异,Bert[3]描述了一类壳色发生变异的伶鼬榧螺个体,其浅棕色的贝壳上伴有纵向的青灰色斑纹,明显有别于正常个体的“之”字形花纹。形态可塑性为分类学家带来极大困难,使其难以区分种内和种间变异的界限。
分子生物学的发展提供了一种新的物种鉴定手段。线粒体细胞色素c氧化酶亚基Ⅰ(cytochromecoxidase subunit Ⅰ,COⅠ)和16S rRNA基因已在贝类的物种鉴定中得到广泛应用[4-7]。本研究旨在通过线粒体COⅠ和16S rRNA基因序列的分析来鉴别两种不同形态的榧螺,以期为资源的开发利用和保护,以及其他学科的研究提供基础资料。
2 材料和方法
2.1 实验材料
实验所用的榧螺标本采自珠江口及汕头南澳岛海域(表1),腹足肌以90%酒精或-80℃冰冻保存。榧螺标本的壳色和花纹相似,但依据螺旋部的差异可分为2种不同的形态类型:类型Ⅰ(Morphotype Ⅰ)个体的螺旋部陷入体螺层(图1a,1b),螺旋部的所有螺层愈合(图1c);类型Ⅱ(Morphotype Ⅱ)个体的螺旋部高出壳顶,缝合线明显(图1d,1e),螺旋部的螺层不愈合(图1f),此类型的个体可鉴定为伶鼬榧螺。共选取两种形态类型的31个个体用于形态学测量分析,其中24个用于分子生物学分析(表1)。

图1 两种不同形态类型榧螺的贝壳Fig.1 Shells of two Morphotypes of Oliva species
表1 用于DNA序列分析的榧螺标本信息
Tab.1 Specimen information ofOlivaspecimens used for DNA sequence analyses

形态类型标本采集时间采集地点GenBank登录号单倍型COⅠ16SrRNACOⅠ16SrRNAMorphotypeⅠXD⁃12010年4月珠江口KF186640KF186631H1L1MorphotypeⅠXD⁃22010年4月珠江口KF186632L2MorphotypeⅠXD⁃32010年4月珠江口KF186641KF186631H2L1MorphotypeⅠXD⁃42013年3月南澳岛KF186642KF186631H3L1MorphotypeⅡLY⁃22010年4月珠江口KF186631L1MorphotypeⅡLY⁃62013年3月南澳岛KF186643KF186633H4L3MorphotypeⅡLY⁃72013年3月南澳岛KF186644KF186634H5L4

续表1
2.2 贝壳形态数据的测量和分析
用游标卡尺(精度0.2 mm)测量两种形态类型榧螺标本的壳高(SL)和壳宽(SW),比较两种形态类型的SL及壳高与壳宽之比(SL/SW)的差异。
2.3 模板DNA制备、PCR扩增和序列分析
采用碱裂解法制备模板DNA,具体步骤为:剪取小块腹足肌,去离子水清洗两次,吸干水分后置于180 μL 0.05 mol/L NaOH溶液中,95℃消化15 min,然后加入20 μL 1 mol/L Tris-Cl(pH7.6),混匀后10 000 r/min离心10 min,上清液作为模板使用。用于扩增COⅠ基因的引物为LCO-1490(5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′)和LCO-2198 (5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′)[8];扩增16S rRNA基因的引物为16sar-L(5′-CGC CTG TTT ATC AAA AAC AT-3′)和16sbr-H(5′-CCG GTC TGA ACT CAG ATC ACG T-3′)[9]。PCR反应体系总体积为50 μL,其中:2×buffer 25 μL,dNTP 400 μmol/L,引物各0.3 μmol/L,KOD FX高保真PCR酶(TOYOBO公司)1.0 U,模板DNA1.2 μL,加去离子水补足至50 μL。PCR反应条件为:94℃预变性2 min;98℃变性15 s,52℃退火30 s,68℃延伸45 s,共35个循环;最后68℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测后回收纯化,然后进行双向测序,测序引物与扩增引物相同。
将所测的序列与GenBank和国际生命条形码数据库BOLD Systems中查得的其他榧螺属序列一起用DAMBE(Version 5.2.65)软件进行多序列比对。使用MEGA 5.10软件计算变异位点、碱基组成和基于Kimura 双参数模型(Kimura 2-parameter,K2P)下各个类群内部及类群之间的遗传距离,并利用该软件中的邻接法(Neighbor-joining,NJ)和非加权配对算术平均法(Unweighted Pair Group Method with Arithmetic mean,UPGMA)构建所有单倍型系统进化树,可靠性经过1 000次自展(Bootstrap)检验。采用DNASP 5.10软件计算单倍型多样性指数(Hd)和核苷酸多样性指数(Pi)。
3 结果
3.1 形态测量数据的分析
从贝壳的SL及SL/SW比值来看,两种形态类型的榧螺是大范围重叠的,通过SL/SW比值无法将两者明确区分(见表2)。

表2 两种不同形态榧螺贝壳的形态测量数据
3.2 线粒体基因的序列分析
共成功获得22条COⅠ基因序列,各序列长度均为658 bp(不含引物区),无插入/缺失位点,A、T、G、C的平均含量分别为23.4%、39.1%、20.5%和17.0%,A+T的含量明显高于G+C的含量。22条COⅠ基因序列定义了18种单倍型(H1~H18,GenBank登录号KF186640~KF186657)。其中,1个Morphotype Ⅰ个体(XD-3)和2个Morphotype Ⅱ个体(LY-13和LY-19)共享单倍型H2。
在COI基因序列上共检测到52个变异位点(22个为简约信息位点),占核苷酸总数的7.9%,其中46个发生在密码子第三位碱基,6个发生在密码子第一位碱基。所有核苷酸的变异未导致氨基酸序列的变化。MorphotypeⅠ个体间的遗传距离为0.019~0.025,Morphotype Ⅱ个体间的遗传距离为0~0.028,两种形态类型之间的遗传距离为0~0.026,同一形态类型内及两种形态类型间的遗传距离完全重叠,无明显的界限。上述两种形态的个体与其他榧螺,如O.spicata(GenBank登录号FM999165)、O.sayana(GenBank登录号U86333)、O.amalda(BOLD序列号NEOGA900-10、NEOGA901-10和NEOGA902-10)、Olivasp.(BOLD序列号NEOGA879-10)、Olivasp.1(BOLD序列号NEOGA877-10)间的遗传距离较大,在0.129~0.241之间。
共成功获得18条16S rRNA基因序列,其长度为508~510 bp,存在2个位点的插入/缺失。A、T、G、C的平均含量分别为34.5%、29.5%、19.9%和16.1%,A+T的含量显著高于G+C的含量。18条16S rRNA基因序列共定义了9种单倍型(L1~L9,GenBank登录号KF186631~KF186639)。其中,2个Morphotype Ⅰ个体和8个Morphotype Ⅱ个体共享单倍型L1,这一单倍型序列与来自日本Ise bay的一条伶鼬榧螺序列[10](GenBank登录号AB121038)完全相同。
在16S rRNA基因序列上共检测到10个变异位点,占核苷酸总数的2.0%,其中4个为简约信息位点。两种形态类型内的遗传距离分别为0~0.008和0~0.012,两种形态类型间的遗传距离为0~0.014,形态类型内和形态类型间的遗传距离完全重叠。而它们与O.spicata(FM999114)间的遗传距离则高达0.104~0.112。
基于COⅠ基因序列计算的单倍型多样性指数(Hd)和核苷酸多样性指数(Pi)分别为Hd=(0.974±0.024)和Pi=(0.015 5±0.001 1);基于16S rRNA基因序列计算的结果分别为Hd=(0.706±0.120)和Pi=(0.003 6±0.001 0)。单倍型多样性指数高于核苷酸多样性指数,且COⅠ基因所反映的单倍型多样性指数和核苷酸多样性指数均高于16S rRNA基因。
3.3 系统发育关系
基于COⅠ和16S rRNA 基因序列分别构建了NJ树和UPGMA树,两种树的拓扑结构基本一致。因此本文仅给出NJ树,其自展支持率(仅大于70%的列出)标注在节点斜线左侧,UPGMA树的自展支持率标注在节点斜线右侧。
基于COⅠ基因序列构建的NJ树(见图2)显示,18个单倍型合在一起形成一个单系,支持率为99%,在系统树中处于较为进化的位置。两种形态类型榧螺的单系性在系统树上均没有得到显现,相互间无法明确区分。
在基于16S rRNA 基因序列构建的NJ树(见图3)中,9个单倍型与GenBank中的一条伶鼬榧螺序列(GenBank登录号AB121038)共同形成一个单系群,支持率为99%。两种形态类型的单系性也未在系统树上得到显现。
4 讨论
贝壳的形态受多种生物和非生物因素的影响,因此仅依据贝壳形态来进行分类学和系统学研究越来越有争议性[11]。腹足类的个体差异、性别差异和以及发育阶段的不同均可能导致贝壳形态特征的变化[7]。

图2 基于COⅠ基因序列通过邻接法构建的系统发育树Fig. 2 Molecular phylogenetic tree based on COⅠ gene sequences using Neighbor-joining method
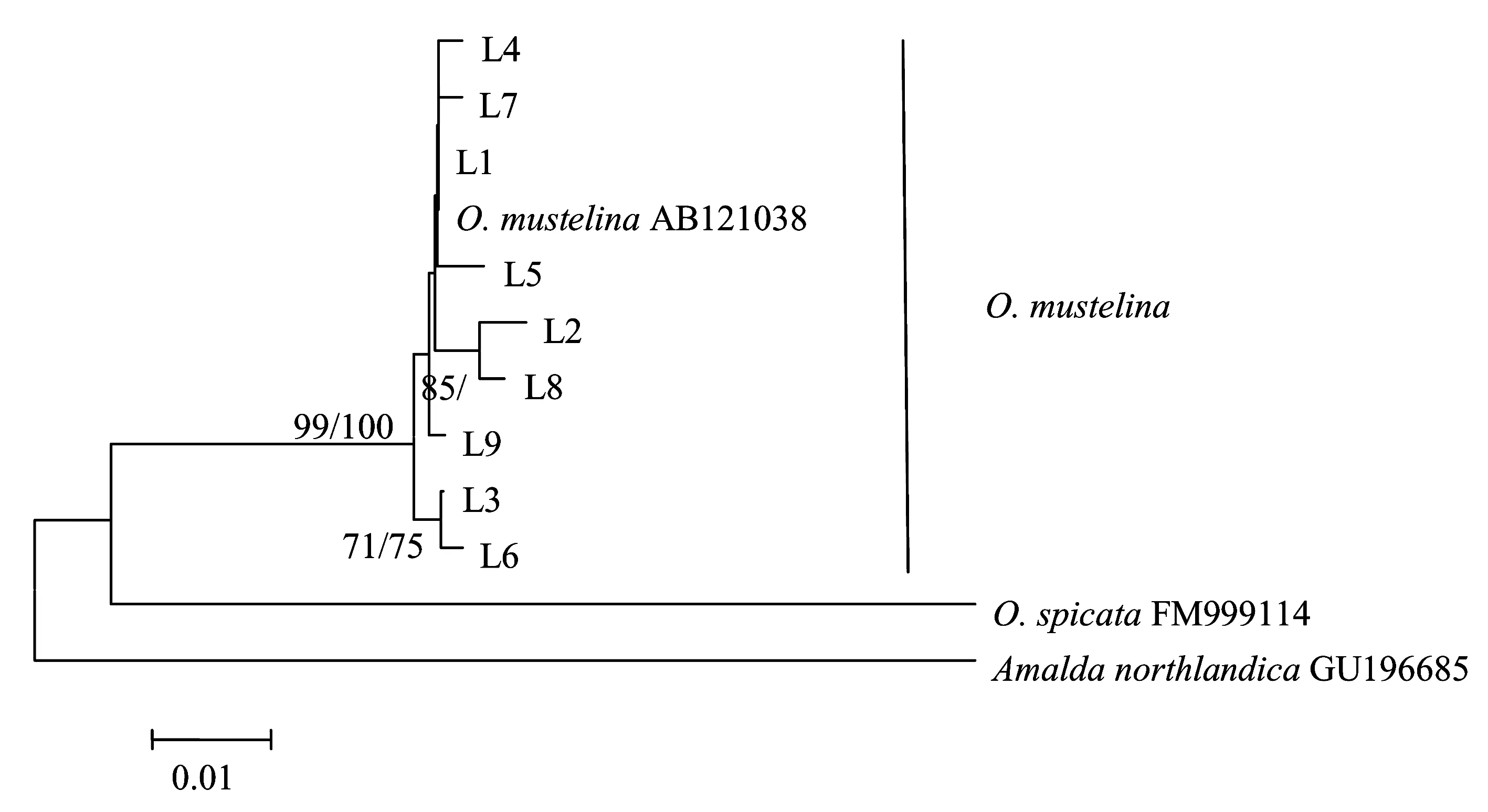
图3 基于16S rRNA基因序列通过邻接法构建的系统发育树Fig.3 Molecular phylogenetic tree based on 16S rRNA gene sequences using Neighbor-joining method
基于线粒体COⅠ和16S rRNA 基因序列的分析结果表明两种形态类型的榧螺之间没有明显的遗传差异:两者享有共同的单倍型;遗传距离完全重叠;各自无法形成单系,而是共同形成一个单系群,且获得很高的支持率。因此,两种类型的个体为同一物种,即伶鼬榧螺。COⅠ基因为蛋白质编码序列,在种内没有发生碱基的插入/缺失,其变异也未造成氨基酸序列的改变。COⅠ基因的进化速率较16S rRNA基因更快,本文的研究结果与其他研究结论[4,12]是一致的。COⅠ基因所揭示的伶鼬榧螺单倍型多样性指数和核苷酸多样性指数均明显高于16S rRNA基因。
榧螺属的分类问题由来已久,其现生种的拉丁学名达500多个,远远超过实际的种类数,同种异名现象十分严重[13]。壳顶是否陷入体螺层是伶鼬榧螺与陷顶榧螺(O.concavospira)的形态区别之一,但伶鼬榧螺中也存在螺旋部陷入体螺层的个体,且其螺旋部的螺层常愈合在一起。因此,这些特征在用于属下种间的鉴别时需要十分慎重,也不宜作为亚种区分的依据。Bert[3]描述的有别于典型伶鼬榧螺形态的个体除颜色和花纹不同之外,其螺旋部也明显低矮。本研究中的Morphotype Ⅰ个体也可能是伶鼬榧螺中一种较为少见的形态变异类型,这类个体在采集标本中的数量不多。
[1] 《中国药用动物志》协作组.中国药用动物志[M].天津:天津科学技术出版社,1983:37.
Cooperative Group of China Medicinal Animal Fauna. China Medicinal Animal Fauna[M]. Tianjin: Tianjin Science and Technology Press,1983: 37.
[2] 傅秀梅,王长云,邵长伦,等.中国海洋药用生物濒危珍稀物种及其保护[J]. 中国海洋大学学报(自然科学版),2009,39(4):719-728.
Fu Xiumei,Wang Changyun,Shao Changlun,et al. Endangered and rare species of marine medicinal organisms and their protection in China[J]. Periodical of Ocean University of China(Natrual Sience),2009,39(4): 719-728.
[3] Bert C. New form ofOlivamustelina Lamarck,1811 form SW Taiwan[J]. Bulletin of Malacology R.O.C.,1984,10: 7-8.
[4] Yu Z N,Kong X Y,Zhang L S,et al. Taxonomic status of fourCrassostreaoysters from China as inferred from mitochondrial DNA sequences[J]. Journal of Shellfish Research,2003,22(1): 31-38.
[5] 张亮,黄艳艳,刘焕章.利用mtDNA 16S rRNA 序列差异鉴定江西青岚湖的河蚌物种[J].水生生物学报,2004,28(3):294-299.
Zhang Liang,Huang Yanyan,Liu Huanzhang. The species identification of Mussels in Qinglan Lake(Jiangxi Province) based on sequence differences of mitochondrial 16S rRNA gene[J]. Acta Hydrobiologica Sinica,2004,28(3): 294-298.
[6] Duda T F,Bolin M B,Meyer C P,et al. Hidden diversity in a hyperdiverse gastropod genus: discovery of previously unidentified members of aConusspecies complex[J]. Molecular Phylogenetics and Evolution,2008,49: 867-876.
[7] 班绍君,薛东秀,张涛,等.三种壳口颜色脉红螺(Rapanavenosa)形态学和线粒体16S rRNA与COⅠ基因片段差异比较分析[J].海洋与湖沼,2012,43(6):1209-1217.
Ban Shaojun,Xue Dongxiu,Zhang Tao,et al. Variance analysis of three color morphs ofRapanavenosabased on morphological traits and mt16S rRNA and COI partial sequences[J]. Oceanologia et Limnologia Sinica,2012,43(6): 1209-1217.
[8] Folmer O,Black M,Hoeh W,et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit 1 from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology,1994,3: 294-299.
[9] Simon C,Frati F,Beckenbach A,et al. Evolution,weighting and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J]. Annals Entomological Society of America,1994,87: 651-701.
[10] Hayashi S. The molecular phylogeny of the Buccinidae (Caenogastropoda: Neogastropoda) as inferred from the complete mitochondrial 16S rRNA gene sequences of selected representatives[J]. Molluscan Research,2005,25(2): 85-98.
[11] Wilke T,Falniowski A. The genusAdriohydrobia(Hydrobiidae: Gastropoda): polytypic species or polymorphic populations?[J]. Journal of Zoological Systematics and Evolutionary Research,2001,39: 227-234.
[12] 苏天凤,黄建华,吴进锋,等.2种东风螺线粒体基因序列多态性研究[J].中国水产科学,2007,14(3):369-376.
Su Tianfeng,Huang Jianhua,Wu Jinfeng,et al. Genetic polymorphism of mitochondrial DNA sequences inBabyloniaareolataandBabyloniaformosae[J]. Journal of Fishery Sciences of China,2007,14(3): 369-376.
[13] Tursch B,Germain L. Studies on Olividae. Ⅰ:A morphometric approach to theOlivaproblem[J]. Indo-Malayan Zoology,1985,2: 331-352.
Molecular identification ofOlivamustelinaand its morphological variation
Li Haitao1,He Wei1,Zhou Peng1,Chen Kaibiao1,Dong Yanhong1
(1.SouthChinaSeaEnvironmentalMonitoringCenter,StateOceanicAdministration,Guangzhou510300,China)
Based on the morphologic difference of spire,Olivasnails with similar shell colors and patterns can be divided into two morphotypes. Morphotype Ⅰ is characterized by the sunk spire in its concavity below the shoulder of the body whorl and the whorls of spire fused together. Morphotype Ⅱ shows the identical conchological characteristics of typical specimens ofO.mustelina. The mitochondrial cytochrome c oxidase subunit Ⅰ (COⅠ) and 16S rRNA gene segments of these two morphotypes were sequenced. Genetic analyses clearly showed many common haplotypes and lack of significant genetic differentiation between the two morphotypes. In addition,further phylogenetic analyses showed that all haplotypes were grouped in a monophyletic clade. Thus,the two morphotypes are not genetically diagnosable,and therefore should be assigned to speciesO.mustelina. Morphotype Ⅰ may be a new form ofO.mustelinathat differ from the typical specimens.
Olivamustelina; COⅠ gene; 16S rRNA gene; morphological variation
10.3969/j.issn.0253-4193.2015.04.011
2014-06-04;
2015-01-03。
海洋公益性行业科研专项(201305010,201105003);南海区海洋环境质量综合评价方法(DOMEP(MEA)-01-03)。
李海涛(1981—),男,湖北省沙洋县人,主要从事贝类分类学研究。E-mail:haitaoli1981@126.com
Q959.212
A
0253-4193(2015)04-0117-07
李海涛,何薇,周鹏,等. 伶鼬榧螺(Olivamustelina)的分子鉴定及其形态变异[J].海洋学报,2015,37(4):117—123,
Li Haitao,He Wei,Zhou Peng,et al. Molecular identification ofOlivamustelinaand its morphological variation[J]. Haiyang Xuebao,2015,37(4):117—123,doi: 10.3969/j.issn.0253-4193.2015.04.011

