一种基因芯片图像的对比度增强方法
李铁军,孙 跃,邵桂芳
(1.重庆大学自动化学院,重庆400044; 2.厦门大学自动化系,福建厦门361005)
一种基因芯片图像的对比度增强方法
李铁军1,孙 跃1,邵桂芳2
(1.重庆大学自动化学院,重庆400044; 2.厦门大学自动化系,福建厦门361005)
为降低对比度对网格定位和基因点分割的影响,提出了一种基因芯片图像的自适应对比度增强方法.基于四阶矩进行图像对比度计算,并统计图像背景灰度,从而实现仅针对基因点的自适应对比度增强.通过6个不同数据集的79幅图像实验表明:该对比度增强方法无需人工干预及参数输入,可自动增强图像对比度,鲁棒性强;对比度增强后的图像网格定位准确率普遍提高,最高可达25%.
基因芯片;图像处理;对比度;网格定位
基因芯片 (cDNA microarray)以高通量、微型化、自动化及低成本等优点成为后基因组时代最重要的基因检测工具之一,是缓解重大疾病预防与控制 (中国大肠癌发病率以4.71%逐年递增[1])的一项新技术[2],能够有效地应用于基因测序和生物学研究[3].基因芯片分析包含样本采集、芯片制备、扫描成像、图像处理、数据分析和生物学应用等步骤[4].基因芯片的图像处理是其中提取基因表达水平信息的关键步骤,其准确性和可靠性将直接影响基因表达谱数据的有效性,并最终决定生物学分析和病理分析的成败.因此,基因芯片图像处理对解读成千上万个基因点的杂交信息,建立可信疾病预测具有重要的意义,其分析的可靠性和准确性将直接影响到芯片的推广与使用.目前,基因芯片图像处理的研究主要在网格定位算法[5-7]、基因点分割算法[8-11]和工具软件三个方面展开.然而,芯片制作过程复杂 (会带来操作失误或仪器误差)、图像质量多变 (会出现噪声等级不同、基因点形态各异和基因点分布疏密不一)以及图像分析方法多样等,对基因芯片图像处理造成巨大挑战.导致目前基因芯片图像处理过程存在如下问题:1)算法精度完全依赖于图像质量;2)单一算法无法满足要求,需要采用新的理论或方法对基因芯片图像处理进行更深入的研究.
图像预处理对提高图像质量,提高图像分析结果精度具有重要意义.现有基因芯片图像预处理着重于基因芯片图像的噪声去除[12-13]或者倾斜校正,很少有人研究对比度增强.目前的基因芯片对比度增强研究,大部分是针对磁共振成像的对比度增强扫描[14].另外有少部分文献研究用直方图均衡化进行基因芯片图像的对比度增强[15-16].实际上,除了常用的直方图均衡化外,一些新方法不断涌现,如小波变换[17]、亮度保持[18]和多尺度top-hat变换[19]等,但这些新方法执行速度较慢.为此,本文提出一种新的自适应对比度增强方法.为仅增强有效基因点信息,本文提出先进行背景灰度统计,然后基于四阶矩自动计算图像自身对比度,最后进行对比度增强.
1 算法原理及流程
通常图像对比度增强采用的方法是灰度变换法,即对图像灰度范围进行线性扩展,如

但该方法涉及到的a,b,c,d参数均需要预先设定,即需要人为给出原始图像的对比度范围以及需要增强的目标对比度范围,不具有适应性.图像整体灰度的改变容易造成过度增强.
像素点与其周围像素点的相似程度,可以说明其对比度变化情况,相似程度高,表明该点所在区域灰度分布比较平坦,也就是具有较低的对比度.四阶矩采用多维方式描述点集分布情况,可以更好地展示像素点与其周围邻近点的灰度对比.此外,由于基因芯片图像的特殊性,即背景灰度值较低,并且只需关注基因点的灰度值.因此,通过背景灰度统计,只放大基因点部分灰度值,从而实现局部对比度增强,是一种有效增强图像对比度的方法,其关键是如何实现背景估计以及对比度增强系数设计.
自适应对比度增强方法主要包括3个关键部分:1)为了只增强基因点,进行背景灰度估计;2)基于四阶矩的图像对比度获取;3)自适应对比度增强.
算法具体流程如下:
步骤1:读入图像f(x,y),为简化计算,将其由16位转换为8位,f(x,y)=f(x,y)/256.
步骤2:采用10×10的Aj窗口在图像边缘位置扫描,取每个窗口内最大灰度值.
步骤3:每个边缘位置分别随机选择两边和中间3个位置,共计12个窗口位置,最后取最大灰度值集合的最小值作为背景灰度值,即

步骤4:为避免随机性及噪声影响,重复步骤2~3,取m=10,即重复10次,取平均值作为最后的背景估计值k.
步骤5:计算图像f(x,y)的水平投影信号x,即将二维信号转换为一维信号.
步骤7:图像对比度计算采用

步骤8:对图像f(x,y)进行扫描,如果判断是背景f(x,y)≤k,则保留图像内容,如果是基因点f(x,y)>k,则进行对比度增强,即

步骤9:为了去除增强后的噪声影响,采用3×3的中值滤波器对图像进行扫描,针对某个像素点g(x,y),取其邻域图像块g(x+n,y+l),其中l,n∈[-1,1].
步骤11:对图像块进行从小到大排序gs=sort(g(x+n,y+l)).
步骤12:取中间值g′(x,y)=gs(5);
步骤13:重复步骤9~12,最后得到滤除噪声后的图像g′(x,y).
步骤14:还原图像g′(x,y)=g′(x,y)×256.
2 实验分析
2.1 实验设置
为验证所提方法的有效性,本文选取了6个数据库进行实验,包括:1)贝勒医学院 (BaylorCollege of Medicine,BCM)提供的图像数据;2)瑞士生物信息研究所的癌症基因组学计算小组提供的图像 (Swiss Institute of Bioinformatics,SIB);3)Joe DeRisi个人网站提供的图像数据;4)加利福尼亚大学 (University of California,San Francisco,UCSF)提供的图像数据;5)基因表达谱数据库(Gene Expression Omnibus,GEO);6)斯坦福大学基因数据库 (Stanford Microarray Database,SMD).数据库详细信息如表1所示.
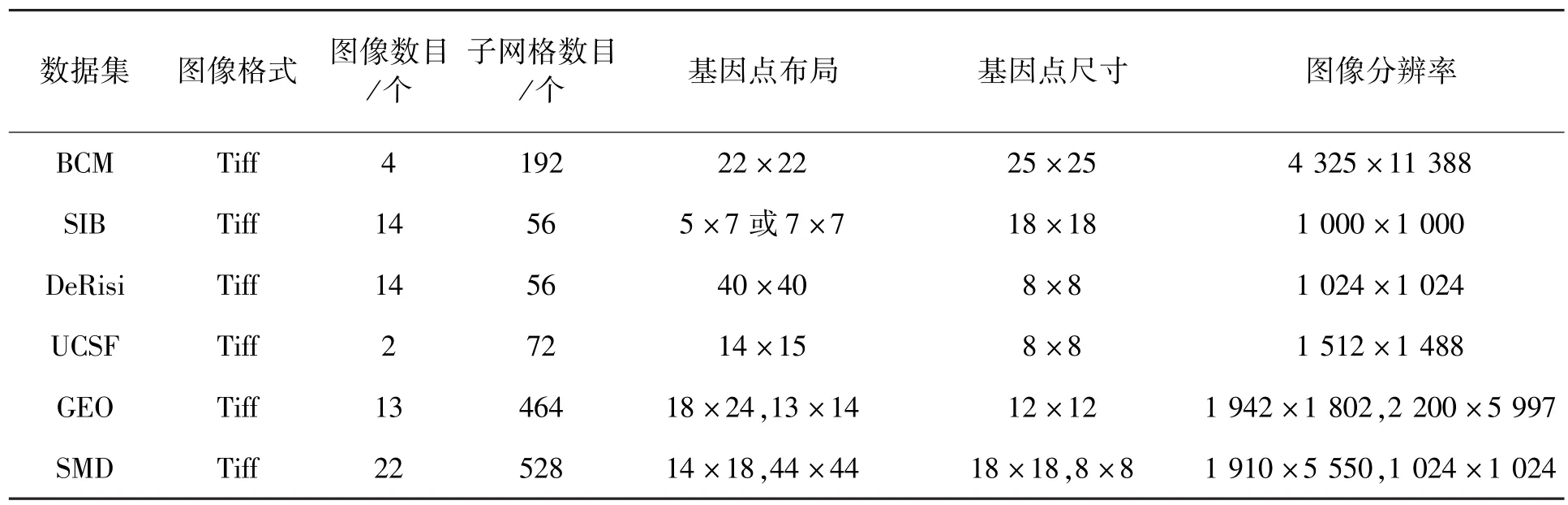
表1 6个数据库的基因芯片图像细节信息Table 1 Information of microarray images on six different data sets
全部实验在Matlab 6.5下实现,为方便实验,利用网格定位方法先将整幅基因芯片图像分割成子网格,对比度增强及后续网格定位效果分析都在子网格图像上进行.
2.2 对比度增强效果
图1展示了子网格图像增强前后的对比度数值.由图1可以看出本文方法在很大程度上提高了图像对比度,可以看出基因点对比度被增强了,证明本文提出的自适应对比度增强方法是有效的.

图1 SMD和GEO数据集对比度增强前后效果对比Fig.1 Comparison of contrast enhancement on SMD and GEO data sets
表2给出了SMD数据库图像的对比度数值统计结果,可以看出对比度增强前图像的平均对比度为2 340.483,增强后平均对比度为6 694.416,图像对比度增强了3倍左右.

表2 对比度增强前后SMD数据集图像对比度值Table 2 Contrast values of SMD data set before and after contrast enhancement
图2给出了其他4个数据库图像对比度增强对比情况,可以看出在6个数据集里,GEO和USCF图像对比度普通较低,BCM和UCSF数据库图像对比度增强倍数最大.实验表明,不论原始图像如何,本文方法都可以有效实现基因点的对比度增强.
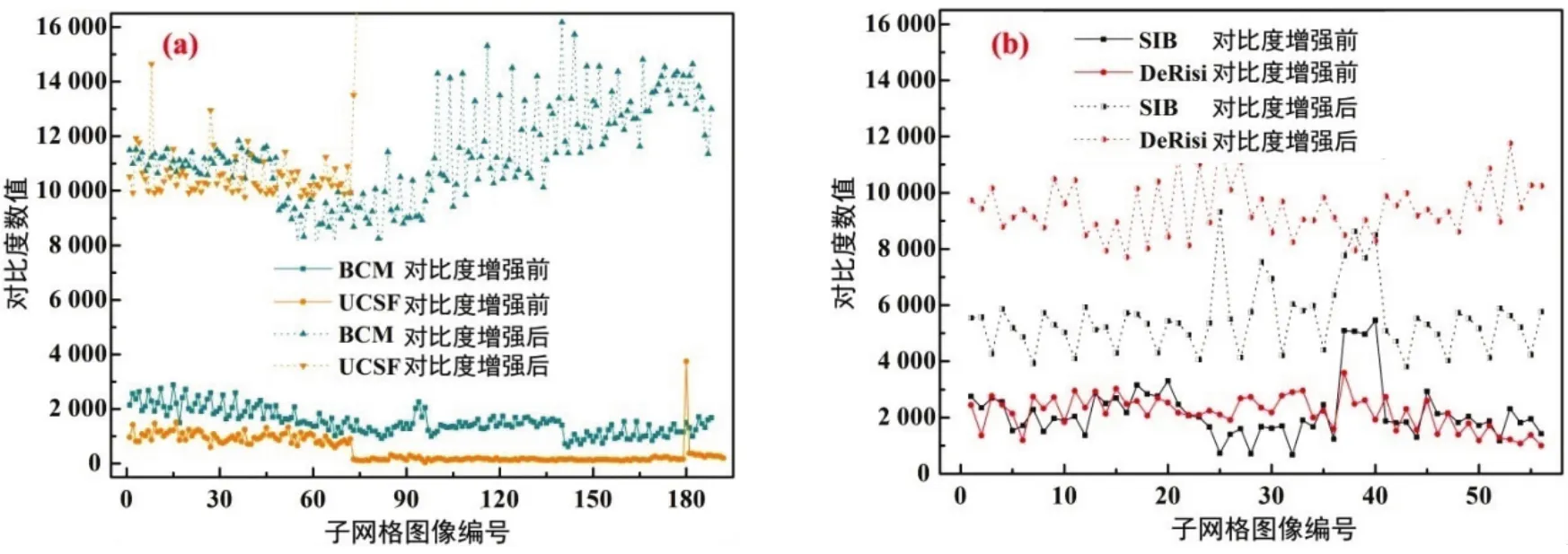
图2 4个数据集增强前后对比度数值变化情况Fig.2 Contrast values vary on four different data sets before and after contrast enhancement
2.3 对比度增强对网格定位的影响
为进一步比较对比度增强对基因芯片图像处理的影响,文中详细分析了6个数据集下不同类型图像的网格定位效果.网格定位方法采用课题组之前提出的基于最大类间方差方法[20].表3给出了对比度增强前后不同数据集下网格定位准确率,可以看出,对比度增强后,网格定位准确率都有所提高.

表3 对比度增强前后网格定位平均准确率Table 3 Average precision of gridding before and after contrast enhancement
图3给出了对比度增强前后网格定位结果,可以看出,对比度较低的图像,都无法精确实现网格定位,而在对比度增强后基本都能准确定位,除了SIB数据库的图像因含有大量噪声导致网格定位失败外.

图3 图像的对比度增强前后网格定位结果对比Fig.3 Gridding results before and after contrast enhancement
上述实验均表明,图像对比度是影响基因芯片图像网格定位准确与否的一大因素,因此,进行对比度增强是有必要的.此外,本文提出的对比度增强方法不需要任何参数输入,完全自动获取图像对比度并进行增强,不仅能用到各种类型的基因芯片图像上,也可以直接移植到其他图像上,具有通用的适用价值.重要的是,利用该方法对基因芯片图像进行对比度增强后,可以大幅度提高后续图像分析处理的准确率,最高可达25%.
3 结语
基因芯片图像预处理对后续网格定位、基因点分割准确率有很大影响,而现有基因芯片图像预处理主要关注图像去噪和倾斜校正.但通过研究发现,图像对比度对网格定位准确率影响非常大.因此,本文提出通过自动计算图像对比度,并估计背景灰度,而有效针对基因点进行对比度增强的方法.该方法可自动实现图像对比度增强,无需任何参数输入.实验结果表明对比度增强后的网格定位准确率可提高5%~25%.通过对BCM,SIB,DeRisi,UCSF,GEO和SMD 6个数据集的图像进行对比度增强实验,发现在6个数据库上,所有图像对比度都得到了增强.就网格定位准确率而言,GEO数据库图像网格定位准确率提高最多,原因是该数据库图像对比度较低,难以区分背景和基因点.而SIB数据库网格定位准确率提高最少,原因是该数据集图像含噪声比较多.此外,本文方法存在噪声点会同时增强及部分图像过度增强的缺陷,后续工作将研究修改对比度增强公式,以避免对比度非常低的图像过度增强,从而导致噪声增多的情况.
[1]王媛媛.大肠癌干细胞基因表达谱特点及其信号通路的筛选鉴定 [D].广州:南方医科大学,2011.
[2]夏俊芳,刘箐.生物芯片应用概述 [J].生物技术通报,2010(7):73-77.
[3]博思数据研究中心.2011—2013年中国生物芯片行业深度调研与投资前景研究报告 [R].北京:博思数据研究中心,2011.
[4]PETER B.An overview of DNA microarray image requirements for automated processing[C]//IEEE Computer Society International Conference on Computer Vision and Pattern Recognition.San Diego:Springer,2005,147.
[5]THAMARAIMANALAN P,KUMAR D,NIRMALAKUMARI K.Efficient gridding and segmentation for microarray images[J].International Journal of Computer Science and Mobile Computing,2014,3(2):353-360.
[6]HARIKIRAN J,AVINASH B,VLAKSHMI D R,et al.Automatic gridding method for microarray images[J].Journal of Theoretical and Applied Information Technology,2014,65(1):235-241.
[7]RUEDA L,REZAEIAN I.A fully automatic gridding method for cDNA microarray images[J].BMC Bioinformatics,2011,12(16):1-17.
[8]ALIAA S E,GHADA E,MOHAMED M E,et al.Segmentation of complementary DNA microarray images using markercontrolled watershed technique[J].International Journal of Computer Applications,2015,110(12):30-34.
[9]MAGULURI L P,RAJAPANTHULA K,SRINIVASU P N.A comparative analysis of clustering based segmentation algorithms in microarray images[J].International Journal of Emerging Science and Engineering,2013,1(5):27-32.
[10]WANGA Zi-dong,ZINEDDIN B,LIANG Jin-ling,et al.cDNA microarray adaptive segmentation[J].Neurocomputing,2014,142(22):408-418.
[11]GIANNAKEAS N,KARVELIS P S,FOTIADIS D I.A classification-based segmentation of cDNA microarray images using support vector machines[C] //The 30th Annual International IEEE EMBS Conference.Vancouver:Springer,2008:875-878.
[12]SHAMS R,RABBANIB H,GAZORC S.A Comparison of x-lets in denoising cDNA microarray images[C]//IEEE International Conference on Acoustic,Speech and Signal Processing.Florence:Springer,2014:2 843-2 847.
[13]HOWLADER T,CHAUBEY Y P.Noise reduction of cDNA microarray images using complex wavelets[J].IEEE Transactions on Image Processing,2010,19(8):1 953-1 967.
[14]CHOYKE P L,DWYER A J,KNOPP M V.Functional tumor imaging with dynamic contrast-enhanced magnetic resonance imaging[J].Journal of Magnetic Resonance Imaging,2003,17(5):509-520.
[15]SAFFARIAN N,ZOU J J.DNA microarray image enhancement using conditional sub-block bi-histogram equalization[C]//IEEE International Conference on Video and Signal Based Surveillance.Sydney:[s.n.],2006:86-91.
[16]尹宁.基因芯片图像识别系统研究 [D].长春:吉林大学,2012.
[17]STARCK J L,MURTAGH F,CANDES E J,et al.Gray and color image contrast enhancement by the curvelet transform[J].IEEE Transactions on Image Processing,2003,12(6):706-717.
[18]IBRAHIM H,KONG N S P.Brightness preserving dynamic histogram equalization for image contrast enhancement[J]. IEEE Transactions on Consumer Electronics,53(4):1 752-1 758.
[19]BAI X Z,ZHOU F G,XUE B D.Infrared image enhancement through contrast enhancement by using multiscale new top-hat transform[J].Infrared Physics&Technology,2011,54(2):61-69.
[20]SHAO G F,YANG F,ZHANG Q,et al.Using the maximum between-class variance for automatic gridding of cDNA microarray images[J].IEEE/ACM Transactions on Computational Biology and Bioinformatics,2013,10(1):181-192.
A Contrast Enhanced Imaging for cDNA Microarray
LI Tie-jun1,SUN Yue1,SHAO Gui-fang2
(1.College of Automation,Chongqing University,Chongqing 400044,China;2.Department of Automation,Xiamen University,Xiamen 361005,China)
An automatic algorithm for enhanced contrast is proposed for microarray imaging to reduce the low precision of gridding and segmentation caused with low contrast images.Contrast was first computed based on the fourth order moment,and a background gray value estimated.Contrast was thus enhanced on the spot only.Experiments on 79 images drawn from six different data sets were then done.The results show that the proposed method can enhance the image contrast with good robustness without any parameter input or other human intervention.In addition,the gridding accuracy is increased with enhanced contrast,up to a maximum of 25%.
cDNA microarray;imaging;contrast;gridding
TP391.4
A
1673-4432(2015)03-0029-06
(责任编辑 李 宁)
2015-04-08
2015-05-12
国家自然科学基金项目 (61403318,6140138);福建省自然科学基金项目 (2013J01255;2013J01246)
李铁军 (1976-),男,工程师,硕士,研究方向为模式识别与智能优化处理.E-mail:litiejunxin@gmail.com

