三种裸腹溞的16S rDNA序列分析与系统分类探讨*
徐 敏,邓道贵,张海军,王文平,晁秋杰
(淮北师范大学生命科学学院,资源植物生物学安徽省重点实验室,淮北 235000)
三种裸腹溞的16S rDNA序列分析与系统分类探讨*
徐 敏,邓道贵**,张海军,王文平,晁秋杰
(淮北师范大学生命科学学院,资源植物生物学安徽省重点实验室,淮北 235000)
为了检验已记录的3种裸腹溞(发头裸腹溞(Moinairrasa)、短型裸腹溞(Moinabrachiata)、微型裸腹溞(Moinamicrura))的系统分类,用试剂盒法分别提取3种裸腹溞的基因组DNA.利用特异性引物,通过PCR扩增了3种裸腹溞的16S rDNA部分序列,并与来自GenBank中每个种类相似度较高的裸腹溞属序列进行分析.结果表明,3种裸腹溞的平均种间相似度为88.7%,碱基中A+T含量均明显高于G+C含量.本研究的发头裸腹溞的16S rDNA序列与GenBank所下载的多刺裸腹溞(Moinamacrocopa)的16S rDNA序列相似度为99%,遗传距离(K2P双参考模型)为0.5%,属种内范围;两个地区的短型裸腹溞测得的16S rDNA序列与GenBank下载的欧洲短型裸腹溞的16S rDNA序列序列相似度相对较低(88%~90%),遗传距离较大(13.2%~13.5%左右),已达到属内种间分化水平.基于16S rDNA构建的NJ树和贝叶斯树也支持以上结论.结果表明,本研究的发头裸腹溞可能为多刺裸腹溞,本研究用的短型裸腹溞与GenBank下载的欧洲短型裸腹溞已经达到种间分化的标准.由于缺乏物种形态资料和其他分子标记的对比,3种裸腹溞的分类地位还需进行更深入的探讨.
裸腹溞; 16S rDNA; 系统分类
裸腹溞属(Moina)是枝角类的重要类群,通称水蚤.本属的描述种类有几十种,我国记载的有十种,存在同物异名现象[1-2].早期的枝角类种类鉴定主要依据其形态特征,由于枝角类种类的分类特征有限以及形态变异十分丰富,加上近源个体之间可以杂交[3],至今只有40%~50%的种类被准确描述,部分种类分类比较含糊,其中一些种类可能以种复合体的形式存在[4].随着分子生物学技术的进步,利用基因标记对枝角类进行分子系统学研究已逐渐成为趋势,Petrusek等以线粒体12S rRNA基因作为分子标记验证了微型裸腹溞在两个大陆间(欧洲中部和澳大利亚)的种类不统一[5];Prosser等用不同引物在不同条件下进行对照,扩增了包括微型裸腹溞在内的不同小型甲壳动物的线粒体COI基因[6],但没有进行系统分化方面的分析.国内学者对薄皮溞属(Leptodora)、西藏拟溞(Daphniopsistibetana)、拟同形溞(Daphniasimiloides)的系统分类进行了研究[7-9],有关裸腹溞属系统分化的研究尚未见报道.本研究通过对安徽巢湖、江苏太湖、洪泽湖、淮北乾隆湖4个湖泊中已发现的短型裸腹溞、发头裸腹溞和微型裸腹溞的16S rDNA序列进行分析,通过与GenBank上下载的与本研究序列相似度最大的裸腹溞属种类的16S rDNA进行比对,探讨他们之间的分子系统关系,提出了一些与传统分类上不一致的问题,以期为裸腹溞属的遗传多样性和物种归属问题提供基础资料.
1 材料与方法
1.1 实验材料
研究用巢湖的短型裸腹溞、发头裸腹溞和微型裸腹溞来自2012年3月巢湖及其支流(南淝河)沉积物中休眠卵的孵化,并分离鉴定[2,10](表1);洪泽湖发头裸腹溞于2013年6月采集于洪泽湖;太湖微型裸腹溞于2013年5月采自太湖;乾隆湖短型裸腹溞于2014年5月采自淮北乾隆湖.几种裸腹溞种类均在智能光照培养箱中用斜生栅藻(Scenedesmusobliquus)培养,培养温度为25℃±1℃、光照时间L∶D=12h∶12h.实验时用吸管吸取单只个体饥饿一昼夜后用双蒸水冲洗,离心管收集备用.
1.2 基因组DNA提取
采用微量样品基因组DNA试剂盒(TIANamp Micro DNA Kit)提取裸腹溞属DNA.由于裸腹溞属种类属于小型甲壳动物,外被甲壳,在提取前用灭菌的牙签捣碎溞体,有利于蛋白酶K对组织进行消化.
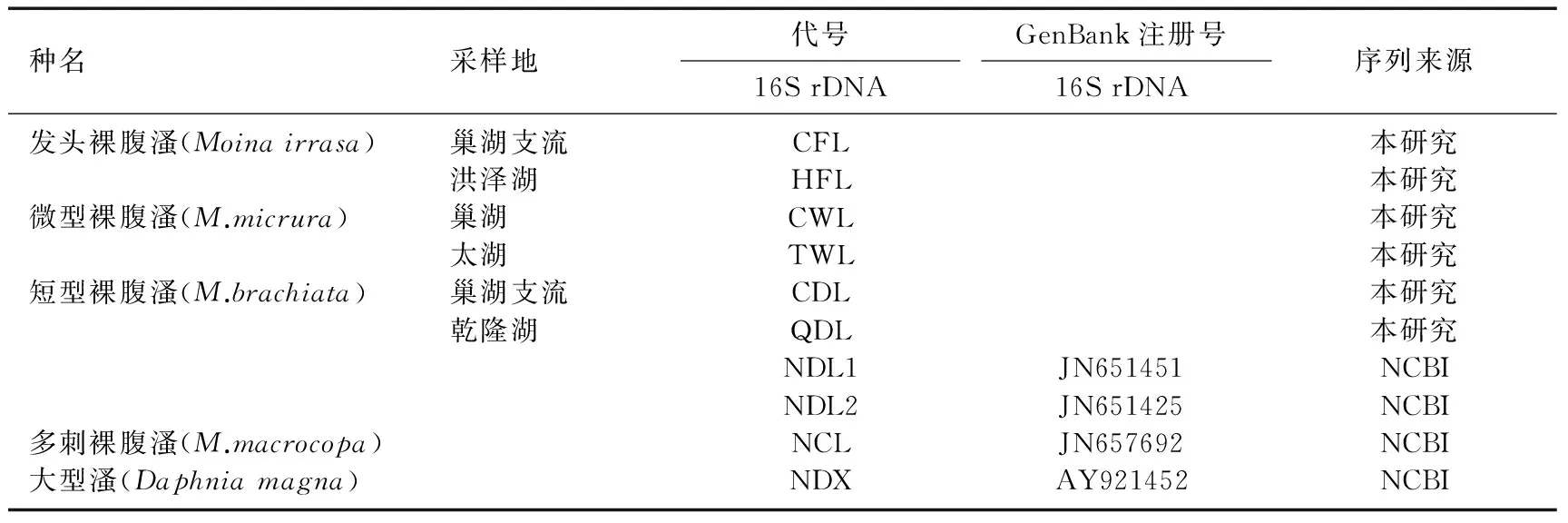
表1 本研究中样本核苷酸序列的来源及编号Tab.1 The origin and number of nucleotide sequence of samples in this study
1.3 PCR扩增及序列测定
用于扩增16S rDNA基因引物[11]:L2510 5′ to 3′(CGCCTGTTTAACAAAAACAT) H3059 5′ to 3′(CCGGTCTGAACTCAGATCATGT)
PCR反应体系25μl,每个反应体系内含10×LA-Taq BufferⅡ 2.5μl、dNTP(2.5 mol)4.0μl、Mg离子(2.5 mol)0.5μl、模板DNA(100 ng/μl左右)1.0μl、Taq酶(5U/μl)0.25μl、上下游引物各(10μm)1.0μl,双蒸水补足到25μl.
扩增条件为95℃预变性3min,95℃变性45s,50℃退火45s,72℃延伸45s,共35个循环,72℃ 10min充分延伸,4℃结束.
扩增产物经电泳检测后,选择目的带清晰且明亮的PCR反应产物送交生工生物工程公司(上海)纯化并测序.所有样品均采用双向测序,正反向对比后确定分析用序列,用DNAStar中的SeqMan比对,并对比峰图修除两端不可靠的碱基,经人工校正后获得CDL有效碱基417个,CFL有效碱基411个,CWL有效碱基417个,HFL有效碱基381个,TWL有效碱基406个,QDL有效碱基491个.其它从GenBank上下载的用于分析的序列见表1.
1.4 系统发生分析
用DNAStar对测序结果进行人工校正,并去除两端冗余序列.使用DNAStar 软件比较序列差异百分比;用Clustalx 1.81软件多重序列比对;用DNAspV5分析变异位点;用Mega 4.1计算不同序列间转换/颠换数,生成序列差异位点表,并计算遗传距离和构建NJ进化树.采用K2P双参模型计算两两遗传距离,系统树设置采用P-distance模型,以大型溞(Daphniamagna)为外群,Bootstrap Replications1000次;用Mrbayes 3.2构建贝叶斯树,替换模型设置参数nst=6(GTR模型),位点速率变异设置invgamma,其余设置为默认值;采用mcmc法运算500000代,每100代取样一次,在舍弃25%老化样本后,用剩余样本构建一致树.
2 结果
CDL、QDL、CFL、CWL、HFL、TWL在DNAStar中经过两两序列比对,TWL与CWL相似度为100%,HFL与CFL相似度为99%,有两个位置发生转换;CDL与QDL相似度为99%,有一个位置发生转换,另一个位置发生颠换.碱基中A+T含量(67.6%)明显高于G+C含量(32.4%);3个物种间平均种间相似度为88.7%,用DNAsp v5进行序列变异位点分析,上述6个裸腹溞属个体可识别位点(包括缺失与丢失的位点)381个,其中保守位点313个,变异位点68个,单一位点3个,简约信息点65个;用MEGA软件分析平均转换/颠换(si/sv)数为0.78,颠换数明显高于转换数.与来自GenBank上Moina的16S rDNA进行序列比对,计算遗传距离见表3;就构建的NJ树(图1a)和贝叶斯树(图1b)来看,两者主要分枝基本一致.
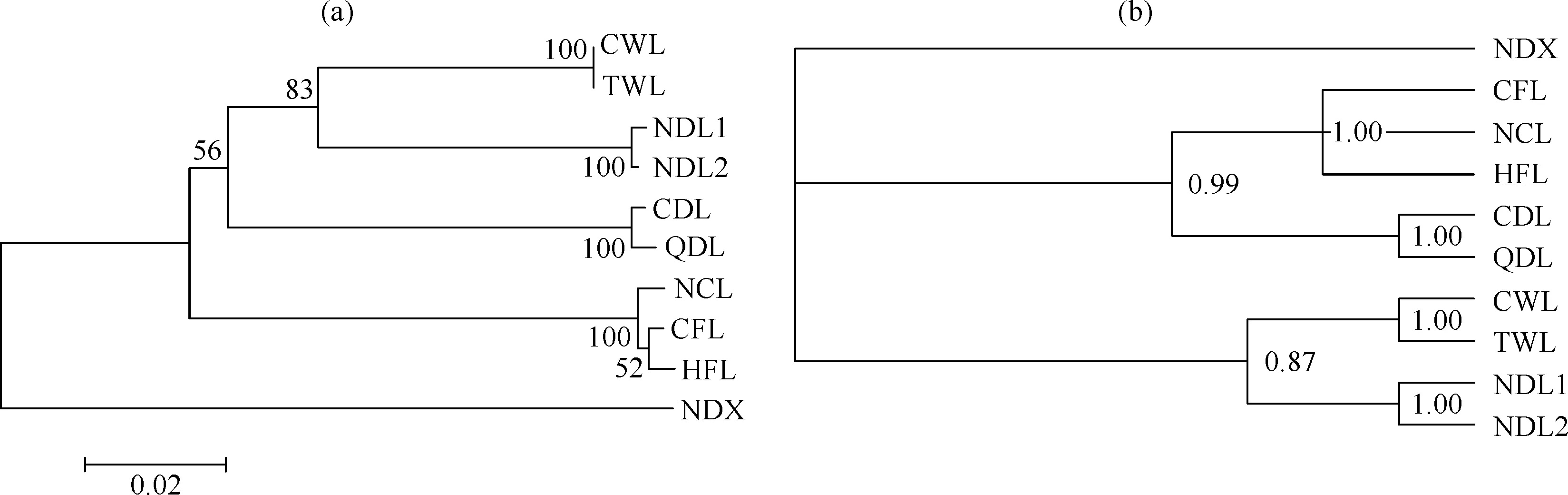
图1 基于9个裸腹溞属个体16S rDNA序列构建的系统发生树(NJ树)(a)和贝叶斯树(b)Fig.1 NJ tree(a) and Bayes tree(b) based on the sequence of 16S rDNA in nine Moina individuals
3 讨论
3.1 3种裸腹溞的16S rDNA序列分析
线粒体rRNA基因是非编码蛋白质的基因,进化过程中未受到密码子编码的选择压力影响,进化速率相对较低且比较保守[12].本研究所测3个裸腹溞属种类的个体中,种间的16S rDNA序列相似度百分比在87%~90%之间(表2),这与臂尾轮虫属的种间序列相似度较一致[13].从已测出的3种裸腹溞的16S rDNA部分碱基序列比对表可以看出,3个裸腹溞属种类的变异区相对比较集中,以序列最短的洪泽湖发头裸腹溞序列作为基准定位,在第1位到第163位碱基之间仅有12处发生碱基的改变,而从164到282之间却有51处发生变异;碱基中A+T(67.6%)与Petrusek等研究的多刺裸腹溞和微型裸腹溞12S rRNA基因的A+T含量(69.4%~72.5%)接近[5],较高的A+T含量是无脊椎动物线粒体DNA中的普遍现象[14-16].
3.2 3个裸腹溞属种类的系统分类探讨
经过DNAStar序列比对,CFL与HFL的16S rDNA序列相似度为99%,遗传距离(K2P双参模型,下同)为0.5%,这可能是地理隔离造成的种内遗传差异.而CFL和HFL的16S rDNA序列与从GenBank下载的NCL的遗传距离也较小(0.5%~1.1%),远低于枝角类线粒体序列种内差异上限5%的分歧度[17].NJ树(P-distance模型)也支持NCL与CFL和HFL聚为一类.因此,就16S rDNA分析看,我国的CFL和HFL应与来自欧洲的NCL为同一个物种.通过查阅文献发现[18-21],发头裸腹溞这一名称仅限国内学者使用,国外学者较少使用,而Goulden把发头裸腹溞归到M.affinis一类[22].本研究的发头裸腹溞雌性体长一般大于1.00mm,孵育囊饱满时,壳高常大于壳长.头部膨大,背侧丛生刚毛,无眼上凹,卵鞍长卵形,内储2个冬卵.后腹部宽大,各侧有10个羽状肛刺.这与《中国动物志·淡水枝角类》[2]中对发头裸腹溞的描述一致.通过形态观察发现,本研究的发头裸腹溞在肛刺数量、卵鞍形状及卵鞍内冬卵的数量等方面与M.affinis存在较大的区别,与多刺裸腹溞的卵鞍形状、卵鞍内冬卵的数量也不同[2,22].Elmoor-Loureiro等报道了一种新发现的多刺裸腹溞,其外形、卵鞍特征和冬卵数量与本研究的发头裸腹溞相似[23].结合16S rDNA序列的分析结果,推测本研究的发头裸腹溞应为多刺裸腹溞.由于缺乏更多的形态和分子对比资料,有关国内发头裸腹溞的归属问题仍需进一步的探讨.
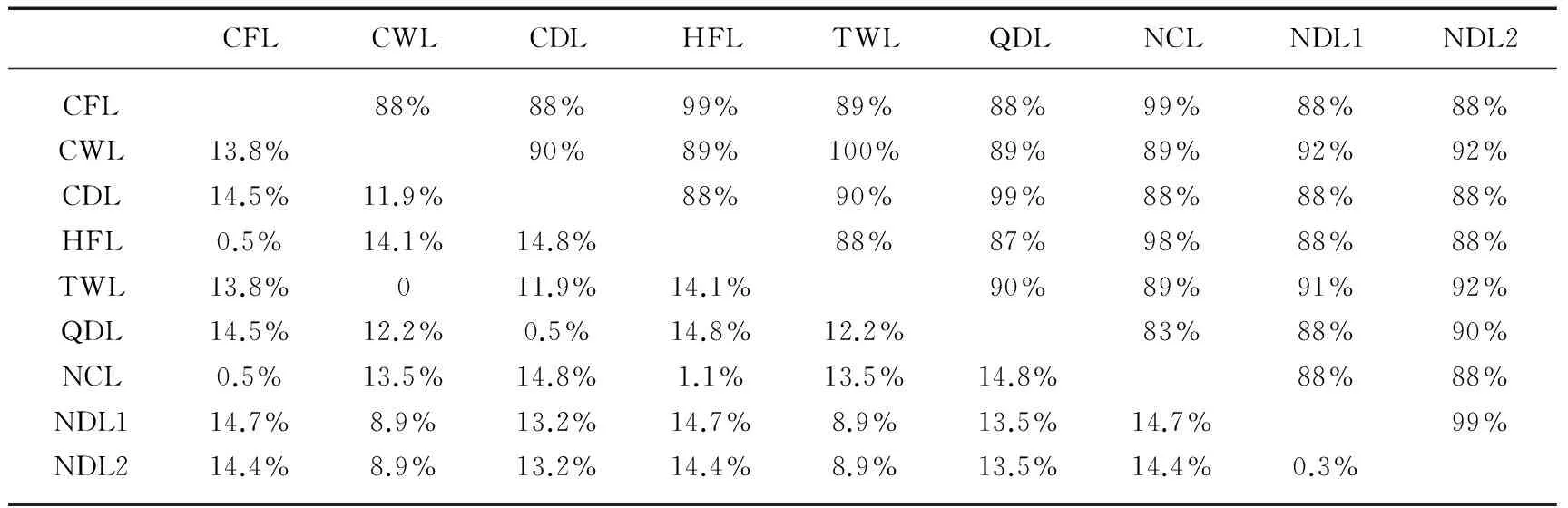
表2 9个裸腹溞属个体16S rDNA的遗传距离(对角线下)和序列相似度(对角线上)Tab.2 The genetic distances(below diagonal) and sequence similarity(above diagonal) of 16S rDNA in nine Moina individuals
本研究的微型裸腹溞雌性体长在0.65~0.83mm之间,复眼很大,位于头顶;头部很大,背侧有较深的眼上凹;后腹部短,侧面有羽状肛刺6个,叉状肛刺1个;卵鞍近乎卵圆型,内储冬卵1个.这些形态特征与《中国动物志.淡水枝角类》[2]中关于微型裸腹溞的描述较一致.来自巢湖与太湖的微型裸溞相似度为100%,但在NCBI的BLAST中未找到与之相似度较高的物种,只有M.brachiata(JN651425)与之有92%的相似度;在GenBank中也没有找到微型裸腹溞16S rDNA序列的数据与本研究进行比对.因此,关于微型裸腹溞的分子系统学仍需做更多的研究.
本研究的短型裸腹溞雌性体长1.00~1.50mm,浅青灰色,壳瓣背缘均匀弓起,头部无毛发,长度超过壳长的一半,向下倾斜,背侧有大而深的眼上凹,后腹部较长,各侧有羽状肛刺8个,叉状肛刺一个,这与《中国动物志·淡水枝角类》[2]中的短型裸腹溞描述较一致.卵鞍内储冬卵一个,这与文献[22]中对短型裸腹溞的描述一致.本研究中,巢湖(CDL)和淮北乾隆湖(QDL)的短型裸腹溞相似度为99%,遗传距离为0.5%.他们与GenBank下载的欧洲短型裸腹溞(NDL1、NDL2)的相似度相对较小(88%~90%),遗传距离(13.2%~13.5%)较大.其遗传距离远远大于枝角类小于5%的种内范围[17],达到端足类钩虾属的种间分化水平[24].就NJ树和贝叶斯树分析,CDL、QDL与下载的欧洲短型裸腹溞(NDL1和NDL2)的分枝较远,也已经不在一个种的界限内.造成这种差距的原因可能有两点:一是长期远距离的地理隔离,不同环境发生的自然选择不同导致遗传上的较大差异;二是存在形态相似而基因有差别的物种或称隐种[25],这种现象已经发生在微型裸腹溞的跨洲(欧洲中部和澳大利亚)分化上[5].圆形盘肠溞曾被看作是枝角类中一个分布较广、生活习性多样化的种,但后来发现它实际上是以一种复合体的形式存在[26-27].其他广泛分布的短钝溞(D.obtusa)、长额象鼻溞(Bosminalongirostris)也依然存在分类上的误区[4].关于枝角类物种的统一性问题一直受到质疑[28-29].近年来,随着分子工具的使用,大量的形态结构和分子研究都证明了非统一性物种的存在,这将会改变我们对枝角类的发展史、生物地理学和物种多样性的认识[30-33].
[1] 赵 文.水生生物学.北京:中国农业大学出版社,2005:181-185.
[2] 蒋燮治,堵南山.中国动物志·淡水枝角类.北京:科学出版社,1979:1-160.
[3] Billiones R, Brehm M, Klee Jetal. Genetic identification ofHyalodaphniaspecies and interspecific hybrids.Hydrobiologia, 2004, 526:43-53.
[4] Korovchinsky NM. How many species of Cladocera are there?Hydrobiologia, 1996, 321:191-204.
[5] Petrusek A, Cerny M, Audenaert E. Large intercontinental differentiation ofMoinamicrura(Crustacea: Anomopoda): one less cosmopolitan cladoceran?Hydrobiologia, 2004, 526:73-81.
[6] Prosser S, Martinez-Arce A, Elias-Gutierrez M. A new set of primers for COI amplification from freshwater microcrustaceans.MolecularEcologyResources, 2013, 13(6):1151-1155.
[7] Xu L, Han BP, Damme KVetal. Biogeography and evolution of the Holarctic zooplankton genusLeptodora(Crustacea: Branchiopoda: Haplopoda).JournalofBiogeography, 2011, 38:359-370.
[8] 赵 文,李 睿.4个品系西藏拟溞12S rRNA基因序列及其分子进化研究.大连海洋大学学报,2012,27(4):300-305.
[9] 顾杨亮,徐 磊,林秋奇等.新亚种——中华拟同形溞(Daphniasimiloidessinensis).生态科学,2013,32(3):308-312.
[10] Deng DG, Xie P, Zhou Qetal. Field and experimental studies on the combined impacts of cyanobacterial blooms and small algae on crustacean zooplankton in a large, eutrophic subtropical Chinese lake.Limnology, 2008, 9:1-11.
[11] Bouchon D, Souty-Grosset C, Raimond R. Mitochondrial DNA variation and markers of species identity in two penaeid shrimp species:PenaeusmonodonFabricius andP.japonicusBate.Aquaculture, 1994, 127(2):131-144.
[12] 窦红霞,张彤晴,许志强等.长臂虾亚科9个种系统发育关系的16S rDNA序列分析.淡水渔业,2008,38(1):40-43.
[13] 程双怀,席贻龙.基于16S rDNA基因序列探讨十种臂尾轮虫的系统关系和分类地位.动物分类学报,2009,34(4):934-940.
[14] Anderson FE. Phylogeny and historical biogeography of theloliginidsquids(Mollusca: Cephalopoda) based on mitochondrial DNA sequence data.MolecularPhylogeneticsandEvolution, 2000, 15(2):191-214.
[15] 高天翔,张秀梅,吉崎悟朗等.日本绒螯蟹线粒体DNA序列研究I.12S rRNA.青岛海洋大学学报,2000,30(1):43-47.
[16] 刘 君,李 琪,孔令锋等.基于线粒体COI的DNA条形码技术在贻贝科种类鉴定中的应用.水生生物学报,2011,35(5):874-881.
[17] Adamowicz SJ, Petrusek A, Colbourne JKetal. The scale of divergence: a phylogenetic appraisal of intercontinental allopatric speciation in a passively dispersed freshwater zooplankton genus.MolecularPhylogeneticsandEvolution, 2009, 50(3):423-436.
[18] 郑又雄.发头裸腹溞卵和胚胎发育形态研究.动物学杂志,1993,28(6):5-8.
[19] 高露娇,赖 伟,陈立侨.发头裸腹溞(MoinairrasaBrehm)种群增长与食物浓度及光照的关系.水产科技情报,1997,24(2):61-64.
[20] 饶小珍,许友勤,陈寅山等.发头裸腹溞——一种新的溞类毒性试验材料.生物技术,2000,10(1):17-19.
[21] 陈丽娜,李玉颖,邓道贵等.种群密度和培养体积对发头裸腹溞生长和繁殖的影响.应用生态学报,2012,23(7):1959-1964.
[22] Goulden CE. The systematics and evolution of the Moinidae.TransactionofAmericanPhilosopnicalSociety, 1968, 58(6):15-41.
[23] Elmoor-Loureiro LMA, Santangelo JM, Lopes PMetal. A new report ofMoinamacrocopa(Straus,1820)(Cladocera, Anomopoda) in South America.BrazilianJournalofMedicalandBiologicalResearch, 2010, 70(1):225-226.
[24] Costa FO, de Waard J, Boutillier Jetal. Biological identifications through DNA barcodes: the case of the Crustacea.CanadianJournalofFisheriesandAquaticSciences, 2007, 64:272-295.
[25] 项贤领,席贻龙,温新利等.萼花臂尾轮虫种复合体遗传分化的空间格局.动物学研究,2010,3(31):205-220.
[26] Frey DG. Questions concerning cosmopolitanism in Cladocera.ArchivfürHydrobiologia, 1982, 145:5-17.
[27] Frey DG. The taxonomy and biogeography of the Cladocera.Hydrobiologia, 1987, 145:19-28.
[28] Dumont HJ, Negrea SV. Introduction to the class Branchiopoda. In: Dumont HJ ed. Guides to the identification of the microinvertebrates of the continental waters of the world 19. Leiden: Backhuys Publishers, 2002.
[29] Frey DG. The taxonomic and phylogenetic significance of the head pores of the Chydoridae(Cladocera).InternationaleRevuedergesamtenHydrobiologie, 1959, 44:27-50.
[30] Adamowicz SJ, Hebert PDN, Marinone MC. Species diversity and endemism in theDaphniaof Argentina: A genetic investigation.ZoologicalJournaloftheLinneanSociety, 2004, 140:171-205.
[31] Cox AJ, Hebert PDN. Colonization, extinction, and phylogeographic patterning in a freshwater crustacean.MolecularEcology, 2001, 10:371-386.
[32] Schwenk KN, Posada D, Hebert PDN. Molecular systematics of European Hyalodaphnia: the role of contemporary hybridization in ancient species.ProceedingsoftheRoyalSocietyofLondon, 2000, B267:1833-1842.
[33] Taylor DJ, Ishikane CR, Haney RA. The systematics of Holarctic bosminids and a revision that reconciles molecular and morphological evolution.LimnologyandOceanography, 2002, 47:1486-1495.
Molecular phylogeny of threeMoinaspecies based on 16S rDNA gene sequences
XU Min, DENG Daogui, ZHANG Haijun, WANG Wenping & CHAO Qiujie
(AnhuiKeyLaboratoryofResourceandPlantBiology,SchoolofLifeScience,HuaibeiNormalUniversity,Huaibei235000,P.R.China)
In order to examine the systematic classification of threeMoinaspecies,M.irrasa,M.brachiataandM.micrurarecorded previously, genomic DNA of threeMoinaspecies were extracted through the kit method. PCR was used to amplify partial sequences of 16S rDNA of threeMoinaspecies by employing specific primers, and same sequence with higher matching rate of eachMoinaspecies from the GenBank were analysed. Based on 16S rDNA, the average interspecific similarity rate among threeMoinaspecies was 88.7%, and the content of A+T was obviously higher than that of G+C.The similarity ofM.irrasain this study andM.macrocopafrom the GenBank was 99%, and the divergence rate was 0.5%, within a range of same species. The similarity ofM.brachiatain this study and the GenBank was relatively low(88%-90%), and the divergence rate was 13.2%-13.5%, without a range of same species. The NJ tree and Bayes tree based on 16S rDNA did also support the above results.The results suggested thatM.irrasain this study andM.macrocopafrom the GenBank might be the same species, andM.brachiatafrom this study and the GenBank had reached the standard of species divergence. Due to the lack of morphology and other genes analysis, the classification status of threeMoinaspecies in this study mentioned need to be further investigated.
Moina; 16S rDNA; systematic classification
*国家自然科学基金项目(31370470)资助.2014-03-18收稿;2014-07-02收修改稿.徐敏(1983~),女,硕士研究生;E-mail:minxuzheng@126.com.
*通信作者;E-mail:dengdg@263.net.

