A 型产气荚膜梭菌青海分离株tetA(P)耐药基因序列分析及蛋白结构预测
冶贵生,马玉花,张 爽,张晓芬,韩志辉,邹 勇,贾跃宁
(1.青海大学农牧学院,青海西宁810016;2.西北农林科技大学动物医学院,陕西杨凌712100)
产气荚膜梭菌(Clostridium perfringens)是一种革兰阳性芽胞杆菌,在污水、食物等中均有分布,是一种重要的人畜共患传染病的病原[1],也是一种引起食物中毒的重要病原菌[2]。随着四环素类抗生素的广泛使用,四环素耐药性细菌也日渐增多,大肠埃希菌[3]、沙门菌[4]、链球菌[5]等多种细菌均表现出对四环素类药物的耐药性。四环素类药物可与细菌核糖体30S亚基结合从而中断细菌蛋白质的生物合成,而细菌对四环素产生耐药性可通过表达核糖体保护蛋白、外排泵等方式进行[6]。
近年来产气荚膜梭菌的耐药性日趋严重,国外学者已经开展了产气荚膜梭菌四环素耐药性以及四环素耐药基因检测等方面的研究[7-10]。产气荚膜梭菌对四环素耐药相关的基因有tetA(P)、tetB(P)和tetM等[11],其中tetA(P)基因编码产气荚膜梭菌外排泵蛋白,其在产气荚膜梭菌主动外排抗药性过程中起着重要作用,而目前国内关于产气荚膜梭菌四环素耐药基因的研究开展的相对较少。因此,本实验通过PCR技术扩增A 型产气荚膜梭菌青海分离株tetA(P)基因,软件分析tetA(P)基因序列,预测TetA(P)蛋白亲水性、二级结构、三级结构、功能位点及跨膜区,以期为进一步研究A 型产气荚膜梭菌TetA(P)蛋白主动外排抗药功能提供参考。
1 材料与方法
1.1 材料
1.1.1 菌株 A 型产气荚膜梭菌青海分离株由青藏高原动物疾病研究室保存。
1.2.1 主要试剂 FT 培养基为北京陆桥技术有限责任公司产品;Ex Taq DNA聚合酶为宝生物工程(大连)有限公司产品;DNA Marker、细菌质粒提取试剂盒为天根生化科技(北京)有限公司产品;其他试剂均为国产分析纯。
1.2 方法
1.2.1 引物设计 根据GenBank中登录的产气荚膜梭菌tetA(P)基因序列(登录号:AB054980.1),设计tetA(P)基因寡核苷酸引物,预期长度为1 263bp。引物 序 列 为:tetA (P)-F:5′-ATGGTTAATAAACTTTCAG-3′、tetA (P)-R:5′-TTAATTATTTTCTTCATAATC -3′。
1.2.2 细菌培养 取超低温保存的A 型产气荚膜梭菌分离株菌液,室温解冻,无菌操作吸取5μL 接种于FT 培养基中,酒精灯融化石蜡无菌封口,37℃厌氧培养16h~17h。
1.2.3 质粒DNA 的提取 收集3mL 37℃厌氧培养的A 型产气荚膜梭菌于1.5 mL 离心管中,12 000r/min室温离心3 min,弃上清,后续操作步骤按试剂盒说明书依次进行。
1.2.4 tetA(P)基因的PCR 扩增 PCR 反应体系:PCR 缓冲液5μL,dNTPs 4μL,tetA(P)-F 1 μL,tetA(P)-R 1μL,质粒DNA 1μL,DNA 聚合酶0.5μL,灭菌超纯水37.5μL。PCR 扩增条件:95℃4min;95℃1min,46℃50s,72℃1min 30s,30个循环;72℃10min,4℃保存。
1.2.5 tetA(P)基因分析、蛋白结构预测 将PCR扩增产物送公司测序,利用生物软件对tetA(P)基因 进 行 基 因 分 析,根 据Garnier-Robson[12]、Chou-Fasman[13]方法分析tetA(P)蛋白的二级结构,根据Kyte-Doolittle[14]方法分析蛋白的亲水性,通过Predictprotein、TMHMM 和I-Tasser服务器预测tetA(P)蛋白的修饰位点、跨膜区和三级结构。
2 结果
2.1 质粒DNA 提取
提取的产气荚膜梭菌分离株质粒DNA 经电泳检测,结果表明,质粒DNA 条带虽然亮度较淡,但基本能够满足作为PCR 扩增的模板(图1)。
2.2 tetA(P)因扩增结果
分离株tetA(P)基因PCR 扩增产物经10g/L琼脂糖凝胶电泳。结果表明,tetA(P)基因扩增良好,大小与预期片段大小长度相符,阴性对照所在泳道无条带(图2)。

图1 质粒DNA 琼脂糖凝胶电泳结果Fig.1 Agarose gel electrophoresis of plasmid DNA
2.3 tetA(P)基因序列分析
2.3.1 核苷酸序列分析 采用DNAStar软件分析tetA(P)基因核苷酸序列,结果表明(表1)tetA(P)基因核苷酸序列长度为1 263nt,A、T 和A+T 含量较高。
2.3.2 氨基酸序列分析 采用DNASTAR 软件推导TetA(P)蛋白氨基酸序列并分析,结果表明,TetA(P)蛋白氨基酸序列长度为420aa,分子质量为45 967.68u,碱性氨基酸和酸性氨基酸的含量较少(表2)。
2.3.3 分离株与参考菌株tetA(P)基因核苷酸序列和氨基酸序列比较结果 将A 型产气荚膜梭菌青海分离株tetA(P)基因与参考产气荚膜梭菌EHE-NE18菌株tetA(P)基因(登录号:JN689220.1)、产气荚膜梭菌Wakayama菌株tetA(P)基因(登录号:AB054980.1)和产气荚膜梭菌CW92菌株tetA(P)基因(登录号:L20800.1)的核苷酸序列与氨基酸序列进行比较,结果表明(图3~图5)分离株tetA(P)基因与EHE-NE18、Wakayama、CW92 参考菌株tetA(P)基因的核苷酸序列同源性依次为100%、99.8%和98.7%,氨基酸序列同源性依次为100%、100%和98.3%。
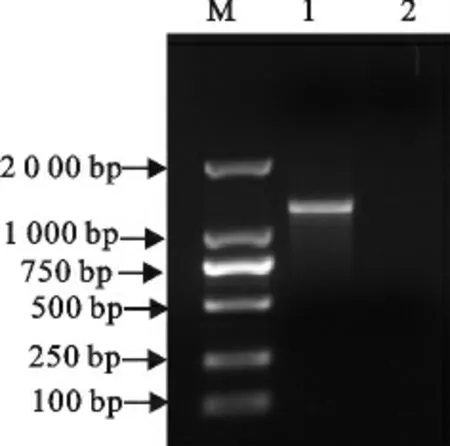
图2 tetA(P)基因PCR 扩增Fig.2 PCR identification of tetA(P)gene

表1 tetA(P)基因核苷酸序列分析结果Table 1 Analysis of nucleotide sequence of tetA(P)gene

表2 TetA(P)蛋白氨基酸序列分析结果Table 2 Analysis of amino acid sequence of TetA(P)

图3 tetA(P)基因核苷酸序列同源百分比Fig.3 Homology percentage of tetA(P)nucleotide sequence
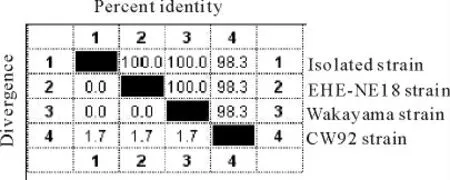
图4 TetA(P)蛋白氨基酸序列同源性百分比Fig.4 Homology percentage of tetA(P)amino acid sequence

图5 tetA(P)基因核苷酸序列同源性比较Fig.5 Homology comparison of tetA(P)nucleotide sequence
2.4 TetA(P)蛋白二级结构预测
Garnier-Robson方法分析结果表明(图6),TetA(P)蛋白二级结构中α螺旋分布较均匀,β折叠分布较多且均匀,β转角和无规则卷曲分布较少,其中无规则卷曲在肽段N-端和中部分布相对较多。Chou-Fasman方法预测结果显示(图6),TetA(P)蛋白二级结构中α螺旋分布少,只有4处且连续氨基酸较短,β折叠分布较多且均匀,β转角分布区段较Garnier-Robson法多。
2.5 TetA(P)蛋白亲水性预测
Kyte-Doolittle方法分析结果显示,TetA(P)蛋白的疏水性氨基酸分布区域较多,且存在7处较大的连续分布区,说明该蛋白的疏水性较高,可能是一种跨膜蛋白(图7)。
2.6 TetA(P)蛋白跨膜区分析
TMHMM 预测结果显示,TetA(P)蛋白具有10个跨膜螺旋的结构,且N-端和C-段均在膜内(图8)。
2.7 TetA(P)蛋白功能位点分析
Predictprotein预测结果显示,TetA(P)蛋白含有4 个N-糖基化位点,分别为156-159位NFSV、253-256 位NLSS、313-316 位NFSL、376-379 位NISV;1个cAMP和cGMP依赖性蛋白激酶磷酸化位点,为71-74位RKLS;5个蛋白激酶C 磷酸化位点,分别为70-72位SRK、158-160位SVR、196-198位TFK、203-205位TFK、326-328位TFR;1 个酪蛋白激酶Ⅱ磷酸化位点,为49-52位TTLE;1个酪氨酸激酶磷酸化位点,为127-133位KDLDEIY;12个N-豆寇酰化位点,分别为78-83位GGVLTG、90-95 位GSISSF、106-111 位GLGSTF、114-119 位GSLEAW、136-141 位GAQAGQ、143-148 位GAFIGI、252-257 位GNLSSV、267-272 位GMILSF、356-361 位GQMNSL、362-367 位GQILGG、371-376位GIIATN、382-387位GIACTS。
2.8 TetA(P)蛋白三级结构预测
I-Tasser服务器对TetA(P)蛋白三级结构进行预测后,结果表明,tetA(P)蛋白存在4种结构模型,这4种模型的C-score值均在[-5,2]的置信区间,空间结构主要是由α-螺旋和无规则卷曲形成,模型1相对于其他3 个模型而言,C-score值最大,可信度较高,说明所肽链可能进行了正确的折叠(图9)。
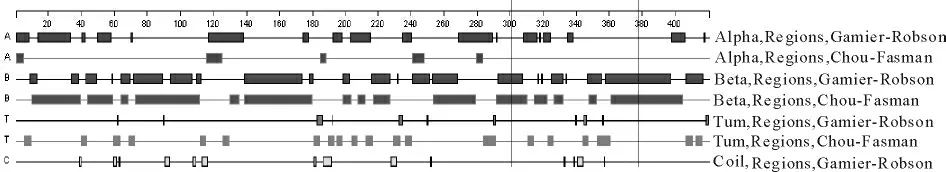
图6 TetA(P)蛋白二级结构Fig.6 Second structure of TetA(P)protein
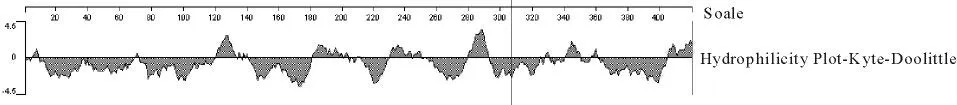
图7 TetA(P)蛋白亲水性Fig.7 Hydrophilicity plot of TetA(P)protein

图8 TetA(P)蛋白跨膜区Fig.8 Transmembrane domain of TetA(P)protein

图9 TetA(P)蛋白三级结构Fig.9 Tertiary structure of TetA(P)protein
3 讨论
主动外排功能是细菌在进化过程中形成的一种防御机制,是细菌产生耐药性的重要途径。当细菌面临抗生素压力时会启动与药物外排相关的基因从而将药物排出菌体细胞,这也是大肠埃希菌、金黄色葡萄球菌等对四环素、β-内酰胺类等抗生素产生多重耐药的重要原因[15]。将产气荚膜梭菌可能编码ABC转运蛋白的基因克隆到野生型菌株中,可降低诺氟沙星和环丙沙星在菌体内的积累。
TetA(P)蛋白作为产气荚膜梭菌与四环素耐药相关的外排泵蛋白,其编码基因一般定位于菌体质粒上[16],TetA(P)蛋 白 可 调 节 四 环 素 的 主 动 外排[17]。A 型产气荚膜梭菌青海分离株tetA(P)基因长度为1263个核苷酸(含起始密码子和终止密码子),编码420个氨基酸。核苷酸序列同源性比对结果说明分离株与EHE-NE18参考菌株无突变位点,与Wakayama参考菌株有2处突变位点,与CW92参考菌株有16处突变位点。氨基酸序列同源性分析显示分离株与EHE-NE18、Wakayama参考菌株均无氨基酸突变,其中与Wakayama参考菌株虽有核苷酸序列的突变,但由于密码子具有简并性的特点,致使分离株与Wakayama参考菌株TetA(P)蛋白的氨基酸序列未见差异。另外,A 型产气荚膜梭菌青海分离株TetA(P)蛋白疏水性氨基酸数量较多,蛋白的疏水性较高,该蛋白具有跨膜蛋白的特征,且分离株TetA(P)蛋白具有10 个跨膜螺旋结构,但这与Bannam TL 等[18]报道的产气荚膜梭菌TetA(P)蛋白含有12个跨膜区有所不同。
[1] 陆承平.兽医微生物学[M].3 版.北京:中国农业出版社,2001:318-320.
[2] Sparks S G,Carman R J,Sarker M R,et al.Genotyping of enterotoxigenic Clostridium perfringens fecalisolates associated with antibiotic associated diarrhoea and food poisoning in north America[J].J Clin Microbiol,2001,39(3):883-888.
[3] 牛建宁,崔恩慧,唐 攀,等.鸡源致病性大肠杆菌对四环素类抗生素耐药性及耐药基因检测[J].西北农业学报,2014,23(11):35-39.
[4] 宁家宝,梁梓森,陈建红,等.鸡白痢沙门菌对四环素类药物耐药性和耐药基因调查分析[J].中国兽医杂志,2014,50(7):39-41.
[5] 黄文明,欧阳昀,张民泽,等.广东地区猪链球菌的耐药性特点及四环素耐药基因携带情况[J].中国畜牧兽医,2013,40(5):54-58.
[6] 何 伟.革兰阳性菌对四环素耐药的生化和遗传机制[J].国外医药:抗生素分册,2005,26(5):201-204.
[7] Gholamiandehkordi A,Eeckhaut V,Lanckriet A,et al.Antimicrobial resistance in Clostridium perfringens isolates from broilers in Belgium[J].Vet Res Commun,2009,33(8):1031-1037.
[8] Park M,Rooney A P,Hecht D W,et al.Phenotypic and genotypic characterization of tetracycline and minocycline resistance in Clostridium perfringens[J].Arch Microbiol,2010,192(10):803-810.
[9] Soge O O,Tivoli L D,Meschke J S,et al.A conjugative macrolide resistance gene,mef(A),in environmental Clostridium perfringens carrying multiple macrolide and/or tetracycline resistance genes[J].J Appl Microbiol,2009,106(1):34-40.
[10] Diarra M S,Silversides F G,Diarrassouba F,et al.Impact of feed supplementation with antimicrobial agents on growth performance of broiler chickens,Clostridium perfringens and Enterococcus counts,and antibiotic resistance phenotypes and distribution of antimicrobial resistance determinants in E.coli isolates[J],Appl Environ Microbiol,2007,73(20):6566-6576.
[11] Sasaki Y,Yamamoto K,Tamura Y,et al.Tetracycline-resistance genes of Clostridium perfringens,Clostridium septicum and Clostridium sordellii isolated from cattle affected with malignant edema[J].Vet Microbiol,2001,83(1):61-69.
[12] Garnier J,Osguthorpe D J,Robson B.Analysis of the accuracy and implications of simple method for predicting the secondary structure of globular proteins[J].J Mol Biol,1978,120(1):97-120.
[13] Chou P Y,Fasman G D.Prediction of the secondary structure of proteins from their amino acid sequence[J].Adv Enzymol Relat Areas Mol Biol,1978,47:45-148.
[14] Kyte J,Doolittle R F.A Simple method for displaying the hydropathic character of a protein[J].J Mol Biol,1982,157(1):105-132.
[15] Li X Z,Nikadio H.Efflux-mediated drug resistance in bacteria:an update[J].Drugs,2009,69(12):1555-1623.
[16] Rafii F,Park M,Carman R J.Characterization of an ATPbinding cassette fromClostridium perfringens with homology to an ABC transporter from Clostridium hathewayi[J].Anaerobe,2009,15(4):116-121.
[17] Lyras D,Rood J.Genetic organization and distribution of tetracycline resistance determinants in Clostridium perfringens[J].Antimicrob Agents Chemother,1996,40(11):2500-2504.
[18] Bannam T L,Johanesen P A,Salvado C L,et al.The Clostridium perfringens TetA(P)efflux protein contains a functional variant of the Motif A region found in major facilitator superfamily transport proteins[J].Microbiology,2004,150(Pt 1):127-134.

