基因芯片检测耐利福平结核分枝杆菌准确性的Meta分析
冉兵 蔡林
·论著·
基因芯片检测耐利福平结核分枝杆菌准确性的Meta分析
冉兵 蔡林
目的 对基因芯片方法在检测耐利福平结核分枝杆菌的准确性进行系统评价,为基因芯片技术应用于临床检测耐药结核分枝杆菌的必要性提供证据。方法 计算机检索The Cochrane Library(2014年第1期)、 PubMed、EMbase、Wanfang Data、CNKI数据库,检索时限均为建库至2014年2月。由2位研究者根据纳入与排除标准独立筛选文献、提取资料,采用评价诊断准确性质量研究(quality assessment of diagnostic accuracy studies,QUADAS)条目评价纳入研究论文的方法学质量,检出相关文献223篇,按照标准纳入22篇文献。采用Meta-DiSc 1.4软件对其敏感度(SEN)、特异度(SPE)、阳性似然比(+LR)、阴性似然比(-LR)、诊断比值比(DOR)进行异质性检验和合并分析,绘制汇总受试者工作特征(SROC)曲线,计算曲线下面积(AUC)。 结果 共纳入22篇文献(包括一篇多中心研究文献),25个研究,包括4879株经传统药敏试验鉴定结核分枝杆菌菌株,其中耐RFP菌株(1314株)、敏感菌株(3565株)。基因芯片技术检测Mtb对RFP耐药的SEN合并为0.89[95%CI(0.87~0.91)]、SPE合并为0.98[95%CI(0.97~0.98)]、+LR为30.89[95%CI(22.15~43.06)]、 -LR为0.10[95%CI(0.07~0.15)]、 DOR合并为354.06[95%CI(212.09~591.07)]、AUC为0.9833。结论 基因芯片方法具有较好的诊断价值;以传统药敏法为金标准,基因芯片方法特异度、敏感度,诊断OR值均较高,说明其在Mtb对RFP耐药检出率较高,可作为临床结核分枝杆菌耐药检测的辅助手段。
结核分枝杆菌; 利福平; 寡核苷酸序列分析; 结核, 抗多种药物性; 细菌蛋白质类; Meta分析
20世纪40~50年代初随着一些抗结核药物的陆续出现,Mtb得到了阶段性的控制。但是由于公共卫生监督力度的不足,在部分抗生素的广泛滥用与合并艾滋病感染等多种因素下,结核病疫情又呈上升趋势,尤其是在发展中国家,如印度、中国等更是成为耐多药结核病的重灾区[1]。在中国约有1/10的结核病患者是耐多药结核病(至少对一线药物异烟肼(INH)和利福平(RFP)耐药,MDR-TB),在所有新发结核病患者中MDR-TB的比例更是达到5.7%,且8%的MDR可被定义为广泛耐药结核病(即至少对一线药物INH和RFP耐药外,还同时对卡那霉素、阿米卡星或卷曲霉素中任何一种注射类药物和任何一种氟喹诺酮类药物耐药,XDR-TB)[2],甚至部分地区已经出现全耐药的Mtb。
Mtb感染、特别是耐药Mtb感染率的增加,已是一个广泛的世界医疗问题[3]。Mtb因生长缓慢,应用传统的药物敏感性试验(简称“药敏试验”)检测,至少需4~8周时间,这对临床的早期诊断和治疗带来了障碍[4]。Mtb及其耐药性的快速鉴定对结核病早期治疗和控制耐药菌的传播具有重要的意义。因此,近年来建立了多种检测Mtb耐药基因的方法用于快速检测耐药Mtb,以弥补传统药敏试验的不足[5]。基因芯片检测方法是利用荧光标记的特定基因探针与Mtb基因杂交,经芯片扫描仪扫描,检测荧光信号,比较与标准株间荧光信号的异同或通过序列分析,判定耐药基因是否存在突变,确定Mtb的耐药情况。基因rpoB已被证明是Mtb耐RFP的相关基因,并已确定了该基因的热突变(突变频率较高的基因位点)位点[6]。即使已有很多有关基因芯片检测Mtb耐药基因rpoB的研究,但因限于实验室资源有限,多为小样本研究,到目前为止还罕有多中心大样本的研究评价该方法与传统药敏试验检测耐RFPMtb的敏感度和特异度,特别是在耐药结核病发生较严重的地区[7]。为了进一步评价基因芯片检测方法在临床的应用前景,笔者带着该问题对国内外文献进行了检索,尝试通过Meta分析的方法得出较为可信的结果供业内人士参考。
资料和方法
一、纳入与排除标准
1. 研究类型:国内外已发表的基因芯片方法诊断Mtb对RFP耐药的诊断性试验论文。文种限中、英文。
2. 研究对象: ① 研究菌株为传统药敏试验确定的Mtb菌株(敏感株和耐RFP菌株),以及用于对照的标准菌株H37Rv。② 能获得基因芯片方法独立诊断Mtb对RFP耐药的真阳性值(TP)、假阳性值(FP)、假阴性值(FN)、真阴性值(TN)等原始测量数据。
3. 诊断方法:待评价试验为基因芯片方法检测,以传统药敏试验结果为金标准。
4. 结局指标:敏感度(SEN)、特异度(SPE)、阳性似然比(+LR)、阴性似然比(-LR)、诊断比值比(DOR),以及汇总受试者工作特征(SROC)曲线下面积(AUC)。
5. 排除标准:① 会议摘要;② 重要资料报告不全者;③ 测量指标不明确者;④ 不同期刊重复发表的文献;⑤ 对同一个机构的重复报道,只纳入其中质量最好者;⑥ 排除仅题目涉及耐药Mtb和rpoB基因或基因序列,而与基因芯片方法诊断Mtb对RFP耐药无关的研究。
二、 检索策略
1. 计算机检索:The Cochrane Library(2014年第1期)、 PubMed、EMbase、Wanfang Data、CNKI数据库,收集采用基因芯片方法诊断Mtb对RFP耐药的诊断性试验,检索时限均为建库至2014年2月。
2. 检索词选择:在 PubMed、EMbase、The Cochrane Library中英文检索词为“tuberculosis、tuberculosis and drug resistance、tuberculosis and drug resistance and gene chip、 tuberculosis and drug resistance and gene chip and rifampicin”。Wanfang Data、CNKI数据库中文检索词为“结核分枝杆菌,耐药结核分枝杆菌,耐利福平结核分枝杆菌,基因芯片,快速检测”。
3. 检索步骤:文献检索分3个步骤:①在上述数据库中检索相关的原始论文,并对文献、文题、摘要、所用的关键词及主题词进行分析,进一步确定文献检索关键词;②运用所有相关关键词及主题词进行检索,若摘要和纳入标准相符合,则阅读全文;③通过所获文献进行进一步手工和电子数据库检索。
三、 文献筛选与资料提取
由2 位研究者根据纳入与排除标准独立筛选文献、提取资料,所有进入筛选的文献均可获得全文资料;如遇分歧讨论解决。提取内容包括研究的一般信息(文献编号、文题、年份、作者、样本量、统计方法),患者信息(性别、年龄等)、是否与所建标准比较TP、FP、FN、TN。初检出相关文献223篇,其中中文117篇,英文106篇。剔除重复发表及明显不符合的文献,纳入标准的文献170篇,经阅读文题和摘要,纳入57篇。进一步查找和阅读全文,排除其中的部分数据不全及未与金标准进行对照的研究论文,最后纳入22篇文献(包括一篇多中心研究文献),25个研究,包括4879株经传统药敏试验鉴定结核分枝杆菌菌株,其中耐RFP菌株(1314株)、敏感菌株(3565株)(图1、表1)。
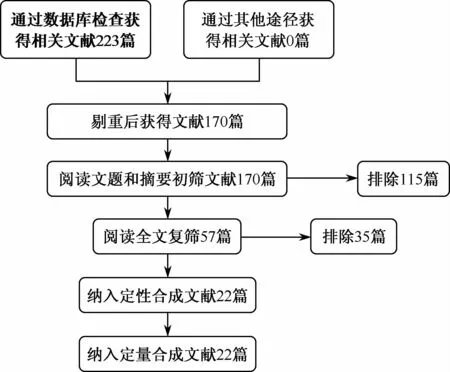
图1 PRISMA文献筛选四阶段流程图

表1 纳入研究论文中基因芯片技术检测Mtb的结果统计
注 所有纳入文献的研究均为前瞻性研究,均以传统药敏试验为金标准,均未描述是否使用盲法。TP:真阳性;FP:假阳性;TN:真阴性;FN:假阴性。文献21为多中心研究文献,分别在不同的城市(a:呼和浩特;b:连云港;c:开封;d:永川)进行了实验
四、纳入研究的方法学质量评价
应用Whiting 等[8]制订的评价诊断准确性质量研究(quality assessment of diagnostic accuracy studies,QUADAS)工具评价纳入文献质量,其中第3、8、9 条条目为非必须评价条目。每一条目以“是”、“否”、“不清楚”评价,“是”为满足此标准,“否”为不满足此标准,部分满足或者从文章中无法得到足够的信息为“不清楚”。 纳入的22篇论文的25个研究中,文献[6-13,15-30]中的24个研究均满足了质量评价的14条标准,文献[14]未满足第10、11条标准(表2)。
五、 统计学分析
参考《超声造影对乳腺肿块良恶性鉴别诊断价值的系统评价》一文对诊断性Meta分析格式[9],Meta 分析采用Meta-DiSc 1.40[10]软件进行。统计学分析指标和内容包括:SEN、SPE、+LR、-LR、DOR、SROC、AUC。
六、 异质性检验
将数据输入Meta-DiSc 1.40软件,采取随机效应模型进行初步合并,利用统计量I2来检验其统计异质性,凭借医学专业知识对临床异质性的来源可能进行分析,当异质性较大时应用Meta-DiSc 1.40软件进行逐一亚组分析,寻找异质性来源、分析原因并剔除导致异质性的纳入研究。如果I2≥50%则接受随机效应模型合并结果,若I2<50%则可采用固定效应模型进行合并分析。
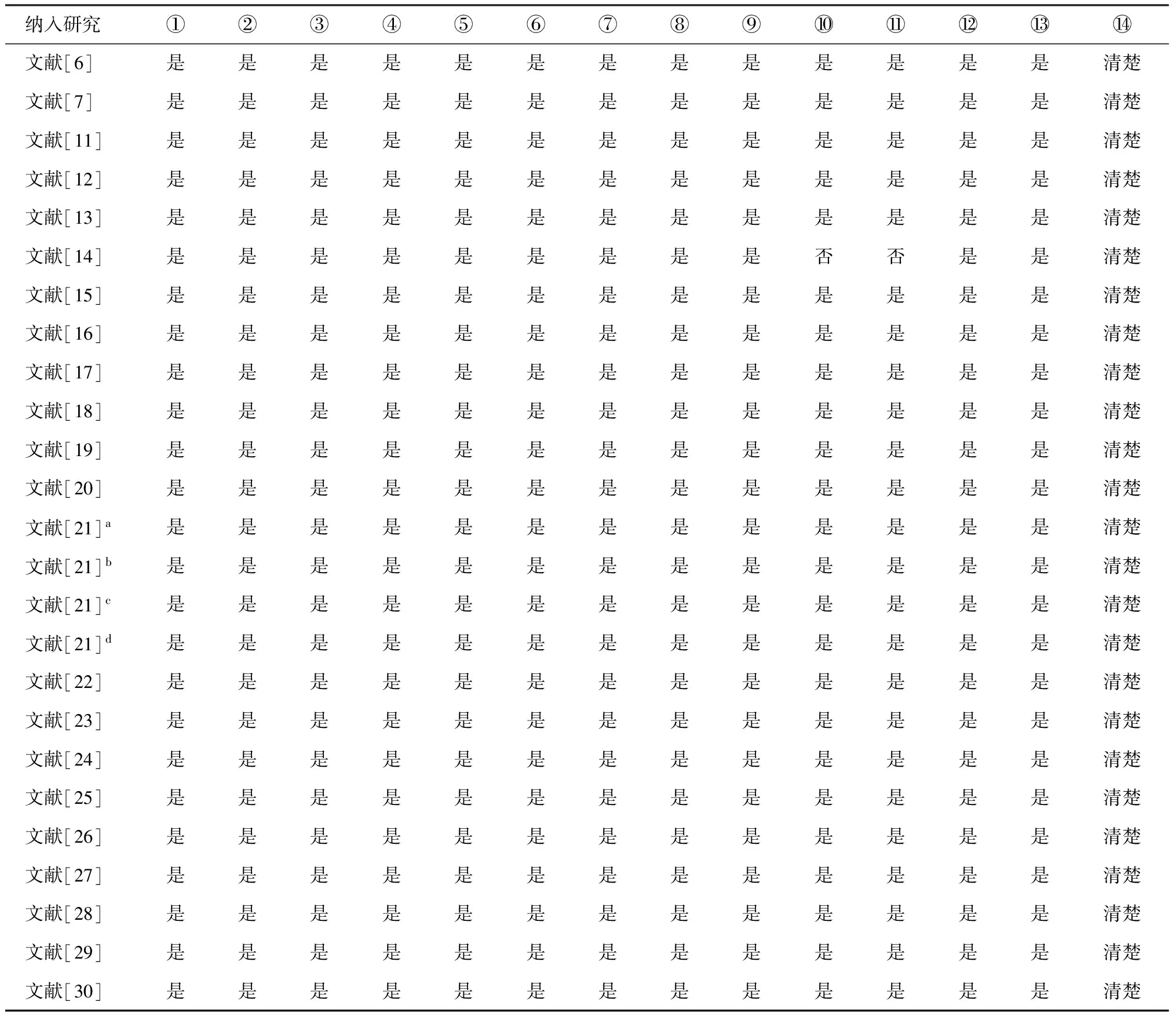
表2 纳入研究文献中的方法学质量评价

结 果
一、 Meta分析结果
ROC平面散点图不呈典型的“肩臂状”,提示无阈值效应;Spearman相关系数=0.192,P=0.380,提示尚不能认为存在阈值效应。DOR的Cochran-Q值为21.77,P=0.474,可认为多个同类研究具有同质性。各项结局指标的I2均<50%,提示各研究由非抽样误差所引起的异质性较小,故采用固定效应模型进行分析。固定效应模型Meta分析结果显示:SEN合并为0.89(95%CI=0.87~0.91)、SPE合并0.98(95%CI=0.97~0.98)、 +LR为30.98(95%CI=22.15~43.06)、 -LR为0.10(95%CI=0.07~0.15)、 DOR合并为354.06(95%CI=212.09~591.07)、AUC为0.9833。若I2>50%则选择随机效应模型进行分析。将每个研究逐一剔除后进行敏感度分析,结果显示SEN和SPE未见明显改变,说明结果稳定性好(图2~6)。
二、异质性结果
本研究应用Meta-diSc 1.4软件检测,结果敏感度I2为42.2%,特异度I2为47.5%,阳性预测值I2为23.3%,阴性预测值I2为24.5%,诊断性比值比I2为43.0%。
讨 论
一、 异质性来源
Higgins等[31]在2003年将异质性分为低、中、高3个程度,分别用I2值25%、50%、75%表示。2011年以后Cochrane标准[32]将异质性分为4个程度:I2值0~40%,轻度异质性,40%~60%中度异质性,60%~75%较大异质性,75%~100%,很大异质性。I2作为一个率,用于描述由各个研究所致的,而非抽样误差所引起的变异(异质性)占总变异的百分比,克服了Q统计量对纳入个数的依赖,可以更好地衡量多个研究结果间异质性程度的大小。在Cochrane系统评价中,只要I2≤50%,起异质性是可以接受的[31],我们可以认为异质性由偶然因素引起,而非各研究间差异。由于本研究的敏感度和特异度I2均>40%,为了进一步分析其异质性来源,推测其异质性是否与国内外研究等差异有关,进行了分析对比:国内研究敏感度略高于国外研究,异质性较国外低;而国外研究特异度、诊断OR值、AUC、Q值较国内研究高,异质性低(表3)。
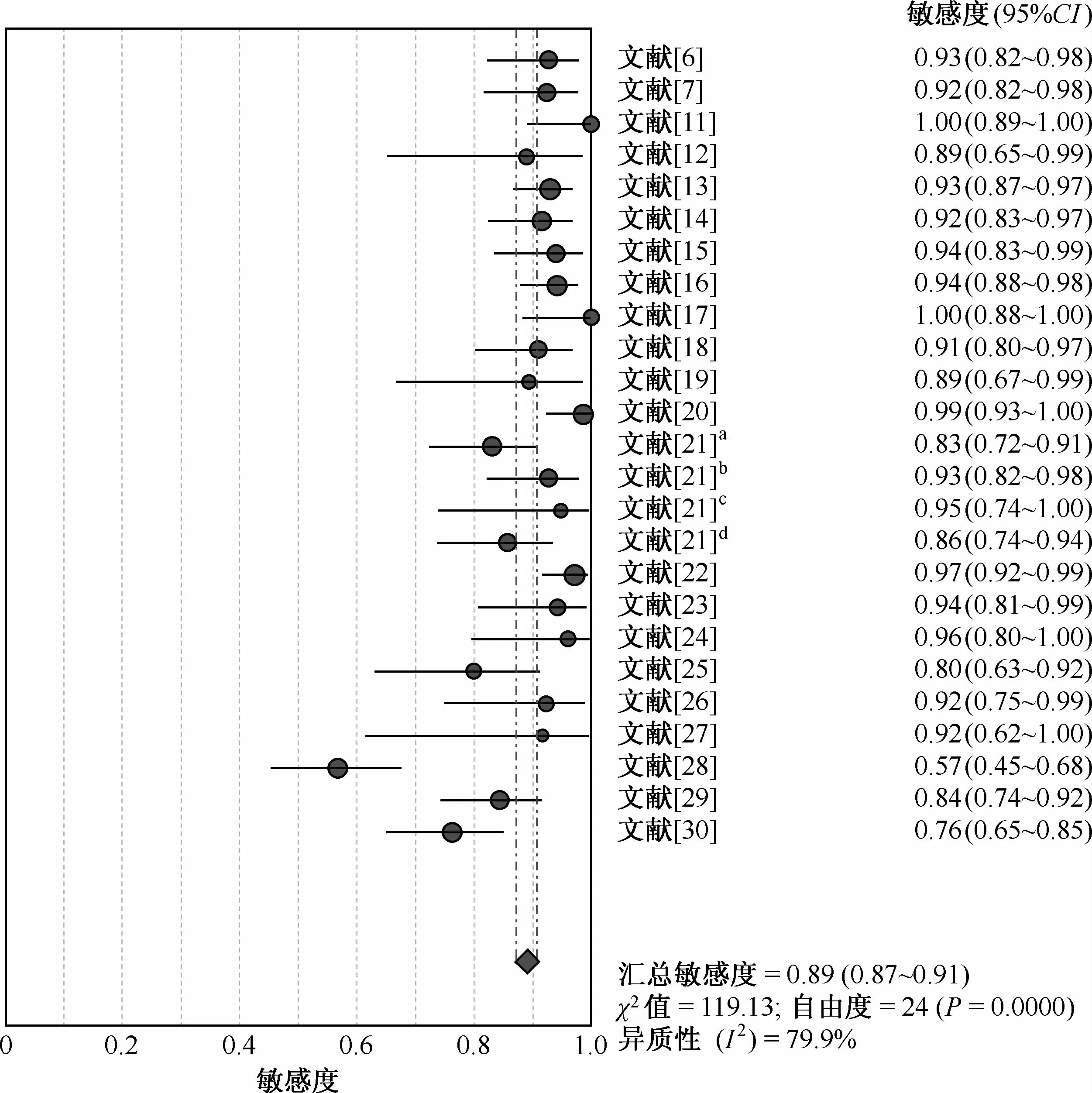
文献[21]为多中心研究文献,分别在不同城市(a:呼和浩特;b:连云港;c:开封;d:永川)进行了研究图2 22篇文献中基因芯片方法诊断Mtb对RFP耐药的敏感度分析
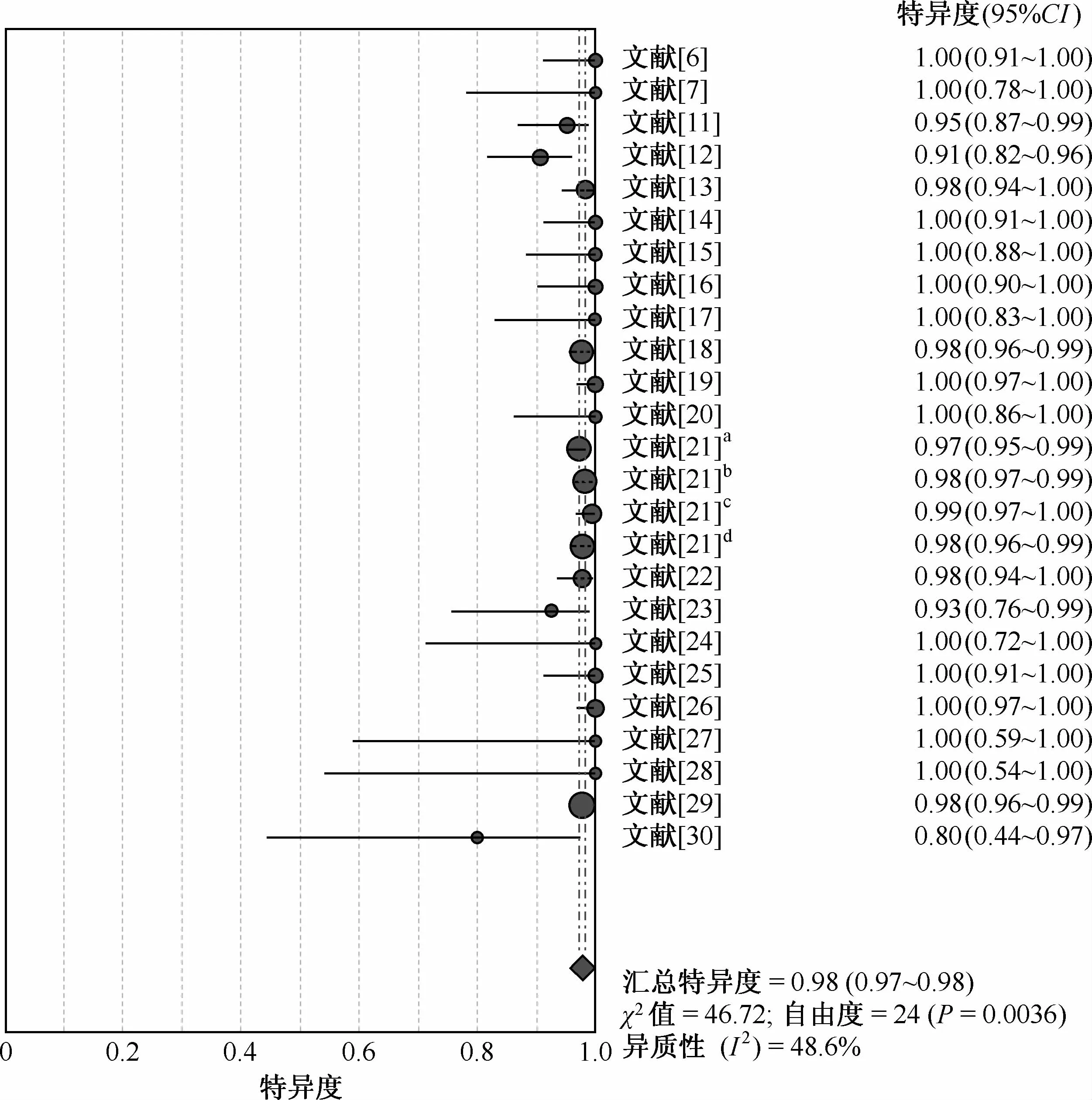
文献[21]为多中心研究文献,分别在不同城市(a:呼和浩特;b:连云港;c:开封;d:永川)进行了研究图3 22篇文献中基因芯片方法诊断Mtb对RFP耐药的特异度分析

文献[21]为多中心研究文献,分别在不同城市(a:呼和浩特;b:连云港;c:开封;d:永川)进行了研究图4 22篇文献中基因芯片方法诊断Mtb对RFP耐药的诊断OR值分析
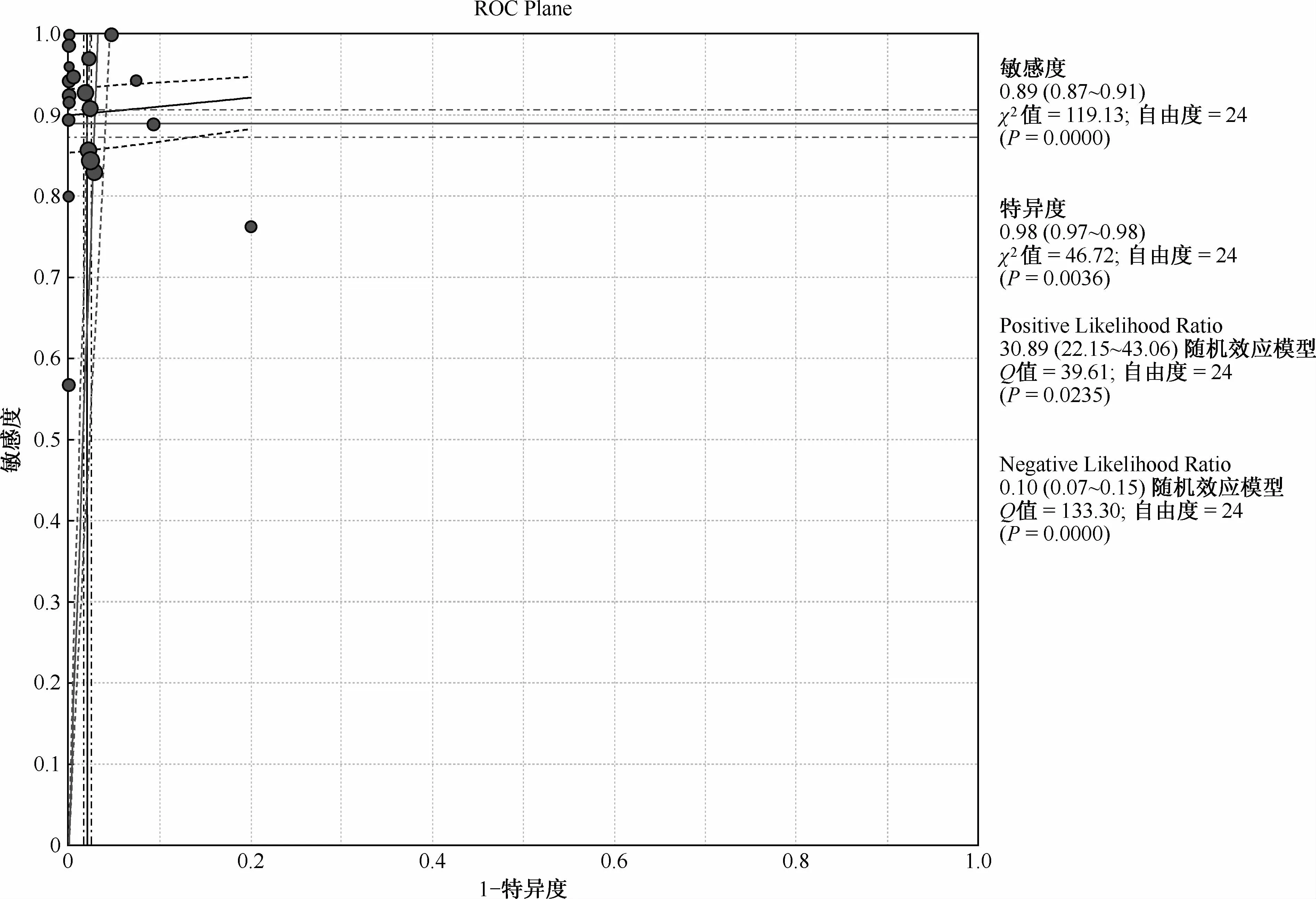
图5 22篇论文中基因芯片方法诊断Mtb对RFP耐药的阳性预测值和阴性预测值分析
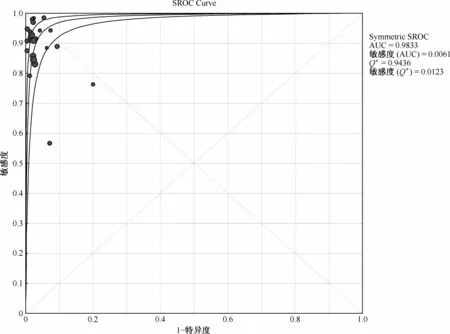
图6 纳入的22篇论文中研究基因芯片方法诊断Mtb对RFP耐药的SROC曲线
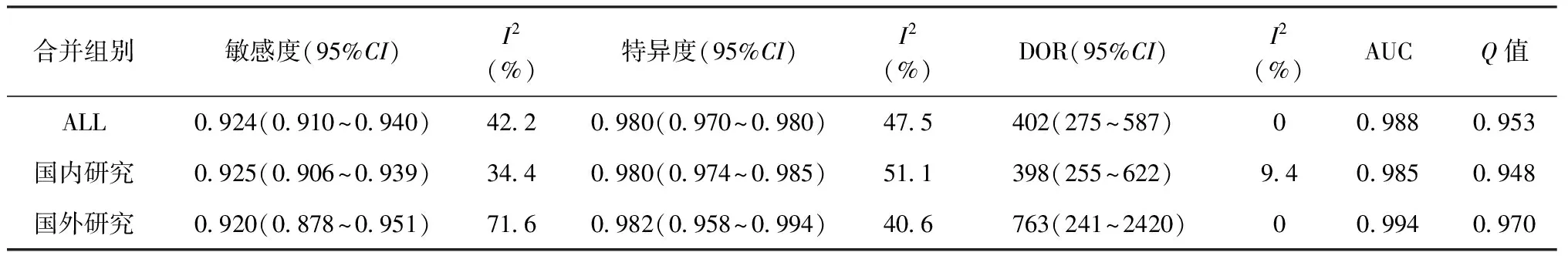
表3 Meta DiSc 1.4软件随机效应模型合并分析结果(敏感度分析和亚组分析)
注 ALL:纳入的所有研究;I2(%):异质性;DOR:诊断OR值;AUC:SROC曲线下面积
Mtb作为一种病原微生物,其表型各异,且易受所处环境的影响。尽管有许多关于Mtb耐药性的研究,但是仍然没有发现可以直接区分是否耐药的表型标记分子或检测手段。正因为如此,使得应用基因芯片方法检测Mtb耐药相关基因得到了更多的关注。不同抗生素作用压力会导致不同位点的基因突变,不同地区抗生素应用情况不一致使得各地区所获得的耐RFP菌株的数量上会有差别;而在同是RFP耐药的菌株其突变的碱基也存在不同,rpoB531和rpoB526是RFP耐药基因(rpoB)的易突变点[33-34],其他的突变点还有512、513、514、515、517、518、522、524[35]。不同的研究应用的基因芯片结构的差异,可能会使部分突变位点检不出,引起各个研究间的敏感度差异,导致异质性增大。
目前仍有5%~35%的耐药分离株未能检测出耐药基因突变,说明可能存在其他的耐药机制。因此,临床标本耐药基因突变检测阴性并不完全意味着对药物敏感,而敏感株的耐药基因可能存在不改变酶活性的同义突变与耐药无关的错义突变而被判定为耐药,则可引起假阳性;或因为在一群体中耐药个体数目尚未达到一定的比例,但因为检测过程中敏感度高的PCR扩增而出现假阳性[36-37]。另外,传统药敏试验检测以临界水平判断耐药,而基因芯片法可能检测到比传统药敏试验临界浓度更低的低度耐药突变菌株,这可能对最终结果产生影响。
基因芯片来源不同、芯片上点样孔数目、预测位点的不同,会使各研究间出现偏差,出现不同程度的假阳性和假阴性;另外,实验操作人员的熟练程度,操作过程中接种菌量、药物浓度、耐药或敏感结果标准的判定差异也是出现异质性的原因之一。
在诊断性实验研究中应用盲法可以得到更为真实的结果,使得结果更有说服力,尽可能地减少假阳性和假阴性。
二、基因芯片技术在耐药性结核分枝杆菌诊断的临床应用
本研究共纳入4159株Mtb菌株,其中耐RFP菌株1161株,敏感Mtb菌株2998株。结果显示:采用基因芯片方法诊断Mtb对RFP耐药的SEN合并为0.89,SPE合并为0.98,说明漏诊率为11%,误诊率为2%。+LR合并为30.89>1,说明基因芯片诊断Mtb对RFP耐药为阳性时,感染对RFP耐药Mtb的可能性较大;-LR合并为0.10<1,说明基因芯片方法检测为阴性时,不能完全排除患者感染对RFP耐药的Mtb的可能性。SROC曲线下面积为0.9878,表明其诊断效能较高。OR值为354.06,提示基因芯片方法对Mtb是否耐RFP具有很高的准确性。本研究结果还显示,在诊断的人群中基因芯片方法检测的SEN和SPE变化范围不大,分别为0.87~0.91、0.97~0.98,提示该诊断方法诊断的稳定性好;又因基因芯片方法检测方便快速,检测效率高,所以基因芯片方法是鉴别诊断Mtb是否耐RFP的比较好的手段。
Steingart等[38]以传统药敏试验为金标准,对应用Xpert®Mtb/RIF试剂盒检测Mtb耐RFP的耐药基因进行了系统评价,纳入研究555株耐RFP菌株和2411株敏感菌株,其敏感度为0.95[95%CI(0.90~0.97)],特异度为0.98[95%CI(0.97~0.99)]。Boehme等[39]用Xpert Mtb/RIF试剂盒对来自秘鲁、阿塞拜疆、南非、和印度等4个结核病高负担国家的Mtb标本进行检测,以传统药敏试验作为金标准,对耐RFP菌株的检出率达97.6%(200/205),对RFP敏感菌株的检出率达98.1%(504/514)。上述研究结果与本研究结果相似。另有报道提示,基因芯片方法与传统药敏试验检测Mtb对RFP耐药的一致性高,但因Mtb生长缓慢,传统药敏试验检测周期需要4周以上,无法为临床提供及时的用药参考,其临床应用存在一定的局限性;而快速培养仪测定系统尽管可以将检测时间缩短到5~7 d,但需特殊仪器设备,价格昂贵;分子生物学方法如PCR-SSCP(单核苷酸链构象多态性)简便、快速、价廉,但只能判断基因有无突变,不能确定具体突变的部位及性质,某些耐药基因呈天然多态性或某些基因突变与耐药无关,可能导致假阳性;PCR-RFLP(基因型之间限制性片段差异)只能用于分析已知序列特定位点的基因突变,应用范围相对局限;DNA序列测定仪器设备价格昂贵,需在专业实验室进行,限制了其推广应用[32]。相比较而言,基因芯片方法能够可靠,可以快速地检测是否为Mtb感染及其菌型,为临床提供明确的诊断,具有较好的应用前景[13]。另外,应用基因芯片方法检测时的通量较高,可以同时将大量探针固定于支持物上,可以一次性对样品大量序列进行检测和分析,从而解决了传统核酸印迹杂交技术的复杂性、自动化低、操作序列少、检测效率低等缺陷;而且通过设计不同的探针阵列,使用特定的分析方法使该技术具有不同的使用价值。将基因芯片技术用于检测分子突变,不仅可以明确突变位点和突变的类型,更重要的是其快速高效是目前其他方法无法比拟的,并且它可以同时检测多个基因乃至整个基因组的突变。总之,基因芯片方法作为新的检测方法,检测Mtb是否对RFP耐药具有广阔的应用前景,它具有简便、快速和敏感度、特异度高等特点。因此,基因芯片方法可作为临床传统药敏试验的补充,用于耐药Mtb的初筛试验,为临床制定合理的治疗方案提供参考,对控制耐药Mtb的传播、降低耐药率和死亡率有重要意义。
笔者对国内与国外实验结果进行了比较,结果显示:国内外研究在应用基因芯片测定Mtb对利福平耐药的敏感度、特异度、DOR、AUC、Q值均较高,异质性低,结果相对一致,说明基因芯片检测Mtb是否对利福平耐药的准确率高、假阳性和假阴性率低。
当然,本研究也存在一定的局限性:①纳入文献为不同国家和地区的研究,可能存在一定的发表偏移;②纳入论文的研究测量仪器之间有所不同,可能会因为仪器的改进更新和系统误差而影响到测量结果;③由于Mtb对培养环境要求较高,实验室条件和操作者水平有一定要求,不同地区研究难以达到技术的统一性。
[1] World Health Organization.Multidrug and extensively drug-resistant TB (M/XDR-TB): 2010 global report on surveillance and response. Geneva:World Health Organization, 2010 (WHO/HTM/TB/2010).
[2] Zhao Y, Xu S, Wang L, et al. National survey of drug-resis-tant tuberculosis in China. N Engl J Med, 2012, 366(23): 2161-2170.
[3] Wei J, Guo N, Liang J, et al.DNA microarray gene expression profile of Mycobacterium tuberculosis when exposed to osthole. Pol J Microbiol, 2013,62(1):23-30.
[4] Koser CU, Bryant JM, Becq J, et al. Whole-genome sequencing for rapid susceptibility testing of M. tuberculosis. New Engl J Med,2013,369(3):290-292.
[5] Almeida Da Sliva PE, Palomino JC. Molecular basis and mecha-nisms of drug resistance in Mycobacterium tuberculosis:classical and new drugs.J Antimicrob Chemother,2011, 66(7):1417-1430.
[6] Mokrousov I, Jiao WW, Sun GZ, et al. Evalution of the rpoB macroarray assay to detect rifampin resistance in Mycobacterium tuberculosis in Beijing China. Eur J Clin Microbiol Infect Dis,2006, 25(11):703-710.
[7] Yue J, Shi W, Xie J, et al. Detection of rifampin-resistant Mycobacterium tuberculosis strains by using a specialized oligonucleotide microarray. Diagn Microbiol Infect Dis,2004,48(1):47-54.
[8] Whiting P,Rutjes AW,Reitsma JB,et al. The development of QUADAS: a tool for the quality assessment of studies of diagnostic accuracy included in systematic reviews. BMC Med Res Methodol, 2003, 3: 25.
[9] 张晓光,张小利,帕丽达·帕尔哈提,等.超声造影对乳腺肿块良恶性鉴别诊断价值的系统评价.中国循证医学杂志,2014,14(1):79-84.
[10] 张天嵩,钟文昭.Meta-DiSc软件在诊断试验Meta分析中的应用.循证医学,2008,8(2):97-100.
[11] Kim SY, Park YJ, Song E, et al. Evaluation of the CombiChip Mycobacterium Drug-Resistance detection DNA chip for identifying mutations associated with resistance to isoniazid and rifampin in Mycobacterium tuberculosis.Diag Microbiol Infect Dis,2006,54(3):203-210.
[12] Zhang Z, Li L, Luo F, et al. Rapid and accurate detection of RMP- and INH- resistant Mycobacterium tuberculosis in spinal tuberculosis specimens by CapitalBioTMDNA microarray: a prospective validation study. BMC Infect Dis,2012,14(12):303-311.
[13] Park H, Song EJ, Song ES, et al. Comparison of a conventional antimicrobial susceptibility assay to an oligonucleotide chip system for detection of drug resistance in Mycobacterium tuberculosis isolates.J Clin Microbiol,2006,44(5):1619-1624.
[14] Jiao WW, Mokrousov I, Sun GZ, et al. Molecular characteristics of rifampin and isoniazid resistant Mycobacterium tuberculosis strains from Beijing, China. Chin Med J(Engl),2007,120(9):814-819.
[15] 张俊仙, 吴雪琼,阳幼荣,等.应用基因芯片方法检测结核分枝杆菌利福平和异烟肼的耐药性.中国防痨杂志,2011,33(10):680-685.
[16] 吴雪琼,张琼,张俊仙,等.应用基因芯片分析结核分枝杆菌常见耐药基因型的研究.中国防痨杂志,2006,28(1):4-10.
[17] 姚春艳, 张立群, 府伟灵. 应用基因芯片技术检测结核分枝杆菌耐药基因. 中华医院感染学杂志,2010,20(11):1501-1504.
[18] 李卫彬,李新旭,张彤群,等. 基因芯片检测技术检测结核分枝杆菌异烟肼和利福平耐药的实际应用效果评价,中华临床医师杂志(电子版),2012,6(16):4666-4670.
[19] 张俊仙,吴雪琼,邢婉丽,等. 应用基因芯片技术检测耐利福平结核分枝杆菌基因型. 中国现代医学杂志, 2007,17(13): 1572-1577.
[20] 潘建华,彭雪峰,邓为之,等.DNA芯片技术检测结核分枝杆菌耐药基因.临床肺科杂志,2010,15(6):825-827.
[21] Pang Y, Xia H, Zhang Z, et al. Multicenter evaluation of gene chip for detection of multidrug-resistant Mycobacterium tuberculosis.J Clin Microbiol,2013,51(6):1707-1713.
[22] Sun AH, Fan XL, Li LW, et al.Rapid detection of ropB gene mutation in Rif- resistance tuberculosis isolates by oligonucleo-tide microarray. Biomed Environ sci,2009,22(3):253-258.
[23] 何敏,曾尔良,郑艳燕,等.利用基因芯片检测结核分支杆菌及其利福平耐药性的研究.中华流行病学杂志,2003,24(5):385-388.
[24] Ao W, Aldous S, Woodruff E, et al. Rapid detection of rpoB gene mutations conferring rifampin resistance in Mycobacterium tuberculosis. J Clin Microbiol,2012,50(7):2433-2440.
[25] Caoili JC, Mayorova A, Sikes D, et al. Evalution of the TB-Biochip oligonucleotide microarray system for rapid detection of rifampin resistance in Mycobacterium tuberculosis.J Clin Microbiol,2006,44(7):2378-2381.
[26] 李芳芳,唐曙明,李爱敏,等.膜芯片技术对结核分枝杆菌耐药性检测的研究.国际检验医学杂志,2013,34(21):2792-2794.
[27] 梁莉,乐军,肖和平,等.利用基因芯片技术检测耐利福平分离株rpoB基因突变的研究.中华医院感染学杂志,2005,15(8):841-844.
[28] Zhang SL, Shen JG, Xu PH,et al. A novel genotypic test for rapid detection of multidrug-resistant Mycobacterium tuberculosis isolates by a multiplex probe array.J Appl Microbiol,2007,103(4):1262-1271.
[29] 赵冰,时金艳,逄宇,等. 基因芯片结核分枝杆菌耐多药检测在地市级实验室的应用性评估.中国防痨杂志,2013,35(9): 718-722.
[30] 李锋,李凫坚,陈园园,等. 应用基因芯片快速检测结核分枝杆菌利福平耐药基因的研究. 中国防痨杂志, 2011,33(3):188-189.
[31] Higgins JP, Thompson SG,Deeks JJ, et al. Measuring inconsistency in a Meta-analyses. BMJ,2003,327(7414):557-560.
[32] Higgins JPT,Green S.Cochrane handbook for systematic reviews of interventions Version5.1.0.[2011-03-01]. http://www.cochrane.org/handbook.org.
[33] Jnawali HN, Hwang SC, Park YK, et al. Characterization of mutations in multi- and extensive drug resistance among strains of Mycobacterium tuberculosis clinical isolates in Republic of Korea. Diagnb Microbiol Infect Dis,2013,76(2):187-196.
[34] 陈效友,黄海荣,马屿,等.荧光定量PCR技术快速检测rpoB基因突变及其临床应用.中国防痨杂志,2007,29(5):386-390.
[35] Mikhailovich V, Lapa S, Gryadunov D, et al. Identification of rifampin-resistant Mycobacterium tuberculosis strains by hybridi-zation, PCR, and ligase detection reaction on oligonucle-otide microchips.J Clin Microbiol,2001,39(7):2531-2540.
[36] 谢士达,刘成永,张凤池,等.基因芯片技术快速检测结核分枝杆菌异烟肼、利福平耐药性.临床肺科杂志,2008,1(2):147-149.
[37] 王峰,朱玉梅,桂静,等.PCR测序和基因芯片快速鉴定分枝杆菌菌种的应用研究.中国防痨杂志,2011,33(11):713-717.
[38] Steingart KR, Schiller I, Horne DJ, et al. Xpert®MTB/RIF assay for pulmonary tuberculosis and rifampicin resistance in adults. Cochrane Database syst Rev,2014, 1: CD009593.
[39] Boehme CC, Nicol MP, Nabeta P, et al. Feasibility, diagnostic accuracy, and effectiveness of decentralized use of the Xpert MTB/RIF test for diagnosis of tuberculosis and multidrug resistance:a multicentre implementation study. Lancet, 2011,377(9776):1495-1505.
(本文编辑:范永德)
Diagnostic accuracy of gene chip in identifying rifampicin resistanceMycobacteriumtuberculosis: a Meta-analysis
RAN Bing, CAI Lin.
Fourth Department of Orthopedics, Zhongnan Hospital of Wuhan University, Wuhan 430071, China
Corresponding author: CAI Lin, Email: guke3559@aliyun.com
Objective To systematically review the diagnostic accuracy of gene chip in identifying rifampicin (RFP) resistance Mycobacterium tuberculosis (Mtb),and to provide the evidence for the clinical use of gene chip in detection of RFP-resistance Mtb. Methods We electronically searched The Cochrane Library (Issue 1, 2014), PubMed, EMbase, Wanfang Data, CNKI from the date that the database was established to February 2014. In order to find any publications which are related to detection of RFP-resistance Mtb by using gene chip, the search key terms in English and Chinese were designed as follows: tuberculosis, tuberculosis and drug resistance, tuberculosis and drug resistance and gene chip, tuberculosis and drug resistance and gene chip and rifampicin. Two reviewers independently screened literatures according to the inclusion and exclusion criteria, extracted data and assessed the quality of methodology of the included studies according to the QUADAS items. The Meta-DiSc software (version 1.4) was used to conduct pooling on sensitivity (SEN), specificity (SPE), positive likelihood ratio (+LR), and negative likelihood ratio (-LR). Heterogeneity test was performed and the summary receiver operating characteristic (SROC) curve was drawn and area under curve (AUC) was calculated. Results A total of 223 publications were found. Finally, 22 articles (including one article from multicenter study) and 25 studies were included into the review,which involved 4 879 Mtb isolates (including 1314 RFP-resistant strains, 3565 susceptible strains and the standard strain H37Rv). The results of Meta-analysis showed that, compared with the traditional drug sensitivity test, SEN of gene chip in detection of RFP-resistance Mtb was 0.89 (95%CI(0.87-0.91)), SPE was 0.98 (95%CI(0.97-0.98)), +LR was 30.89 (95%CI(22.15-43.06)), -LR was 0.10 (95%CI(0.07-0.15)), diagnostic OR was 354.06 (95%CI(212.09-591.07)), AUC was 0.9833. Conclusion Gene chip has a good value in detection of RFP-resistance Mtb. If the traditional drug sensitivity test is regarded as a gold standard, gene chip has high sensitivity, high specificity and high diagnosticORvalue. It can be used as a clinical supplementary method of drug sensitivity test.
Mycobacterium tuberculosis; Rifampin; Oligonucleotide array sequence analysis; Tuberculosis, multidrug-resistant; Bacterial proteins; Meta-analysis
10.3969/j.issn.1000-6621.2015.01.012
430071 武汉大学中南医院骨四科
蔡林,Email:guke3559@aliyun.com
2014-05-30)

