四倍体彩色马铃薯分子遗传连锁图谱构建研究
崔阔澍,陈 龙,于肖夏,鞠天华,于 卓,肖 特,聂利珍,姜 超
(1.内蒙古农业大学农学院,内蒙古 呼和浩特 010019;2.赤峰天泽生物科技有限公司,内蒙古 赤峰 025457)
四倍体彩色马铃薯分子遗传连锁图谱构建研究
崔阔澍1,陈 龙1,于肖夏1,鞠天华2,于 卓1,肖 特1,聂利珍1,姜 超1
(1.内蒙古农业大学农学院,内蒙古 呼和浩特 010019;2.赤峰天泽生物科技有限公司,内蒙古 赤峰 025457)
以四倍体彩色马铃薯(‘黑美人’,‘MIN-021’)杂交F1代单株无性株系群体为材料,利用SSR分子标记技术及作图软件(TetraploidMap)进行了彩色马铃薯分子遗传连锁图谱的构建研究.结果表明:从255对SSR引物中筛选出34对条带清晰稳定、多态性丰富的适宜引物,对‘黑美人’בMIN-021’杂种F1的210个单株无性系及其双亲的基因组DNA进行PCR扩增共得到201个多态性标记.用这201个SSR标记对双亲分别作图,首次构建了四倍体彩色马铃薯的分子遗传连锁框架图谱.母本图谱含129个标记,12个连锁群总长度为1 167 cM,标记间平均距离为9.04 cM;父本图谱含131个标记,12个连锁群总长度为1 187 cM,标记间平均距离为9.06 cM.
彩色马铃薯;四倍体;SSR标记;遗传图谱构建
彩色马铃薯是指块茎薯肉为紫、红等颜色的马铃薯(SolanumtuberosumL.),简称彩薯.[1]彩色马铃薯不仅含有普通黄肉、白肉马铃薯品种的营养成分,还富含抗氧化物质——植物花青素(anthocyanin),具有抗氧化、抗病毒、护肝、抗癌等多种保健功能.[2]在国外,彩薯作为功能食品已被大众所接受,彩薯育种、栽培等研究备受关注.随着我国人民生活水平的提高,彩薯开始逐渐为人们所认识,近年来一些研究单位及企业开始引种彩薯,并开展了新品种的选育工作,彩薯产业将会越来越受到重视.[3]分子遗传连锁图谱的建立是作物重要性状QTLs定位分析及分子标记辅助育种的重要基础.国外关于马铃薯遗传连锁图谱的构建研究起步较早,自Bonierbale等(1988)采用RFLP分子标记首次构建了二倍体马铃薯分子遗传连锁图谱以来[4],已有不少学者开展了马铃薯的遗传作图研究,并构建了多张马铃薯图谱[5],其中最为著名的是荷兰瓦赫宁根大学Han van Os等(2006)利用SSR,RFLP和AFLP标记构建的马铃薯“超密图谱”[6].Meyer 等(1998)采用AFLP分子标记技术最先构建了四倍体马铃薯的遗传连锁图谱,其母本图谱含231个标记,父本图谱含106个标记[7].我国马铃薯遗传连锁图谱的构建研究起步较晚,迄今只有金黎平等(2007)构建了含170个AFLP标记的二倍体马铃薯遗传连锁图谱[8],以及单友蛟(2010)构建的含86个SSR标记的遗传图谱[9].
上述遗传连锁图谱的构建均以普通黄肉和白肉的马铃薯为材料,而对彩色马铃薯遗传连锁图谱的构建研究尚未见报道.近几年来,为培育彩色马铃薯新品种,我们根据优缺点互补、生态型差异大的亲本选配原则,将美国引进的四倍体彩色马铃薯材料‘MIN-021’与国内育成的彩色马铃薯品种‘黑美人’相组配,人工去雄授粉杂交后成功获得了杂种F1代群体.本试验以该四倍体彩色马铃薯杂种F1代分离的210个单株无性株系为材料,利用SSR分子标记构建了彩色马铃薯的分子遗传连锁图谱,旨为下一步开展彩色马铃薯高密度分子连锁图谱的构建、花青素色素含量等重要性状的QTL定位及分子标记辅助育种提供依据.
1 材料与方法
1.1 供试材料及其来源
材料为四倍体(2n=4x=48)彩色马铃薯‘黑美人’בMIN-021’杂种F1代的210个单株无性株系及其亲本.母本‘黑美人’是兰州陇神航天育种研究所育成品种,其薯块椭圆形,芽眼浅,紫皮紫肉,产量和花青素含量中等,适应性强;父本‘MIN-021’于2009年引自美国加州大学,其薯块近圆形,芽眼浅,红皮红肉,产量和花青素含量较高.2011年夏季组配亲本,通过人工授粉杂交获得杂交种子;2012年春季利用杂交种子在温室内育苗获得F1代实生苗210株,深秋收获单株块茎形成F1分离群体无性系一代,观察发现F1代群体单株薯块的薯皮薯肉性状及地上部植株形态性状分离明显,变异较大;2013年春季田间播种,出苗后按单株提取基因组DNA,用于彩色马铃薯分子遗传图谱的构建研究.
1.2 研究方法
1.2.1 DNA的提取与检测
苗期取亲本及其杂种F1代各无性系单株材料的幼嫩叶片,采用植物基因组试剂盒(北京天根公司生产)提取DNA,用1.6%琼脂糖凝胶电泳检测DNA纯度,将DNA浓度稀释至约50 ng/μL后,置于-20℃冰箱中冷冻保存备用.
1.2.2 SSR-PCR反应体系及程序
SSR分析参照Feingold等提出的方法[10],并在张自强等[11]建立的马铃薯SSR-PCR反应体系和反应程序的基础上进行了优化.SSR-PCR反应体系为:含Mg2+的10×Buffer 2.0 μL,0.225 mmol/L的dNTPs 1.6 μL,0.5 μmol/L的上、下游引物各1.0 μL,50 ng/μL的模板DNA 1.5 μL,5 U的Taq酶0.15 μL,ddH2O补足至20 μL.反应程序为:95℃预变性3 min;94℃变性30 s,57℃退火30 s,72℃延伸1 min,35个循环;最后72℃延伸10 min.
1.2.3 PCR扩增产物检测
用7%变性聚丙烯酰胺凝胶电泳检测扩增产物,上样量为5 μL,电泳恒定功率为70 W,电泳时间为1.2 h.胶板用蒸馏水漂洗1次,在染色液中银染16 min,取出蒸馏水中速漂10 s,放入显影溶液中直至条带清晰后用蒸馏水漂洗2 min,风干后进行扫描.[12]
1.2.4 SSR引物来源
从NCBI基因库中获得马铃薯基因组序列,用Primer 5.0网页版及DNAMAN软件自主设计马铃薯EST-SSR引物碱基序列54对,编号设定为C系列.并利用NCBI网站公布的201马铃薯SSR特异性引物碱基序列,委托上海生工有限公司合成255对SSR引物.
1.2.5 SSR多态性位点统计
采用0/1赋值法,即在等位位点上出现条带记为1,无条带记为0,条带缺失或不清记为9,按条带从大到小的顺序记录,建立0,1型二元数据矩阵,统计多态性位点.多态性位点百分率P=(K/N)×100%,式中K为多态位点数目,N为所测位点总数.[13]
1.2.6 SSR标记偏分离统计
根据孟德尔遗传分离规律和卡方检验原理,当P<0.05时视为偏分离标记.[14]
1.2.7 SSR分子遗传图谱构建
采用TetraploidMap软件构建彩色马铃薯分子遗传连锁图谱[15].
2 结果与分析
2.1 SSR引物筛选及多态性分析
用双亲及其8个表型差异明显的F1代单株基因组DNA为模板进行PCR扩增,从255对SSR引物中选出条带清晰、重复性好、多态性丰富的适宜引物34对,其中C系列引物8对,S系列引物28对.利用这34对引物对F1代分离群体210个单株无性系及其双亲的DNA进行扩增,得到235个清晰、稳定的SSR位点,平均每个引物扩增出6.91个位点,其中多态性位点201个,多态性位点百分率为85.53%,表明供试材料的SSR多态性丰富(见表1、图1).
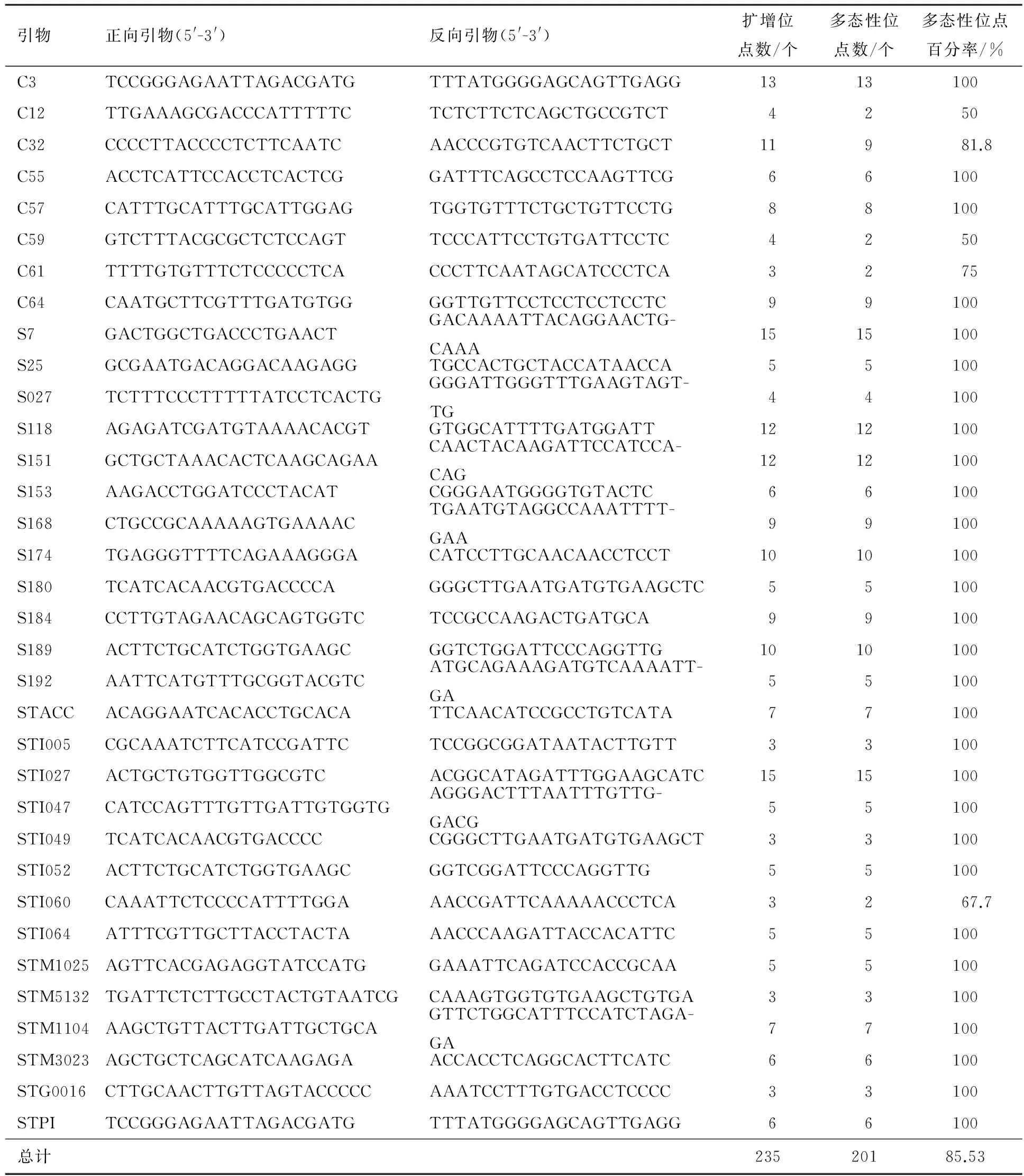
表1 SSR引物及供试材料的PCR扩增结果
M:Maker;1:♀黑美人;2:♂MIN-021;3~93:F1单株无性株系
图1 引物C3对亲本及F1分离群体部分单株无性株系的SSR扩增结果
2.2 群体中SSR标记的分离分析
由表2可知,F1分离群体中来自双亲的SSR标记的比例接近1∶1,表明后代均衡分离且双亲在后代遗传中占有同等重要的地位.经卡方检验,母本有27个标记、父本有28个标记呈现不同程度的偏分离,其平均偏分离比例分别为20.93%和21.37%(P<0.05),符合作物一般分子标记遗传作图时偏分离比例小于30%的要求.[16]

表2 SSR分子标记的偏分离分析
129个标记中有69个标记来自母本黑美人,131个标记中有71个标记来自父本MIN-021,另有60个标记为双亲共有标记.
2.3 遗传图谱的构建及其基本特征分析
用TetraploidMap for Windows作图软件按SSR标记的亲本来源进行连锁分析,分别得到母本(黑美人)和父本(MIN-021)的遗传连锁图,母本的129个SSR标记(含27个偏分离标记)和父本的131个SSR标记(含28个偏分离标记)均全部分布在各自的12个连锁群上(见图2、图3、表3、表4).
母本的连锁图总长度为1 167 cM,标记间平均间距9.04 cM,12个连锁群长度范围在27~224 cM之间,各连锁群平均间距变幅为6.75~21.60 cM.其中,最长的连锁群LGMa1覆盖基因组224 cM,有29个标记;最短的连锁群LGMa4覆盖基因组27 cM,仅有4个标记.
父本图谱的总长度为有1 187 cM,标记间平均间距为9.06 cM,各连锁群长度变幅为27~215 cM,平均图距变幅为 5.53~18.00 cM.连锁群LGFe1覆盖基因组215 cM,有23个标记,为最长连锁群;连锁群 LGFe5为27 cM,有4个标记,为最短的连锁群.
由表3、表4还可以看出,双亲遗传连锁图谱的平均间距均<10 cM,各标记在图谱中分布较均匀,只有个别密集区,表明图谱的总体质量较好,可用于彩色马铃薯花青素含量等重要性状的QTL定位研究.
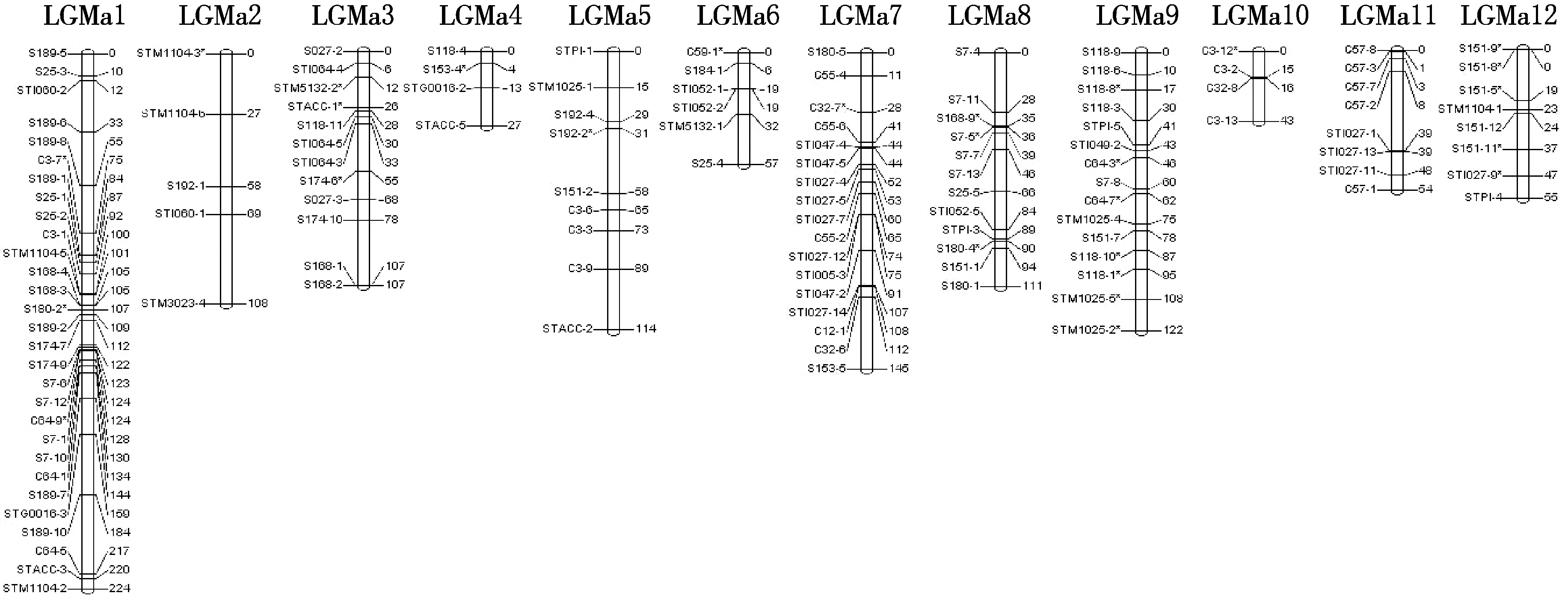
图2 ♀‘黑美人’的SSR分子遗传连锁图谱
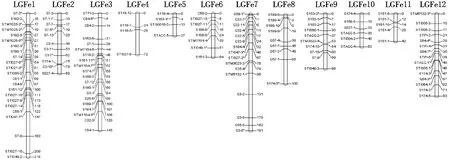
图3 ♂‘MIN-021’的SSR分子遗传连锁图谱
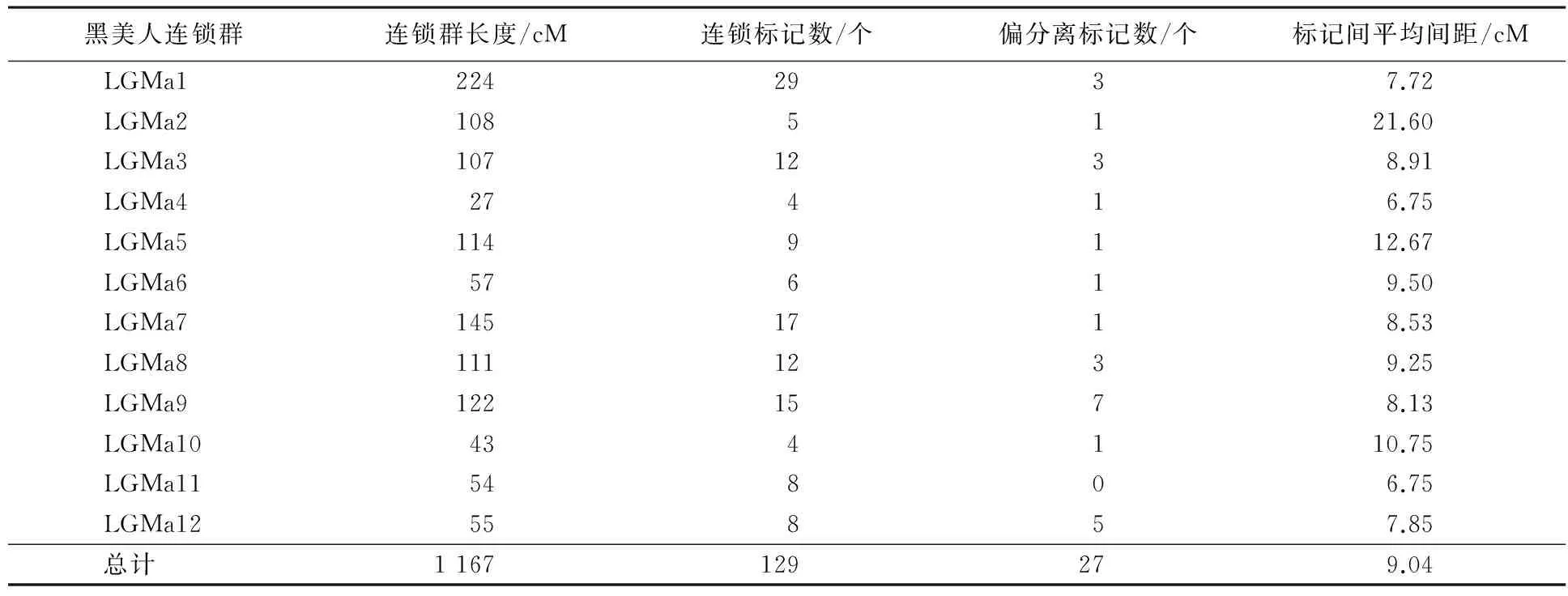
表3 ♀‘黑美人’分子遗传图谱的基本特征
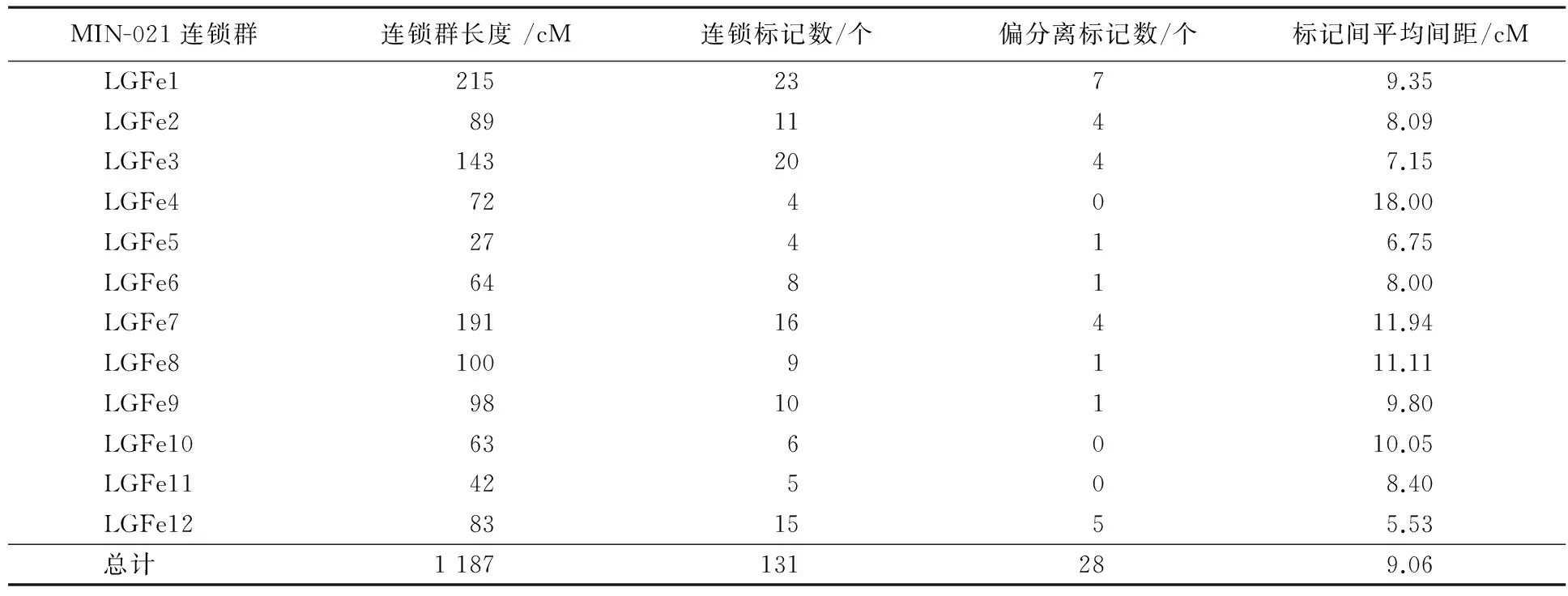
表4 ♂‘MIN-021’分子遗传图谱的基本特征
3 讨论
3.1 作图群体的选择
构建作物遗传连锁图谱的群体通常有两类:第一类是单株分离的暂时性群体,如杂种F2代及其自交家系F3代、F4代等;第二类为株系分离的永久性群体,如RILs,BILs,DH等[17].栽培马铃薯是同源四倍体(2n=4x=48),基因型高度杂合,其两个不同亲本杂交后的杂种F1代浆果中每粒种子都是不同的基因型,且马铃薯以无性繁殖为主,每粒种子形成的植株其无性后代的基因型是稳定的,一般使用F1代群体进行马铃薯分子连锁图谱的构建,群体的数量多在90~200个之间.[18]本试验以‘黑美人’与‘MIN-021’两个四倍体彩色马铃薯杂种F1代的210个单株无性株系为材料,田间观察发现该群体的株高、花色、叶片、薯形、薯皮、薯肉等表型性状分离明显,适合彩色马铃薯遗传图谱的构建研究.
3.2 标记的偏分离
等位基因的偏分离是生物进化的重要原因之一[19].目前已在玉米、番茄、苜蓿等多种作物中报道了遗传标记的偏分离现象[20-22].偏分离现象可能是由配子体选择、遗传漂变、物种的细胞学属性或一些生物学原因引起的,它可能会改变重组估计结果.[23]分子标记在连锁图中偏分离的比例一般在30%以内,但也有远高出这一比例的,这与所用标记类型、不同作物及群体有关.[8,16]Hackett[24]和Zhang等[25]的研究均表明偏分离标记并不影响分子遗传连锁图的构建准确性.本研究中,双亲遗传连锁图谱的偏分离标记比例均小于30%,其中母本图谱上有27个SSR标记呈现偏分离(占20.93%),父本图谱上有28个标记呈现偏分离(占21.37%),在对SSR标记进行连锁遗传分析时,每个偏分离标记均进入到双亲各自的遗传连锁群上,表明偏分离标记可以用于彩色马铃薯的遗传作图.另外,母本和父本各连锁群的偏分离标记数目不同,有的连锁群上偏分离标记数目较多,有的连锁群上较少或没有(见表3、表4),这可能是由于偏分离区域与一些孢子体致死或配子体致死基因相关,尚有待进一步的深入研究.
3.3 作图软件选择与连锁图谱构建
目前常用的遗传作图软件是JoinMap.本研究采用的作图软件为国际上同源四倍体专用作图软件TetraploidMap for Windows,TetraploidMap软件是Hackett等(2003)根据同源四倍体遗传规律开发的专用作图软件,已应用于四倍体苜蓿和四倍体马铃薯等遗传连锁图的构建[15].与JoinMap软件相比,该软件构建的分子连锁图谱不仅适合四倍体马铃薯的遗传分离规律,增加了标记量,也提高了作图的精确度;JoinMap软件的作图亲本遵循纯合或杂合二倍体分离规律,软件仅提供CP模式来计算重组率和遗传距离,且默认染色体假定出现交换干涉现象,从而导致其绘制的连锁图长度比TetraploidMap 软件偏小;[26]TetraploidMap软件首先根据分离群体的标记多态性、分离比等因素检测父、母本基因型,判断双亲基因型后通过极大似然估计(MLE)进行两点双边检验,通过聚类分析确定标记排列最适顺序,显著提高了双亲作图的精确度.[27]另外,计算图距时JoinMap软件仅提供单一的两点测验;而TetraploidMap软件不仅用两点测验进行聚类,还提升了作图的准确度,虽然计算机运行时间略长,但Stimulated annealing(模拟退火算法)计算出的图距更加精确.[28]Bradshaw等利用AFLP和SSR两种分子标记研究构建了具有代表性的四倍体非彩色马铃薯的分子遗传连锁图谱,其父本‘Stirling’连锁群总长度1 234 cM,母本‘12106ab’连锁群总长度1 203 cM.[29]本试验利用SSR标记首次构建了四倍体彩色马铃薯的分子遗传连锁图谱,其中母本连锁群总长度为1 167 cM,父本连锁群总长度为1 187 cM,覆盖基因组长度与Bradshaw的研究相近,但本图谱仅用了SSR一种分子标记,标记数相对较少,标记间仍存在较大空隙,目前我们正在筛选AFLP遗传标记拟对该图谱进行进一步加密.
4 结论
试验筛选出条带清晰、重复性好、多态性丰富的彩色马铃薯适宜引物34对.用这些SSR引物对‘黑美人’בMIN-021’杂种F1代的210个单株无性株系及其双亲的基因组DNA进行了PCR扩增,得到201个多态性标记.采用TetraploidMap 软件对双亲分别作图,首次构建了四倍体彩色马铃薯的分子遗传连锁框架图谱,其中母本图谱含129个标记,12个连锁群总长度1 167 cM,标记间平均间距9.04 cM;父本图谱含131个标记,12个连锁群总长度1 187 cM,标记间平均间距9.06 cM.
[1] 张晓萌,于肖夏,于卓,等.彩色马铃薯杂种F1无性株系花粉育性及染色体配对构型分析[J].内蒙古农业大学学报,2012,34(5):32-34.
[2] 石少穿,高亦珂,张秀梅,等.植物花青素生物合成相关基因的研究及应用[J].植物研究,2011,31(5):633-640.
[3] 石景,宋波涛,金开建,等.SSR标记的彩色马铃薯遗传多样性分析及指纹图谱构建[J].农业生物技术学报,2012,20(4):362-371.
[4] BONIERBALE M W,PLAISTED P L,TANKSLEY S D.RFLP maps based on a common set of clones reveal modes of chromosome evolution in potato and tomato[J].Genetics,1988,120(2):1095-1103.
[5] FELCHER K J,COOMBS J J,MASSAET A N,et al.Integration of two diploid potato linkage maps with the potato genome sequence[J].The Public Library of Science,2012,7(4):1-11
[6] HANS VAN OS,ANDRZEJEWSKI S,BAKKER E,et al.Construction of a 10000 marker ultra-dense genetic recombination map of potato:providing a framework for accelerated gene isolation and a genome-wide physical map[J].Genetics,2006,173(2):1075-1087.
[7] MAYER R.C,MILBOURNE D,HACKETT C A,et al.Linkage analysis in teraploid potato and association of makers with quantitive resistance to late blight(Phytophthorainfestans)[J].Molecular Genetics and Genomics,1998,59(2):150-160.
[8] 金黎平,刘杰,H DE JONG,等.二倍体马铃薯分子连锁图谱的构建[J].园艺学报,2007,34(2):397-402.
[9] 单友蛟,刘杰,卞春松,等.马铃薯SSR遗传连锁图谱构建及3个重要农艺性状的QTLs定位[J].中国蔬菜,2010,18:10-14.
[10] FEINGOLD S,LLOYD J,NORERO N,et al.Mapping and characterization of new EST-derived microsatellites for potato(SolanumtuberosumL.)[J].Theoretical and Applied Genetics,2010,111(2):456-466.
[11] 张自强,于肖夏,鞠天华,等.2个马铃薯杂交组合F1的SSR 鉴定[J].中国生态农业学报,2013,21(11):1411-1415.
[12] MORGANTE M,OLIVIERI A M.PCR-amplified microsatellites in plant genetics[J].Plant Journal,1993,3(1):175-182.
[13] NEI M.Analysis of gene diversity in subdivided populations[J].Proceedings of the National Academy of Sciences of the USA,1973,70(12):3321-3323.
[14] 严建兵,汤华,黄益勤,等.玉米F2群体分子标记偏分离的遗传分析[J].遗传学报,2003,30(10):913-918.
[15] HACKETT C A,PANDE B,BRYAN G J.Constructing linkage maps in autotetraploid species using simulated annealing[J].Theoretical Applied Genetics,2003,106(6):1107-1115.
[16] 宋宪亮,孙学振,张天真.偏分离对植物遗传作图的影响[J].农业生物技术学报,2006,14(2):286-292.
[17] 席章营,朱芬菊,台国琴,等.作物QTL分析的原理与方法[J].中国农学通报,2005,21(1):88-93.
[18] COSTANZO S,SIMKO I,CHRIST B J,et al.QTL analysis of late blight resistance in a diploid potato family ofSolanumphureja×S.stenotomum[J].Theoretical Applied Genetics,2005,111(3):609-617.
[19] BRADSHAW H D,STETTLER R F.Molecular genetics of growth and development in populus: II.Segregation distortion due to genet-ics load[J].Theoretical Applied Genetics,1994,89(5): 551-558.
[20] LU H,ROMERO-SEVERSON J,BERNARDO R.Chromosomal regions associated with segregation distortion in maize[J].Theoretical Applied Genetics,2002,105(4):622-628.
[21] PATERSON A H,DAMON S,HEWITT J D,et al.Mendelian factors underlying quantitative traits in tomato:comparative across species,generations,and environments[J].Genetics,1991,127(1):181-197.
[22] BRUMMER E C,BOUTON J H,KOCHERT G.Development of an RFLP map in diploid alfalfa[J].Theoretical Applied Genetics,1993,86(2):329-332.
[23] SHAPPLEY Z W,JENKINS J N,MEREDITH W R,et al.An RFLP linkage map of upland cotton,GussypiumhirsutumL [J].Theoretical Applied Genetics,1998,97(5/6):756-761.
[24] HACKETT C A,BROADFOOT L B.Effects of genotyping errors,missing values and segregation distortion in molecular marker data on the construction of linkage maps[J].Heredity,1992,69:315-324.
[25] ZHANG L Y,WANG S Q,LI H H,et al.Effects of missing marker and segregation distortion on QTL mapping in F2populations[J].Theoretical Applied Genetics,2010,121(6):10-14.
[26] STAM P.Construction of integrated genetic linkage maps by means of a new computer package JoinMap[J].Plant Journal,1993,3(5):739-744.
[27] LUO Z W,HACKETT C A,BRADSHAW J E,et al.Construction of a genetic linkage maps in tetraploid species using molecular makers [J].Genetics,2001,157(3):1369-1385.
[28] LUO Z W,ZHANG Z,LEACH L,et al.Constructing genetic linkage maps under a tetrasomic model[J].Genetics,2006,172(4):2635-2645.
[29] BRADSHAW J E,HACKETT C A,PANDE B,et al.QTL mapping of yield,agronomic and quality traits in tetraploid potato(Solanumtuberosumsubsp.tuberosum)[J].Theoretical Applied Genetics,2008,116(2):193-211.
(责任编辑:方 林)
Construction of SSR genetic linkage map for tetraploid colored potato
CUI Kuo-shu1,CHEN Long1,YU Xiao-xia1,JU Tian-hua2,YU Zhuo1,XIAO Te1,NIE Li-zhen1,JIANG Chao1
(1.Agronomy College,Inner Mongolia Agricultural University,Huhhot 010019,China;2.Chifeng Tianze Bio-Technique Co. Ltd,Chifeng 025457,China)
Using the hybrid F1population from a cross between ‘Hei meiren’ and ‘MIN-021’ as materials,constructed a genetic linkage map of colored potato by SSR molecular marker technology and tetraploid mapping software TetraploidMap. The results showed that a total of 34 suitable SSR primer pairs with clear,stable and high polymorphic bands were screened from 255 tested SSR primers. Then these suitable SSR primers were used to amply genome DNA of 210 hybrid F1individuals from a cross ‘Hei meiren’בMIN-021’ and their parents,and a total of 201 polymorphic markers were amplified. Using the 201 SSR markers,two genetic linkage maps for parents ‘Hei meiren’ and ‘MIN-021’ were constructed,respectively. The maternal map contained 129 SSR markers,which distributed on 12 linkage groups,and had a map length of 1 167 cM,with an average marker distance of 9.04 cM;the paternal map included 131 SSR markers,which distributed on 12 linkage groups,and had a map length of 1 187 cM,with an average marker distance of 9.06 cM.
colored potato;tetraploid;SSR marker;construction of genetic linkage map
1000-1832(2015)04-0116-07
10.16163/j.cnki.22-1123/n.2015.04.025
2014-11-26
国家科技支撑计划资助项目(2012BAD02B05);内蒙古自然科学基金重大项目(2013ZD03).
崔阔澍(1987—),女,博士研究生;通讯作者:于卓(1958—),男,博士,教授,博士研究生导师,主要从事马铃薯及饲用作物遗传育种研究.
Q 943.2 [学科代码] 180·5155
A

