基于SRAP分子标记构建的菊苣遗传连锁图谱
梁小玉,季杨,白史且,黄琳凯,张新全*
(1.四川省畜牧科学研究院,四川 成都 610066;2. 四川省草原科学研究院,四川 成都 611731;3. 四川农业大学,四川 雅安 625014)
基于SRAP分子标记构建的菊苣遗传连锁图谱
梁小玉1,季杨1,白史且2,黄琳凯3,张新全3*
(1.四川省畜牧科学研究院,四川 成都 610066;2. 四川省草原科学研究院,四川 成都 611731;3. 四川农业大学,四川 雅安 625014)
基于SRAP标记,以遗传关系和表型差异大的菊苣亲本PI 651947和PI 652007杂交获得的84个F1单株为作图群体,进行连锁图谱的构建。采用Map Manager QTX b20软件进行连锁分析,分别构建了PI 651947和PI 652007的分子连锁框架图,共获得77个SRAP标记,其中父本遗传图谱涉及4个连锁群,包含19个标记,图谱总长为450.9 cM,标记间平均图距为23.7 cM。母本遗传图谱涉及13个连锁群,包含58个标记,图谱总长为1404.8 cM,标记间平均图距为24.2 cM。研究结果可为菊苣重要农艺性状QTL定位奠定基础,为菊苣分子育种研究提供了基础信息。
菊苣;遗传连锁图谱;SRAP分子标记
菊苣(Cichoriumintybus)为菊科(Asteraceae)温带多年生草本植物,在食品、医药、畜牧业及化工等多个领域发挥重要的作用,是当今世界最具发展潜力的经济植物之一[1]。菊苣是自然界中菊粉含量最高的植物,而菊粉是目前国际食品界受到高度重视的集医疗、保健于一体的优秀的功能性食品基料[2-3]。菊苣不仅富含菊粉,而且具有高产、优质、高抗、适应性范围广等优点,在美国、新西兰等畜牧业发达国家是非常重要的优质牧草。因此,提高菊苣产量和菊粉含量是当前菊苣研究和育种的首要目标。菊苣产量和菊粉含量均是受多基因控制的复杂的经济数量性状,遗传基础复杂,容易受遗传背景和环境因子的共同影响。传统遗传改良方法进展较慢,效率低,分子技术则是一种快速有效的改良数量性状的遗传改良方法。菊苣分子遗传图谱的构建将有助于重要的农艺性状基因的定位和克隆[4],从而大幅度提高菊苣的育种水平和育种效率,为优良新品种选育奠定基础。
国外学者对菊苣遗传图谱构建及QTL定位进行了研究,迄今为止,已公布的菊苣图谱仅有4张,而国内尚未开展这方面的研究。1997年Matteo等[5]采用单假测交方法,总计371个标记(包括16个RAPD、283个AFLP和72个SAMPL)构建了第1张菊苣分子遗传连锁图。研究者采用菊苣和苦苣(Cichoriumendivia)种间杂交的杂合材料和苦苣种自交产生的纯合材料为亲本,构建了46个单株的F1群体,最后获得了13个连锁群,图谱总长1330 cM。2010年,Cassan等[6]选择营养生长期对N肥敏感差异很大的两个食用型菊苣为亲本构建了302个重组自交系群体,采用两种标记绘制其遗传连锁图谱,最终上图标记包括9个SSR和73个RAPD,标记位点间平均图距为13.5 cM,并进行QTL定位分析。结果显示,控制N转化的基因对菊苣黄化芽球产量和品质的性状影响是截然相反的,与产量呈正相关而品质反之。认为通过分子标记辅助选择育种对农艺性状和生理性状QTLs基因分析,可以帮助选择适宜施N量获得优质的菊苣黄化芽球。同年,Cadalen等[7]选择2个工业菊苣和1个食用型菊苣为亲本,基于SSR和STS总计734个标记分别构建了3个群体的3张遗传连锁图。利用检测到的193个“桥标记”构建了9个同源连锁群,涉及472个标记,覆盖图距为878 cM。2013年,Gonthier等[8]采用工业用菊苣品种处理后获得的雄性不育的K28和花粉可育的K59 两份材料为亲本构建了389个单株的F1群体,基于13个与雄性不育位点相关,6个与自交不亲和位点相关的AFLP 标记转化的8个SCAR标记及256对AFLP引物构建了高密度的菊苣遗传连锁图谱。研究结果显示,包含NMS1-locus和S-locus的染色体区域的图距都局限于0.8 cM范围内。现有研究表明,菊苣遗传图谱构建及QTL定位研究较少,遗传研究相对滞后,而且主要是集中在以菊苣种和苦苣种为亲本的食用型和工业型菊苣图谱构建,以饲用型菊苣为亲本的菊苣图谱尚未见报道,也未见基于SRAP标记的菊苣遗传图谱构建的报道。由于已构建的菊苣图谱较少,而且构建图谱的亲本来源不同,涉及种间和种内的杂交材料,很难将现有的图谱信息进行整合分析和利用,在一定程度上限制了菊苣分子生物学研究的进程,制约了菊苣育种水平的提高。而SRAP是针对外显子区域进行扩增的一种新型标记,与RFLP、SSR、RAPD以及AFLP标记相比,操作更简单、成本低廉、稳定性好、遗传多态性高,可直接切胶回收用于测序,便于克隆目标片段,目前已经被成功应用于多种植物的图谱构建[4,9-15]。
本研究以饲用型菊苣种的栽培材料和野生材料为作图亲本,采用“双假测交”方法,利用SRAP标记构建菊苣遗传图谱,为饲用菊苣产量及其相关重要农艺性状QTL定位、菊苣杂种优势机理等研究搭建一个良好的平台,同时也为菊苣及其近缘种比较分析奠定一定基础。
1 材料与方法
1.1 F1作图群体的构建
菊苣材料由美国国家基因库提供,选择来源和农艺性状差异很大的两份材料为作图亲本,母本PI 651947为源自北荷兰的晚熟、饲用型栽培材料,直立株型、植株高大、叶片宽大、叶缘为全缘或者浅裂;父本PI 652007为源自波兰彼得哥什的晚熟野生材料,莲座期匍匐株型、植株低矮、叶片窄、叶缘为深裂[16](图1)。2010年进行人工杂交,杂交前分别针对父、母本,选择健壮、饱满即将张开并已露色的花序除去周围的小花序后套袋。观察到父本花序有大量花粉时开始杂交,直接从父本单株上剪下花序,然后轻轻把花粉抖落在母本花序上,授完粉之后,即刻套上杂交袋,并且挂上标签纸牌,注明杂交组合和授粉时间。母本植株杂交种子呈褐色时开始收获成熟种子。当年将杂交种子在温室大棚中利用育苗盘穴播育苗,9周后将F1群体移栽到试验基地中。提取单株DNA用于分子标记杂交后代鉴定,真杂种用于F1作图群体构建。

图1 父本PI 652007和母本PI 651947植株Fig.1 The plant male parental PI 652007 and female parental PI 651947 of parents
1.2 基因组DNA的提取及纯度检测
2011年3月,选取菊苣F1代杂种及亲本材料单株健康幼嫩叶片采用CTAB方法提取基因组DNA[9]。利用0.8%琼脂糖凝胶电泳和紫外分光光度计检测DNA浓度和纯度,合格的DNA稀释成10 ng/μL后在-20℃冰箱内保存备用。
1.3 SRAP-PCR 反应体系及扩增程序
参照Li和Quiros[9],Mishra和Nishani[17]及Guo和Luo[18]发表的引物分别设计了SRAP标记的20条上游和下游引物,由北京六合华大基因科技股份有限公司合成。PCR反应体系为15 μL,包括1 μL DNA,0.3 μL(2.5 U/μL) Taq DNA 酶,0.8 μL 10 mmol/L上游引物,0.8 μL 10 mmol/L下游引物,2 μL 10×PCR buffer,2 μL 25 mmol/L Mg2+,2.4 μL 2.5 mmol/L dNTPs,5.7 μL无菌水。PCR反应程序参考Li和Quiros[9]的方法进行优化,具体如下:94℃预变性4 min;94℃变性1 min,35℃退火1 min,72℃延伸1 min,共5个循环;94℃变性1 min,50℃退火1 min,72℃延伸1 min,共35个循环;72℃延伸10 min,4℃保存。PCR扩增产物在6.0%聚丙烯酰胺凝胶中电泳,点样6 μL,电泳缓冲液为1×TBE,稳压400 V,时间为2.0 h。电泳完成后银染,最后用数码相机拍照保存。
1.4 数据分析及遗传图谱构建
采用“双假侧交”(two-way pseudo-testcross)策略,按照显性标记统计方法人工统计双亲中存在多态性以及在子代中发生分离的条带。有带记为“1”,无带记为“0”,缺失或者无法判断的记为“-”。根据孟德尔分离规律,对F1群体所有条带进行卡方检验,符合1∶1分离比率的标记用于遗传图谱的构建,显著水平为0.05。采用Map Manager QTX b20软件进行连锁遗传分析,分别构建父本和母本的遗传连锁图谱。构图概率值设为0.001 (LOD=3), 采用 Kosambi函数[19]将重组率转换为图距(cM),最后用MapDraw V 2.1软件绘制遗传连锁图谱。
2 结果与分析
2.1 菊苣杂种鉴定
从SRAP引物中随机选取了40对引物对4个F1子代及其亲本的多态性进行扩增,筛选出带型清晰、稳定性好的11对引物作为检测菊苣杂交种纯度的特异引物,分别为:E17M12,E6M12,E8M15,E10M15,E8M16,E6M17,E6M18,E4M19,E3M20,E7M20,E9M20。筛选出的引物在F1与其亲本之间均呈多态性。对89个杂种的扩增图谱进行分析,84个F1植株均具有双亲特征带,鉴定为真杂种,5个只有母本特征带则鉴定为假杂种。结果表明,菊苣自交率很小,为5.62%。其中,引物对E10M15鉴定效率最高,一次性能鉴定出69粒真杂种,达到真杂种比例的82.14%,E17M12鉴定效率最差,一次性只鉴定出28粒真杂种,仅占真杂种比例的33.33%。
2.2 引物筛选
用400对SRAP 引物组合对双亲进行多态性筛选,共有384对引物能扩增出清晰、明亮条带,再分别对亲本及89株F1代个体筛选,根据后代的分离情况,选出了扩增稳定、能够用于图谱构建的SRAP 引物143对,引物入选率为35.75%(图2为引物E3M3对部分群体的标记分离情况)。
2.3 标记的多态性和偏分离分析
143对多态性引物在亲本和F1群体间共扩增出535条多态性条带,主要分布在90~500 bp之间。平均每个组合产生3.74个标记,最多达10个。经卡方(χ2)测验(P=0.05),535个标记中,偏离孟德尔分离规律的标记有121个,占22.6 %,符合1∶1的标记有216个,符合3∶1的标记有198个。利用1∶1的标记作图,共有56对引物的78个标记位点用于遗传图谱构建,其中59个标记来自母本PI651947,19个标记来自父本PI652007。
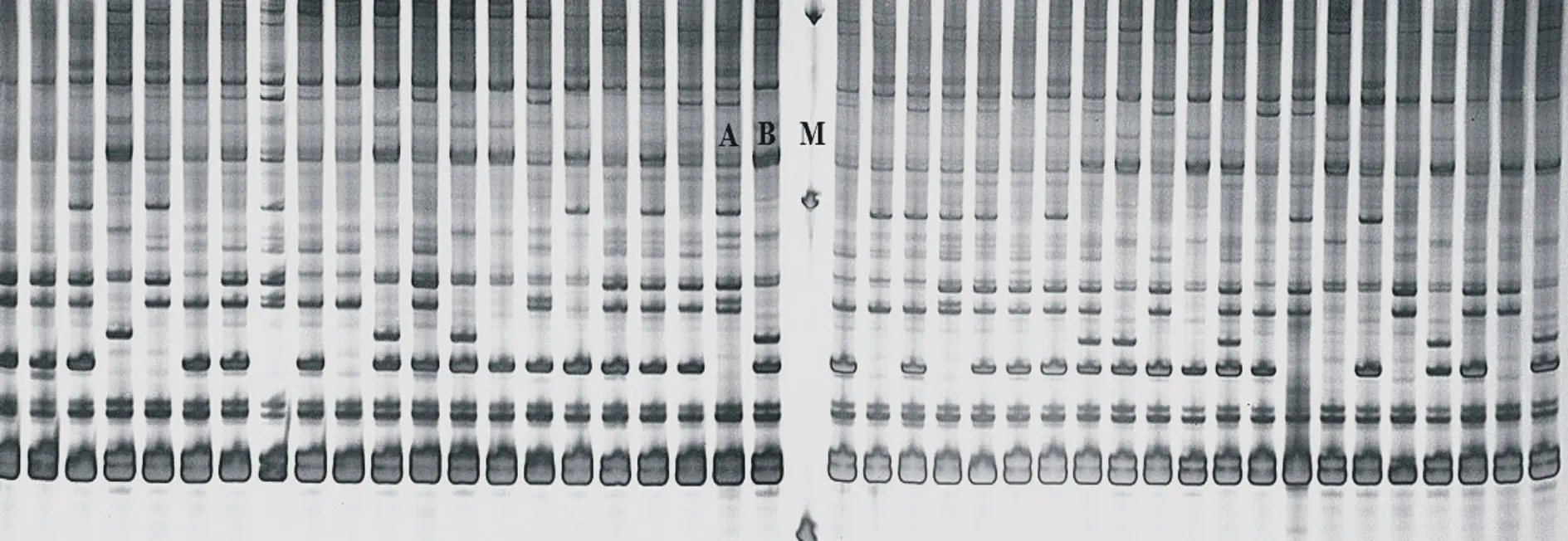
图2 SRAP(E3M3)标记在部分F1群体的分离 Fig.2 Segregation of SRAP(E3M3)marker in the partial F1 population A:PI651947; B:PI652007; M:标记Marker;其他Others:F1个体 F1 individuals.
2.4 遗传图谱构建
利用Map Manager QTX b20软件对216个SRAP多态性标记进行连锁遗传分析(表1)。共有77个标记进入连锁群,最终得到2张包含17个连锁群的菊苣分子遗传图谱(图3和4)。其中,父本的遗传图谱包含4个连锁群(图3),涉及19个SRAP标记位点,该图谱总长450.9 cM,标记间平均图距为23.7 cM,每个连锁群长度位于22.1~129.4 cM范围内,标记数介于2~8个;母本的遗传图谱包含13个连锁群(图4),涉及58个SRAP标记位点,该图谱基因组总长度1404.8 cM,每个连锁群的长度位于22.9~480.4 cM范围内,长度最长的连锁群LG1为480.4 cM,有17个位点。每个连锁群的标记位点数2~17个,每个标记位点间平均图距为24.2 cM。
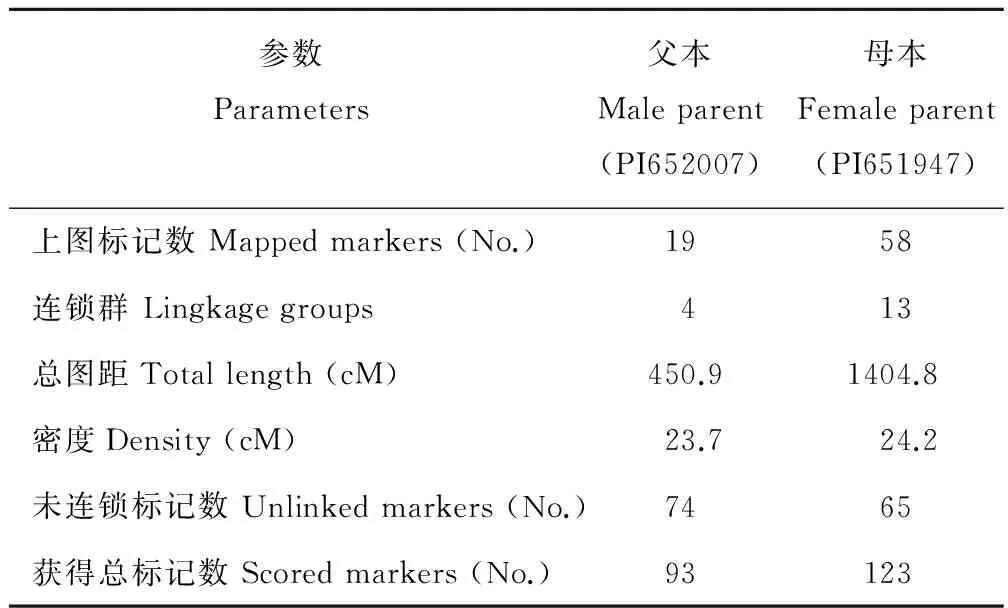
表1 用于连锁分析的位点及连锁分析结果

图3 基于SRAP标记构建的菊苣父本(PI652007)遗传连锁图谱Fig.3 PI 652007 genetic linkage map of chicory based on SRAP markers

图4 基于SRAP标记构建的菊苣母本(PI 651947)遗传连锁图谱Fig.4 PI 651947 genetic linkage map of chicory based on SRAP markers
3 讨论
本研究采用SRAP标记构建菊苣遗传连锁图谱,结果表明,SRAP标记操作简单、经济成本低、扩增谱带清晰,多态性好,对于构建高密度菊苣遗传连锁图谱是有效的。
遗传作图群体的类型与群体大小直接关系到遗传图谱的精度和应用范围。菊苣是一个自然杂交的二倍体物种,存在严重自交不亲和、花粉竞争和雄性先熟等防止自花受粉的冗余系统,具有高度杂合性,许多基因位点在F1代即发生分离,较难获得纯系,利用高世代群体构建遗传图谱很难[20]。从目前已经公布的4个二倍体菊苣分子遗传图谱看,群体样本数在40~400以内,种间或种内杂交所产生的F1代群体就会有高频率的分离位点,作图群体主要是F1杂交群体。理论上本研究中选择的亲本为亲缘关系远且多态性高的材料,获得了84个F1真杂种单株群体,检测到重组最小图距为13.4 cM,表明亲本和群体样本数量上可以满足构建菊苣连锁框架图的需要。“双拟测交”被认为是解决多倍性异花授粉植物遗传图谱构建的有效方法[21],该方法对F1群体作图已经被广泛应用于异花授粉植物中。与其他作图群体比较,F1群体作图不仅节约时间而且操作简单,也避免了自交不亲和所造成的阻碍。因此,本研究采用“双假侧交”策略对F1群体进行作图,可视为回交(BC)群体模型来进行图谱构建[22]。
本研究构建了饲用菊苣的父本和母本2张遗传连锁图谱,共包含了77个基于PCR扩增技术的SRAP分子标记。其中,母本图谱包含13个连锁群,涉及58个标记,总图距1404.8 cM,虽然上图的标记不多,但与Matteo等[5]构建的连锁图谱的总图距(1405 cM)一致,比较接近实际基因组,可以反映出双亲间遗传距离大,差异位点多。父本图谱仅包含了4个连锁群,总图距450.9 cM,远远小于已经构建的菊苣图谱总长度,主要原因可能是因为父本图谱上图标记太少仅有19个,因此对整个基因组的覆盖率也较小。此外,父、母本图谱上均存在较大的标记空隙区,连锁群数目与染色体组的数目不一致,而通常情况下,分子连锁群的数目与相应物种染色体的数目是一致的。除了上图标记少外,分子标记在染色体上分布的随机性及染色体不同区段交换值的异质性存在也会造成连锁群上存在较大空隙,严重的则出现小片段的连锁群[23]。
总的来看,总标记数目相对较少、作图软件的局限性以及作图群体较小是导致本研究所获得的连锁标记不多图距相对较大的主要原因,当然,数据统计的失误也可能造成标记图位差异的现象[24],有待于进一步增加标记数量,甚至利用不同分子标记特性的互补来减小遗传图谱中的空隙[25]。
致谢:本试验曾得到兰州大学谢文刚老师和四川农业大学蒋林峰、汪霞、赵欣欣等同学的支持,特此致谢!
[1] Bais H P, Ravishankar G A.CichoriumintybusL. - cultivation, processing, utility, value addition and biotechnology, with an emphasis on current status and future prospects. Journal of the Science of Food and Agriculture, 2001, 81: 467-484.
[2] Kikuchi H, Inoue M, Saito H,etal. Industrial production of difructose anhydride III (DFA III) from crude inulin extracted from chicory roots using Arthrobacter sp H65-7 fructosyltransferase. Journal of Bioscience and Bioengineering, 2009, 107: 262-265.
[3] Park K J, Oliveira de R A, Brod F P R. Drying operational parameters influence on chicory roots drying and inulin extraction. Food and Bioproducts Processing, 2007, 85(3): 184-192.
[4] Jin M Y, Liu L Z, Fu F Y,etal. Construction of a genetic linkage map inBrassicanapusbased on SRAP, SSR, AFLP and TRAP. Molecular Plant Breeding, 2006, 4(4): 520-526.
[5] Matteo De S, Michele M, Margherita L,etal. A first linkage map ofCichoriumintybusL. using a one-way pseudo-testcross and PCR-derived markers. Molecular Breeding, 1997, 3: 415-425.
[6] Cassan L, Moreau L, Segouinc S,etal. Genetic map construction and quantitative trait loci (QTL) mapping for nitrogen use efficiency and its relationship with productivity and quality of the biennial crop Belgian endive (CichoriumintybusL.). Journal of Plant Physiology, 2010, 167: 1253-1263.
[7] Cadalen T, Mörchen C, Blassiau A,etal. Development of SSR markers and construction of a consensus genetic map for chicory (CichoriumintybusL.). Molecular Breeding, 2010, 25: 699-722.
[8] Gonthier L, Blassiau C, Mörchen M,etal. High-density genetic maps for loci involved in nuclear male sterility (NMS1) and sporophytic self-incompatibility (S-locus) in chicory (CichoriumintybusL., Asteraceae). Theoretical and Applied Genetics, 2013, 126: 2103-2121.
[9] Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics, 2001, 103: 455-461.
[10] Li Y Y, Shen J X, Wang T H,etalConstruction of a linkage map using SRAP, SSR and AFLP markers inBrassicanapusL. Scientia Agricultura Sinica, 2007, 40(6): 1118-1126.
[11] Ou C G, Deng B T, Bao S Y,etal. QTL mapping for contents of main carotenes and lycopene in carrot (DaucuscarotaL.). Hereditas (Beijing), 2010, 32(12): 1290-1295.
[12] Aneja B, Yadav N R, Chawla V, Yadav, R C. Sequence-related amplified polymorphism (SRAP) molecular marker system and its applications in crop improvement. Molecular Breeding, 2012, 30(4): 1635-1648.
[13] LI J Q, Wang L H, Zhan Q W,etal. Genetic diversity of 20 ryegrass accessions by SRAP markers . Acta Prataculturae Sinica, 2013, 22(2): 158-164.
[14] Gu X Y, Guo Z H, Zhang X Q,etal. Genetic diversity ofElymussibiricusgermplasm resources revealed by SRAP markers. Acta Prataculturae Sinica, 2014, 23(1): 205-216.
[15] Chen Q, Yuan X J, He Y L.Screening molecular markers for heat tolerance and its relation to summer tolerance in tall fescue single plants. Acta Prataculturae Sinica, 2013, 22(5): 84-95.
[16] Liang X Y, Zhang X Q, Bai S Q,etal. Multiple statistical analysis of the phenotypic characters ofCichoriumintybus.Acta Prataculturae Sinica, 2013, 22(6): 257-267.
[17] Mishra K M, Nishani S. Molecular identification and genetic relationships among coffee species (CoffeaL.) inferred from ISSR and SRAP marker analyses. Archives of Biological Sciences, 2011, 63: 667-679.
[18] Guo D L, Luo Z R. Genetic relationships of some pcna persimmons (Diospyroskakithunb.) from China and Japan revealed by SRAP analysis. Genetic Resources and Crop Evolution, 2006, 53: 1597-1603.
[19] Kosambi D D. The geometric method in mathematical statistics. The American Mathematical Monthly, 1944, 51(7): 382-389.
[20] Zheng Y Q, Liu J X. Advances in construction and application of turfgrass molecular genetic maps. Acta Prataculturae Sinica, 2009, 18(1): 155-162.
[21] Humphreys M W, Yadav R S, Cairns A J,etal. A changing climate for grassland research. New Phytologist, 2006, 169(1): 9-26.
[22] Yu C, Jin M Y, Zhang B Z,etal. Genetic linkage map of anthurium andraeanum based on SRAP molecular markers. Acta Horticulturae Sinica, 2012, 39(6): 1151-1158.
[23] Grattapaglia D, Sederoff R. Genetic linkage maps ofEucalyptusgrandisandEucalyptusurophyllausing a pseudo-testcross: Mapping strategy and RAPD markers. Genetics, 1994, 137: 1121-1137.
[24] Yang X, Yu Y J, Zhang F L,etal. Linkage map construction and quantitative trait loci analysis for bolting based on a double haploid population ofBrassicarapa. Journal of Intergrative Plant Biology, 2007, 49(5): 664-671.
[25] Bi C, Lu J N, Yin X G. Molecular linkage map constructed by SSR markers in castor. Journal of Inner Mongolia University for Nationalities(Natural Sciences), 2013, 28(5): 532-564.
参考文献:
[4] 金梦阳, 刘列钊, 付福友, 等. 甘蓝型油菜SRAP、SSR、AFLP和TRAP标记遗传图谱构建. 分子植物育种, 2006, 4(4): 520-526.
[10] 李媛媛, 沈金雄, 王同华, 等. 利用SRAP、SSR和AFLP标记构建甘蓝型油菜遗传连锁图谱. 中国农业科学, 2007, 40(6): 1118-1126.
[11] 欧承刚, 邓波涛, 鲍生有, 等. 胡萝卜(DaucuscarotaL.)中主要胡萝卜素和番茄红素含量的QTL分析. 遗传, 2010, 32(12): 1290-1295.
[13] 李杰勤, 王丽华, 詹秋文, 等. 20个黑麦草品系的SRAP遗传多样性分析. 草业学报, 2013, 22(2): 158-164.
[14] 顾晓燕, 郭志慧, 张新全, 等. 老芒麦种质资源遗传多样性的SRAP分析. 草业学报, 2014, 23(1): 205-216.
[15] 陈群, 袁晓君, 何亚丽. 高羊茅单株耐热性相关分子标记的筛选及其与越夏性的关系研究. 草业学报, 2013, 22(5): 84-95.
[16] 梁小玉, 张新全, 白史且, 等. 菊苣主要表型性状的多元统计分析. 草业学报, 2013, 22(6): 257-267.
[20] 郑轶琦, 刘建秀. 草坪草分子遗传图谱的构建与应用研究进展. 草业学报, 2009, 18(1): 155-162.
[22] 于翠, 金茂勇, 张宝珠, 等. 基于SRAP分子标记的安祖花遗传连锁图谱的构建. 园艺学报, 2012, 39(6): 1151-1158.
[25] 毕川, 陆建农, 殷学贵. 蓖麻遗传图谱构建初报.内蒙古民族大学学报(自然科学版), 2013, 28(5): 532-564.
Construction of a genetic map for chicory usingsequence-related amplified polymorphism markers
LIANG Xiao-Yu1, JI Yang1, BAI Shi-Qie2, HUANG Lin-Kai3, ZHANG Xin-Quan3*
1.SichuanAcademyofAnimalSciences,Chengdu610066,China; 2.SichuanAcademyofGrasslandSciences,Chengdu611731,China; 3.DepartmentofGrasslandSciences,SichuanAgriculturalUniversity,Yaan625014,China
Sequence-related amplified polymorphism (SRAP) markers were used to construct a draft linkage map using an F1 population (84 individual plants) derived from a cross between PI 651947 and PI 652007; these lines possess different agronomic traits and DAN levels. Draft linkage maps of PI 651947 and PI 652007 were constructed using Map Manager QTX b20 software. 77 markers were used to construct parental linkage maps.The male map included 4 linkage groups with 19 molecular markers covering a total length of 450.9 centimorgans (cM). The average genetic distance between markers was 23.7 cM. The female map included 13 linkage groups with 58 molecular markers and covered a total length of 1404.8 cM. The average genetic distance between markers was 24.2 cM. This information established a foundation for extending genetic mapping in chicory and will serve as a framework for mapping QTLs and provide information required for further molecular studies.Key words: chicory (Cichoriumintybus); genetic linkage map; SRAP markers
10.11686/cyxb20150518
http://cyxb.lzu.edu.cn
2014-05-21;改回日期:2014-06-30
四川省畜牧科学研究院基本科研业务费专项资金(SASA2014A02)和国家科技支撑计划项目(2011BAD17B03)资助。
梁小玉(1976-),女,四川合江人,副研究员,博士。E-mail:liangxiaoyucao@163.com *通讯作者Corresponding author. E-mail: zhangxq@sicau.edu.cn
梁小玉, 季杨, 白史且, 黄琳凯, 张新全.基于SRAP分子标记构建的菊苣遗传连锁图谱. 草业学报, 2015, 24(5): 153-158.
Liang X Y, Ji Y, Bai S Q, Huang L K, Zhang X Q. Construction of a genetic map for chicory usingsequence-related amplified polymorphism markers. Acta Prataculturae Sinica, 2015, 24(5): 153-158.

