基于科学计量方法的生物实体研究方案
,
1 生物实体研究问题的提出
人类基因组计划的提出和完成,全面改变了生命科学的面貌,开辟了基因组学发展的新纪元[1-2]。科学家开始融合信息科学、计算机科学以及数学等学科的理论和技术,整体研究一个生物系统中所有生物实体(Gene、Disease、Phenotype、Chemical Compound、Protein、Drug和Virus等)的构成,以及特定条件下这些生物实体间的相互关系。这些研究对于揭示细胞内和细胞间的作用机理、疾病标靶基因的发现以及新药的研制等具有重大意义。
随着科技的不断进步及基因组学的迅速发展,生物医学领域海量的新生物实体不断被科研人员发现[3-4],与这些生物实体相关的知识形成了大量与人类健康密切相关的科研成果。
这些成果大都以论文形式发表,并被数字化成电子文献。由于这些文献都是非结构化的自然语言,因此为了获取研究所需的实体关系知识,科研人员需要花费大量的时间和精力来阅读数量众多的文献。于是,一种新的需求应运而生,那就是如何从海量的生物文献中迅速地找到生物实体之间可能存在的关系。
2 国内外研究现状
2.1 基于文献搜索引擎的生物实体关系发现
文献搜索引擎可以帮助科研人员解决部分问题。在PubMed中,我们以“diabetes[MeSH Major Topic], drug*, gene*”为检索式查询了讨论糖尿病、基因和药物3种生物实体关系的所有文献,返回3 473条摘要记录。说明存在大量探讨糖尿病、基因和药物之间关系的文献,但靠人工阅读无法从大规模的文献记录中迅速获取相关知识。
2.2 基于数据挖掘技术的生物实体识别
近年来,随着数据挖掘技术的不断发展,批量文本的自动化处理已成为现实,为生物实体的挖掘提供了新的契机。不过就国内外相关研究仍仅局限于对海量文献中生物医学实体的识别[5-6]。最早的生物医学实体识别方式是基于字典,如Proux等人于1998年第一次应用英语词典对基因和蛋白质进行了识别[7]。
基于启发式规则的方式是早期被广泛使用的一种方法,Fukuda等最早利用基于规则的系统判定文献中的蛋白质名称[8];Tsuruoka等采用启发式规则以最小化相关术语的歧义性和变化性,实现了术语名称的标准化,从而提高了查找字典的效率[9]。
基于机器学习的方式则是目前主流的方法,它主要利用统计方法从大量数据中估算相关参数和特征进而建立识别模型,具有客观、移植性好等特点[10]。
随后,生物信息领域的科研人员开始尝试以生物医学实体共现的手段从大规模的生物文本中探测隐藏的关系,并取得了一定的效果[11-14]。这种方法假设同一篇文献中出现的两个生物医学实体之间存在某种关系,如果两个生物医学实体同时出现于多篇文献中,则二者之间存在关系的可信度增强。然而,这些研究仅局限于同种生物实体之间的关系(如蛋白质—蛋白质)或两种不同生物实体之间的关系(基因—蛋白质)。
2.3 基于知识库的生物实体揭示
为了便于研究人员迅速获取生物实体相关知识,一些发达国家投入大量的人力、物力和财力,以人工标注的方式构建相关生物实体关系知识库。Nucleic Acids Research (NAR)期刊在每年一度的特刊中会对全球所有高质量的、有价值的生物实体关系知识库进行分类和描述,现已介绍1 552个数据库[15],其他数据库集合还包括MetaBase[16]和Bioinformatics Links Collection[17]等。由于这些知识库中的知识大都以结构化的方式存储,故在一定程度上满足了科研人员快速获取知识的需求。但随之产生的问题是数据生产耗费巨大,因而增长速度缓慢[18]。有研究表明,目前仅有20%的生物医学知识以结构化的形式存储于生物实体关系知识库中,剩余的80%则为非结构化数据,以自由文本的形式隐藏在科学文献中[19-21],有待挖掘。
3 基于科学计量方法的生物实体评价研究方案
科学计量学关注的对象主要分为两类:一类为宏观特征,如作者、机构、国家、期刊等,它们用于评价文献的非内容特征;另一类为中观特征,如关键词、题目和参考文献等,主要用于文献主题的分析和评价。事实上,科研文献中还存在一种微观特征,即概念实体,专指科研文献中出现的某一事物的概念或对象,例如文献中所使用的某一理论的名称、某一方法的名称或本文所研究的生物实体。文献中的概念实体对象也是科学计量学应关注的一种文献特征(图1)。
由于概念实体隐藏在科研文献中,而早期数据挖掘技术不成熟,主要依靠手工标识的方式实现。因此方法的推广性较差,相关研究也较少。如有学者以“研究理论”(theory)为对象,人工对信息科学研究和家庭治疗研究领域文献中使用的基本理论进行标识,进而分析这些理论被使用的情况[22-23]。虽然有关生物实体的研究已取得一些成果,但仍局限于生物实体的识别研究和基于共现关系的生物实体关系研究。因此,本文拟基于科学计量方法进行生物实体评价研究,即选取科研文献中出现的生物实体为研究对象,利用科学计量方法对其影响力进行评估,以期对生物实体之间的关系进行分类和预测,将其所代表的知识快速准确地展现给科研人员,加速科研假说的生成,加快科学研究进程。生物实体评价研究的具体方案如下。
3.1 生物实体的识别
科研文献中生物实体的有效识别是实现生物实体研究的前提。传统的生物实体识别方法包括基于字典、基于启发式规则和基于机器学习等方法。基于字典的识别方法受字典本身的限制,会产生一些假阳性和假阴性数据,需引入一些简单的规则来辅助;基于规则的识别方法由于需要人工处理,推广性较差;基于机器学习的方法对训练语料的规模和质量依赖性较大。故可结合3种方法开展生物实体识别:以机器学习方法为主线,将字典特征形式整合至机器学习方法的第一步(生物实体特征选择),接着进行机器学习的第二步(采用分类方法对生物实体进行分类),最后将基于启发式规则的方法融入机器学习方法的后期处理中。
3.2 基于“引用”关系的生物实体关系建模
我们基于引文分析理论提出了概念实体的“引用”关系,其实质上是通过文献之间的引用关系建立概念实体之间的联系,即做出如下假设:如果文献P1引用了文献P2(P1→P2),P1中提及了实体K1和K2,P2中提及了实体K3和K4,则认为K1“引用”了K3和K4(K1→K3、K1→K4),K2“引用”了K3和K4(K2→K3、K2→K4)(图2)。显然,基于大数据建立的这种“引用”关系具有一定的必然性。
3.3 基于“引用”网络的生物实体研究
与生物实体共现网络研究相似,通过网络直径、最小路径、密度和最大Component等网络宏观指标可研究生物医学实体“引用”网络的拓扑特征;通过K-core、Clique等网络中观指标可挖掘生物医学实体“引用”网络的社团结构;通过中心度、PageRank等网络微观指标可分析生物医学实体之间的相互关系。此外,通过生物实体“引用”网络还可以跟踪生物实体所代表知识的流动轨迹,探索生物医学知识转移和扩散的规律。
我们在前期的研究中,基于上述假设构建生物信息数据库“引用”网络[24]和生物医学实体“引用”网络(Gene、Disease和Drug)[25]。对前一个网络的拓扑特征和主路径分析发现,通过引文建立的生物医学信息数据库之间的关联有助于探索数据库的使用规律;对后一个网络中生物医学实体按照中心度指标进行排序,发现该方法能够检测出绝大多数在Comparative Toxicogenomic Database数据库中手工注释的生物医学实体关系。
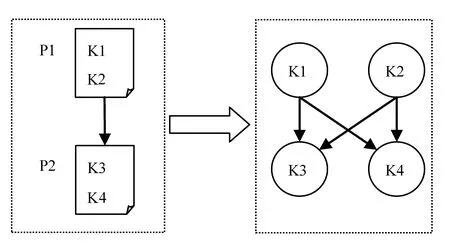
图2 通过文献引用建立的生物实体"引用"关系
4 总结
4.1 发展和完善科学计量学理论和实践研究
科学计量学所关注科研文献中的宏观特征和中观特征大都以题录形式储存于文献数据库,其所代表的均为文献的外显知识,可免费获取。由于概念实体大都蕴藏于文献的全文之中,文献的内含知识只有通过文本挖掘方可获取。此外,概念实体与文献中观特征最大的区别在于其专指性更强。对生物实体这一微观特征对象的评价扩展了科学计量学的内涵,有利于该学科理论的进一步发展和完善。
4.2 开辟知识管理研究的新视角
科学计量方法为生物实体等非结构化数据提供了一种“自上而下(Top-down)”的管理方式,即从海量文本入手分析,一方面帮助生物医学科研人员快速准确地发现隐藏于文献中的生物实体关系,通过合理假设、实验验证,大大节省知识发现的周期;另一方面对生物知识的流动、转移、扩散和利用等规律进行深入探索。因而,生物实体评价开辟了知识管理方式研究的全新视角,并帮助生物医学科研人员迅速、准确地获取隐藏于海量科学文献文本中的相关生物医学知识,加快了科研假说的提出,从而进一步推动生物医学相关学科的发展。

