中华绒螯蟹两种微卫星DNA分型方法的应用比较
刘 皓, 刘 青, 吴旭干, 何 杰, 董鹏生, 成永旭
(1. 上海海洋大学 水产种质资源发掘与利用教育部重点试验室,上海 201306;2. 上海海洋大学 上海高校知识服务平台水产动物遗传育种中心,上海 201306)
中华绒螯蟹两种微卫星DNA分型方法的应用比较
刘 皓1, 2, 刘 青1, 吴旭干1, 何 杰1, 董鹏生1, 成永旭1, 2
(1. 上海海洋大学 水产种质资源发掘与利用教育部重点试验室,上海 201306;2. 上海海洋大学 上海高校知识服务平台水产动物遗传育种中心,上海 201306)
为对比荧光标记毛细管电泳和聚丙烯酰胺凝胶电泳在微卫星等位基因分型上的效果,试验选择6对微卫星引物,对60个中华绒螯蟹个体基因组DNA进行PCR扩增,荧光标记法共检测出等位基因129个,平均每个位点21.5个等位变异,聚丙烯酰胺凝胶电泳共检测出等位基因174个,平均每个位点29个等位变异。对位点Esin67的PCR产物分别进行重复检测,发现荧光标记法的重复率为100%,而聚丙烯酰胺凝电泳法两次结果存在较大差异,证实了荧光标记法检测的可靠性显著高于聚丙烯酰胺凝胶电泳。对两种方法的检测效率和经济成本的分析表明,荧光标记法虽然成本较高,但效率高于聚丙烯酰胺凝胶电泳法。建立单重PCR的多重毛细管电泳技术是提高效率、降低成本的有效途径。
中华绒螯蟹;微卫星;PAGE;荧光标记;等位基因
微卫星又称为简单序列重复(Simple Sequence Repeat,SSR)或简单序列长度多态(Simple Sequence Length Polymorphism,SSLP)或短串联重复(Short Tandem Repeats,STR)。微卫星标记作为一种较为理想的分子标记,早在1994年联合国粮农组织(FAO)在其持续发展和管理动物遗传资源的战略计划中, 就将SSR标记作为优先推荐的分析工具[1]。SSR具有分布广泛、数量充足、多态性高、共显性、重复性强、操作简便等优点,已广泛应用于物种鉴定,种群遗传多样性和遗传结构分析、亲权鉴定、基因定位、基因组作图、数量性状位点解析等领域[2-6]。SSR的技术路线主要包含4个步骤:微卫星序列的获得与引物合成,PCR扩增,产物的检测分型,数据分析。
在已获得SSR序列信息转而进入大规模应用时,主要工作量便集中在PCR扩增和产物检测上,SSR等位基因分型方法的选择直接决定着工作效率、结果的可靠性及可重复性。SSR等位基因分型的方法有琼脂糖凝胶电泳、聚丙烯酰胺凝胶电泳(PAGE)、荧光标记毛细管电泳、高分辨率熔解曲线(HRM)等,但应用最为广泛的是PAGE和荧光标记法[7]。PAGE法因其操作较为简单,成本较低,且具有高于琼脂糖凝胶电泳的分辨率[8],被认为能够满足一般的研究需求,但又存在着工作量大、数据收集完全依赖人工、同一板胶的等位基因变异易出现识别误差,以及不同批次不同胶片之间数据的统一较为复杂等缺点,往往导致实验结果不够可靠[9]。荧光标记毛细管电泳具有分析快速、数据收集自动化、结果可靠及重复性高等优点,可以分辨出最低1个bp的序列长度差异,因而精确度更高,但因成本较高,在一定程度上限制了其广泛应用。为了得到更为精确可靠的SSR等位基因信息,很多研究者都选用了荧光标记法进行等位基因分型[10-11]。然而,有关PAGE和荧光标记法在SSR分型中的重复性和可靠性的研究,尚未见报道。
本研究利用非变性聚丙烯酰胺凝胶电泳(Native-PAGE)和荧光标记毛细管电泳技术,选择6对SSR引物对中华绒螯蟹60个野生个体进行PCR扩增(表1),对产物进行分型检测,以比较两种分型方法的准确性以及成本和效率。
1 材料与方法
1.1 材料
实验用长江野生中华绒螯蟹成蟹采集自江苏镇江江段,雌雄各半,合计60只,编号为YW1-60,体重在100~150 g之间。取其附肢肌肉固定于95%乙醇中,-20℃保存。
1.2 DNA提取
采用海洋动物基因组DNA提取试剂盒(DP324-03,购于TIANGEN公司)抽提中华绒螯蟹基因组DNA,琼脂糖凝胶电泳配合核酸蛋白检测仪检测质量和浓度,ddH2O稀释为50 ng/μL待PCR用。
1.3 引物筛选和PCR扩增
从已报道的中华绒螯蟹SSR引物[12-13]中选择6对多态性高、扩增效果好的引物分别合成为普通引物和正向5′端荧光标记(FAM或HEX)引物(见表1)。10 μL PCR体系包含50 ng 模板,1 μL 10×PCR buffer,200 μM dNTP,各0.5 μM正反向引物,0.5 U Taq 聚合酶,ddH2O补充体积至10 μL(聚合酶、buffer和dNTP均购于Takara公司)。降落PCR(Touch-down PCR)程序设置为:94℃预变性3 min;93℃变性1 min,56~61℃退火30 s,72℃延伸1 min,共2个循环;93℃变性1 min,54~59℃退火30 s,72℃延伸1 min,共2个循环;89℃变性1 min,52~57℃退火30 s,72℃延伸1 min,共5个循环;89℃变性1 min,50~55℃退火30 s,72℃延伸1 min,共20~25个循环;循环结束后,72℃再延伸10 min,4℃终止反应。PCR产物-20℃保存待用。

表1 6个中华绒螯蟹微卫星位点的特征
*—荧光标记法中的上游引物 5′端加上 FAM或HEX 荧光标记。
1.4 扩增产物的检测分型
1.4.1 PAGE检测
配制8%的聚丙烯酰胺凝胶,根据PCR产物的丰度,调整加样量,每板凝胶舍去两端的若干个加样孔,仅在中间31个加样孔加入DL1000Mraker和PCR产物。300 V电泳80 min。快速银染法步骤如下:1)分离凝胶放入托盘,双蒸水洗5 min;2)倒掉双蒸水,加入1‰硝酸银溶液染色8 min;3)倒掉硝酸银溶液,加入双蒸水迅速洗涤2次;4)加入适量显色液(每1000 mL双蒸水加入0.5 g碳酸钠,20 g氢氧化钠,5 mL甲醛溶液)显色至条带清晰可见;5)拍照。用Gel-Pro analyzer-4分析照片,读取目的条带的长度,数据导入Microsoft excel中,待下一步分析。
1.4.2 荧光标记检测
每两个加入了不同颜色荧光标记的位点的PCR产物混合后,使用甲酰胺变性,以ROX-500作为分子量内标,使用ABI-3730XL全自动DNA测序仪进行检测分析。GeneMaper-3.5扫描峰图(见图1),读取目标峰所代表的等位基因片段长度,将数据导入Microsoft excel中。

图1 PCR产物混合后的荧光标记毛细管电泳结果
2 结果与分析
2.1 聚丙烯酰胺凝胶电泳与荧光标记法检测效果比较
对原始数据进行校准,校准的依据是基因库中报道的微卫星的重复类型。使用CONVERT软件将数据转换为Popgene32所需的格式,通过Popgene32分别得出两种方法检测到的等位基因个数(见表2)。在6个中华绒螯蟹微卫星座位中,聚丙烯酰胺电泳共检测出174个等位变异,平均每个位点29个;荧光毛细管电泳则仅检测出129个等位变异,平均每个位点21.5个。
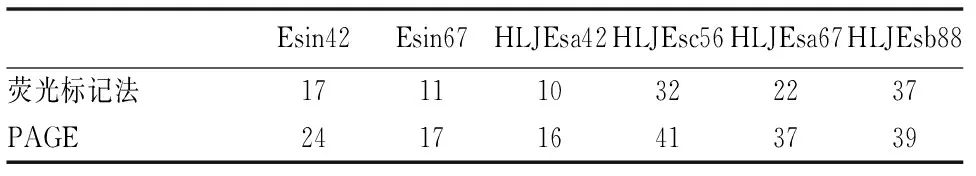
表2 两种分型方法在6个SSR座位中检测出的等位基因数

图2 位点Esin67在YW17中扩增产物的2次荧光标记法检测图谱
Fig 2 Sample YW17′SSR profile of loci Esin67 after two batches capillary electrophoresis detection
选择SSR位点Esin67的30个PCR产物,再次分别使用两种方法进行分型检测,作为不同批次条件下检测结果的对照。PAGE法第1次检测出的等位变异个数为17个,第2次仅检测到13个等位变异,荧光标记法2次检测得到的等位变异个数均为11个,每个样本的检测结果均能重复(见图2)。就每个样本中先后检测到的基因型来看,在本研究中PAGE检测结果重复率较低(见图3和图4)。

图3 位点Esin67扩增产物的2次PAGE检测图谱

图4 Esin67 PCR产物2个批次下PAGE分型结果
Fig 4 Detection results in loci Esin67 by PAGE of two batches
选取位点Esin67的同一个PCR产物,在聚丙烯酰胺凝胶多个点样孔上样后进行电泳,电泳后读取条带,以了解聚丙烯酰凝胶电泳时同一个PCR产物在同一板胶中条带的变异情况(见图5和图6)。

图5 同一PCR产物多个加样的PAGE检测图谱
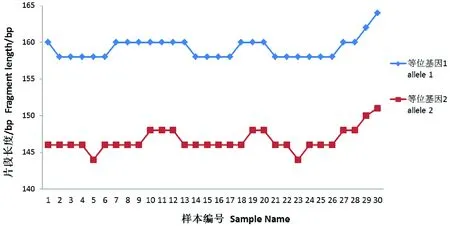
图6 同一PCR产物的2个等位基因在同一板胶中显示的变异
2.2聚丙烯酰胺凝胶电泳与荧光标记法成本及时效比较
在不考虑仪器购置和仪器损耗的前提下,仅就试剂成本来看,经过计算,PAGE方法检测一个PCR产物的成本为0.13元,荧光标记方法的耗费为5.40元(见表3)。在本研究中,荧光标记法的试剂成本是PAGE法的数十倍,其中,荧光引物合成费用占成本的大半。

表3 PAGE和荧光标记法检测360个PCR产物样本所需试剂成本
对大量样本而言,工作效率也非常重要,试验结果表明两种方法的工作效率差异很显著。在本研究的实验流程和实验规模下,以检测360个PCR产物为例,聚丙烯酰胺凝胶电泳从制胶到最终读出目的条带长度耗时约为14.5 h;荧光毛细管电泳从样品前处理到读出目的片段长度耗时约为3.5 h。从时效上看,本研究中荧光毛细管电泳的工作效率约为聚丙烯酰胺凝胶电泳的4倍。
3 讨论
本研究对360份PCR扩增产物进行两种方法分型比较,得到了一个有趣的结果:分辨率较低的PAGE方法检测到的等位基因个数反而比分辨率很高的荧光标记毛细管电泳要多,且在6对引物中表现出相同的结果。对同一批PCR产物进行重复检测,荧光标记法具有很强的重复性,每个样本的检测结果均能重复,而PAGE法则重复率较低,同一个PCR产物经不同批次的电泳后显示出了不同的结果;以同一个PCR产物在同一聚丙烯酰胺凝胶上加多个样的方式进行电泳,则证明了同一等位基因片段在同一板PAGE凝胶上的迁移率也出现一定差异,这就解释了在本研究中为什么PAGE所检测到的等位基因数量比荧光标记法多。结合以上分析,我们推断: PAGE法用于SSR等位基因检测时,容易受到凝胶配制和电泳过程中各种理化条件等因素的影响,同时量大而繁琐的目的条带数据读取和收集工作同样会对结果产生影响,也增加了不同批次、不同胶板间结果的不一致性,这样就会导致同一等位基因片段在不同批次中检测出的片段长度不同;而荧光标记法实现了SSR等位基因分型的高通量和自动化,其检测结果相对精确和可靠。
当面临大批量样本的SSR等位基因变异的分型检测时,工作效率是研究者必须要考虑的因素。就本文中检测360个样本的时效来说,荧光标记法是PAGE法的4倍,而在郝晨阳等的研究中[9],这个值高达7.8,这可能是因为在样本量更大的情下,加之多重PCR体系的应用,荧光标记法能够节约更多的时间和人工成本,凸显更高的工作效率。
SSR分型成本是评价分型方法的一个重要指标。以本研究结果为例,荧光标记法的试剂成本是PAGE法的数十倍。在许多SSR应用的研究中,样本量足以千计,而荧光引物合成后可用于更多样本的PCR扩增,这样就可以有效地减小荧光引物合成对总成本的贡献率。但总体来说,荧光标记法的使用成本较高,一定程度上限制了这种SSR分型方法的使用。探索一种廉价高效的SSR检测方法,是科研工作者和该技术广泛应用必需考虑的重要问题。多重PCR体系的应用对荧光标记法成本的控制及SSR分型的工作效率的提高提供了可能。多重PCR技术是指在同一PCR体系中同时对多对引物进行扩增,需要保证所有引物能够同时扩增,引物间不能有相互作用,不同SSR位点的扩增产物长度范围不能重叠,产物浓度相当等条件[14]。然而,多重PCR体系的成功建立是一个复杂的摸索过程,需要大量的前期工作。基于多重PCR方法,单重PCR产物混合后的毛细管电泳可以大大简化PCR体系建立的摸索过程,只需要将目标片段长度范围不同的位点的PCR产物进行混合,然后进入毛细管电泳仪进行检测[15],可以达到多重PCR荧光标记毛细管电泳相同的效果,是一种有效降低荧光标记法成本的简便途径。
综上所述,荧光标记毛细管电泳法与聚丙烯酰胺凝胶电泳相比,分型效果更加高效可靠,为大规模的SSR标记应用提供了可能,还可以通过多重PCR或单重PCR产物混合后电泳以降低分型成本。虽然荧光标记法试剂价格相对较高,但显著节约了人工成本。建立单重PCR产物混合后的毛细管电泳分型技术较多重PCR相对易于实现,这是提高分型效率、降低成本的有效途径。在实际科研工作中,根据不同物种及等位基因数量和长度等具体情况,应该综合考虑结果可靠性、分型效率和成本等多方面因素,选择一种合适的SSR等位基因分型方法。
[1]Baker J S F. A global protocol for determining genetic distance among domestic livestock breeds[C]. Proceedings of the 5th World Congress on Genetics Applied to Livestock Production, 1994, 21: 501-508.
[2]李晓晖,许志强,潘建林,等.中华绒螯蟹人工选育群体的遗传多样性[J].中国水产科学,2010, 17(2):236-242.
[3]欧阳磊,陈金慧,郑仁华等.杉木育种群体SSR分子标记遗传多样性分析[J].南京林业大学学报:自然科学版,2014,38(1):21-26.
[4]Yang M, Tian C X, Liang X F, et al. Parentage determination of mandarin fish (Sinipercachuatsi) based on microsatellite DNA markers[J]. Biochemical Systematics and Ecology, 2014, 54:285-291.
[5]Chail L J, Biswas M K, Yi H L, et al. Transferability, polymorphism and effectiveness for genetic mapping of the Pummelo (CitrusgrandisOsbeck) EST-SSR markers[J]. Scientia Horticulturae, 2013, 155(29):85-91.
[6]Li Y F, Liu Z Y, Wang Y S, et al. Identification of quantitative trait loci for yellow inner leaves in Chinese cabbage (BrassicarapaL. ssp.pekinensis) based on SSR and SRAP markers[J]. Scientia Horticulturae, 2012, 133(6):10-17.
[7]戴 剑,张云辉. SSR扩增产物检测方法比较及其实验操作注意事项[J].种子,2013,32(9):120-123.
[8]霍金龙,罗古月,张 娟,等.猪微卫星多态性两种检测方法的比较研究[J].云南农业大学学报,2004,19(3):314-317.
[9]郝晨阳,王兰芬,贾继增,等.SSR荧光标记和银染技术的比较分析[J].作物学报,2005,31(2):144-149.
[10]朱泽远,王亚菊,施用晖,等.荧光标记微卫星分析人工饲养中华绒螯蟹的遗传多样性[J].中国海洋大学学报,2007,37(4):591-596.
[11]傅建军,李家乐,沈玉帮,等.草鱼野生群体遗传变异的微卫星分析[J].遗传,2013,35(2): 192-201.
[12]Hanfling B, Weetman D. Characterization of microsatellite loci for the Chinese mitten crab,Eriocheirsinensis[J]. Molecular Ecology Notes, 2003(3): 15-17.
[13]Chang Y M, Liang L Q, Li S W, et al. A set of new microsatellite loci isolated from Chinese mitten crab,Eriocheirsinensis[J]. Molecular Ecology Notes, 2006(6): 1237-1239.
[14]公维华,张宁波,程佳月,等.小型猪微卫星标记多重PCR体系的建立与应用[J]. 中国比较医学杂志,2009, 19(2):22-25.
[15]马雪霞,王 凯,郭旺珍,等.棉花SSR多重PCR技术的初步研究和利用[J].分子植物育种,2007,5(5):648-654.
Comparison of two detection systems of microsatellite markers in the research of Chinese mitten crabs (Eriocheirsinensis)
LIU Hao1,2, LIU Qing1, WU Xu-gan1, HE Jie1, DONG Peng-sheng1, CHENG Yong-xu1,2
(1. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education,shanghia ocean University, Shanghai 201306; 2. Shanghai Universities Knowledge Service Platform Aquatic Animal Breeding Center, Shanghai Ocean University, Shanghai 201306, China)
360 PCR products of 6 loci among 60 Chinese Mitten Crabs(Eriocheirsinensis)were analyzed by capillary electrophoresis with fluorescent-labeled primers and polyacrylamide gel electrophoresis (PAGE), respectively. Total 129 alleles were obtained and 21.5 alleles were detected for every pairs of primers by capillary electrophoresis system, while aggregate 174 and average 29 alleles were detected with PAGE. The repetition rates were at 100% when loci Esin67 was detected for the second time by capillary electrophoresis method, while for PAGE the rates were getting really close to 0, which confirmed that capillary electrophoresis is more reliable than PAGE significantly. More ever, analysis showed that capillary electrophoresis′s material costs and efficiency is higher than PAGE. Establishing a single PCR multiple capillary electrophoresis technology is an effective way to improve efficiency and reduce cost.
Chinese mitten crabs (Eriocheirsinensis); SSR; PAGE; fluorescent-labeled; allele
2014-05-05
2014-06-22
国家863高技术研究发展计划项目 (2012AA10A409-5);上海市科委科技崇明专项 (13231203504);上海高校水产学一流学科建设项目(沪教科2012-62);上海高校创新团队(第二期)项目(沪教科2009-26)
刘 皓,硕士研究生,研究方向为中华绒螯蟹遗传育种,E-mail:siyuewuyv @sina.com;
成永旭,博士,教授,研究方向为甲壳动物营养繁殖,E-mail: yxcheng@shou.edu.cn。
Q959.1;Q343.1+2;S917.4
B
2095-1736(2015)01-0090-05
doi∶10.3969/j.issn.2095-1736.2015.01.090

