水稻叶片氮含量反演偏最小二乘模型设计
王奕涵,石铁柱,刘会增,王俊杰,邬国锋
(1.武汉大学资源与环境科学学院,武汉430079;2.海岸带地理环境监测国家测绘地理信息局重点实验室,广东深圳518060;3.空间信息智能感知与服务深圳市重点实验室,广东深圳518060;4.深圳大学生命科学学院,广东深圳518060)
水稻叶片氮含量反演偏最小二乘模型设计
王奕涵1,石铁柱1,刘会增1,王俊杰1,邬国锋2,3,4
(1.武汉大学资源与环境科学学院,武汉430079;2.海岸带地理环境监测国家测绘地理信息局重点实验室,广东深圳518060;3.空间信息智能感知与服务深圳市重点实验室,广东深圳518060;4.深圳大学生命科学学院,广东深圳518060)
针对高光谱偏最小二乘模型(PLSR)反演作物氮含量时易出现数据冗余和模型复杂的问题,尝试结合波段深度分析和遗传算法(GA)建立水稻氮含量PLSR反演模型。基于去包络线处理的水稻高光谱数据(350nm~750nm),选取波段深度(BD)、波段深度比(BDR)、归一化面积波段深度(BNA)和归一化面积波段指数(NBDI)4种波段深度指数分别建立BDA-PLSR模型,进而采用遗传算法波段选择选取最适宜波段深度指数建立GA-PLSR模型,并将GA-PLSR模型与BDA-PLSR模型进行对比。结果显示,基于BNA的GA-PLSR模型在反演水稻氮含量中获得了最佳的结果(Adj.R2=0.67,RMSEP=0.20,RPD=1.84)。研究证明,利用波段深度分析建立的PLSR模型能一定程度上解决数据冗余问题,进一步采用遗传算法进行波段选择能更有效挖掘光谱信息,提高模型精度。
水稻;氮含量;偏最小二乘回归;波段深度分析;遗传算法
0 引 言
氮素含量是作物营养状况的重要指标之一,影响着作物的产量与品质。实时快速地获取氮含量信息是评价作物长势、估测产量和品质以及精确管理氮素营养的前提。传统的作物氮素测定方法通过大量野外调查抽样和昂贵实验室分析获得,时效差且难以在宏观尺度上推广[1]。遥感技术,尤其是高光谱遥感技术的出现为快速、高效、无损检测作物氮含量提供了一种有效途径[2]。
大量研究通过建立遥感原始波段或植被指数与生化组分含量之间的多元统计回归模型,如逐步线性回归,来估测植物叶片生化组分含量。然而在使用这些回归方法时,容易出现“过度拟合”的现象,缺少增强标准化吸收特征及减小与光谱变化无关的噪声的预处理[3]。针对这些问题,Kokaly[4]提出了改进方法,使用连续统去除之后的光谱进行波段深度分析,成功地对干枯植物落叶的生化组分含量作出了估算。此后,波段深度分析方法(BDA)得到了广泛应用,如Curran等[3]使用波段深度分析方法估测干燥针叶松树的12种生化组分,取得了较为理想的效果;Mutanga和Skidmore等[5-7]将波段深度分析与逐步多元线性回归以及神经网络结合用于预测植被的生化指标含量。近年来,部分学者开始将波段深度分析与偏最小二乘回归(PLSR)法结合进行植物叶片生化组分反演[8-9],此方法应用于水稻新鲜叶片氮含量估算还鲜有探索。全波段高光谱数据具有高数据冗余性、共线性以及噪声问题,影响模型的预测能力以及计算效率,因此合适的波段选择方法对于提高模型精度和降低模型复杂度有很重要的作用[10]。已有研究将遗传算法(Genetic Algorithm,GA)用于波段选择提高高光谱反演模型的精度[11-12],此方法在植物生化组分反演上仍有很大潜力。
基于实验室水稻光谱数据,本文旨在:结合波段深度分析,构建孕穗期水稻高光谱波段深度分析指数与叶片氮含量定量估算的BDA-PLSR模型;采用GA选择敏感波段,建立基于敏感波段的PLSR模型(GA-PLSR),并对比BDA-PLSR和GA-PLSR模型,探究GA波段选择方法在降低数据冗余和提高模型精度方面的潜力。
1 数据采集和方法
1.1 光谱测量及样本测定
钟祥位于湖北省中部,汉江中游,气候温暖湿润,年降雨量952.6mm,年平均气温15.9℃[13]。在2012年8月8日~12日(水稻孕穗期),随机选取不同区域的水稻田80个点作为实验数据采集点,采集冠层水稻鲜叶带回实验室进行光谱和氮含量测量。
光谱测量在暗室环境下进行。将样本平铺在黑色纸质托盘上,使用ASD公司的FieldSpec 3地物光谱仪进行测定。光谱仪光谱测定范围为350nm~2500nm,其中350nm~1000nm区间光谱分辨率为3nm,采样间隔为1.4nm;在1000nm~2500nm区间光谱分辨率为10nm,采样间隔为2nm。每个样本各测10个光谱,取平均值作为该样本的光谱反射值;每隔6个样本进行白板校正。光谱测量完毕后,将叶片烘干、粉碎后用凯氏定氮法[14]测定氮含量。
1.2 数据预处理
将测得的氮含量和原始光谱生成原始数据集,使用主成分分析法[15]检测并去除异常点。剩余的样本进行建模集和验证集的划分,2/3作为建模集,1/3作为验证集。为保证模型对验证集有效,将样本中含氮率测定值最大与最小者归入验证集,其余样本随机抽取。
1.3 模型建立
(1)波段深度分析
350nm~750nm波段包含叶绿素强吸收的蓝、红光区和及“红边”波段范围,此区域光谱反射率与植物叶片氮含量具有密切相关性[3,16],加上新鲜叶片的近红外光谱特征主要被液态水控制,叶片的生化组分信息易被液态水信息干扰[17],所以研究中只选取此区域进行波段深度分析。首先对光谱进行包络线去除。包络线去除法(Continuum Removal)是一种有效增强吸收特征的光谱分析方法[18]。“包络线”定义为逐点直线连接光谱曲线突出的峰值点而形成的曲线,从直观上来看就像包住光谱曲线的“外壳”(图1)。以原始光谱曲线上的值除以包络线上对应的值,即为光谱去包络线(记为R′)。包络线去除法处理后提取出如下光谱吸收特征:波段深度(Band Depth,BD)、波段深度比(Band Depth Ratio,BDR)、归一化面积波段深度(Band Depth Normalized to Band Area,BNA)、归一化面积波段指数(Band Depth Normalized to Band Area,NBDI)[6]。各表达式见表1。
(2)遗传算法
遗传算法(Genetic Algorithm)是一类借鉴生物进化规律(适者生存,优胜劣汰)演化而来的搜索方法[19]。本文中使用遗传算法进行波段选择的主要步骤为:①编码。每一个波段序号为一个基因,对基因进行二进制编码,若基因为1,建模时包括此波段;若为0,不包括此波段。一种编码组合称为一条染色体,染色体的长度m为被编码的波段个数。②选择初始种群。随机选取初始种群n,即初始种群的选择方法为随机产生n个m位的0-1二进制数作为初始群体。③适应性评价。这里将选取波段指数进行偏最小二乘拟合,采用交叉验证均方根误差(RMSECV)[20]作为精度评价指标。④复制。根据评价因子舍弃一半个体,剩余一半个体进行交叉繁殖,选取双点交叉法,重复此过程直至最大繁殖代数时停止,最终获得最优后代。研究预设参数:种群大小(64),最大繁殖代数(100),变异概率(0.005),循环次数(3),交叉方式为双点交叉。
(3)模型校准和验证
PLSR是一种多元统计方法,其吸取了主成分回归法中提取主成分的思想,从因变量出发,选择与因变量相关性较强而又方便运算的自变量的线性组合,同时克服了自变量之间多重相关性的问题[21]。由于对变量的综合,可能克服多重相关性造成的信息重叠,而由于对变量系统中的信息进行筛选,有效地区分系统的信息与噪声,提高系统建模的准确性[22]。表达式如下:
Y=Xb+E (1)
其中,Y指经过均值中心化的因变量矩阵(本研究中为n×1),X为经过均值中心化的自变量矩阵(本研究中为高光谱波段变量及波段深度分析指数变量),b为回归系数矩阵,E为残差矩阵。
基于建模集的BD、BDR、BNA和NBDI,分别建立氮含量估算BDA-PLSR及GA-PLSR模型,采用留一交叉验证法[21]确定模型中采用的最优因子数(factors)。为了防止过拟合同时保持模型较好的预测能力,仅当均方根误差(RMSECV)减少大于2%时,加入新的因子。将建立的模型应用于验证集,进行模型的验证。采用模型的校正决定系数(Adj.R2)检验模型的拟合程度,用均方根误差(RMSEP)和剩余预测偏差(RPD)评价模型预测能力。使用PLS toolbox 7.5.2(Eigenvector,USA)进行BDA-PLSR与GA-PLSR模型的建立,全部工作均在平台Matlab 2010b中实现。

图1 350nm~750nm原始光谱、包络线和去包络线曲线图

表1 波段深度分析表达式
2 结果与分析
2.1 含氮量测定结果
异常点检测去除了5组具有显著异常的光谱及氮含量数据,表2为剩余75个样点的含氮量统计结果。全集介于1.89%和4.02%之间,均值为2.69%,标准差为0.39%。建模集和验证集均值差异较小,说明建模集和验证集可以有效地代表全集。

表2 样本含氮率测定统计表(n=75)
2.2 GA波段选择结果
4种波段分析处理方法的选择结果如图2所示。由图2可以看出,波段选取数目和波段选取分布随不同处理方式有较大不同。例如,波段深度比(BDR)被选取62个波段,较均匀分布在350nm~750nm之间,而归一化面积波段深度(NBDI)仅被选取49个波段,较集中于354nm~356nm、683nm~697nm等区域。

图2 遗传算法选取波段指数分布
4种波段分析处理下:351nm、375nm、389nm、454nm、455nm、607nm、644nm、686nm处的指数有较高被选取的频率(≥3次)。氮素是植物叶绿素的重要成成分,研究[23-24]已知叶绿素在430nm、460nm、640nm和660nm波长处存在吸收特征,420nm和620nm、660nm分别对应叶绿素a和叶绿素b的吸收。这些波段中,454nm、455nm、644nm和686nm处部分与叶绿素吸收敏感波段相临近,一定程度上证实了氮素与叶绿素的密切关系。
2.3 波段深度分析与PLSR结合的氮含量估算
表3为BDA-PLSR及GA-PLSR模型精度校准和验证结果。BDA-PLSR 4种模型中,BNA与全波段模型精度最高(Adj.R2=0.59)。与全光谱模型比较,BNA模型只利用400波段,数据量大幅减小。可见,此模型可在一定程度上改善PLSR全光谱模型数据冗余问题。
将数据进一步进行遗传算法波段选择后,建模模型及验证模型精度均在之前基础上有了较大程度的提高,所有模型的校正决定系数均在0.6左右;参与运算的波段数目大幅减少,全波段GA-PLSR模型波段数目由2151减少至500左右,波段深度结合GA-PLSR模型波段数目由401减少最少到50左右。最优建模集模型为波段选择后的BNA模型,将此模型用于预测集验证得到预测模型校正决定系数为0.67,RPD达到1.84(表3、图3)。

表3 水稻叶片氮含量BDA-PLSR和GA-PLSR模型的校准和验证结果
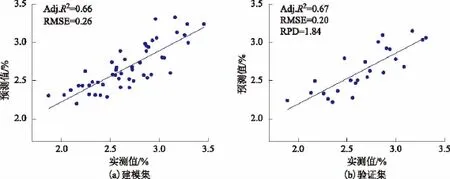
图3 最佳GA-PLSR模型的氮含量预测值与实测值散点图
所有模型在GA波段选择后大大减少了参与建模的变量数目,模型精度有所提高,说明被选取波段包含反演水稻叶片氮含量的足够信息,原始全波段高光谱数据无关信息及噪声较多。GA用于波段选择来提高PLSR模型精度是可行的。
3 结束语
为了解决高光谱反演水稻叶片氮素时存在的数据冗余、模型复杂的问题,本文使用实验室数据,尝试将波段深度分析与PLSR结合建立孕穗期水稻叶片氮含量估算模型,同时进一步采用遗传算法进行波段选择,对比了波段深度结合模型与全光谱模型,以及BDA-PLSR模型与GA-PLSR模型的精度。波段深度分析与PLSR结合建立的水稻叶片氮含量估算模型能一定程度解决PLSR全光谱模型的数据冗余问题,将遗传算法应用于波段深度分析数据进行选择后,可以进一步减小数据冗余,极大的缩减了模型运算的数据量,且模型精度较选择前有很大程度提高。
[1]FOLEY W J,MCILWEE A,LAWLER I,et al.Ecological applications of near infrared reflectance spectroscopy-a tool for rapid,cost-effective prediction of the composition of plant and animal tissues and aspects of animal performance[J].Oecologia,1998,116(3):293-305.
[2]INOUE Y,SAKAIYA E,ZHU Y,et al.Diagnostic mapping of canopy nitrogen content in rice based on hyperspectral measurements[J].Remote Sensing of Environment,2012,126:210-221.
[3]CURRAN P J,DUNGAN J L,PETERSON D L.Estimating the foliar biochemical concentration of leaves with reflectance spectrometry:Testing the kokaly and clark methodologies[J].Remote Sensing of Environment,2001,76(3):349-359.
[4]KOKALY R F,CLARK R N.Spectroscopic determination of leaf biochemistry using band-depth analysis of absorption features and stepwise multiple linear regression[J].Remote Sensing of Environment,1999,67(3):267-287.
[5]MUTANGA O,SKIDMORE A K,WIEREN S V.Discriminating tropical grass(cenchrus ciliaris)canopies grown under different nitrogen treatments using spectroradiometry[J].ISPRS Journal of Photogrammetry and Remote Sensing,2003,57(4):263-272.
[6]MUTANGA O,SKIDMORE A K.Hyperspectral band depth analysis for a better estimation of grass biomass(cenchrus ciliaris)measured under controlled laboratory conditions[J].International Journal of Applied Earth Observation and Geoinformation,2004,5(2):87-96.
[7]MUTANGA O,SKIDMORE A K.Integrating imaging spectroscopy and neural networks to map grass quality in the Kruger National Park,South Africa[J].Remote Sensing of Environment,2004,90(1):104-115.
[8]CHEN J,GU S,SHEN M,et al.Estimating aboveground biomass of grassland having a high canopy cover:an exploratory analysis of in situ hyperspectral data[J].International Journal of Remote Sensing,2009,30(24):6497-6517.
[9]RAMOELO A,SKIDMORE A K,SCHLERF M,et al.Water-removed spectra increase the retrieval accuracy when estimating savanna grass nitrogen and phosphorus concentrations[J].ISPRS Journal of Photogrammetry and Remote Sensing,2011,66(4):408-417.
[10]XIAO B Z,JIEWEN Z,POVEY M J W,et al.Variables selection methods in near-infrared spectroscopy[J].Analytica Chimica Acta,2010,667(1/2):14-32.
[11]GUO Z,ZHAO C,HUANG W,et al.Nondestructive quantification of foliar chlorophyll in an apple orchard by visible/near-infrared reflectance spectroscopy and partial least squares[J].Spectroscopy Letters,2013,47(6):481-487.
[12]WANG J,CUI L,GAO W,et al.Prediction of low heavy metal concentrations in agricultural soils using visible and nearinfrared reflectance spectroscopy[J].Geoderma,2014,216:1-9.
[13]SHI T,LIU H,WANG J,et al.Monitoring arsenic contamination in agricultural soils with reflectance spectroscopy of rice plants[J].Environmental Science &Technology,2014,48(11):6264-6272.
[14]BREMNER J M,MULVANEY C S.“Total Nitrogen”in methods of soil analysis.Part 2.chemical and microbiological properties[D].American Society of Agronomy,Soil Science Society of America,1982.
[15]VERBOVEN S,HUBERT M.LIBRA:a MATLAB library for robust analysis[J].Chemometrics and Intelligent Laboratory Systems,2005,75(2):127-136.
[16]PEUELAS J,GAMON J A,FREDEEN A L,et al.Reflectance indices associated with physiological changes in nitrogenand water-limited sunflower leaves[J].Remote Sensing of Environment,1994,48(2):135-146.
[17]ELVIDGE C D.Visible and near infrared reflectance characteristics of dry plant materials[J].International Journal of Remote Sensing,1990,11(10):1775-1795.
[18]CLARK R N,ROUSH T L.Reflectance spectroscopy:quantitative analysis techniques for remote sensing applications[J].Journal of Geophysical Research:Solid Earth,1984,89(B7):6329-6340.
[19]JARVIS R M,GOODACRE R.Genetic algorithm optimization for pre-processing and variable selection of spectroscopicdata[J].Bioinformatics,2005,21(7):860-868.
[20]GOICOECHEA H C,OLIVIERI A C.A new family of genetic algorithms for wavelength interval selection in multivariate analytical spectroscopy[J].Journal of Chemometrics,2003,17(6):338-345.
[21]GELADI P,KOWALSKI B R.Partial least-squares regression:a tutorial[J].Analytica Chimica Acta,1986,185:1-17.
[22]THOMAS E V,HAALAND D M.Comparison of multivariate calibration methods for quantitative spectral analysis[J].Analytical Chemistry,1990,62(10):1091-1099.
[23]FERWERDA J G,SKIDMORE A K,MUTANGA O.Nitrogen detection with hyperspectral normalized ratio indices across multiple plant species[J].International Journal of Remote Sensing,2005,26(18):4083-4095.
[24]CURRAN P J.Remote sensing of foliar chemistry[J].Remote Sensing of Environment,1989,30(3):271-278.
Partial Least Square Regression Model for Retrieving Paddy Rice Nitrogen Content with Band Depth Analysis and Genetic Algorithm
WANG Yi-han1,SHI Tie-zhu1,LIU Hui-zeng1,WANG Jun-jie1,WU Guo-feng2,3,4
(1.School of Resource and Environmental Sciences,Wuhan University,Wuhan 430079;2.Key Laboratory for Geo-environment Monitoring of Coastal Zone(GEMCO)of the National Administration of Surveying,Mapping and GeoInformation,Shenzhen518060;3.Shenzhen Key Laboratory of Spatial Smart Sensing and Services,Shenzhen518060;4.College of Life Sciences,Shenzhen University,Shenzhen518060)
To reduce the data redundancy and complexity of partial least square regression(PLSR)model in retrieving nitrogen content of crops,this article tries to combine band depth analysis(BDA)and genetic algorithm(GA)to build PLSR models for rice nitrogen content retrieval.Based on the continuum-removed spectrum over 350nm~750nm of paddy rice,BDA is employed to derive band depth indexes,including band depth(BD),band depth ratio(BDR),normalized band depth index(NBDI)and band depth normalized to area(BNA),and they are used to build BDA-PLSR models.GA is then utilized to select BDA-derived index most highly correlated with the nitrogen content to build GA-PLSR models,and it is then compared with the BDAPLSR models.Results show that the nitrogen contents are best estimated by the GA-PLSR model based on BNA(Adj.R2=0.67,RMSEP=0.20,RPD=1.84).It is concluded that the combination of BDA and PLSR could reduce the data redundancy,and further selection by GA could explore spectral information effectively and improve the nitrogen content estimation accuracy.
paddy rice;nitrogen content;PLSR;band depth analysis;genetic algorithm
10.3969/j.issn.1000-3177.2015.06.008
TP79
A
1000-3177(2015)142-0042-06
2014―11―21
2015―04―01
测绘地理信息公益性行业科研专项经费项目(20141207)。
王奕涵(1988—),女,硕士研究生,主要从事植被高光谱遥感研究。
E-mail:wyh520915@163.com
邬国锋(1969—),男,教授,博士,主要从事遥感技术在水质、土壤质量和植被质量参数反演及湖泊、海岸带生态系统方面的研究。
E-mail:guofeng.wu@szu.edu.cn

